青海湖裸鲤FXYD基因家族鉴定、分子进化及表达
2023-09-27周秉正陈生学刘思嘉祁得林祁洪芳
周秉正,陈生学,刘思嘉,祁得林,祁洪芳,汪 洋,赵 凯,田 菲
( 1.中国科学院 西北高原生物研究所, 高原生物适应与进化重点实验室, 高原鱼类进化与功能基因组学实验室,青海 西宁 810001;2.青海省动物生态基因组学重点实验室,青海 西宁 810001;3.中国科学院大学,北京 100049;4. 青海大学,省部共建三江源生态与高原农牧业国家重点实验室,青海 西宁 810001;5.青海湖裸鲤繁育重点实验室,青海湖裸鲤救护中心,青海 西宁 810001 )
青海湖裸鲤(Gymnocyprisprzewalskiissp.przewalskii)属鲤形目鲤科裂腹鱼亚科。裂腹鱼亚科鱼类是鲤科鱼类中唯一一种适应青藏高原极端环境的自然群体。青海湖裸鲤是裂腹鱼类中研究最为广泛的物种之一,主要栖息于我国最大的咸水湖泊青海湖(盐度12~14、pH 9.0~9.2)[1]。前期研究表明,青海湖裸鲤起源于生活在黄河淡水环境中的花斑裸鲤(G.ecklonissp.eckloni)[2],由于地理隔离和对自然环境的长期适应,使青海湖裸鲤演化出其特有的形态、生理以及遗传特征。因此,青海湖裸鲤是研究青藏高原极端环境下物种形成和适应性进化的良好素材,而其适应高盐碱环境的机制也成为了国内外学者关注的焦点[3]。青海湖裸鲤的扩张基因主要富集于离子转运相关通路中[4],推测可能是其对青海湖高盐环境长期适应的结果。
苯丙氨酸-X-酪氨酸-天冬氨酸(FXYD)家族是一类小分子跨膜蛋白,不仅是钠钾ATP酶(Na+-K+-ATPase)的组成部分[5],也是钠钾ATP酶活性调控的重要因子之一,参与维持细胞离子稳态、调节渗透压平衡和调控细胞体积等生物学功能。已有研究表明,FXYD基因家族通过调节离子通道和水通道活性响应渗透压变化,广泛参与了鱼类盐度的适应性调节。FXYD基因包含6~9个外显子,编码60~160个氨基酸,其中在脊椎动物中高度保守的FXYD基序位于胞质外[6]。青海湖裸鲤FXYD基因家族有12个家族成员(FXYD1~ FXYD12),在多种组织中表达并对组织的生理功能起到积极作用,如维持肾的钠离子重吸收[7]、肌肉收缩和神经元兴奋性[8]。在该家族成员中,FXYD8~FXYD12基因被认为是鱼类特有的FXYD基因家族成员,例如在大西洋鲑(Salmosalar)中发现有8个FXYD基因成员,包括FXYD2、FXYD5~FXYD9、FXYD11和FXYD12基因,而斑马鱼(Daniorerio)包含了FXYD1、FXYD2、FXYD3、FXYD5~FXYD7、FXYD11和FXYD12基因共8个成员[9-11]。
在多种鱼类中的研究已证明,FXYD1、FXYD2、FXYD4、FXYD7、FXYD10~FXYD12等在结构和功能上与钠钾ATP酶相互作用使得机体离子浓度发生变化,从而引起一系列的生理变化[12-15]。在咸水环境中,大西洋鲑、绿河鲀(Tetraodonnigroviridis)等鱼类鳃组织的FXYD基因被激活,与淡水环境相比其转录水平显著提高;同时,在进行盐度处理时发现,斑马鱼(Daniorerio)、鳗鲡(Anguillajaponica)鳃和皮肤组织中的FXYD11基因以及青鳉(Oryziaslatipes)肾脏组织中FXYD12基因的转录水平也有明显的提高[12,15-16]。另有研究表明,FXYD1可调节水通道蛋白(AQP2)在肾脏组织中的功能,进而影响对水的重吸收[17]。综上所述,FXYD基因家族作为钠钾ATP酶基因表达和调控因子,在鱼类盐度适应和盐度变化响应中发挥至关重要的作用[11]。
笔者基于实验室组装的青海湖裸鲤基因组数据,对青海湖裸鲤FXYD基因家族成员进行鉴定,通过与斑马鱼等物种FXYD基因家族成员的序列比对及共线性分析,揭示其分类与基因加倍事件,同时,利用不同盐度条件下青海湖裸鲤转录组数据探讨FXYD基因家族在青海湖裸鲤盐度适应中的潜在功能。
1 材料与方法
1.1 青海湖裸鲤 FXYD 基因家族成员鉴定
基于PacBio和Hi-C测序得到青海湖裸鲤基因组为2.03 Gb,挂载到46条染色体,scaffold N50达到44.93 Mb,contig N50达到386.94 kb。染色体数目与已报道的青海湖裸鲤核型分析结果[18-19]一致。根据序列的同源性和功能结构域的保守性共同鉴定青海湖裸鲤FXYD基因家族成员。通过Pfam数据库(http://pfam.xfam.org/)获得FXYD保守域HMM模型(PF03106),利用HMMER 3.0在青海湖裸鲤蛋白序列中搜索包含有FXYD保守域HMM模型的候选序列(cutoff为E值<10-5)。在Pfam数据库和CDD数据库(http://www.ncbi.nlm.nih.gov/cdd/)中对鉴定得到的候选蛋白序列进行手动分析,确保候选基因含有FXYD保守结构域。利用验证后的青海湖裸鲤候选蛋白,使用Hmmscan软件,构建青海湖裸鲤FXYD的隐马尔科夫模型(HMM),在基因组水平进行搜索,以确定其包含FXYD的保守结构域。同时,利用Ensembl数据库(http://asia.ensembl.org/index.html)获得斑马鱼FXYD蛋白质序列,以斑马鱼FXYD氨基酸序列为检索序列(query),采用BLASTP的比对方法通过比对获得青海湖裸鲤FXYD家族成员(cutoff为E值<10-5),并进行手动验证。使用ExPASy蛋白质组学服务器(https://web.expasy.org/protparam/)和scansite (https://scansite4.mit.edu)计算gpFXYD家族成员的分子质量、等电点等信息,并预测磷酸化位点。
1.2 青海湖裸鲤FXYD基因家族成员蛋白质序列分析及染色体定位
利用mafft软件对已获取的青海湖裸鲤FXYD蛋白序列和Ensembl数据库中斑马鱼、鲫(Carassiusauratus)、哲罗鲑(Huchotaimen)、金线鲃(Sinocyclocheilusgrahami)、鲤(Cyprinuscarpio)、花斑剑尾鱼(Xiphophorusmaculatus)等6种硬骨鱼类的FXYD蛋白质序列进行多序列比对,并使用 RAxML 软件构建进化树。根据实验室测序获得的青海湖裸鲤基因组注释文件中获取的FXYD基因家族所在染色体位置信息,对FXYD基因家族成员在染色体分布进行可视化分析,通过MapChart软件绘制FXYD家族在染色体上的定位分布图。利用MEME 4.12.0(http://meme-suite.org/tools/meme)在线程序,对所鉴定青海湖裸鲤FXYD蛋白氨基酸序列中保守基序进行预测分析,参数设置如下:结构区块最大值设为5,宽度最小值设为6,最大值设为10,并利用SMART在线工具预测保守结构域(http://smart.embl-heidelberg.de)。
1.3 青海湖裸鲤FXYD共线性及进化分析
通过BLASTALL方法,以青海湖裸鲤氨基酸序列为检索序列,分别与青海湖裸鲤和斑马鱼进行比对。为分析青海湖裸鲤中FXYD基因是否经过基因复制以及复制方式,利用McscanX软件进行青海湖裸鲤自共线性分析以及青海湖裸鲤基因组与斑马鱼基因组之间的共线性分析,并利用KaKs Calculator软件[20-21]计算基因对之间的非同义突变率、同义突变率和进化选择压力值。
1.4 青海湖裸鲤FXYD基因结构分析
从青海湖裸鲤基因组中下载每个预测基因对应的DNA和cDNA序列,使用基于网络的方法分析内含子等分布模式和拼接阶段生物信息学工具GSDS(http://gsds.cbi.pku.edu.cn/)进行基因结构分析[22]。
1.5 试验材料以及处理
试验对象为野生青海湖裸鲤实验室繁育F1代。试验前期完成了青海湖裸鲤在不同盐度条件下的转录组测序,方法为:将体长、体质量接近的8月龄的青海湖裸鲤饲养于相同条件下 2 周,随后随机分为2组,其中对照组盐度为 13,与青海湖盐度一致;试验组为淡水条件,盐度为 0.1;处理 96 h 后,试验组和对照组各3个平行,对青海湖裸鲤的鳃、肾组织进行采样,保存于液氮,用于 RNA 提取和转录组测序。
1.6 基于转录组数据分析青海湖裸鲤FXYD基因表达模式
利用实验室产生的青海湖裸鲤转录组(心脏、肝胰脏、脑组织、肾脏、鳃、骨骼肌、肠道、精巢和卵巢组织)数据,以及实验室产生的不同盐度处理的鳃组织的转录组数据(表1),获得青海湖裸鲤FXYD基因家族成员的基因表达量每1 000 000单位比对片段中,比对到目的基因1000碱基单位的片段数(FPKM),利用R语言Pheatmap包对表达量进行可视化,探讨FXYD基因家族成员的组织表达规律和在不同盐度条件下的表达情况。

表1 试验使用的转录组数据来源
2 结果与分析
2.1 青海湖裸鲤FXYD基因家族成员的鉴定
利用FXYD结构域作为检索序列在青海湖裸鲤本地数据库进行同源比对搜索,结合青海湖裸鲤基因组数据库注释信息,共获得16个青海湖裸鲤FXYD(gpFXYD)基因。基于青海湖裸鲤与斑马鱼、鲫、哲罗鲑、金线鲃、鲤、花斑剑尾鱼、大西洋鲑FXYD的氨基酸序列(表2)构建了最大似然树(图1),结果显示,青海湖裸鲤包含5个FXYDs亚型,即gpFXYD1、gpFXYD5、gpFXYD6、gpFXYD7和gpFXYD11,且每个成员中均存在多个同源基因,并据此对gpFXYD基因进行了命名,即:gpFXYD11a、gpFXYD11b、gpFXYD11c、gpFXYD1a、gpFXYD1b、gpFXYD1c、gpFXYD5a、gpFXYD5b、gpFXYD5c、gpFXYD5d、gpFXYD5e、gpFXYD6a、gpFXYD6b、gpFXYD6c、gpFXYD7a、gpFXYD7b。对gpFXYD基因在青海湖裸鲤染色体上进行定位,可以看出gpFXYD基因主要分布于青海湖裸鲤5条染色体中(图2)。其中gpFXYD5a、gpFXYD5c和gpFXYD11c分布在7号染色体,gpFXYD5b和gpFXYD5d分布在8号染色体,gpFXYD5e分布在10号染色体,gpFXYD1a、gpFXYD1b、gpFXYD6a、gpFXYD7a和gpFXYD11a分布在29号染色体,gpFXYD1c、gpFXYD6b、gpFXYD6c、gpFXYD7b和gpFXYD11b分布在30号染色体,第29和30号染色体上分布最多。利用Scansite(https://scansite4.mit.edu)Calculate Amino Acid Composition工具对gpFXYD蛋白质的分子质量、等电点和疏水性等理化参数进行分析。16个gpFXYD蛋白分子中氨基酸为42~343个,平均139个;分子质量为4563.91~38 777.88 u,平均15 495.23 u;理论等电点3.89~9.78(表3)。多重序列比对结果显示,所有gpFXYD均具有FXYD结构域或FXYD样结构域(图3)。

图1 青海湖裸鲤与其他鱼类FXYD 基因家族系统进化树Fig.1 Phylogenetic tree of gpFXYD gene family of G. przewalskii ssp. przewalskii and other fishes

图3 青海湖裸鲤FXYDs结构域的多序列比对Fig.3 Multi-sequence alignment of the prediction phosphorylation site domain of FXYD gene family in G. przewalskii ssp. przewalskii

表2 本试验使用的FXYD蛋白序列
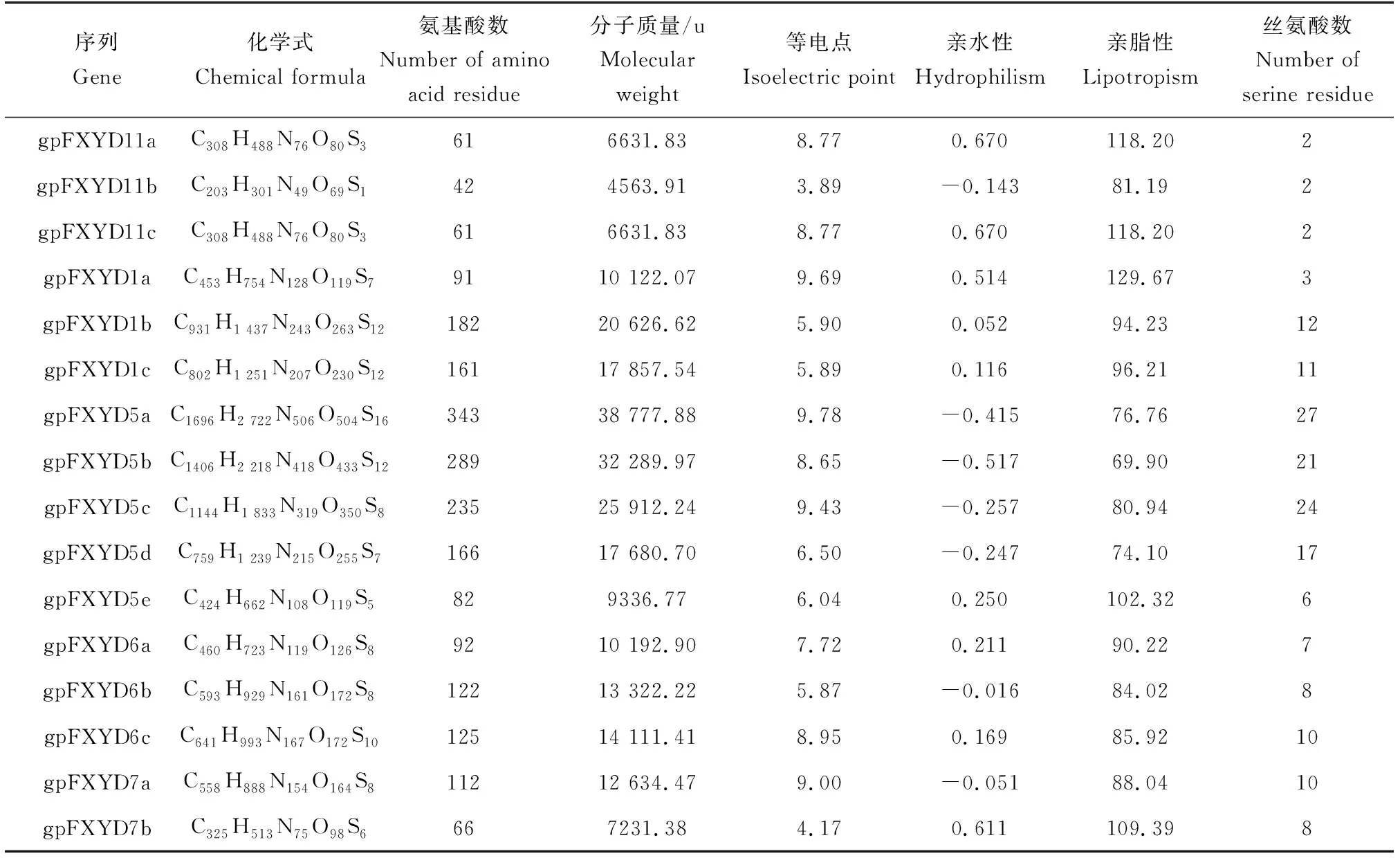
表3 青海湖裸鲤FXYD基因家族理化性质
2.2 青海湖裸鲤 FXYD 基因家族成员基因结构、保守基序和功能结构域分析
使用GSGD软件对gpFXYD家族基因每个成员的基因结构进行可视化分析,结果显示,除gpFXYD 7亚型片段较短,其他亚型的 gpFXYDs 基因具有相似的序列结构,且内含子与外显子数目以及每个外显子的长度也较为接近,都含有5个以上外显子(EXON)区,其中gpFXYD5以及gpFXYD1亚型含有较长的非编码区(图4a)。保守基序分析显示gpFXYD家族共包含10种蛋白质基序(motif)(表4),每个亚型的成员具有相同或类似的motif和排列顺序(图4b)。其中,gpFXYD1均包含motif1和motif10,gpFXYD5包含motif1、motif2和motif4,gpFXYYD6包含motif1、motif3、motif9,gpFXYD7和gpFXYD11都包含motif1,这表明相同亚型的gpFXYD成员具有相似的motif组成。虽然不同亚型基因的motif组成不同,但它们都具有重要的活性位点motif1,且共有的 motif 以基本相同的顺序排列。Motif在不同亚型中的分布也有所不同,如motif4和motif7在gpFXYD1是分布于motif1之后,而在gpFXYD1和gpFXYD6均分布于motif1之前。功能结构域分析发现,所有的gpFXYDs成员都具有FXYD结构域和N-肉豆蔻酰转移酶结构域(NMT),在gpFXYD5、gpFXYD6、gpFXYD7中分布有酪蛋白激酶II(CK-2)磷酸化结构域(CK2_PHOSPHO_SITE)、N-糖基化结构域(ASN_GLYCOSYLATION)、蛋白激酶C磷酸化结构域(PKC_PHOSPHO_SITE)。而gpFXYD1家族还含有cAMP和cGMP依赖性蛋白激酶磷酸化结构域(CAMP_PHOSPHO_SITE)(表4)。
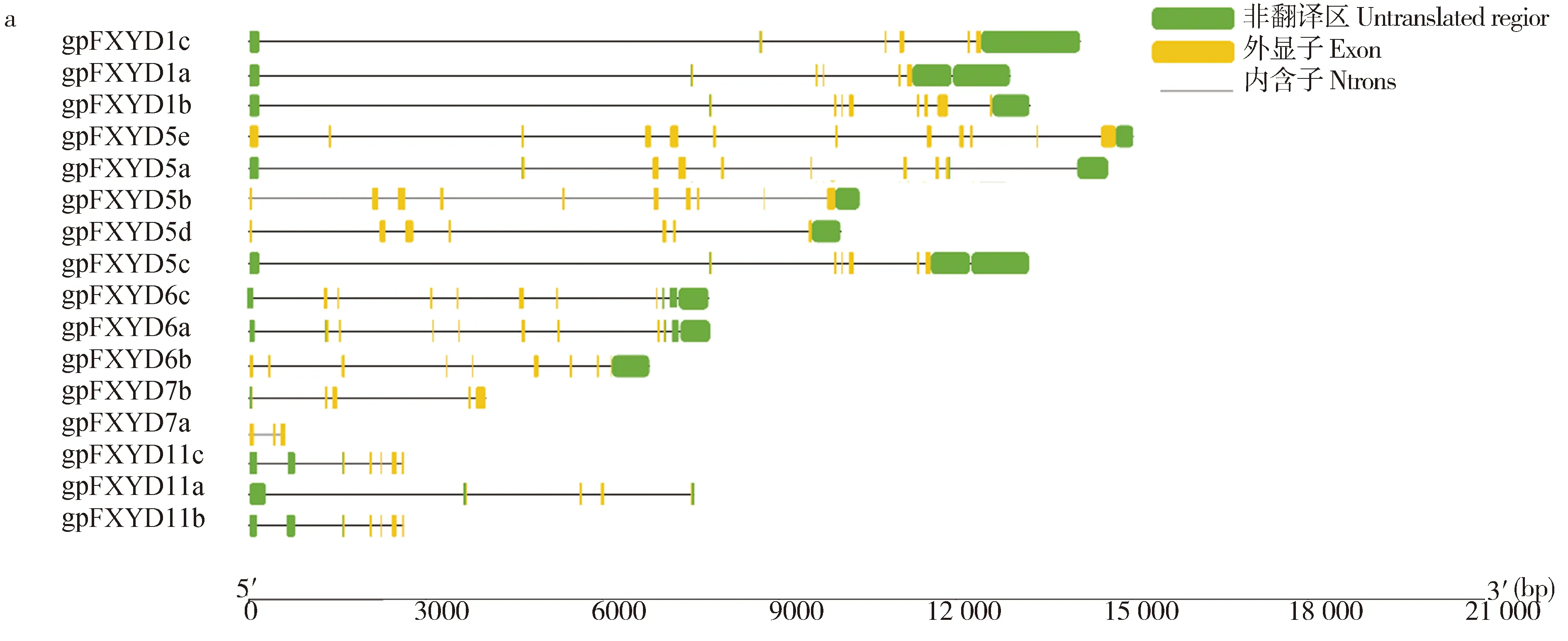
图4 青海湖裸鲤gpFXYD基因结构和氨基酸保守基序分析Fig.4 gpFXYD gene structure and conserved amino acid motif analysis in G. przewalskii ssp. przewalskii

表4 青海湖裸鲤gpFXYDs结构域分布
2.3 青海湖裸鲤FXYD共线性分析
利用MCScanX软件对gpFXYDs进行了共线性分析,结果显示,在16个gpFXYD基因中有12个基因与斑马鱼的6个基因存在共线性关系。其中,位于青海湖裸鲤7号和8号染色体的gpFXYD5a/gpFXYD5b以及gpFXYD5c/gpFXYD5d与斑马鱼15号染色体的FXYD5呈现共线性。位于29号和30号染色体的gpFXYD6a/gpFXYD6b、gpFXYD11a/gpFXYD11b、gpFXYD7a/gpFXYD7b、gp-FXYD1b/gpFXYD1c分别与斑马鱼16号染色体的FXYD1、FXYD6、FXYD7和FXYD11呈共线性关系(图5)。青海湖裸鲤自共线性分析显示,gpFXYD6a/gpFXYD6b和gpFXYD11a/gpFXYD11b呈共线性关系,共线性分析表明全基因组复制事件引起了gpFXYD基因加倍。通过各个基因对非同义突变率(Ka)、同义突变率(Ks)以及进化选择压力值(Ka/Ks),可以估算进化选择模式。进化选择压力值(Ka/Ks)>1表示正选择,进化选择压力值(Ka/Ks)<1表示纯化选择,进化选择压力值(Ka/Ks)=1表示中性选择。分析显示,gpFXYD6a/gpFXYD6b、gpFXYD5a/gpFXYD5b、gpFXYD7a/gpFXYD7b以及gpFXYD11a/gpFXYD11b的进化选择压力值(Ka/Ks)< 1,表明它们都经过了纯化选择(表5)。 gpFXYD1c/ gpFXYD1b以及gpFXYD5c/gpFXYD5d的进化选择压力值(Ka/Ks)> 1,表明gpFXYD1c和gpFXYD5c受到了正选择。
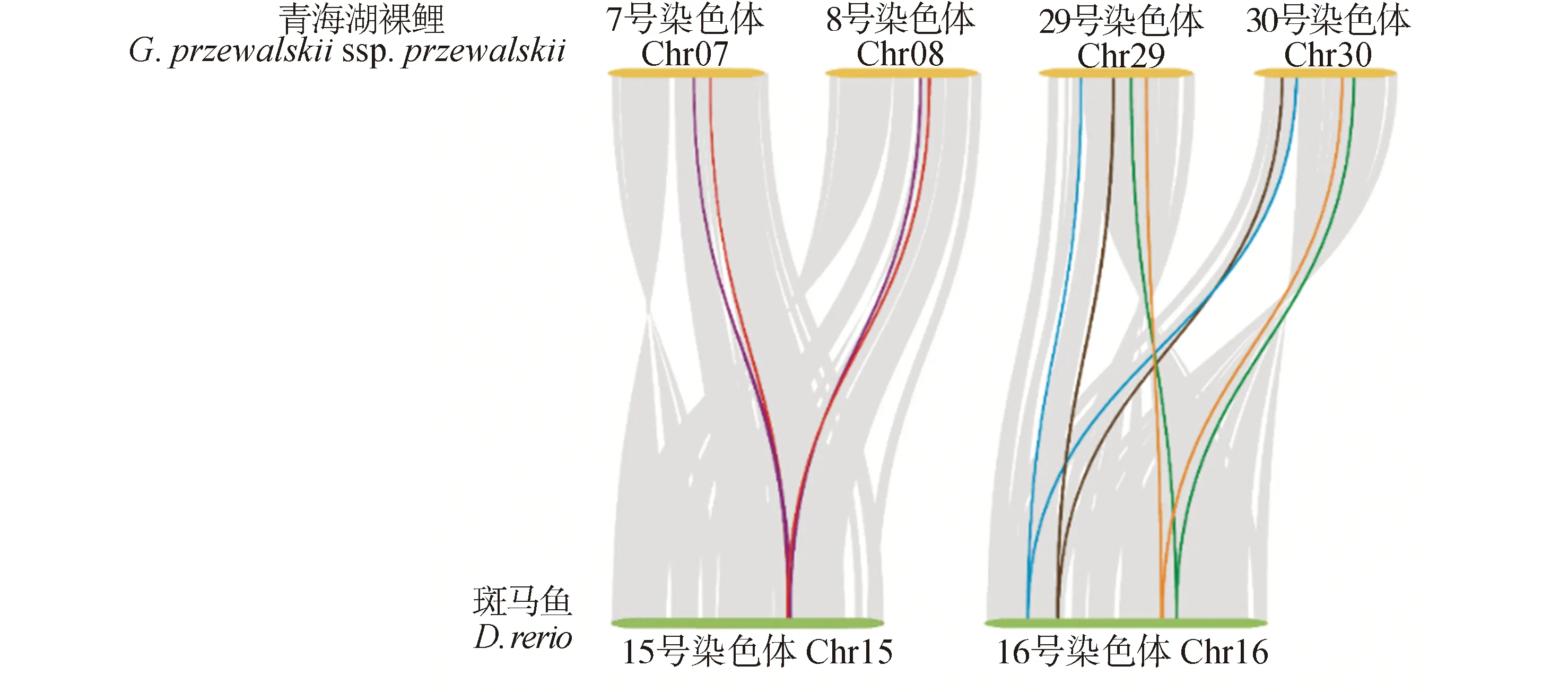
图5 青海湖裸鲤和斑马鱼FXYD 基因的共线关系Fig. 5 Syntenic analysis of FXYD genes in G. przewalskii ssp. przewalskii and Zebrafish D.rerio
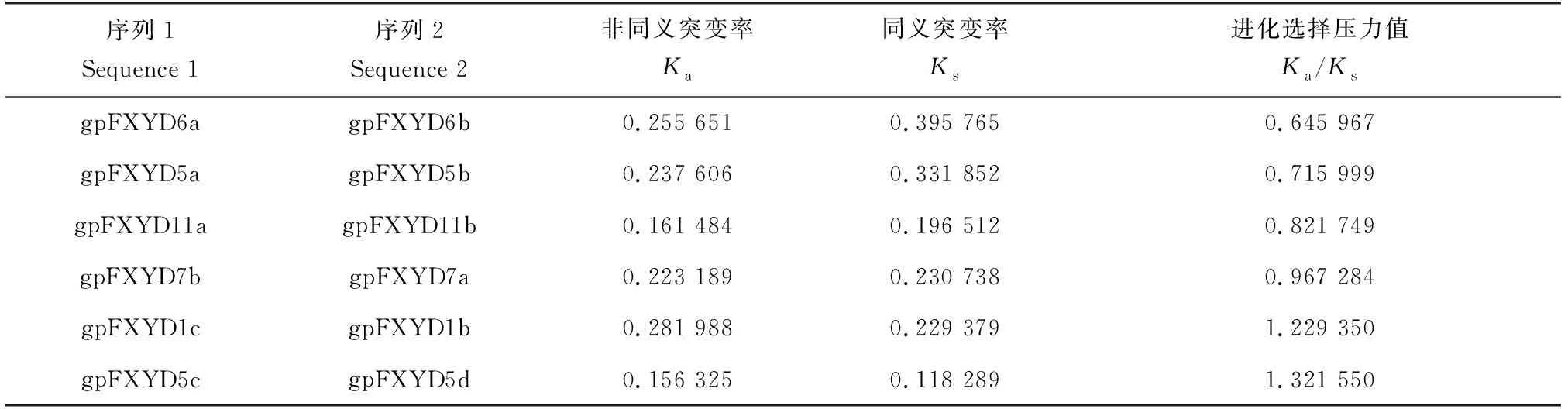
表5 青海湖裸鲤 FXYD 复制基因对的进化选择压力值
2.4 青海湖裸鲤 FXYD基因表达模式分析
gpFXYD基因在青海湖裸鲤心脏、肝胰脏、脑组织、肾脏、鳃、骨骼肌、肠道、精巢和卵巢等不同组织中的表达结果显示,gpFXYD同一亚型成员间的表达模式相似且具有组织特异性(图6a)。其中,gpFXYD1基因在心脏的表达高于其他组织,在脑、鳃、肝胰脏和骨骼肌中表达量相似,在肾脏和性腺组织中表达量最低。gpFXYD5亚型在肾脏中的表达量最高,除了gpFXYD5e基因在精巢组织中表达较高,其他gpFXYD5成员在性腺中的表达量均较低。同时发现,gpFXYD5a和gpFXYD5b基因以及gpFXYD5c和gpFXYD5d基因具有更为相似的表达模式,即gpFXYD5a和gpFXYD5b基因在心脏中的表达量高于肌肉和肝胰脏组织,而gpFXYD5c和gpFXYD5d基因则与之相反。肌肉组织中gpFXYD6基因的表达量整体显著高表达,其次是脑组织和精巢的表达,在其他组织中gpFXYD6基因的表达量均较其他家族成员表达量低。gpFXYD7亚型中,gpFXYD7a基因在脑组织中的表达量最高,其他组织表达水平相似,未在转录组的结果中发现gpFXYD7b基因的表达,这种现象极有可能是同源基因对中其中一个基因发生了非功能化。gpFXYD11亚型在鳃组织的表达较高,gpFXYD11b基因在肠道和卵巢组织中表达较高,gpFXYD11a基因肾脏组织中表达较高。据此推测不同亚型可能在不同组织中发挥作用,同一亚型不同基因拷贝转录表达的差异可能与基因加倍后的新功能化和功能分化有关。
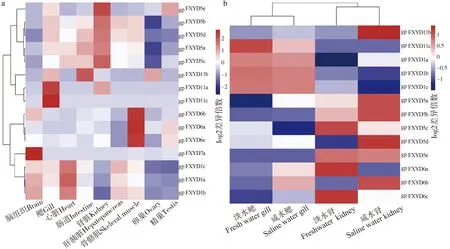
图6 青海湖裸鲤FXYD基因家族表达图谱 Fig. 6 Expression profiles of FXYD genes in G.przewalskii ssp. przewalskii
青海湖裸鲤gpFXYD基因在淡水和咸水条件下鳃和肾脏组织中的表达存在差异。gpFXYD基因各亚型在鳃、肾脏组织中的表达具有明显的组织特异性,且表达水平在淡水组和咸水组之间差异较大(图6b)。在鳃组织中,gpFXYD5a、gpFXYD6a和gpFXYD6b基因在咸水组中的表达显著上调。肾脏组织中,gpFXYD6a基因在淡水组中的表达量较高,而gpFXYD11b、gpFXYD5d和gpFXYD6b基因在咸水组中表达上调。
3 讨 论
3.1 青海湖裸鲤FXYD 基因家族进化
青海湖裸鲤是生活在青海湖中唯一的鲤科鱼类,对高盐碱水环境有良好的适应性,而维持青海湖裸鲤的细胞内外环境之间离子梯度是保持细胞稳态和组织正常功能的首要条件。FXYD基因家族是一类小分子跨膜蛋白,其与钠钾 ATP 酶相互作用可以调控钠钾ATP酶的表达与活性,被认为是重要的离子调节因子,在生物响应盐度胁迫和适应盐度变化中具有重要作用[16,23-24]。
笔者利用青海湖裸鲤基因组数据鉴定了16个完整的FXYD基因, 且每个gpFXYD基因中均包含了起始密码子和终止密码子,暗示其均可能具有翻译能力,而非假基因。基于蛋白质序列分析,gpFXYD成员具有完整的FXYD蛋白结构域,目前已发现的FXYD在N端均包含1个由6个氨基酸序列组成的保守序列由保守的苯丙氨酸-X-酪氨酸-天冬氨酸以及跨膜区的两个保守的甘氨酸组成,C端(-COOH)和N端(-NH2)的氨基酸序列可变。
因为鱼类的基因变化更频繁,在一些情况下,它们的基因比其他脊椎动物有更多的亚型[25],且由于膜外序列的保守性有限,笔者主要比对了青海湖裸鲤和其他硬骨鱼类的系统发育情况,并对每个家族的亚型进行命名。结果显示,gpFXYD不仅具有FXYD特征序列且具有保守的甘氨酸以及亮氨酸残基,同时,磷酸化的丝氨酸存在于所有的gpFXYD中[26]。有研究表明,磷酸化是FXYD与钠钾ATP酶相互作用的主要调控机制[27-31],本研究结果显示,gpFXYD每个亚家族具有几乎相同的预测磷酸化位点以及蛋白结构域,说明青海湖裸鲤与其他硬骨鱼成员[斑马鱼、青鳉和虹鳟(Oncorhynchusmykiss)]的FXYD家族同源[11,32],且氨基酸序列具有较高相似性,表明了其结构和功能的相似性。已有的研究表明,1个和2个丝氨酸残基的磷酸化会产生不同的效果[33],而磷酸化位点的存在表明可能通过磷酸化调控其功能。本研究结果显示,青海湖裸鲤每个FXYD家族成员的丝氨酸个数不一样,说明每个家族成员可能存在不同的磷酸化调控机制。
3.2 青海湖裸鲤FXYD基因家族分析
全基因组复制是进化的主要驱动力之一,也是产生新基因的来源[34]。硬骨鱼类不同系谱在其长期的进化过程中经历了2~6轮数目不等的大规模基因组复制事件,硬骨鱼类的全基因组复制事件和鲤科鱼类特有的全基因组复制事件与该类群物种辐射适应以及对环境有较高适应性密切相关[35-38]。为探讨gpFXYD基因复制与复制方式,笔者选取斑马鱼的FXYD基因与gpFXYD基因进行共线性分析。与斑马鱼相比,gpFXYD基因家族中gpFXYD1、gpFXYD5、gpFXYD6、gpFXYD7和gp-FXYD11都发生了加倍[34]。在16个gpFXYD基因中有12个基因与斑马鱼的6个基因存在共线性关系,表明他们起源于全基因组复制事件,由于青海湖裸鲤为四倍体(4n=92),斑马鱼为二倍体(2n=50),因此,大部分青海湖裸鲤与斑马鱼基因数目为2∶1关系。多倍体生物由于基因组的复制,使得同源基因拥有更多向不同功能分化的潜能,在青海湖裸鲤中,gpFXYD6a/gpFXYD6b、gpFXYD5a/gpFXYD5b、gpFXYD7a/gpFXYD7b以及gpFXYD11a/gpFXYD11b的进化选择压力值(Ka/Ks)<1,同时gpFXYD6a/gpFXYD6b和gpFXYD11a/gpFXYD11b呈自共线性关系,一般认为纯化选择通常使得基因功能更保守。gpFXYD1c/ gpFXYD1b以及gpFXYD5c/gpFXYD5d进化选择压力值(Ka/Ks)>1,表明gpFXYD1c和gpFXYD5c受到了正选择,其变异可能与青海湖裸鲤适应青海湖高盐碱水环境相关,从而保证青海湖裸鲤能更好地适应盐度的变化,推测这些基因可能是在复制过程中获得了新功能[34,39-41]。
3.3 青海湖裸鲤FXYD基因的组织表达和功能分析


本研究结果丰富了青海湖裸鲤gpFXYD基因家族的信息,不仅为研究青海湖裸鲤盐度适应机制提供了参考,同时也为研究耐盐鱼类品种提供了素材。
4 结 论
(1)青海湖裸鲤中有16个gpFXYD基因,分为5种亚型,分别是gpFXYD1(3个)、gpFXYD5(5个)、gpFXYD6(3个)、gpFXYD7(2个)、gpFXYD11(3个),基因家族成员具有相似的motif分布和基因结构。
(2)gpFXYD基因的5个亚家族都发生了扩张,gpFXYD6a/gpFXYD6b、gpFXYD5a/gpFXYD5b、gpFXYD7a/gpFXYD7b以及gpFXYD11a/gpFXYD11b共4对复制基因发生了纯化选择;gpFXYD1c/gpFXYD1b以及gpFXYD5c/gpFXYD5d这2对复制基因发生了正选择。
(3)gpFXYD基因家族中,gpFXYD7基因在青海湖裸鲤脑组织中特异性表达,gpFXYD1、gpFXYD6基因主要在青海湖裸鲤骨骼肌中表达,gpFXYD5基因主要在青海湖裸鲤肾组织中表达,而gpFXYD11基因主要在青海湖裸鲤鳃和肾脏组织中特异表达。这表明gpFXYD基因家族的亚型具有相似的表达模式且显现出不同的组织表达特异性。
(4)在高盐条件下,gpFXYD5、gpFXYD6以及gpFXYD11基因在青海湖裸鲤肾脏和鳃组织中出现显著的表达变化。相比淡水条件,咸水条件下,gpFXYD5a、gpFXYD6a和gpFXYD6b基因在鳃组织中表达较高,gpFXYD5d、gpFXYD6b和gpFXYD11b基因在肾脏组织中表达量较高。而淡水条件下,gpFXYD5c和gpFXYD6a基因在肾脏组织中被激活,表明gpFXYD5、gpFXYD6以及gpFXYD11基因可能与青海湖裸鲤在高盐度条件下的离子运输和渗透压调节密切相关。
