浓香型酒醅中活性梭菌群落多样性及其与挥发性有机酸代谢的关联性
2023-08-30胡晓龙冯大鸿杨雨惠王亚平朱文优孙继祥
胡晓龙, 冯大鸿, 杨雨惠, 迟 雷, 张 勇,王亚平, 朱文优, 孙继祥, 曾 田
(1.郑州轻工业大学 食品与生物工程学院,河南 郑州 450000;2.固态发酵资源利用四川省重点实验室,四川 宜宾 644000;3.河南皇沟酒业有限责任公司,河南 永城 476600;4.河南省五粮浓香固态酿造发酵工程技术研究中心,河南 淮滨 464400)
浓香型白酒是中国白酒的重要代表之一,酒醅是其发酵的主体,也是发酵微生物的主要载体,酒醅中微生物群落的多样性和组成与浓香型白酒的呈味物质密切相关[1]。Clostridia(梭菌纲,简称梭菌)是浓香型白酒发酵微生态系统中的重要组成部分。胡晓龙等[2]在浓香型酒醅中共检测到39个梭菌纲中的属,其中Caproiciproducens(产己酸菌属)是优势属;马箭等[3]也在酒醅中鉴定到152个属于梭菌纲的OTU,且预测发现梭菌主要参与氨基糖和核苷酸糖代谢、磷酸戊糖代谢、果糖和甘露糖代谢等途径;蒲秀鑫[4]通过预测梭菌培养基从窖泥中分离得到31株梭菌。梭菌还是一类产有机酸的主要微生物,其产生的己酸、丁酸和乙酸等能直接影响白酒的呈香和呈味程度。如Clostridiumkluyveri是典型的己酸菌,能以乙醇、琥珀酸和丙醇为底物产生乙酸、丁酸和己酸等[5]。由于酒醅中梭菌群落及代谢途径复杂,目前对酒醅中有机酸形成相关的梭菌及其代谢途径尚不明晰。
宏转录组学技术是直接分析环境样本中的mRNA,可有效检测具有生理或转录活性的微生物。诸多研究证明了宏转录组学的优势且较DNA水平的结果具有较大差异。Hu等[6]通过宏转录组学测序和16S rRNA/ITS 基因扩增子测序在浓香型酒醅的研究中发现,在RNA水平上共检测到1331个微生物属,而DNA水平上仅获得332个;Duan等[7]发现Lactobacillus(乳酸杆菌)、Enterococcus(肠球菌)、Streptococcus(链球菌)仅在宏转录中被发现,而16S rRNA检测的一部分细菌在RNA水平上没有基因表达。宏转录组学技术已经被应用到浓香型大曲[8]、酱香型大曲[9]和泡菜[10]等发酵产品之中,均从基因转录水平揭示了微生物组成、功能及酶谱,这为认识微生物群落提供了一个新的角度。目前,宏转录组学技术应用于酒醅活性梭菌群落演替的研究鲜有报道。
本研究以浓香型白酒整个发酵周期(60 d)的酒醅为研究对象,采用宏转录组学方法揭示酒醅中具有生理活性的梭菌群落多样性及其相关功能,并探究梭菌群落与浓香型白酒重要有机酸的关联性,以期为进一步阐明浓香型白酒发酵过程中梭菌群落的演替规律、代谢功能和发酵机理等提供理论依据。
1 材料与方法
1.1 材料与试剂
酒醅样品取自河南省某浓香型白酒酿造车间。
Ribo-ZeroTM rRNA Removal Kits(Bacteria)和Ribo-ZeroTM Magnetic Gold Kit(Yeast)试剂盒,上海麦克林生化科技有限公司,水饱和酚(pH值 4.5),上海源叶生物科技有限公司;Tris、RNase-Free Water、2×Taq Master Mix,北京索莱宝科技有限公司;二氯甲烷,色谱纯,天津市大茂化学试剂厂;叔戊醇、2-乙基丁酸、乙酸正戊酯,均为色谱纯,上海麦克林生化科技有限公司。
1.2 仪器与设备
DYY-8C型电泳仪,北京市六一仪器厂;WD-9403C型紫外分析仪,北京六一生物科技有限公司;NanoDrop 2000型微量分光光度计,赛默飞世尔科技(中国)有限公司;2100型生物分析仪,北京爱立斯生物科技有限公司;7890B-5977B型气相色谱-质谱联用仪,美国安捷伦公司;KQ3 200DA型数控超声波清洗器,昆山市超声仪器有限公司;TGL-20M型高速离心机,上海卢湘仪离心机仪器有限公司。
1.3 实验方法
1.3.1样品采集
随机选取正常发酵的3口平行窖池,对每口窖池中发酵第3、7、15、25、45、60天的酒醅进行跟踪取样。对每口窖池下层(距窖底约50 cm)平面上任一对角线的1/2、1/4及其距窖壁20 cm处3个不同位置的酒醅分别采集并混匀作为该窖池的代表性酒醅样品。共获得18个酒醅样品,3d-1、3d-2、3d-3、7d-1、7d-2、7d-3、15d-1、15d-2、15d-3、25d-1、25d-2、25d-3、45d-1、45d-2、45d-3、60d-1、60d-2、60d-3。以3d-1、3d-2、3d-3为例,其中3d代表发酵时间为第3天,-1、-2、-3代表3个平行样品。
1.3.2酒醅RNA提取
按照胡晓龙等[11]的SDS-苯酚法对酒醅进行总RNA提取,然后采用质量浓度1 g/100 mL琼脂糖凝胶电泳、微量分光光度计和生物分析仪评估总RNA的纯度和完整性。
1.3.3宏转录组学测序文库准备和Illumina测序
RNA样本检测合格后,使用Ribo-ZeroTM rRNA Removal Kits(Bacteria)和Ribo-ZeroTM Magnetic Gold Kit(Yeast)试剂盒分别去除细菌和真菌核糖体RNA。文库的构建和测序在北京奥维森基因科技有限公司Illumina HiSeq 2500(Illumina,San Diego,CA,United States)平台上完成。
1.3.4挥发性有机酸检测
样品预处理:酒醅样品预处理参照王康丽[12]的方法进行。
色谱条件:色谱柱为DB-FFAP毛细管柱(30 m×0.25 mm×0.25 μm),升温程序为初始温度45 ℃保留3 min,以5 ℃/min 的速率升温至220 ℃后保持5 min,再以250 ℃运行2 min。进样口温度250 ℃;柱前压50 754.095 Pa;载气为高纯 He(体积分数99.999%),载气流量1.0 mL/min;不分流模式;溶剂延迟时间3.6 min;进样量1 μL。
质谱条件:电子轰击离子源,离子源温度230 ℃,四极杆温度150 ℃,电子能量70 eV,激活电压1.5 V,扫描范围m/z为35~550 amu。
挥发性有机酸相对含量分析:根据挥发性有机酸的质谱特征离子峰(m/z=60),对其特征离子进行提取,然后通过GC-MS标准质谱数据库检索,得到各色谱峰对应的物质。最后将各酸的峰面积与内标中的2-乙基丁酸峰面积的比值作为半定量结果。
1.4 数据处理
测序后获得原始数据,通过过滤带有测序接头、未知碱基含量大于1%和低质量碱基(Q≤20)含量大于50%的reads后得到clean reads,采用Trinity(v0.27)对clean reads进行拼接,然后使用prodigal软件对拼接组装得到的contig序列进行开放阅读框预测,再通过CD-HIT软件对预测的基因序列以95%相似度去除冗余基因从而得到非冗余基因集。利用diamond将非冗余基因集与NR数据库比对分析,并利用megan进行微生物分类学解析得到物种信息,筛选出梭菌数据进行后续分析。通过FPKM(fragments per kilobase of exon model per million mapped fragments,每百万片段中来自某一基因每千碱基长度的片段数目)值对梭菌基因进行表达丰度水平计算。用KEGG(https:∥www.kegg.jp/)和CAZy(http:∥www.cazy.org)数据库将梭菌的非冗余基因集序列进行功能注释和碳水化合物酶注释。用Canoco5软件进行主成分分析(principal component analysis,PCA),基于KEGG注释结果和KEGG数据库绘制代谢途径,其余图均使用Origin 9.0绘制。
2 结果与分析
2.1 酒醅中活性梭菌群落组成分析
通过NR数据库注释,不同发酵节点酒醅中纲水平的细菌和科水平的梭菌组成如图1。所有酒醅样品共注释到79个细菌界中的纲,其中优势纲(平均相对丰度>1%)共4个,分别为Bacilli(芽孢杆菌纲,87.23%)、Clostridia(梭菌纲,3.04%)、Gammaproteobacteria(γ-变形菌纲,1.59%)和Actinobacteria(放线菌纲,1.33%)[图1(a)]。梭菌纲由6个目、27个科及148个属组成,其相对丰度在0.34%~9.95%,在发酵3 d时最低,发酵15 d最高。在科水平下分析梭菌组成,共检测到1个可鉴定的优势菌科Heliobacteriaceae(太阳杆菌科,1.49%)以及3个次优势菌科(平均相对丰度0.1%~1.0%),Ruminococcaceae(瘤胃菌科,0.87%)、Clostridiaceae(梭菌科,0.34%)和Lachnospiraceae(毛螺旋菌科,0.15%)[图1(b)]。Heliobacteriaceae、Ruminococcaceae和 Clostridiaceae的相对丰度在发酵15 d增加至最高,Lachnospiraceae在发酵45 d增加至最大值。梭菌的变化可能与酒醅理化性质和其他微生物有关,如发酵3 d时酒醅中氧气的存在会抑制梭菌的生长,导致其丰度仅为0.34%;随发酵的进行氧气逐渐消耗,给梭菌提供了合适的生长环境,其相对丰度增加直至9.95%。随后梭菌的丰度开始减少,这可能和乳酸杆菌的丰度在发酵过程中不断上升有关,其代谢产生的乳酸链球菌素和乳酸菌素等细菌素会抑制革兰氏阳性菌的生长,导致酒醅中梭菌较少[13]。在不同的发酵节点酒醅中梭菌科组成具有明显差异,在发酵3 d时,Clostridiaceae具有最高的丰度(0.10%);发酵7~15 d的酒醅中Heliobacteriaceae快速增加成为主要的优势菌科,其次为Ruminococcaceae;随着发酵的继续进行,Clostridiaceae和Lachnospiraceae成为主要的梭菌。

图1 不同发酵节点酒醅中纲水平细菌组成和科水平梭菌组成
梭菌群落属水平上β多样性分析见图2。属水平上中共检测到148种梭菌,基于这148个属的相对丰度进行PCA分析[图2(a)]。PC1和PC2分别可以解释76.69%和12.76%的梭菌群落结构差异,累计解释了酒醅样品中梭菌群落组成变化的89.45%。酒醅样品在第1轴和第2轴上可以明显被分为3组,其中发酵3 d样品聚集在第二象限,7 d和15 d样品主要聚集在第一象限,而25 d、45 d和60 d样品聚集在第三象限。同一象限内的梭菌群落较为类似,即表明梭菌群落的变化能通过发酵时间很好地区分开,且主要的变化发生在发酵前25 d。这也表明,酒醅发酵过程可分为3个阶段,即发酵前期(3~7 d)、发酵中期(7~25 d)和发酵后期(25~60 d),与Hu等[14]的研究结果一致。此外,相对于钱玮等[15]和胡晓龙等[2]基于DNA水平的研究,本研究检测到更多梭菌属(148个),丰富了对酒醅中梭菌的认识。
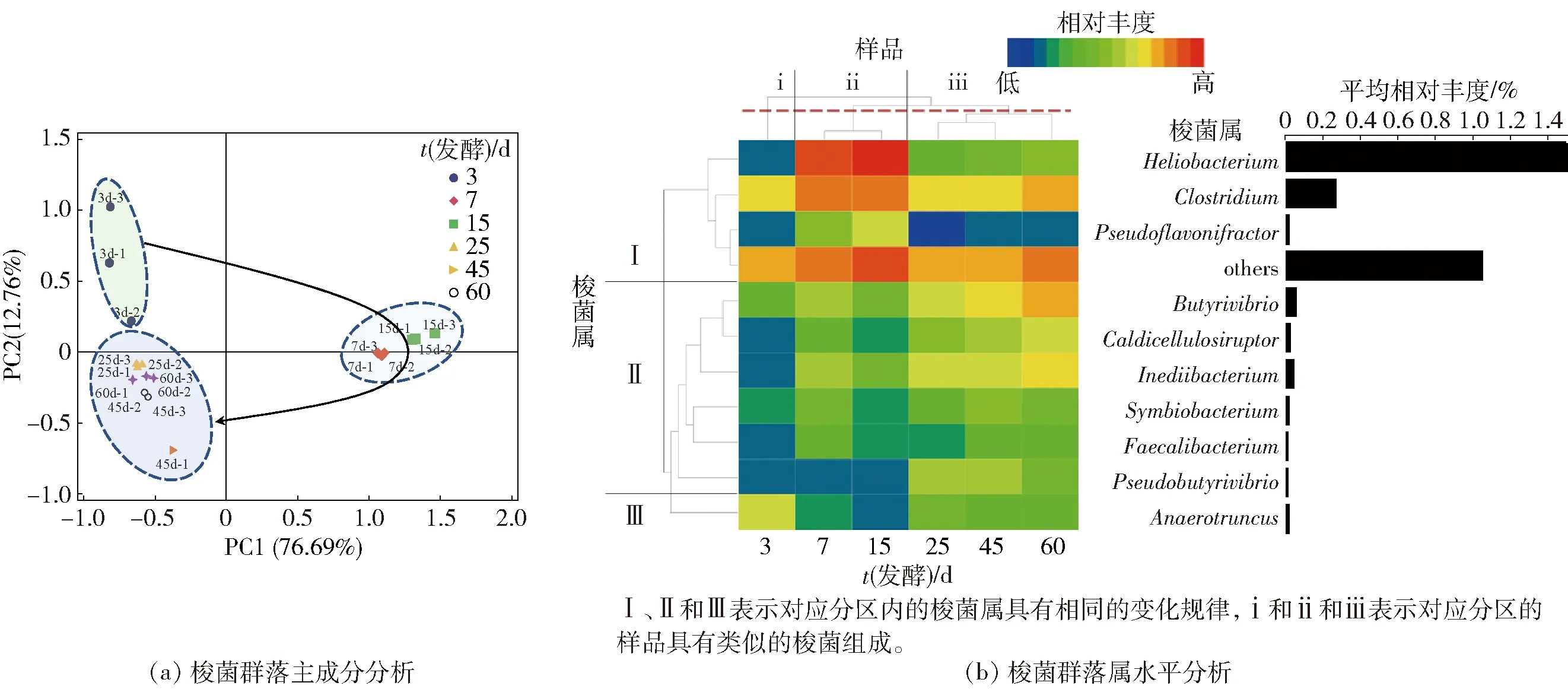
图2 梭菌群落β多样性分析
为进一步了解酒醅样品聚类的原因,挑选相对丰度前10(top10)的可鉴定属进行分析,其他的合并为others,图2(b)展示了这10个属的相对丰度随时间的变化。这10个属的总平均相对丰度占梭菌纲的65.44%,因此可以反映酒醅中梭菌群落水平的整体变化规律,其中仅Heliobacterium(太阳杆菌属)为优势属(1.49%)。从发酵天数上看,所有样品聚为3类,即3 d样品被分为i组,7 d和15 d被分为ii组,其余样品被分为iii组,结果与PCA类似,再次表明发酵时间是影响酒醅梭菌群落组成的重要因素。梭菌群落变化同样可聚为3类,其中,Ⅰ包含Heliobacterium、Clostridium(梭菌属)和Pseudoflavonifractor(假黄铜单胞菌),其相对丰度呈现先增加(3~15 d)后减少的趋势(15~60 d),而马箭等[3]在DNA水平上发现与Clostridium相似的OTU在发酵第7天显著高于其他发酵节点,与本研究结果类似。Ⅱ中6种梭菌的相对丰度在发酵25~45 d时较高,在发酵过程中呈现增加的趋势。Ⅲ中仅包含Anaerotruncus(厌氧棍状菌属),其相对丰度在发酵3 d时最高,随后开始减少。Clostridium是浓香型白酒中研究较多的梭菌纲中的属,研究表明,Clostridium的相对丰度会随着窖泥质量的增加而增加,因此推断其在维护窖泥质量和发酵环境等方面有重要作用[2],Clostridiumsp.W1还能将乙酸和乙醇转换为丁酸和己酸[13]。Pseudoflavonifractor是一种产丁酸盐的细菌,常见于肠道微生物的研究中[16],但很少作为优势菌在白酒研究中有报道,说明到发酵中期的时候,酒醅已经具备了产丁酸和己酸的潜力。Anaerotruncus属于瘤胃菌科,其分类学下的物种,如Anaerotruncuscolihominis、Anaerotruncusmassiliensis能产生乙酸、丁酸和琥珀酸作为最终代谢产物[17-18]。Pseudobutyrivibrio(假丁酸弧菌属)和Butyrivibrio(丁酸弧菌属)是厌氧环境中常见的梭菌,其相对丰度分别在发酵60 d和45 d达到最高。Pidcock等[19]对Pseudobutyrivibrio和Butyrivibrio的71个基因组进行EggNOG注释发现,其功能条目主要集中在碳水化合物转运和代谢以及细胞壁/膜/包膜生物发生上,它们能将大部分遗传能力用于复杂多糖的分解和重组,由此产生的单糖经过发酵产生丁酸盐。
2.2 酒醅梭菌群落的功能特性分析
图3表示使用KEGG数据库进行6个发酵节点酒醅中梭菌富集的基因功能注释结果。在一级代谢通路上,代谢注释到的基因数量最多(3 306个),其次是基因信息(965个)、环境信息过程(711个)、细胞过程(546个)和生物体系统(299个)。在二级通路上,梭菌在碳水化合物代谢(1 013个基因)、全球和概览地图(648个)、翻译(552个)和能量代谢(463个)4个方面的能力最强。酒醅原料中富含大量的碳水化合物,如淀粉、葡萄糖和蔗糖等发酵底物,而梭菌具有广泛的底物利用谱,能通过淀粉和蔗糖代谢、糖酵解和丁酸代谢等途径利用这些底物产生乙酸、丁酸和己酸等重要有机酸,此外碳水化合物代谢通常伴随着能量代谢,从而满足梭菌自身需求[15-16]。梭菌基因的富集结果说明其参与部分或全部次级代谢物的生物合成、碳代谢和脂肪酸代谢等11种途径,对白酒中微量元素和有机酸的产生可能有积极作用。翻译是合成蛋白质的关键步骤,蛋白质合成后经各种化学修饰,从而会形成不同功能的蛋白质参与到梭菌生理代谢中。能量代谢是代谢中的核心问题,贯穿梭菌的整个生命周期,其作用主要是将有机物或无机物转化为通用能源ATP从而维持梭菌的生长代谢。此外,梭菌基因还注释到膜转运基因(401个),说明酒醅中梭菌较为活跃,通过细胞膜进行物质交换的代谢较为频繁;氨基酸代谢也注释到394个基因,其代谢作用主要用于合成自身独特的蛋白质、多肽和其他含氮物质,以产生相应的氮源为微生物利用,同时可利用α-酮酸通过胺化反应转化为糖、脂质或非必需氨基酸,也可参与三羧酸循环完全氧化成CO2和水,并释放出大量能量[20]。酒醅中梭菌活动较为活跃,表达的基因主要参与代谢活动,这可能对浓香型白酒的发酵起到积极作用,特别是对碳水化合物的分解产生重要的有机酸过程。

图3 酒醅中梭菌KEGG功能注释分析
2.3 酒醅中挥发性有机酸变化规律及与梭菌的关联性分析
在酒醅样品共检测到乙酸、丁酸、戊酸、己酸、苯甲酸、苯丙二酸、3-甲基丁酸和2-甲基丙酸8种碳原子数小于10的挥发性有机酸,具有乳酪酸香、奶酪香和甜香等风味,对浓香型白酒的风味均有较大贡献[21]。图4表示这8种酸的相对含量随时间的变化。半定量分析结果表明,苯甲酸是酒醅中相对含量较高的有机酸,且在发酵过程中保持较高的水平。相关研究表明,苯甲酸在白酒中属于相对含量极微的第3类有机酸,质量浓度一般仅为1 mg/L[22];但其在浓香型白酒中的产生机制和影响也未见详细报道。此外,虽然自发发酵产生的微量苯甲酸能给酒体带来芳香味,但其在食品工业上常常添加用作广谱抑菌剂进行防腐[23]。己酸是相对含量第2的有机酸,但其对浓香型白酒风味的贡献最大,经酯化反应形成的己酸乙酯是衡量浓香型白酒质量的重要指标之一,主要呈现出窖香和水果香,其相对含量在发酵第7天增加至最高,发酵15 d和45 d时相对含量也较多。相对含量较多的还有乙酸、丁酸和戊酸,其对应的酯呈现菠萝香、奶酪香和水蜜桃香,但本研究中丁酸和戊酸的相对含量较少,都在发酵7 d达到最大值,发酵25 d最少,而乙酸主要在发酵中后期具有较高含量,特别是发酵45 d。从发酵节点上看,酒醅中有机酸在发酵3 d时含量最低,随发酵的进行各有机酸的相对含量不断发生动态变化,直至发酵60 d时有机酸总相对含量达到最高。乙酸、丁酸、己酸和乳酸通常认为是浓香型白酒的四大酸,但是供试样品中并没有检测到乳酸,这可能是因为乳酸是属于含量较高但是不挥发的酸,在GC-MS的氢离子火焰检测器上无响应,一般需要前衍生或者使用顶空进样,而本研究直接进样无法分析酒醅中的乳酸[24]。

图4 不同发酵节点酒醅中有机酸组成
尽管酒醅中风味物质的产生是多种微生物共同作用的结果,但仍然可以通过spearman相关性分析初步解析top10梭菌对挥发性有机酸形成的贡献,见表1。乙酸与Butyrivibrio、Inediibacterium和Caldicellulosiruptor呈显著正相关(P<0.05)。丁酸分别与Pseudobutyrivibrio和Pseudoflavonifractor呈显著负相关和正相关(P<0.05)。己酸与Heliobacterium和Clostridium呈显著正相关(P<0.05),与Anaerotruncus呈显著负相关(P<0.05)。戊酸与Clostridium和Pseudoflavonifractor呈显著正相关,与Pseudobutyrivibriofractor呈显著负相关。从微生物水平上看,Heliobacterium和Clostridium仅与苯甲酸呈负相关,Pseudobutyrivibrio与苯甲酸和乙酸呈正相关,而Anaerotruncus仅与苯甲酸呈正相关。结合苯甲酸的相对含量以及与梭菌属的关系看,苯甲酸在本研究的样品中很可能是主要由梭菌之外的细菌或真菌产生,而Heliobacterium和Clostridium对有机酸的代谢有重要贡献。
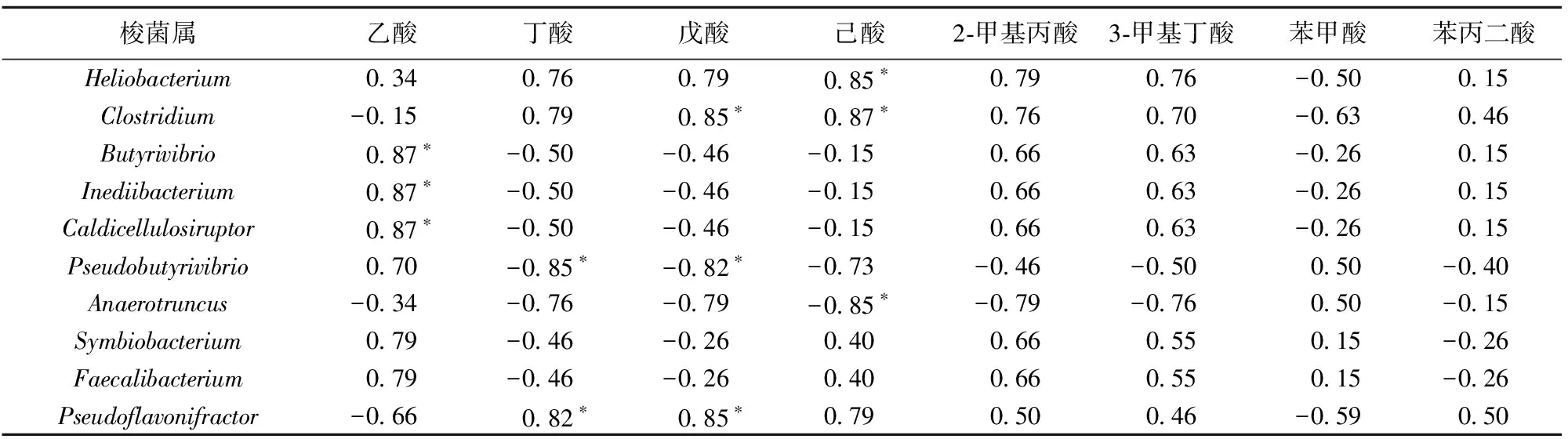
表1 有机酸与优势梭菌相关性分析
2.4 重要有机酸代谢途径分析
有机酸类化合物是浓香型白酒中重要的呈味物质,供试样品中检测到的乙酸、己酸、丁酸是浓香型白酒的主要有机酸[21],因此本研究结合梭菌物种注释结果和KEGG数据库绘制了梭菌参与这3种酸合成的主要途径(图5)。其主要涉及ko00010(糖酵解)、ko00620(丙酮酸代谢)、ko00650(丁酸代谢)和ko00062(脂肪酸链延长)4条途径,涉及了梭菌中12种可鉴定的属及1种不可鉴定属中的25个基因、22个关键酶。其中Clostridium(9个基因)、Ruminiclostridium(2个基因)和Proteiniclasticum(2个基因)是整个代谢过程中主要的贡献菌,图5中热图展示了途径中每一步参与梭菌基因的表达量(以FPKM值衡量)。在糖酵解途径中,来自Herbinix、Proteiniclasticum和Clostridium等7种梭菌的磷酸葡萄糖变位酶、6-磷酸葡萄糖异构酶、6-磷酸果糖激酶和丙酮酸激酶等11种碳水化合物酶将葡萄糖转化为丙酮酸。其中6-磷酸果糖激酶和丙酮酸激酶是2种关键的限速酶,分别对应糖酵解途径中2步不可逆的反应,因此这2种酶具有调节糖酵解速度的作用,不可逆的步骤也保证梭菌能最大程度地利用葡萄糖[25]。在能量代谢方面,1分子葡萄糖分解后净产生2分子ATP,这是微生物在无氧条件下获得能量的重要途径,同时产生一些重要中间体和其他代谢途径关联,其中丙酮酸是重要的中间体之一,可参与到乳酸、乙酸和乙醇等重要的代谢产物的合成途径中。如在丙酮酸脱氢酶和丙酸辅酶A转移酶的催化作用下生成乙酸,这一步反应主要是由Clostridiisalibacter和Clostridium参与完成。丙酮酸在氧化还原酶的作用下还原生成另一个必要的中间体乙酰辅酶A。乙酰辅酶A与酰基转移酶偶联生成双乙酰辅酶A从而进入丁酸代谢途径,这一步仅Oscillibacter(颤杆菌克属)具有相关的基因和酶,表明该梭菌可能是链接丙酮酸代谢和丁酸代谢2个途径的重要梭菌,但Oscillibacter在之前的浓香型白酒研究中鲜有报道。双乙酰辅酶A在一系列酶促反应的作用下得到产生丁酸的关键中间体丁酰辅酶A。丁酰辅酶A在乙酰/双乙酰辅酶A转移酶的作用下将一个CoA转移到乙酸,同时生成丁酸并释放乙酰辅酶A开始乙酸盐延伸。随后乙酰辅酶A与丁酰辅酶A偶联,开始丁酸延伸为己酸的脂肪酸延长途径。在本研究的注释结果中,缺失丁酸代谢途径中的巴豆酰胺辅酶A到丁酰辅酶A的巴豆酰胺辅酶A还原酶及己酸合成相关的酶,这可能是1)受到测序原理和RNA提取误差的影响,从而造成梭菌信息的缺失;2)任聪等[26]和朱晓军[27]的研究发现浓香型白酒产己酸的主体菌并不一定属于梭菌属,甚至可能存在一些尚未报道的产己酸途径;3)这可能与供试酒醅样本中相应梭菌含量较低有关,进而导致不能有效提取其RNA。
在糖酵解途径的5个主要反应步骤中,梭菌属的基因表达量基本在发酵45 d时增加至最大,Clostridium虽然参与到图5中第5步的反应中,但是其基因表达量较低,对丙酮酸的形成可能无明显的促进作用。在乙酸的形成步骤中,Clostridium同样在发酵45 d时具有最大的表达量,乙酸的相对含量也在发酵45 d时最高,表明Clostridium对乙酸的形成具有积极作用,之前的研究也表明Clostridium能将淀粉转化为各种有机酸[13]。在图5中第7步、第8步和丁酸代谢的反应途径中,梭菌的基因表达量均在发酵45 d最高。由此可见,在浓香型白酒发酵45 d时梭菌对乙酸和丁酸的贡献较大,但是对己酸形成的贡献暂不明确。
3 结 论
采用宏转录组学技术解析了浓香型白酒6个发酵节点酒醅中活性梭菌群落及其功能,并结合GC-MS探究酒醅中挥发性有机酸的变化规律及与优势活性梭菌之间的相关性。研究发现:酒醅中活性梭菌群落由6个目、27个科及148个属组成,梭菌群落主要参与到碳水化合物代谢、翻译和能量代谢等过程,极大地丰富了对酒醅中梭菌群落的认识;酒醅中共检测到8种挥发性有机酸,其中乙酸、丁酸和己酸等重要有机酸在发酵7 d和45 d时具有较高的含量;通过KEGG数据库注释发现梭菌纲中的12种可鉴定属及不可鉴定属中的25个基因、22个关键酶参与到糖酵解、丙酮酸代谢和丁酸代谢的途径中,Clostridium是主要的贡献菌,发酵45 d时梭菌具有较大的贡献。本研究从转录层面解析了浓香型发酵酒醅中梭菌的群落多样性以及功能,揭示了酒醅中梭菌群落的主要贡献菌(Clostridium)和贡献节点(45 d),旨在为提高浓香型白酒的质量提供理论支持。
