外显子拼接复合体塑造m6A表观转录组的形成
2023-07-05宋鹏辉马丽娟严冬
宋鹏辉,马丽娟,严冬
外显子拼接复合体塑造m6A表观转录组的形成
宋鹏辉1,马丽娟2,严冬1
1. 复旦大学生命科学学院,遗传工程国家重点实验室,上海 200438 2. 中国科学院分子植物科学卓越创新中心,中国科学院大学,上海 200032
6-甲基腺嘌呤(6-methyladenosine,m6A)是mRNA中含量最丰富的化学修饰之一,在各种生理病理过程中发挥关键性的作用。m6A修饰主要位于mRNA终止密码子附近和长的内部外显子上,然而导致这一特异性分布的机制却一直不清楚。近期发表的3篇论文揭示了外显子拼接复合体(exon junction complexes,EJCs)作为m6A修饰的抑制蛋白suppressors,塑造了m6A表观转录组的形成,解决了这一重大问题。由此,本文简要介绍了m6A修饰通路,并结合这些研究成果阐述了EJC对m6A修饰形成的作用和机理,进而说明外显子-内含子结构通过m6A修饰影响mRNA的稳定性,以期理解m6A这一RNA表观修饰领域的最新进展。
RNA修饰;m6A;外显子拼接复合体;剪接;RNA稳定性
核酸上存在着多种化学修饰,在DNA中,已知有超过17种不同的修饰;RNA中有超过160种化学修饰被发现,而这些修饰的生物学功能才刚开始被揭示,因此RNA修饰成为目前国际研究的热点并形成了一个新的领域,表观转录组学(epitranscriptomics)[1~3]。RNA的化学修饰存在于细菌、古菌和真核生物中,包括对rRNA、tRNA、mRNA以及snRNA等的修饰。常见的RNA修饰包括6-甲基腺嘌呤(6-methyladenosine,m6A)、1-甲基腺嘌呤(1-methyladenosine,m1A)、5-甲基胞嘧啶(5-methylcytidine,m5C)、5-羟甲基胞嘧啶(5-hydroxymethylcytidine,hm5C)、4-乙酰基胞嘧啶(4-acetylcytidine,ac4C)、7-甲基鸟嘌呤(7-methylguanosine,m7G)、假尿嘧啶(pseudouridine,Ψ)和2′-O-甲基化修饰(2′-O-methylation,Nm)等[4,5]。虽然RNA上的修饰早在1957年就被检测到[6],但多年来研究主要集中在含量丰富的rRNA和tRNA修饰[7]。这一领域直到最近十几年才取得了突破性的进展,而m6A作为真核生物mRNA中含量最丰富的内部修饰,在RNA修饰的研究中起着矛头的作用[8,9]。一个重要的进展是发现了RNA修饰通路的核心成员,特别是鉴定了m6A的甲基转移酶和去甲基化酶,使人们意识到RNA修饰是一个动态可逆的过程[10~12]。对这些基因的功能研究发现m6A修饰在干细胞分化、动植物发育与应激、病毒感染和癌症发生等生理病理过程中发挥着关键的作用[13]。另一个主要突破来自于使用高通量测序对全转录组水平上的表观修饰进行定位,从而为关联该RNA修饰与其生物学功能提供了可能[14~16]。m6A修饰在mRNA上的分布具有很强的特异性,在大部分物种中主要富集在mRNA终止密码子(stop codon)附近和内部的长外显子上。由于m6A修饰的共识序列DRACH (D=A/G/U;R=A/G;H=A/C/U)在转录组上均匀分布且只有5%左右的DRACH会被甲基化[13],因此领域里长期的一个谜团就是m6A修饰的特异性是如何达到的。近期发表的3篇论文从不同的途径出发得到类似的结论,基本解决了这一领域中长期存在的重大问题,本文将根据这些论文所报道的研究结果解读外显子拼接复合体如何塑造m6A表观转录组的形成。
1 m6A修饰的调控通路
m6A由甲基转移酶(写入器“writers”)所添加,主要被含有YTH结构域的readers蛋白识别,并且可以被FTO和ALKBH5等去甲基化酶(擦除器“erasers”)去除[17,18]。在m6A writers中,ZCCHC4和METTL5-TRMT112复合物分别负责28S和18S rRNA的m6A甲基化[19,20]。METTL4负责U2 snRNA上m6A的添加[21,22],而METTL16负责U6 snRNA和少数mRNA (如负责S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)生物合成的酶)上的m6A修饰[23~25]。大部分mRNA上的m6A修饰由一个多组分的甲基转移酶复合物(multicomponent methyltransferase complex,MTC)所添加,其中的核心成员为互相稳定的METTL3-METTL14异二聚体[26,27]。METTL3是MTC中唯一具有甲基转移酶催化活性的组分,而METTL14在RNA底物识别中起着结构性的作用[28~30]。由于METTL3的关键作用,目前针对它的小分子抑制剂已被开发出来,在急性髓细胞白血病(acute myeloid leukaemia,AML)小鼠模型中可以抑制白血病细胞的增殖和生长[31],其中STC-15在最近进入临床I期试验用于治疗晚期肿瘤(clinicaltrials.gov,NCT05584111)。此外,蛋白纯化和遗传筛选发现了另外5个在进化中保守的MTC成员,WTAP (Wilms’ tumor 1-associated protein)[32~34]、VIRMA (KIAA1429)[35]、Hakai (E3 ubiquitin ligase CBLL1)[36]、ZC3H13 (zinc finger CCH domain-containing protein)[37~39]和RBM15/15B (RNA- binding motif protein 15/15B)[40]。这些蛋白的缺失会导致mRNA上m6A水平的下降,以及生物体内类似的表型,然而它们在MTC中的确切功能仍不完全清楚。WTAP、VIRMA、Hakai和ZC3H13能形成互相稳定的复合物,WTAP、VIRMA或Hakai的缺失会导致另外3个组分的降解[41,42],而ZC3H13的缺失却引起其他MTC成员的出核[37,41]。最近的冷冻电镜实验解析了包含多个MTC成员的复合物结构,发现WTAP形成同源二聚体,并与VIRMA及ZC3H13的一部分紧密结合[43]。RBM15/15B是RNA结合蛋白,被显示结合在靠近m6A的U-rich区域,因此可能为m6A writer提供一定的特异性[44]。除此之外,组蛋白修饰、转录因子、RNA聚合酶II都被提出可能募集METTL3-METTL14到特定位置提供选择性修饰[45~48],然而所有这些模型都无法充分解释m6A修饰在mRNA终止密码子附近和内部长外显子上的强烈富集。
2 外显子拼接复合体抑制m6A修饰
2023年1月27日,美国芝加哥大学何川课题组在发表论文报道了m6A修饰特异性分布的调控机制[49]。首先,为了无偏差地研究m6A修饰位点周围序列对它的影响,作者开发了可以同时评估数千个设计序列m6A修饰状态的大规模并行检测(massively parallel assay for m6A,MPm6A)手段。他们选取了HeLa细胞中内源的6897个被m6A修饰的DRACH和3058个未被m6A修饰的DRACH,连同每个位点周围的102个核苷酸序列,将它们合成并克隆到报告基因GFP的3′UTR,这些报告质粒被转染至细胞中并通过m6A-seq来检测m6A修饰。92.8%的内源m6A修饰序列在报告系统中仍然被甲基化修饰,有趣的是,90.2%的内源未被m6A修饰的序列在报告系统中展现出显著的甲基化修饰,说明这些位点的m6A修饰在内源情况下被抑制。对这些被抑制的m6A位点分析发现它们富集在编码区(coding sequences,CDS)和3′UTR远离终止密码子的区域,并且在较短的内部外显子上,与内源m6A富集的特征相反。这些结果意味着m6A分布的特异性很可能是选择性抑制,而非过去各种模型提出的选择性激活所造成的。对120个RNA结合蛋白的结合位点分析发现一些剪接体的成员富集在被抑制的m6A附近,暗示剪接可能抑制m6A修饰。随后,作者选择了基因内部外显子上一个被抑制的m6A位点进行了深入分析,通过将该m6A位点周围不同长度的片段放置于报告基因的不同位置,确定了在平均长度外显子中,mRNA的剪接抑制m6A的甲基化。
剪接如何抑制m6A的修饰?外显子拼接复合体(exon junction complexes,EJCs)通过剪接体被组装到mRNA外显子-外显子连接处上游的24个核苷酸处,对mRNA的定位、翻译、降解和剪接都发挥重要的调控作用[50,51](图1)。EJC包含四个核心蛋白:EIF4A3、RBM8A/Y14、Magoh、MLN51/Barentsz以及一系列具有不同功能的外围相关因子。作者推断平均长度的内部外显子中被抑制的m6A位点与EJC接近,而长内部外显子和终止密码子附近的m6A位点距离EJC较远,因此对EJC在m6A修饰中的作用展开了研究。在HeLa细胞中使用siRNA对EJC核心因子EIF4A3敲低(knock down,KD)产生了惊人的结果,m6A-MeRIP-seq显示24,350个区域在KD中出现显著的高甲基化,而只有3140个区域出现低甲基化。同样,超高效液相色谱-串联质谱检测发现KD导致mRNA上的m6A总体修饰水平增加一倍。另一个EJC核心因子KD展现出与一致,尽管较弱的对m6A修饰的影响。大部分在KD或KD中新出现的m6A峰与内源性的m6A峰并不重合,并且富集在内部平均长度的外显子上,说明它们是从原来那些被抑制的m6A位点活化而来。这些结果证实了之前的假设,说明剪接体招募的EJC广泛抑制附近m6A位点的甲基化,从而导致m6A修饰分布的特异性。
m6A修饰影响着mRNA加工代谢的各个方面,如剪接、定位、翻译和稳定性等[13,52]。在KD细胞中,绝大部分(~90%)高甲基化的mRNA半衰期降低,与m6A主要促进mRNA降解的功能相一致。这一功能主要是通过reader蛋白YTHDF2实现的,YTHDF2 CLIP-seq实验显示YTHDF2与高甲基化mRNA的结合普遍增加,同时伴随着mRNA半衰期的降低。有趣的是,虽然KD导致绝大部分高甲基化的mRNA稳定性降低,却有一小部分高甲基化的mRNA稳定性增加,例如,这是由于与另一个m6A reader蛋白IGF2BP结合造成的。随后,作者分析了25个不同人体组织及小鼠组织数据,发现表达水平与整体m6A修饰水平呈负相关,例如脑组织具有最低的表达和最高的m6A水平。最后,他们研究了EJC外围相关因子RNPS1,以及它所介导的mRNA高维度包装功能[53,54]在m6A修饰中的作用。KD也造成大量的m6A甲基化增强,虽然效果弱于两个EJC核心蛋白,但是这些高甲基化区域和KD高甲基化区域之间高度重叠(45%)。相反,敲低另一个控制无义介导的mRNA降解(nonsense-mediated mRNA decay,NMD)的EJC因子UPF1则不会引起m6A修饰的变化。
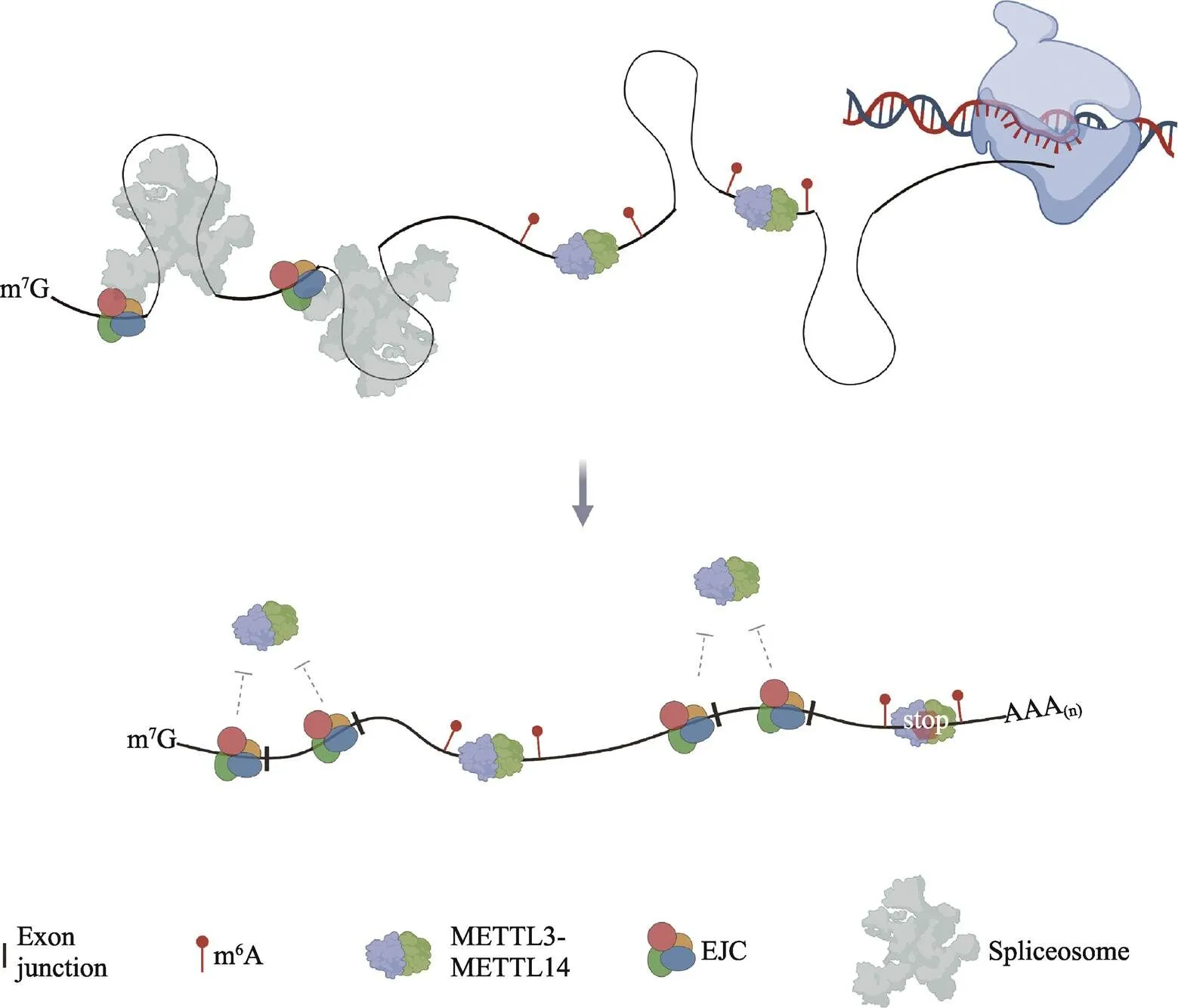
图1 EJCs选择性抑制m6A修饰的模型图
在mRNA成熟过程中,剪接体(spliceosome)将EJC放置于外显子连接处(exon junction)上游的24个核苷酸处。在平均长度内部外显子上,EJC的结合阻止了m6A MTC的作用;在长内部外显子和终止密码子附近,m6A MTC可以添加m6A。为简单起见,m6A MTC只显示了核心成员METTL3-METTL14。本图通过BioRender.com绘制。
2022年12月23日,在另一篇美国波士顿儿童医院Richard I. Gregory课题组发表的论文中,作者从富集新生RNA的染色质相关RNA (chromatin-associated RNA,caRNA)入手进行研究[55]。他们发现与成熟的polyA+RNA相比,caRNA上的m6A分布更加均匀,不具有成熟mRNA特有的终止密码子附近的强富集,说明mRNA在成熟过程中m6A修饰发生了动态变化。作者推断这可能与m6A去甲基化酶有关,接下来的免疫沉淀与质谱实验发现ALKBH5,而不是FTO,与多个剪接因子相互作用,而其中与ALKBH5作用最强的就是EJC核心蛋白EIF4A3。随后,他们也使用siRNA对ALKBH5和EIF4A3进行了敲低,有趣的是,虽然KD对m6A的总体水平影响很小,KD却导致m6A水平的大幅增加。MeRIP-seq显示KD引起43,744个区域的高甲基化和8392个区域的低甲基化,而高甲基化的区域主要富集在短的内部外显子上及靠近剪接位点的位置。这些结果与He等[49]的非常一致,表明EIF4A3在外显子拼接处抑制m6A的添加,而这一过程与m6A去甲基化酶无关。
为进一步揭示EIF4A3作用的机理,他们又进行了METTL3 eCLIP-seq实验,发现METTL3的结合在KD细胞中增加,并且METTL3结合增加的区域与KD细胞中m6A水平增高的区域存在正相关关系。他们选取了其中的几个区域构建了报告系统,证实了EIF4A3对METTL3结合和m6A水平的影响取决于剪接。最后,通过MS2技术直接将EIF4A3定位到外显子拼接位置可以抑制METTL3的结合和m6A的添加。这些结果说明EJC,特别是EIF4A3,在外显子-外显子边界附近通过阻断METTL3介导的m6A修饰,从而塑造m6A表观转录组的形成。
2023年1月19日,在以色列魏茨曼科学研究所Schraga Schwartz和Boris Slobodin课题组发表的论文中,作者通过大规模平行的报告基因检测,系统地探究了7584个不同序列的m6A甲基化状态,发现DRACH共识序列似乎是m6A修饰既必要又充分的条件[56]。因此作者生成了一个预测m6A分布的计算模型“m6Apred-1”,其中每个符合条件的DRACH序列(共7种)都会获得固定水平的m6A甲基化。有趣的是,m6Apred-1可以很好地预测无内含子基因的m6A甲基化,但是不能预测含有内含子基因的m6A甲基化,暗示内含子抑制m6A的生成。对多个公开发表的单核苷酸分辨率m6A- miCLIP数据分析发现离外显子拼接位点~200核苷酸内存在m6A禁区(m6A exclusion zone)。将这一点考虑进来(在外显子/内含子连接处设置100核苷酸不能被m6A修饰的禁区),他们更新了计算模型“m6Apred-2”,这一新模型的预测结果与实验测量的m6A修饰峰具有很好的吻合度,证实了外显子-内含子结构在塑造m6A分布中的重要作用。
接下来作者利用m6Apred-2的预测结果,以及已有的和新生成的数据,分析了外显子密度、RNA稳定性、和m6A修饰水平的关系。m6A水平与RNA稳定性呈负相关,外显子密度与m6A水平呈负相关,而外显子密度与RNA稳定性呈正相关,这一相关性在METTL3敲除或药物干扰情况下被剥夺,说明m6A修饰是外显子密度和RNA稳定性之间的联系机制。最后,他们通过另一种手段dTAG degron对RBM8A/Y14蛋白进行降解,发现m6A禁区内,而不是禁区外的m6A水平增加。在Y14降解情况下,m6Apred-2的预测准确性下降,而m6Apred-1的预测准确性上升,表明EJC蛋白负责在外显子边界建立m6A禁区。总之,Uzonyi等[56]提出m6A的分布依赖于共识序列和外显子-内含子结构,DRACH序列默认为被m6A修饰,除非它们靠近EJC所结合的外显子拼接位点。
3 结语与展望
上述三篇论文各具特色,共同解析了m6A修饰分布的机制问题。过去已知的m6A通路成员主要分为三类:writers,readers和erasers。这几篇论文提出了一类全新的m6A效应蛋白:suppressors,为m6A修饰的研究提供了新的方向。EJC作为主要的suppressor,对m6A表观转录组的塑造起着关键性的作用。在其他物种,特别是植物中,EJC对m6A修饰是否起着类似的作用?EJC影响着mRNA生命周期的各个方面,在表型分析中如何将EJC对m6A修饰的作用与其他功能分开?过去研究表明m6A修饰可以影响mRNA剪接[40,57],那m6A修饰和mRNA剪接发生的先后顺序如何?m6A修饰是否影响EJC介导的其他过程,例如NMD?除了EJC之外,还有哪些suppressor蛋白?这些都是未来值得探究的问题。
从这些论文中得到的另一个启示是基因结构,如外显子的数量和长度以及UTR长度,对基因表达具有重要的调控作用。这一发现具有很强的理论和实际意义,例如在科学实验和基因治疗中,使用cDNA作为表达载体可能与内源性的基因表达产生不同的效果;另一方面,改变外显子-内含子结构,5′和3′ UTR长度可以影响m6A修饰,并最终调节mRNA的稳定性。在不同物种中,基因结构如外显子长度和数量存在差异,m6A修饰图谱也有不同之处,今后可以进一步解析进化中m6A修饰图谱与基因结构的关系。
[1] Hsu PJ, Shi HL, He C. Epitranscriptomic influences on development and disease.,2017, 18(1): 197.
[2] Zhao LY, Song JH, Liu YB, Song CX, Yi CQ. Mapping the epigenetic modifications of DNA and RNA.,2020, 11(11): 792–808.
[3] Yang Y, Chen YS, Sun BF, Yang YG. RNA methylation: regulations and mechanisms., 2018, 40(11): 964–976. 杨莹, 陈宇晟, 孙宝发, 杨运桂. RNA甲基化修饰调控和规律. 遗传, 2018, 40(11): 964–976.
[4] Jonkhout N, Tran J, Smith MA, Schonrock N, Mattick JS, Novoa EM. The RNA modification landscape in human disease.,2017, 23(12): 1754–1769.
[5] Boccaletto P, Stefaniak F, Ray A, Cappannini A, Mukherjee S, Purta E, Kurkowska M, Shirvanizadeh N, Destefanis E, Groza P, Avşar G, Romitelli A, Pir P, Dassi E, Conticello SG, Aguilo F, Bujnicki JM. MODOMICS: a database of RNA modification pathways. 2021 update.,2022, 50(D1): D231–D235.
[6] Davis FF, Allen FW. Ribonucleic acids from yeast which contain a fifth nucleotide., 1957, 227(2): 907–915.
[7] Suzuki T. The expanding world of tRNA modifications and their disease relevance.,2021, 22(6): 375–392.
[8] Sendinc E, Shi Y. RNA m6A methylation across the transcriptome.,2023, 83(3): 428–441.
[9] Zhang X, Jia GF. RNA epigenetic modification:N-methyladenosine., 2016, 38(4): 275–288. 张笑, 贾桂芳. RNA表观遗传修饰:N-甲基腺嘌呤. 遗传, 2016, 38(4): 275–288.
[10] Bokar JA, Shambaugh ME, Polayes D, Matera AG, Rottman FM. Purification and cDNA cloning of the AdoMet- binding subunit of the human mRNA (N6-adenosine)- methyltransferase.,1997, 3(11): 1233–1247.
[11] Jia GF, Fu Y, Zhao X, Dai Q, Zheng GQ, Yang Y, Yi CQ, Lindahl T, Pan T, Yang YG, He C.6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO.,2011, 7(12): 885–887.
[12] Zheng GQ, Dahl JA, Niu YM, Fedorcsak P, Huang CM, Li CJ, Vågbø CB, Shi Y, Wang WL, Song SH, Lu ZK, Bosmans RPG, Dai Q, Hao YJ, Yang X, Zhao WM, Tong WM, Wang XJ, Bogdan F, Furu K, Fu Y, Jia GF, Zhao X, Liu J, Krokan HE, Klungland A, Yang YG, He C. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility.,2013, 49(1): 18–29.
[13] He PC, He C. m6A RNA methylation: from mechanisms to therapeutic potential.,2021, 40(3): e105977.
[14] Meyer KD, Saletore Y, Zumbo P, Elemento O, Mason CE, Jaffrey SR. Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons.,2012, 149(7): 1635–1646.
[15] Dominissini D, Moshitch-Moshkovitz S, Schwartz S, Salmon-Divon M, Ungar L, Osenberg S, Cesarkas K, Jacob-Hirsch J, Amariglio N, Kupiec M, Sorek R, Rechavi G. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq.,2012, 485(7397): 201–206.
[16] Schwartz S, Agarwala SD, Mumbach MR, Jovanovic M, Mertins P, Shishkin A, Tabach Y, Mikkelsen TS, Satija R, Ruvkun G, Carr SA, Lander ES, FinkGR, Regev A. High-resolution mapping reveals a conserved, widespread, dynamic mRNA methylation program in yeast meiosis.,2013, 155(6): 1409–1421.
[17] Yang Y, Hsu PJ, Chen YS, Yang YG. Dynamic transcriptomic m6A decoration: writers, erasers, readers and functions in RNA metabolism.,2018, 28(6): 616–624.
[18] Meyer KD, Jaffrey SR. Rethinking m6A readers, writers, and erasers.,2017, 33: 319–342.
[19] van Tran N, Ernst FGM, Hawley BR, Zorbas C, Ulryck N, Hackert P, Bohnsack KE, Bohnsack MT, Jaffrey SR, Graille M, Lafontaine DLJ. The human 18S rRNA m6A methyltransferase METTL5 is stabilized by TRMT112.,2019, 47(15): 7719–7733.
[20] Ma HH, Wang XY, Cai JB, Dai Q, Natchiar SK, Lv R, Chen K, Lu ZK, Chen H, Shi YG, Lan F, Fan J, Klaholz BP, Pan T, Shi Y, He C.N-Methyladenosine methyltransferase ZCCHC4 mediates ribosomal RNA methylation.,2019, 15(1): 88–94.
[21] Chen H, Gu L, Orellana EA, Wang YY, Guo JJ, Liu Q, Wang LF, Shen ZF, Wu H, Gregory RI, Xing Y, Shi Y. METTL4 is an snRNA m6Am methyltransferase that regulates RNA splicing.,2020, 30(6): 544–547.
[22] Goh YT, Koh CWQ, Sim DY, Roca X, Goh WSS. METTL4 catalyzes m6Am methylation in U2 snRNA to regulate pre-mRNA splicing.,2020, 48(16): 9250–9261.
[23] Warda AS, Kretschmer J, Hackert P, Lenz C, Urlaub H, Höbartner C, Sloan KE, Bohnsack MT. Human METTL16 is aN-methyladenosine (m6A) methyltransferase that targets pre-mRNAs and various non-coding RNAs.,2017, 18(11): 2004–2014.
[24] Pendleton KE, Chen BB, Liu KQ, Hunter OV, Xie Y, Tu BP, Conrad NK. The U6 snRNA m6A methyltransferase METTL16 regulates SAM synthetase intron retention.,2017, 169(5): 824–835.e814.
[25] Mendel M, Delaney K, Pandey RR, Chen KM, Wenda JM, Vågbø CB, Steiner FA, Homolka D, Pillai RS. Splice site m6A methylation prevents binding of U2AF35 to inhibit RNA splicing.,2021, 184(12): 3125–3142.e3125.
[26] Liu JZ, Yue YN, Han DL, Wang X, Fu Y, Zhang L, Jia GF, Yu M, Lu ZK, Deng X, Chen WZ, He C. A METTL3- METTL14 complex mediates mammalian nuclear RNAN-adenosine methylation.,2014, 10(2): 93–95.
[27] Bokar JA, Rath-Shambaugh ME, Ludwiczak R, Narayan P, Rottman F. Characterization and partial purification of mRNA N6-adenosine methyltransferase from HeLa cell nuclei. Internal mRNA methylation requires a multisubunit complex.,1994, 269(26): 17697–17704.
[28] Wang P, Doxtader KA, Nam YS. Structural basis for cooperative function of Mettl3 and Mettl14 methyltransferases.,2016, 63(2): 306–317.
[29] Wang X, Feng J, Xue Y, Guan ZY, Zhang DL, Liu Z, Gong Z, Wang Q, Huang JB, Tang C, Zou TT, Yin P. Structural basis ofN-adenosine methylation by the METTL3- METTL14 complex.,2016, 534(7608): 575–578.
[30] Śledź P, Jinek M. Structural insights into the molecular mechanism of the m6A writer complex.,2016, 5: e18434.
[31] Yankova E, Blackaby W, Albertella M, Rak J, De Braekeleer E, Tsagkogeorga G, Pilka ES, Aspris D, Leggate D, Hendrick AG, Webster NA, Andrews B, Fosbeary R, Guest P, Irigoyen N, Eleftheriou M, Gozdecka M, Dias JML, Bannister AJ, Vick B, Jeremias I, Vassiliou GS, Rausch O, Tzelepis K, Kouzarides T. Small-molecule inhibition of METTL3 as a strategy against myeloid leukaemia.,2021, 593(7860): 597–601.
[32] Zhong SL, Li HY, Bodi Z, Button J, Vespa L, Herzog M, Fray RG. MTA is an Arabidopsis messenger RNA adenosine methylase and interacts with a homolog of a sex-specific splicing factor.,2008, 20(5): 1278–1288.
[33] Ping XL, Sun BF, Wang L, Xiao W, Yang X, Wang WJ, Adhikari S, Shi Y, Lv Y, Chen YS, Zhao X, Li A, Yang Y, Dahal U, Lou XM, Liu X, Huang J, Yuan WP, Zhu XF, Cheng T, Zhao YL, Wang XQ, Rendtlew Danielsen JM, Liu F, Yang YG. Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase.,2014, 24(2): 177–189.
[34] Horiuchi K, Kawamura T, Iwanari H, Ohashi R, Naito M, Kodama T, Hamakubo T: Identification of Wilms' tumor 1-associating protein complex and its role in alternative splicing and the cell cycle.,2013, 288(46): 33292–33302.
[35] Yue YN, Liu J, Cui XL, Cao J, Luo GZ, Zhang ZZ, Cheng T, Gao MS, Shu X, Ma HH, Wang FQ, Wang XX, Shen S, Wang YZ, Feng XH, He C, Liu JZ. VIRMA mediates preferential m6A mRNA methylation in 3'UTR and near stop codon and associates with alternative polyadenylation.,2018, 4: 10.
[36] Růžička K, Zhang M, Campilho A, Bodi Z, Kashif M, Saleh M, Eeckhout D, El-Showk S, Li HY, Zhong SL, De Jaeger G, Mongan NP, Hejátko J, Helariutta Y, Fray RG. Identification of factors required for m6A mRNA methylation in Arabidopsis reveals a role for the conserved E3 ubiquitin ligase HAKAI.,2017, 215(1): 157–172.
[37] Wen J, Lv RT, Ma HH, Shen HJ, He CX, Wang JH, Jiao FF, Liu H, Yang PY, Tan L, Lan F, Shi YG, He C, Shi Y, Diao JB. Zc3h13 regulates nuclear RNA m6A methylation and mouse embryonic stem cell self-renewal.,2018, 69(6): 1028–1038.e1026.
[38] Guo J, Tang HW, Li J, Perrimon N, Yan D. Xio is a component of the Drosophila sex determination pathway and RNAN-methyladenosine methyltransferase complex.,2018, 115(14): 3674–3679.
[39] Knuckles P, Lence T, Haussmann IU, Jacob D, Kreim N, Carl SH, Masiello I, Hares T, Villasenor R, Hess D, Andrade-Navarro MA, Biggiogera M, Helm M, Soller M, Bühler M, Roignant JY. Zc3h13/Flacc is required for adenosine methylation by bridging the mRNA-binding factor Rbm15/Spenito to the m6A machinery component Wtap/Fl(2)d.,2018, 32(5–6): 415–429.
[40] Lence T, Akhtar J, Bayer M, Schmid K, Spindler L, Ho CH, Kreim N, Andrade-Navarro MA, Poeck B, Helm M, Roignant JY. m6A modulates neuronal functions and sex determination in Drosophila.,2016, 540(7632): 242–247.
[41] Wang YH, Zhang LF, Ren H, Ma LJ, Guo J, Mao DC, Lu ZW, Lu LJ, Yan D. Role of Hakai in m6A modification pathway in Drosophila.,2021, 12(1): 2159.
[42] Bawankar P, Lence T, Paolantoni C, Haussmann IU, Kazlauskiene M, Jacob D, Heidelberger JB, Richter FM, Nallasivan MP, Morin V, Kreim N, Beli P, Helm M, Jinek M, Soller M, Roignant JY. Hakai is required for stabilization of core components of the m6A mRNA methylation machinery.,2021, 12(1): 3778.
[43] Su SC, Li SS, Deng T, Gao MS, Yin Y, Wu BX, Peng C, Liu JZ, Ma JB, Zhang KM. Cryo-EM structures of human m6A writer complexes.,2022, 32(11): 982–994.
[44] Patil DP, Chen CK, Pickering BF, Chow A, Jackson C, Guttman M, Jaffrey SR. m6A RNA methylation promotes XIST-mediated transcriptional repression.,2016, 537(7620): 369–373.
[45] Huang HL, Weng HY, Zhou KR, Wu T, Zhao BS, Sun ML, Chen ZH, Deng XL, Xiao G, Auer F, Klemm L, Wu HZ, Zuo ZX, Qin X, Dong YZ, Zhou YL, Qin HJ, Tao S, Du J, Liu J, Lu ZK, Yin H, Mesquita A, Yuan CL, Hu YC, Sun WJ, Su R, Dong L, Shen C, Li CY, Qing Y, Jiang X, Wu XW, Sun M, Guan JL, Qu LH, Wei MJ, Müschen M, Huang G, He C, Yang JH, Chen JJ. Histone H3 trimethylation at lysine 36 guides m6A RNA modification co-transcriptionally.2019, 567(7748): 414–419.
[46] Slobodin B, Han RQ, Calderone V, Vrielink JAF, Loayza-Puch F, Elkon R, Agami R. Transcription impacts the efficiency of mRNA translation via co-transcriptionalN-adenosine methylation.,2017, 169(2): 326– 337.e312.
[47] Barbieri I, Tzelepis K, Pandolfini L, Shi JW, Millán- Zambrano G, Robson SC, Aspris D, Migliori V, Bannister AJ, Han N, De Braekeleer E, Ponstingl H, Hendrick A, Vakoc CR, Vassiliou GS, Kouzarides T. Promoter-bound METTL3 maintains myeloid leukaemia by m6A-dependent translation control.,2017, 552(7683): 126–131.
[48] Bertero A, Brown S, Madrigal P, Osnato A, Ortmann D, Yiangou L, Kadiwala J, Hubner NC, de Los Mozos IR, Sadée C, Lenaerts AS, Nakanoh S, Grandy R, Farnell E, Ule J, Stunnenberg HG, Mendjan S, Vallier L. The SMAD2/3 interactome reveals that TGFbeta controls m6A mRNA methylation in pluripotency.,2018, 555(7695): 256–259.
[49] He PC, Wei JB, Dou XY, Harada BT, Zhang ZJ, Ge RQ, Liu C, Zhang LS, Yu XB, Wang S, Lyu R, Zou ZY, Chen MJ, He C. Exon architecture controls mRNA m6A suppression and gene expression.,2023, 379(6633): 677–682.
[50] Le Hir H, Saulière J, Wang Z. The exon junction complex as a node of post-transcriptional networks.,2016, 17(1): 41–54.
[51] Boehm V, Gehring NH. Exon junction complexes: supervising the gene expression assembly line.,2016, 32(11): 724–735.
[52] Zaccara S, Ries RJ, Jaffrey SR. Reading, writing and erasing mRNA methylation.,2019, 20(10): 608–624.
[53] Singh G, Kucukural A, Cenik C, Leszyk JD, Shaffer SA, Weng ZP, Moore MJ. The cellular EJC interactome reveals higher-order mRNP structure and an EJC-SR protein nexus.,2012, 151(4): 750–764.
[54] Mabin JW, Woodward LA, Patton RD, Yi ZX, Jia MX, Wysocki VH, Bundschuh R, Singh G. The exon junction complex undergoes a compositional switch that alters mRNP structure and nonsense-mediated mRNA decay Activity.,2018, 25(9): 2431–2446.e2437.
[55] Yang X, Triboulet R, Liu Q, Sendinc E, Gregory RI. Exon junction complex shapes the m6A epitranscriptome.,2022, 13(1): 7904.
[56] Uzonyi A, Dierks D, Nir R, Kwon OS, Toth U, Barbosa I, Burel C, Brandis A, Rossmanith W, Le Hir H, Slobodin B, Schwartz S. Exclusion of m6A from splice-site proximal regions by the exon junction complex dictates m6A topologies and mRNA stability.,2023, 83(2): 237–251.e237.
[57] Haussmann IU, Bodi Z, Sanchez-Moran E, Mongan NP, Archer N, Fray RG, Soller M. m6A potentiates Sxl alternative pre-mRNA splicing for robust Drosophila sex determination.,2016, 540(7632): 301–304.
Exon junction complex modulates the formation of the m6A epitranscriptome
Penghui Song1, Lijuan Ma2, Dong Yan1
6-methyladenosine (m6A) is one of the most abundant chemical modifications in mRNA and plays essential roles in diverse physiological and pathological processes. m6A is highly enriched near stop codons and in long internal exons of mRNA, but the mechanism leading to this specific distribution has been unclear. Recently, three papers have solved this major problem by revealing that exon junction complexes (EJCs) act as m6A suppressors and shape the formation of the m6A epitranscriptome. Here, we briefly introduce the m6A pathway, elaborate the roles of EJC on the formation of m6A modification based on these results, and describe the effect of exon-intron structure on mRNA stabilitym6A, which will help us better understand the latest progress in the m6A RNA modification field.
RNA modification; m6A; exon junction complexes; splicing; RNA stability
2023-03-09;
2023-05-17;
2023-05-22
国家自然科学基金项目(编号:31970786,32270868)资助[Supported by the National Natural Science Foundation of China (Nos. 31970786, 32270868)
宋鹏辉,在读博士研究生,专业方向:遗传学。E-mail: 21110700039@m.fudan.edu.cn
马丽娟,在读博士研究生,专业方向:生物化学和分子生物学。E-mail: malijuan@cemps.ac.cn
宋鹏辉和马丽娟并列第一作者。
严冬,博士,研究员,研究方向:发育生物学。E-mail: yandong@fudan.edu.cn
10.16288/j.yczz.23-051
(责任编辑: 韩玉波)
