金鱼草RADIALIS-like 1基因克隆与功能研究
2023-07-05李菲菲郝燕敏崔敏龙朴春兰
李菲菲,郝燕敏,崔敏龙,朴春兰
金鱼草RADIALIS-like 1基因克隆与功能研究
李菲菲,郝燕敏,崔敏龙,朴春兰
浙江农林大学园艺科学学院,浙江省山区农业高效绿色生产协同创新中心,杭州 311300
MYB是植物中最大的转录因子家族之一,其中R3-MYB转录因子()在金鱼草()花发育过程中具有十分重要的作用。本研究对金鱼草基因组进行分析,发现1个与结构相似的R3-MYB基因,将其命名为()。进一步通过生物信息学预测基因功能,利用qRT-PCR分析在野生型金鱼草不同组织器官中的相对表达量,对过表达的转基因金鱼草进行形态学观察和组织学染色分析。结果表明,基因开放阅读框(open reading frame,ORF)长度为306 bp,编码101个氨基酸,具有典型的SANT结构域,C末端含有CREB motif,与番茄()同源性较高。qRT-PCR结果表明,在根、茎、叶和花中均有表达,其中在花中表达量较高;进一步分析其在不同花器官中的表达量差异,发现在心皮中表达最高。转基因金鱼草植株的组织学染色分析结果显示,与野生型相比,转基因植株的心皮细胞大小没有明显的变化,但心皮中胎座区域变小,细胞数目减少。综上所述,可能参与调控心皮发育,但在心皮中的具体作用机制还有待进一步研究。
金鱼草;like基因;花发育;表达分析;组织学染色
开花是被子植物生长发育的关键步骤,花发育分子调控网络精密且复杂,其中转录因子发挥着重要作用[1]。MYB是植物中最大的转录因子基因家族之一,因含有不同数量的重复结构域,而具有结合不同DNA的能力,在植物生长发育等生物学过程中具有广泛的功能[2]。研究发现,MYB转录因子主要通过调控上下游靶基因的转录表达,参与相应的生理生化反应,进而影响植物的花发育过程[3]。
金鱼草()是重要的观赏植物,又是分子生物学研究中重要的模式植物之一。金鱼草由四轮花器官组成,同一轮花器官根据位置和形态的不同呈现出不同的特征,花冠整体表现为两侧对称。前人通过对金鱼草突变体的研究,发现了转座子诱变体系,并提出控制花器官发育的ABC模型,总结了花不对称性的调控网络,因此金鱼草是研究花发育的理想植物[4~6]。研究表明,MYB转录因子在金鱼草花色形成和花形态发育中具有重要的调控作用,其中R2R3-MYB转录因子主要参与调控花色变化,、和基因通过影响花青素生物合成中不同的结构基因进而控制花青素色素沉着的空间分布和强度[7];类基因调控花瓣中圆锥形表皮细胞形状和毛状体的形成,影响金鱼草的花色[8]。R3-MYB是维持金鱼草两侧对称花形成的关键调节因子。()基因编码一种I-box like蛋白,是金鱼草中首个被报道的R3-MYB转录因子,作为调控花不对称性的关键基因,在维持花的形态建成方面具有重要作用[5,9]。基因最早在裸子植物中被发现,随着基因在进化过程中经历广泛的重复,类基因出现新的表达模式,功能趋于多样化。
金鱼草基因组序列的测定,为金鱼草的生长发育、进化起源和遗传调控等相关分子研究提供了理论依据[10]。是金鱼草中唯一确定的参与花发育的R3-MYB基因,但目前-like基因相关研究较少。因此,为了解-like基因的功能多样性,本研究通过对金鱼草基因组数据库进行分析,发现1个与高度相似的基因,其具有典型的R3-MYB结合域,命名为()。进一步利用系统进化树、多序列比对和组织特异性表达等方法,分析了在金鱼草选择进化过程中功能的保守性和差异性,并通过构建过表达载体及遗传转化等方法获得转基因植株,对转基因金鱼草进行DNA鉴定、qRT-PCR分析、表型观察及组织切片染色等,初步探究的功能。本研究旨在完善-like基因的功能研究,为探索金鱼草花发育提供新的思考。
1 材料与方法
1.1 植物材料
以实验室保存的金鱼草种质资源JI7和Am223为实验材料,其中JI7为基因组测序品种,用于生物信息学分析、基因克隆和表达分析;Am223遗传转化体系较为完善,用于金鱼草的遗传转化。植物材料种植于24℃、光周期16 h/8 h的人工气候室。分别取生长状态一致的JI7金鱼草不同组织部位(根、茎、叶、花)、stage13[11]各花器官(萼片、花瓣、雄蕊、心皮),取材后液氮速冻,保存于–80℃备用。
1.2 AmRADL1基因克隆
利用Eastep®Super总RNA提取试剂盒(上海普洛麦格生物产品有限公司)分别提取JI7金鱼草不同组织部位及花器官的总RNA,并用质量分数为1.5%琼脂糖凝胶电泳以及溴化乙锭染色鉴定分析RNA样品提取质量,利用NanoDrop2000超微量紫外分光光度计(美国赛默飞世尔科技公司)对提取RNA样品的浓度和纯度进行检测,获得的RNA样品保存于–80℃。利用®First-Strand cDNA Synthesis SuperMix试剂盒(北京全式金生物技术有限公司)反转录获得cDNA,储存于–20℃备用。
通过金鱼草基因组测序分析获得基因序列,根据基因的开放阅读框(open reading frame,ORF)设计扩增引物(表1),并利用NEBcutter V2.0 (http://nc2.neb.com/NEBcutter2/)网站分析基因序列中潜在酶切位点,在基因克隆引物5'端和3'端分别添加H I (GGATCC)和I (GAGCTC)酶切位点及保护碱基。以JI7金鱼草叶片cDNA为模板,进行PCR扩增。PCR扩增体系均为:95℃预变性3 min;95℃变性40 s,60℃退火40 s,72℃延伸1 min,35个循环;72℃总延伸10 min。PCR扩增片段利用Wizard®SV. Gel and PCR Clean-Up System试剂盒(上海普洛麦格生物产品有限公司)进行胶回收与分离纯化,连接至pEASY-Blunt Simple载体(北京全式金生物技术有限公司),而后转化到大肠杆菌()DH5α感受态细胞中(上海唯地生物技术有限公司),挑单菌落进行菌落PCR验证,若目标条带大小与预期相符,将相应的菌液进行测序以获得扩增基因的CDS序列。
1.3 AmRADL1生物信息学分析
将的CDS序列在NCBI的BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)子程序中进行同源基因检索,并下载同源性较高基因的氨基酸FASTA格式文件。利用Clustal W(2.0)[12]进行多序列比对,在线网站NCBI Conserved Domains Database (https://www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb.cgi)和MEME(https://meme-suite.org/meme/)预测保守结构域(domain)和基序(motif)。通过MEGA-X软件[13]的邻接法(Neighbor-Joining,NJ)构建系统进化树,步长检验参数Bootstrap为1000次。
1.4 AmRADL1表达模式分析
选择金鱼草泛素延伸蛋白基因()为内参基因,根据实时荧光定量PCR (qRT-PCR)引物设计原则进行引物设计(表1)。参考®Tip Green qPCR SuperMix试剂盒(北京全式金生物技术有限公司)说明书,利用Eva Green荧光染料法,以金鱼草不同组织及花器官的cDNA (稀释3倍)为模板进行qRT-PCR反应。反应体系:1 μL cDNA、5 μL Eva Green Mix、上下游引物(10 μmol/L)各0.2 μL、Nuclease-free water 3.6 μL。运用高扩增效率三步法,qRT-PCR反应程序为95℃预变性10 min,95℃变性10 s,60℃退火15 s,72℃延伸25 s,45个循环。每个样品进行3次技术重复,利用Excel进行数据分析,基因相对表达量使用2–∆∆Ct法计算,使用SPSS 25.0软件进行单因素方差分析(ANOVA)计算基因间差异的显著水平,默认置信区间为95%。

表1 本研究所用的基因克隆及荧光定量PCR引物序列
1.5 AmRADL1过表达载体构建及金鱼草遗传转化
利用H I和I限制性内切酶酶切pBI121-GFP载体和目的基因,经酶连、大肠杆菌感受态转化及菌落PCR鉴定等,将载体的绿色荧光蛋白(GFP)基因片段替换成基因的CDS序列,获得pBI121-35S::表达载体。将表达载体转化至根癌农杆菌() GV2260,通过农杆菌介导的方法进行金鱼草的遗传转化[14],获得转基因植株。
1.6 转基因植株鉴定与表达分析
采用SDS法提取野生型及转基因植株基因组DNA,使用卡那霉素抗性基因引物Kan-F、Kan-R和特异性引物AmRADL1-F、AmRADL1-R(表1)进行分子鉴定,获得阳性植株。反应体系为10 μL:DNA 1 μL,Easy0.1 μL,10×EasyBuffer 1 μL,dNTP 1 μL,上下游引物各1 μL,ddH2O 4.9 μL。PCR反应程序:95℃预变性3 min;95℃变性30 s,60℃退火30 s,72℃延伸1 min,35个循环;72℃总延伸10 min。
以为内参基因(表1),选取同一生长时期野生型及不同转基因株系金鱼草成熟叶片,提取总RNA,反转录获得cDNA,使用Eva Green荧光染料法进行qRT-PCR。反应体系及反应程序参考1.4,每个样品进行3次技术重复。选择野生型金鱼草为对照,利用Excel进行数据分析,基因相对表达量使用2–∆∆Ct法计算,使用SPSS 25.0软件进行ANOVA法计算不同转基因株系间的表达差异,默认置信区间为95%。
1.7 转基因植株形态观察及组织学染色
以同一时期播种的野生型及过表达的转基因金鱼草为材料,使用尼康相机(CoolPix P7100)和Leica体视显微镜(M165FC)对转基因植株阳性苗的生长发育进行观察拍照,包括转基因植株的根、茎、叶和成熟花的整体形态的表型变化。
选取野生型及不同转基因株系中stage13的心皮进行组织学染色分析。参照实验室改良的石蜡切片操作方法[15]:分别取样后将材料浸入FAA固定液,通过真空泵抽真空一段时间后于4℃冰箱放置至少24 h;利用不同浓度的二甲苯和乙醇进行脱水透明;将脱水透明后的材料置于熔融的蜡中浸蜡1~2天,保证植物材料浸蜡充分;对材料进行包埋,而后通过调整Leica轮转式切片机(RM2235)进行切片,厚度为8 μm。通过甲苯胺蓝染色法对石蜡切片进行染色、中性树胶封片后,使用体视显微镜观察并拍照。
使用Photoshop 2020中的计数工具,对野生型和转基因金鱼草心皮切片中胎座不同区域的细胞进行计数,其结果通过Excel统计分析并作图,利用SPSS 25.0软件中独立样本检验对数据进行显著性分析,置信区间为99%。
2 结果与分析
2.1 AmRADL1基因克隆及生物信息学分析
以JI7金鱼草叶片cDNA为模板,根据基因组测序所获得的基因序列设计特异性引物(表1)进行目的基因片段扩增,得到300 bp左右的单条带,与目的片段大小符合。测序结果显示:基因编码区长度为306 bp,编码101个氨基酸。将该氨基酸序列与AmRAD[5]、拟南芥()AtRL2[16]、番茄()SlFSM1[17]、蓝猪耳()TfRAD1[18]等氨基酸序列进行比对。多序列比对结果表明(图1),目的蛋白与其他物种的RAD-like蛋白序列高度相似,均含有SANT结构域,属于MYB转录因子家族,将其命名为。AmRADL1与AmRAD、TfRAD1、SlFSM1和AtRL2氨基酸序列间的相似性分别为70.07%、74.17%、77.58%和75.29%,表明该家族的蛋白质结构和功能相对保守。这些蛋白的C末端均具有较短且相对保守的CREB motif,调节基因的表达[19]。
将克隆的基因序列在NCBI-blastp中进行分析,筛选并下载与其相似性较高的序列,构建系统进化树(图2)。结果表明:单子叶植物和双子叶植物-like基因可能由同一祖先进化而来,后又各自独立进化;与[5]、[18]、黄金羽花()[20]等在同一进化分支上,亲缘关系接近;又与拟南芥[16]、番茄[17]、海岛棉()[21]、流苏马兜铃()[22]等物种的-like聚为一大类,而-like基因在细胞增殖方面具有重要的调控作用,推测也具有类似的功能,在进化过程中功能较为保守。
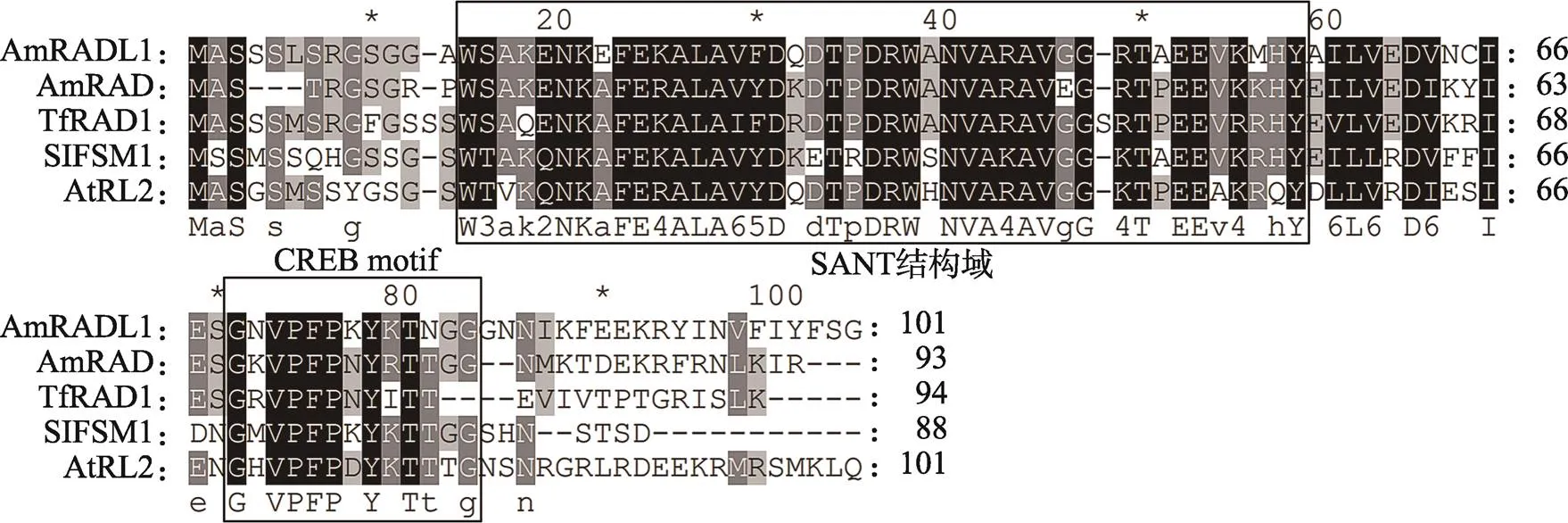
图1 金鱼草AmRADL1及其同源蛋白氨基酸比对
Am:;Tf:;Sl:;At:。
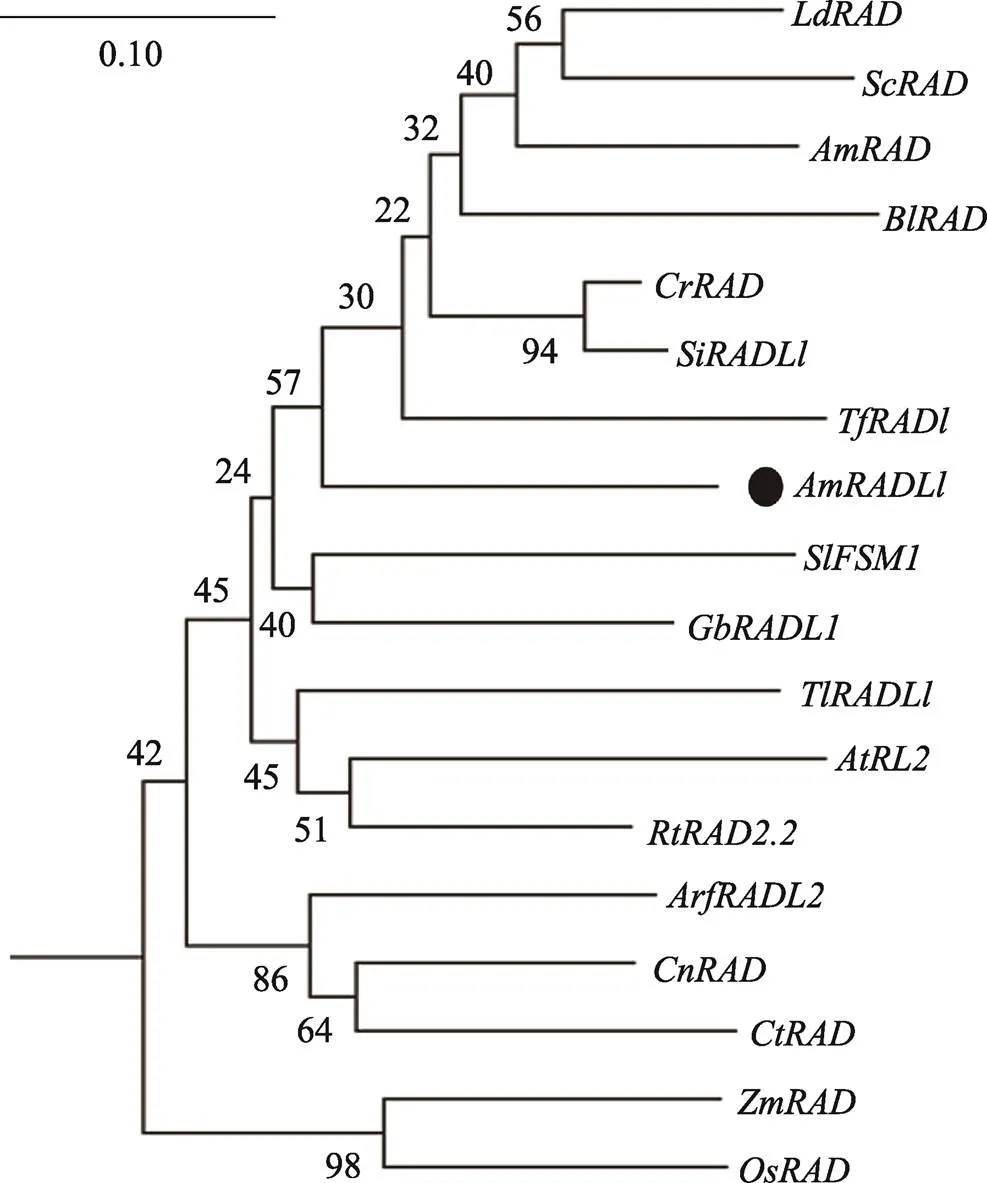
图2 AmRADL1基因系统进化树
Ld:;Sc:;Am:;Bl:;Cr:;Si:;Tf:;Sl:;Gb:;Tl:;At:;Rt:;Arf:;Cn:;Ct:;Zm:;Os:。
2.2 AmRADL1基因表达模式分析
为了解在金鱼草中的组织特异表达模式,本研究通过qRT-PCR分析该基因在JI7金鱼草不同组织的表达量差异。结果显示:在营养器官和生殖器官中均有表达,其中在根、茎和叶中表达相对较弱,在花中表达较高(图3A)。分别以stage13的萼片、花瓣、雄蕊和心皮的cDNA为模板,进一步分析在不同花器官的表达模式。qRT-PCR结果显示:在心皮中特异性表达,在萼片、花瓣和雄蕊中几乎不表达(图3B)。
2.3 转基因植株鉴定与表达分析
金鱼草遗传转化共获得3个独立的过表达株系,以Am223野生型植株为对照,进行DNA鉴定。结果表明,所有转基因株系均携带Kan抗性条带及特异性条带,而野生型均未扩出条带。进一步对转基因阳性植株中的表达进行qRT-PCR分析。结果显示:与对照植株相比,3个株系中的表达量均升高,其中RADL1-7-1和RADL1-7-2过表达株系中表达量基本一致,RADL1-7-3株系表达量最高(图4)。
2.4 转基因金鱼草的形态观察
对金鱼草过表达株系进行表型观察,结果发现:与同一时期野生型金鱼草相比,转基因植株的茎、叶和花序均未观察到明显变化。进一步对过表达株系的成熟花进行分析研究,结果显示3个转基因株系中成熟花的两侧对称性没有发生明显的变化(图5)。
A:不同组织的表达分析;B:不同花器官的表达分析。不同小写字母表示差异显著(<0.05),相同字母表示差异不显著。
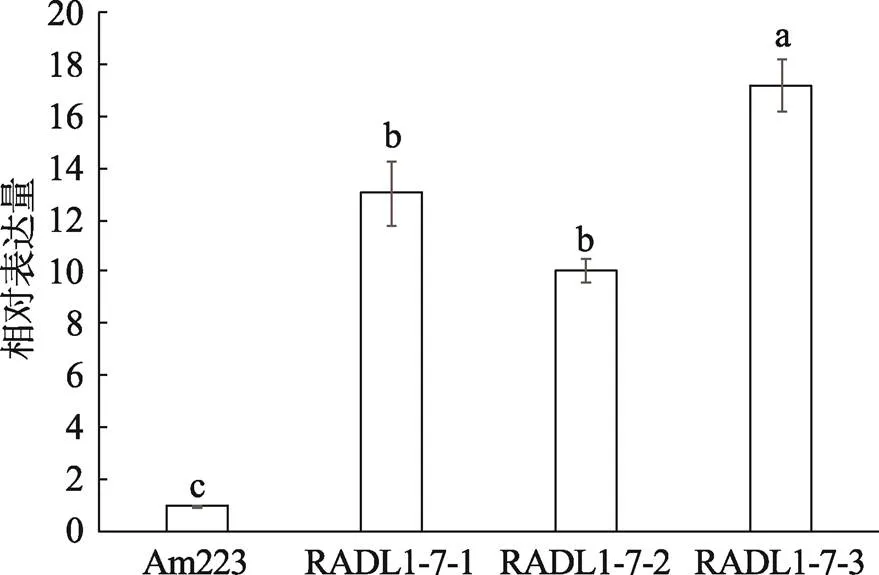
图4 野生型和AmRADL1过表达植株的表达分析
不同小写字母表示差异显著(<0.05),相同字母表示差异不显著。
2.5 转基因金鱼草的组织学染色分析
结合生物信息学预测及表达部位分析推测可能影响心皮的发育,为进一步探究转基因金鱼草心皮内部细胞结构是否发生变化,选择野生型和3个过表达株系stage13的单花(图6A),进行石蜡切片观察。结果显示,转基因植株心皮的整体形状与野生型相比没有区别,但中轴的胎座存在差异。过表达株系心皮内部细胞大小并未发生明显变化,胎座区域明显变小,其中RADL1-7-3株系变化显著,RADL1-7-1和RADL1-7-2株系变化次之(图6,B和C),与转基因植株qRT-PCR结果一致。通过对心皮中胎座区域的细胞进行初步统计发现,3个转基因株系与野生型相比胎座区域的细胞数目均明显减少(图7)。

图5 野生型和AmRADL1过表达植株的表型观察
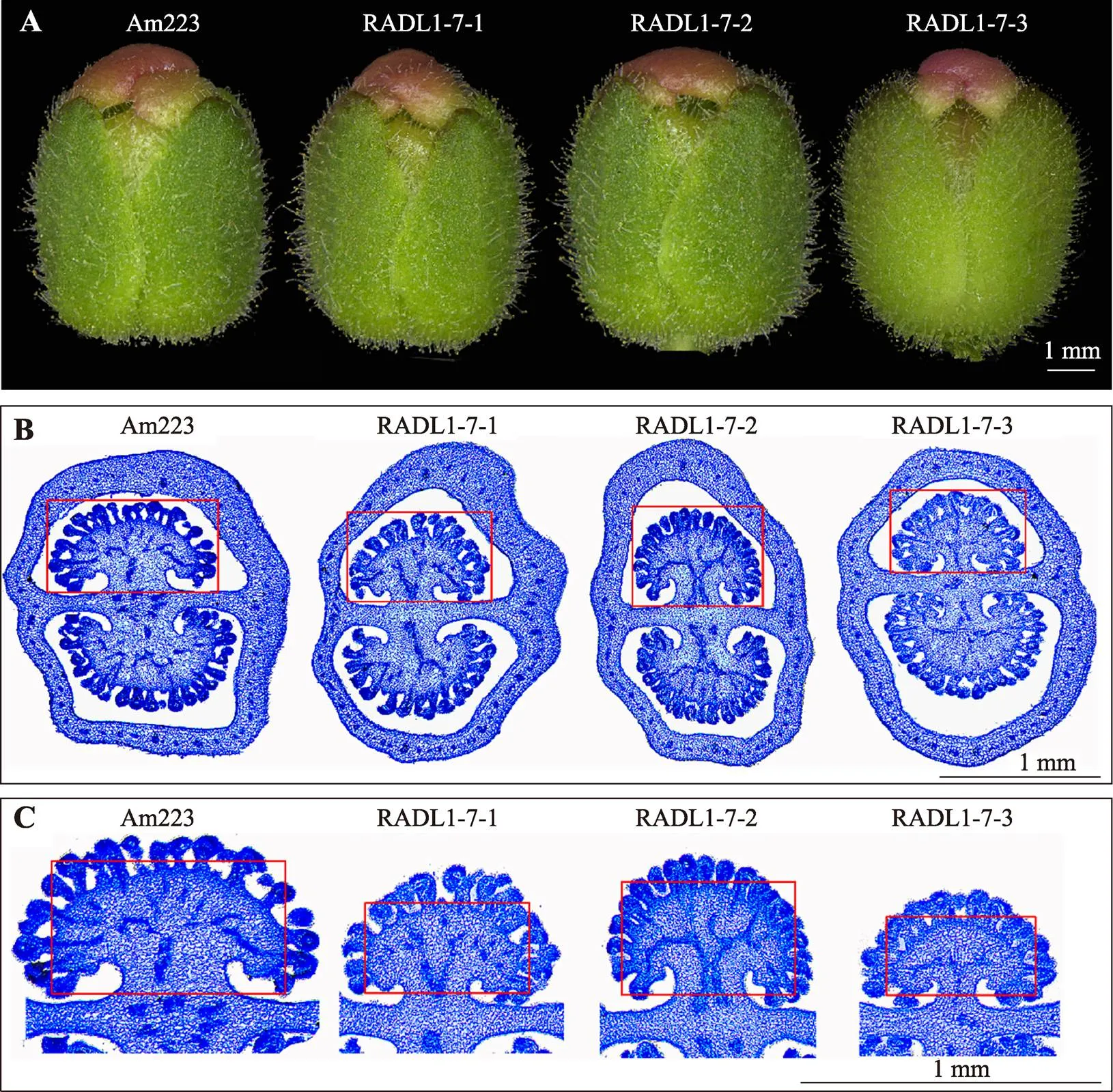
图6 野生型与过表达植株心皮的石蜡组织切片
A:石蜡切片选择时期;B:整体心皮组织切片;C:放大的胎座区域。红色方框为胎座变化区域。
3 讨论
R3-MYB转录因子在植物中广泛参与不同的生长发育过程,如生物钟的调节[23]、细胞形态建成[24]以及次生代谢产物生物合成[25]等,其主要特征是由单个MYB DNA结合域组成,在不同的物种中表现出高度的保守性[2]。基因的功能多样性除了和基因本身的DNA结合域有关外,基因的复制和表达模式同样重要[26]。-like基因编码R3-MYB蛋白,因其表达模式不同进而影响植物不同组织器官的细胞增殖过程,导致器官的形态发生变化。如金鱼草、木油桐()、水八角()以及意大利红门兰()等基因均在两侧对称花的花瓣中高表达,在花发育早期阶段通过调节花瓣的细胞分裂影响对称性的变化,功能相对保守[5,27,28]。研究表明,-like基因在两侧对称花的发育过程中发挥重要作用,而在其他物种中,-like基因同样影响细胞的增殖过程。海岛棉()具有控制棉纤维生长的潜在功能[21];椰子基因可能参与光形态下细胞发育及代谢的相关过程,影响果实表皮特征[29];流苏马兜铃()在花被中具有不同的表达,维持花冠的形态,与细胞增殖有关[22]等。除此之外,拟南芥虽然在雌蕊中高表达,但并不参与雌蕊发育,而是与HY5/HYH相互作用,调节拟南芥种子萌发与幼苗发育过程中对ABA和非生物胁迫的反应[16,30]。综上所述,-like基因影响细胞增殖过程,除参与花发育外,可能还具有不同的非花特异性功能。
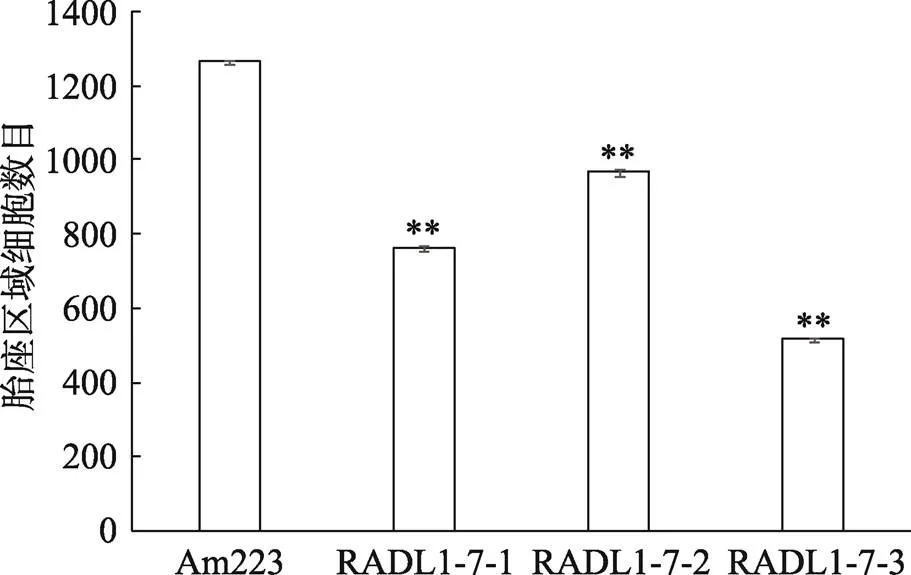
图7 野生型与过表达植株心皮的胎座区域细胞数目统计
Am223为野生型金鱼草;RADL1-7-1、RADL1-7-2、RADL1-7-3为不同的转基因株系;**代表具有统计学显著差异(<0.01,-test)。
本研究从金鱼草基因组中克隆获得基因,通过生物信息学分析发现,AmRADL1与SlFSM1、AtRL2和AmRAD等RAD-like蛋白序列相似度高,且在同一进化分支上;其氨基酸序列在N端含有保守的SANT结构域,在染色质重构过程中发挥重要的作用,C末端包含CREB motif,可以诱导本身基因的表达以及调控靶基因的表达[31,32]。相关研究显示,在不同植物中的同源基因可能影响心皮发育。例如:金鱼草基因在发育后期出现心皮的表达高峰,可能影响心皮及果实发育[33];在心皮中表达,而后在幼果发育中特异性表达,影响果皮的细胞扩增[17,34];柿子()在拟南芥中过表达导致雌蕊过度生长,与细胞的增殖有关[35]。表明-like基因可能在发育中控制心皮微结构的形成,也证明-like基因在调控细胞增殖方面的重要作用。通过的组织特异性表达分析发现,在萼片、花瓣和雄蕊中几乎不表达,心皮中特异性表达,可能参与心皮的发育,区别于在花瓣中高表达进而影响花瓣的发育。金鱼草过表达株系与野生型相比,成熟花的两侧对称性没有发现明显的变化。通过对心皮的组织切片染色分析可知,与野生型相比,金鱼草转基因株系心皮中胚珠着生的胎座区域明显变小,区域内细胞数目减少,细胞大小没有明显的变化,过表达可能抑制金鱼草中轴胎座的细胞扩增,但并不影响胚珠的发育,这与番茄在果实发育前期影响果皮的作用类似。即-like基因在植物发育过程中可能通过控制细胞的增殖参与不同的调控途径,从而发挥不同的功能。
目前关于心皮的发育主要集中在ABCDE模型,如A类基因控制心皮壁分化和果实发育[36]、C类基因()在胚珠中表达,在心皮的发育过程中起关键作用[37];除此之外,生长素和细胞分裂素等植物激素的信号传导,在雌蕊群的形态发生中也发挥主要作用[38],心皮的进化与发育是十分复杂的遗传调控网络。过表达植株并未发生花器官同源异型转变以及花对称性的变化,而是在心皮发育中影响胎座的细胞增殖,可能参与一种新的调控途径进而影响心皮的发育。本研究共获得3个转基因金鱼草株系,转基因材料相对较少,因此后续仍需要对转基因材料进行观察分析。为了深入探索在金鱼草中的具体功能,还需要进一步研究突变体以及与其他基因的相互作用的分子机制。
[1] Thomson B, Wellmer F. Molecular regulation of flower development., 2019, 131: 185–210.
[2] Ambawat S, Sharma P, Yadav NR, Yadav RC. MYB transcription factor genes as regulators for plant responses: an overview., 2013, 19(3): 307–321.
[3] Qian JH, Li ZQ, Liao XF, Tang DF, Shi QQ, Zhou RY, Chen P. Advance on MYB transcription factors in regulating plant flower development., 2016, 27(2): 283–288.钱景华, 李增强, 廖小芳, 汤丹峰, 史奇奇, 周瑞阳, 陈鹏. 调控植物花发育的MYB类转录因子研究进展. 生物技术通讯, 2016, 27(2): 283–288.
[4] Coen ES, Meyerowitz EM. The war of the whorls: genetic interactions controlling flower development., 1991, 353(6339): 31–37.
[5] Corley SB, Carpenter R, Copsey L, Coen E. Floral asymmetry involves an interplay between TCP and MYB transcription factors in., 2005, 102(14): 5068–5073.
[6] Wang SS, Koide Y, Kishima Y. How to establish a mutually beneficial relationship between a transposon and its host: lessons from Tam3 in., 2022, 97(4): 177–184.
[7] Schwinn K, Venail J, Shang YJ, Mackay S, Alm V, Butelli E, Oyama R, Bailey P, Davies K, Martin C. A small family of MYB-regulatory genes controls floral pigmentation intensity and patterning in the genus., 2006, 18(4): 831–851.
[8] Perez-Rodriguez M, Jaffe FW, Butelli E, Glover BJ, Martin C. Development of three different cell types is associated with the activity of a specific MYB transcription factor in the ventral petal offlowers., 2005, 132(2): 359–370.
[9] Cui ML, Copsey L, Green AA, Bangham JA, Coen E. Quantitative control of organ shape by combinatorial gene activity., 2010, 8(11): e1000538.
[10] Li MM, Zhang DF, Gao Q, Luo YF, Zhang H, Ma B, Chen CH, Whibley A, Zhang YE, Cao YH, Li Q, Guo H, Li JH, Song YZ, Zhang Y, Copsey L, Li Y, Li XX, Qi M, Wang JW, Chen Y, Wang D, Zhao JY, Liu GC, Wu B, Yu LL, Xu CY, Li J, Zhao SC, Zhang YJ, Hu SN, Liang CZ, Yin Y, Coen E, Xue YB. Genome structure and evolution ofL., 2019, 5(2): 174–183.
[11] Vincent CA, Coen ES. A temporal and morphological framework for flower development in., 2004, 82(5): 681–690.
[12] Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG. Clustal W and Clustal X version 2. 0., 2007, 23(21): 2947–2948.
[13] Kumar S, Stecher G, Li M, Knyaz C, Tamura K. MEGA X: molecular evolutionary genetics analysis across computing platforms., 2018, 35(6): 1547–1549.
[14] Cui ML, Handa T, Ezura H. An improved protocol for-mediated transformation ofL., 2003, 270(4): 296–302.
[15] Hao YM, Chen KL, Feng LJ, Li FF, Cui ML, Piao CL. Cloning and functional analysis ofin., 2022, 39(4): 821–829. 郝燕敏, 陈柯俐, 冯丽君, 李菲菲, 崔敏龙, 朴春兰. 欧洲千里光基因的克隆及功能分析. 浙江农林大学学报, 2022, 39(4): 821–829.
[16] Yang BC, Song ZH, Li CN, Jiang JH, Zhou YY, Wang RP, Wang Q, Ni C, Liang Q, Chen HD, Fan LM. RSM1, anMYB protein, interacts with HY5/HYH to modulate seed germination and seedling development in response to abscisic acid and salinity., 2018, 14(12): e1007839.
[17] Barg R, Sobolev I, Eilon T, Gur A, Chmelnitsky I, Shabtai S, Grotewold E, Salts Y. The tomato early fruit specific genedefines a novel class of plant-specific SANT/MYB domain proteins., 2005, 221(2): 197–211.
[18] Su SH, Xiao W, Guo WX, Yao XR, Xiao JQ, Ye ZQ, Wang N, Jiao KY, Lei MQ, Peng QC, Hu XH, Huang X, Luo D. The CYCLOIDEA-RADIALIS module regulates petal shape and pigmentation, leading to bilateral corolla symmetry in(Linderniaceae)., 2017, 215(4): 1582–1593.
[19] Boyer LA, Latek RR, Peterson CL. The SANT domain: a unique histone-tail-binding module?, 2004, 5(2): 158–163.
[20] Zhong JS, Preston JC, Hileman LC, Kellogg EA. Repeated and diverse losses of corolla bilateral symmetry in the Lamiaceae., 2017, 119(7): 1211–1223.
[21] Zhang F, Liu X, Zuo KJ, Zhang JQ, Sun XF, Tang KX. Molecular cloning and characterization of a novelL.-like gene., 2011, 29(2): 324–333.
[22] Madrigal Y, Alzate JF, González F, Pabón-Mora N. Evolution ofandgene lineages in flowering plants with an expanded sampling in non-core eudicots., 2019, 106(3): 334–351.
[23] Lu SX, Knowles SM, Andronis C, Ong MS, Tobin EM.andfunction synergistically in the circadian clock of., 2009, 150(2): 834–843.
[24] Pesch M, Schultheiß I, Digiuni S, Uhrig JF, Hülskamp M. Mutual control of intracellular localisation of the patterning proteins AtMYC1, GL1 and TRY/CPC in., 2013, 140(16): 3456–3467
[25] Dubos C, Le Gourrierec J, Baudry A, Huep G, Lanet E, Debeaujon I, Routaboul JM, Alboresi A, Weisshaar B, Lepiniec L.is a new regulator of flavonoid biosynthesis in., 2008, 55(6): 940–953.
[26] Boyden GS, Donoghue MJ, Howarth DG. Duplications and expression of-like genes in Dipsacales., 2012, 173(9): 971–983.
[27] Preston JC, Kost MA, Hileman LC. Conservation and diversification of the symmetry developmental program among close relatives of snapdragon with divergent floral morphologies., 2009, 182(3): 751–762.
[28] Valoroso MC, Paolo SD, Iazzetti G, Aceto S. Transcriptome-wide identification and expression analysis of- and-like genes of the mediterranean Orchid., 2017, 9(6): evx101.
[29] Guo SK, Zeng CR, Wu Y, Li J, LI XG, Yang YD. Cloning and expression analysis of coconut CnRADIALIS-like transcription factor., 2021, 42(09): 2478–2486. 郭树宽, 曾春茹, 吴翼, 李静, 李新国, 杨耀东. 椰子CnRADIALIS-like转录因子的克隆与表达分析. 热带作物学报, 2021, 42(09): 2478–2486.
[30] Baxter CEL, Costa MMR, Coen ES. Diversification and co-option of-like genes in the evolution of floral asymmetry., 2007, 52(1): 105–113.
[31] Stevenson CE, Burton N, Costa MMR, Nath U, Dixon RA, Coen ES, Lawson DM. Crystal structure of the MYB domain of the RAD transcription factor from., 2006, 65(4): 1041–1045.
[32] Ge J, Zhang Y, Zheng ZZ, Huang YW, Song HS. Research progress on CREB and the signal transduction pathway of its phosphorylation., 2010, 38(30): 16769–16771, 16774. 葛军, 张玉, 郑增长, 黄延旺, 宋红生. CREB转录因子及其磷酸化信号通路的研究进展. 安徽农业科学, 2010, 38(30): 16769–16771, 16774.
[33] Sengupta A, Hileman LC. A CYC-RAD-DIV-DRIF interaction likely pre-dates the origin of floral monosymmetry in Lamiales., 2022, 13(1): 3.
[34] Machemer K, Shaiman O, Salts Y, Shabtai S, Sobolev I, Belausov E, Grotewold E, Barg R. Interplay of MYB factors in differential cell expansion, and consequences for tomato fruit development., 2011, 68(2): 337–350.
[35] Masuda K, Ikeda Y, Matsuura T, Kawakatsu T, Tao R, Kubo Y, Ushijima K, Henry IM, Akagi T. Reinvention of hermaphroditism via activation of a-like gene in hexaploid persimmon., 2022, 8(3): 217–224.
[36] Müller BM, Saedler H, Zachgo S. The MADS-box genefromis involved in the regulation of floral meristem identity and fruit development., 2001, 28(2): 169–179.
[37] Callens C, Tucker MR, Zhang DB, Wilson ZA. Dissecting the role of MADS-box genes in monocot floral development and diversity., 2018, 69(10): 2435–2459.
[38] Zúñiga-Mayo VM, Gómez-Felipe A, Herrera-Ubaldo H, de Folter S. Gynoecium development: networks inand beyond., 2019, 70(5): 1447–1460.
Cloning and functional analysis ofgene from
Feifei Li, Yanmin Hao, Minlong Cui, Chunlan Piao
MYB is one of the largest transcription factor families in plants. Among them, the R3-MYB transcription factor() plays a very important role in the flowers development in. In this study, a R3-MYB gene similar towas found by analyzing the genome of, which was named().The gene function was predicted through bioinformatics. The relative expression levels in different tissues and organs of wild-typewere analyzed by qRT-PCR.was overexpressed in, and the transgenic plants were analyzed by morphological observation and histological staining. The results showed that the open reading frame (ORF) ofgene was 306 bp in length, encoding 101 amino acids. It has typical SANT domain, and the C-terminal contains a CREB motif, which was highly homologous to tomato. The results of qRT-PCR showed thatwas expressed in roots, stems, leaves and flowers, and the expression level was higher in flowers. Further analysis of its expression in different floral organs showed thathad the highest expression in carpel. The results of histological staining analysis of the transgenic plants showed that compared with the wild type, although the size of the carpel cells of the transgenic plants did not change significantly, the placenta area in the carpel became smaller and the number of cell decreased. In summary,may be involved in the regulation of carpel development, but the specific mechanism of action in carpel remains to be further studied.
;like gene; flower development; expression analysis; histological stain
2023-03-14;
2023-04-11;
2023-05-29
浙江省农业科学院合作研发项目(编号:H20220312)资助[Supported by the Cooperative R & D Project of Zhejiang Academy of Agricultural Sciences (No. H20220312)]
李菲菲,在读硕士研究生,专业方向:植物花发育。E-mail: 1399745127@qq.com
朴春兰,硕士,助理研究员,研究方向:生物技术与分子育种。E-mail: 2488335116@qq.com
10.16288/j.yczz.23-057
(责任编委: 孔凡江)
