N6-甲基腺苷修饰对女性生殖系统功能的影响
2023-07-05何山赵健宋晓峰
何山,赵健,宋晓峰
6-甲基腺苷修饰对女性生殖系统功能的影响
何山,赵健,宋晓峰
南京航空航天大学自动化学院,南京 211106
近年来女性不孕不育率不断攀升,已成为我国提高生育率亟需解决的困境。生殖系统的健康是保证生育能力的前提条件。6-甲基腺苷(6-methyladenosine,m6A)是真核生物中最常见的化学修饰,在细胞生命活动中发挥着极其重要的作用。近来,m6A修饰被证实在女性生殖系统的各种生理和病理过程中起着关键作用,但其调控机制及生物学功能仍不清楚。本文首先介绍了m6A修饰的可逆调节机制及其功能,随后讨论了其在女性生殖功能和生殖系统疾病中的作用,最后对m6A修饰的检测技术和方法及其最新进展进行了归纳总结,以期为后续女性生殖系统发病机制和治疗研究提供参考。
m6A;甲基化修饰;女性生殖系统;发育和衰老;疾病
女性不孕不育发病率近年来稳步上升,已成为我国应对人口老龄化挑战急需解决的不利因素。生殖系统的衰老和病变是造成女性生育能力缺失的重要原因[1]。研究发现表观遗传修饰在多种生理过程和病理过程中发挥着重要的调节作用,例如胚胎发育、神经系统发育和肿瘤发生等。其中,6-甲基腺苷修饰是真核生物中最为常见的修饰形式,在人体发育和衰老过程中扮演着重要的角色。最近研究报道m6A修饰在女性生殖系统中扮演着重要角色,其异常调控与卵巢早衰、发育不良等生殖系统疾病密切相关[2]。然而,m6A修饰的具体作用及确切机制仍不清楚。据此,本文综述了m6A修饰影响女性生殖功能的最新研究进展,并汇总归纳了m6A检测新技术及相关生物信息学方法。
1 m6A修饰的分子调控机制
m6A修饰在进化上非常保守,偏向于发生在RNA中的5′-RRACH-3′(R = A或G;H=A、C或U)基序,且具有组织和细胞类型特异性[3]。研究发现m6A修饰在终止密码子附近的编码区(coding sequence,CDS)及3′端非翻译区域(3′-untranslated region,3′-UTR)显著富集[4]。与DNA甲基化类似,m6A修饰是一个动态可逆的过程,受到甲基转移酶(写入器“writers”)、去甲基化酶(擦除器“erasers”)和m6A阅读蛋白(阅读器“readers”)的严格调节[5]。
1.1 甲基转移酶(m6A写入)
m6A修饰的“写入”由甲基转移酶复合物(methyltransferase complex,MTC)完成,其核心成分是甲基转移酶样3(METTL3)[6]和甲基转移酶样14(METTL14)[7]。METTL3是首个被发现的m6A甲基转移酶,是MTC中唯一具有催化活性的亚基。METTL14则在促进RNA底物识别和结合方面起着重要作用。研究表明,METTL3或METTL14耗竭可大大降低全转录组m6A修饰水平,进而改变mRNA稳定性,导致细胞功能失调[8]。动物实验显示,METTL3/14在性腺发育和卵母细胞成熟等方面发挥着重要的调节作用,其表达缺失将导致性腺或配子发育不良,使得生育能力受损。除METTL3/14外,MTC还包含一系列辅因子WTAP[9]、VIRMA[10]、ZC3H13[11]、RBM15/15B[12]等,以此调节其催化活性、细胞定位和底物募集等功能。例如,WTAP作为调节亚基,可辅助MTC定位到富含mRNA底物的核斑上,进而增强甲基化效率。
1.2 去甲基化酶(m6A擦除)
相比于甲基化酶,去甲基化酶近年来才逐渐为人所知。去甲基化酶的发现为mRNA转录后的甲基化修饰提供了有力且突破性的证据。FTO是首个被证实的RNA去甲基化酶,对m6A修饰具有很高的亲和力,可在细胞内和体外环境“擦除”m6A修饰[13]。ALKBH5作为第二个被证实的m6A去甲基化酶,显示出与FTO相当的去甲基化活性。和的表达模式均具有组织特异性,并在细胞质和细胞核中有着不同的底物偏好。虽然两者在全身组织中都有表达,但在脑组织中表达量最高,而则在睾丸及女性生殖系统中富集[14]。近来研究发现,低表达引发的m6A修饰障碍使得卵巢加速衰老,而的失活则会导致精子发生和生育能力受损。
1.3 m6A结合蛋白(m6A阅读)
m6A修饰可被特定结合蛋白“阅读”,进而靶向调控RNA加工过程。m6A阅读蛋白主要有三类:YTH家族、hnRNP家族和IGF2BP家族。YTH家族包含五个成员:YTHDC1、YTHDC2、YTHDF1、YTHDF2和YTHDF3。YTHDC1位于细胞核中,可调控RNA的核输出和亚细胞定位[15]。YTHDC2是一种RNA解旋酶,可促进靶mRNA的降解或提高其翻译效率[16]。YTHDF1则与翻译起始因子相互作用,调控mRNA的翻译效率[17]。YTHDF2能够影响靶mRNA的稳定性,诱导其降解[18]。YTHDF3的功能具有细胞类型特异性,可诱导RNA降解,介导其亚细胞定位和选择性剪接[19]。hnRNP家族属于RNA结构依赖性蛋白,hnRNPA2B1被证明与METTL3一起调节可变剪接和microRNA代谢;hnRNPC和hnRNPG则被报道参与RNA变构及转录调控过程[20]。IGF2BP家族包含IGF2BP1~3三种蛋白,以m6A依赖的方式结合靶mRNA,增强其稳定性[21]。值得一提的是,不同的阅读器会跟据不同的细胞环境呈现不同甚至相反的功能,m6A阅读蛋白的结构和功能仍有待进一步阐明。
2 m6A修饰的生物学功能
作为转录本水平上最普遍和保守的碱基修饰,m6A几乎参与调控RNA代谢的所有阶段,包括从细胞核中的加工到细胞质中的翻译和衰变等。m6A甲基化修饰可通过热力学效应改变RNA的二级结构,进而影响RNA基序与RNA结合蛋白(RNA-binding protein,RBP)相互作用的可及性,这种现象被称为m6A开关[22]。图1展示了m6A及其作用元件对mRNA生命历程的典型调控作用。
2.1 m6A影响mRNA成熟
近来研究发现带有poly(A)的mRNA在生长期卵母细胞中聚集在细胞核特定区域,即核多聚腺苷酸结构域(nuclear polyadenylation domains,NPADs)中。该区域富集RNA结合蛋白,调控母源mRNA的剪接、加尾、储存和降解[23]。已有研究表明,相较于成熟mRNA,m6A在pre-mRNA中更加富集,并且m6A位点集中在内含子区域,因此m6A修饰很早被认为是一种剪接调控机制。研究发现,METTL3的消耗通常有利于外显子跳跃和内含子保留事件发生[24]。另一方面,m6A甲基化还被发现参与可变多聚腺苷酸化(alternative polyadenylation,APA)。m6A写入器的敲除会引起APA事件的发生,且基因最后一个外显子中的m6A位点大多位于APA位点所在的3′UTR区域。最近对mRNA聚腺苷酸尾的研究发现,m6A修饰的转录异构体更倾向于使用近端APA位点,使得3′UTR长度更短[25]。
mRNA需从细胞核输出到细胞质才能进行翻译。m6A修饰也参与此过程,通过影响mRNA的核输出调节基因表达水平。m6A写入蛋白METTL3的耗尽被发现可抑制mRNA转运。与之相对,m6A擦除蛋白ALKBH5的耗尽则被发现可增强mRNA的出核能力。进一步研究发现,m6A阅读蛋白YTHDC1也可参与mRNA的出核调控。被甲基化修饰的mRNA在核蛋白YTHDC1的介导下,与剪接因子和核输出衔接蛋白相互作用,进而被递送到核输出受体,使其核输出能力得到增强[26]。这些结果表明,m6A甲基化与pre-mRNA加工和成熟密切相关。
2.2 m6A调节mRNA翻译效率
卵母细胞减数分裂恢复后,储存的母本mRNA会在多聚腺苷酸聚合酶的作用下延长ployA尾的长度,并重新激活翻译[27],这一过程由丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)触发细胞质聚腺苷酸化元件的磷酸化和降解,将母体mRNA翻译与卵母细胞减数分裂偶联[28,29]。鉴于m6A修饰对APA的控制作用,有理由认为m6A参与了mRNA翻译效率的控制。一些证据显示m6A修饰可以通过阅读蛋白与翻译所需因子结合等方式调节翻译效率。YTHDF1与m6A修饰的mRNA结合,招募翻译起始因子eIF3,进而诱导核糖体与mRNA结合,提升翻译效率。另一方面,3′UTR长度的延伸可能导致miRNA结合位点的增加,从而抑制翻译过程[30]。此外,METTL3则可通过识别5′UTR或3′UTR区域的m6A,促进mRNA以非帽依赖方式翻译蛋白。METTL3在翻译起始期间招募eIF3,增强eIF4E依赖的mRNA特定亚群的翻译[31]。总之,m6A可通过多种作用机制调节mRNA翻译,而且位于不同RNA功能区域的m6A修饰的作用方式通常也各不相同。
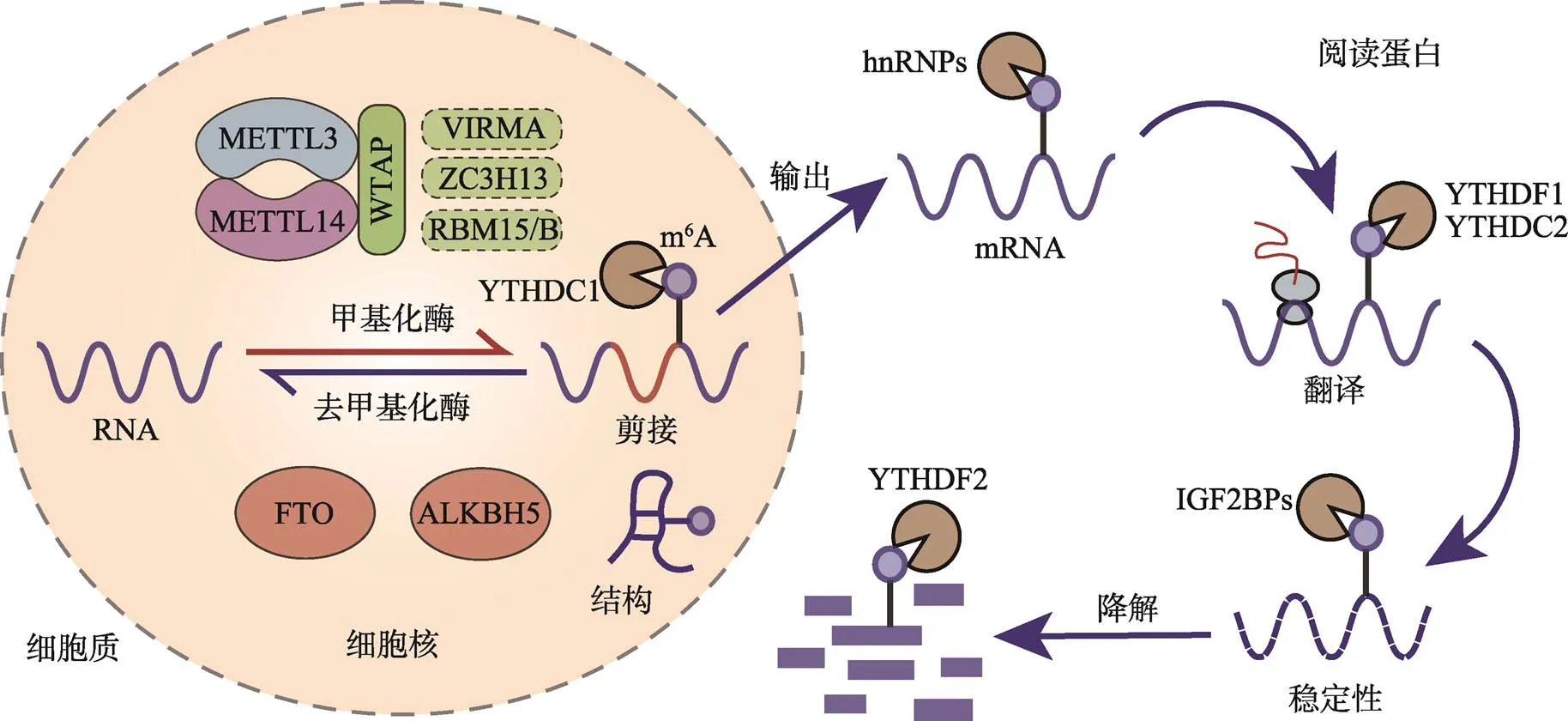
图1 m6A修饰的生物学功能
2.3 m6A改变mRNA稳定性
衰变是mRNA代谢的最后一步,在此期间mRNA结构逐渐不稳定并最终降解。在卵母细胞成熟过程中,母本mRNA会出现两波大规模降解:第一波被称为M-衰变,发生在减数分裂期间,意味着母本调节因子衰变;第二波被称为Z-衰变,发生在着床前胚胎2~4个细胞阶段,意味着合子基因组激活。在卵母细胞中,母本因子会触发聚腺苷酸核酸酶聚合物和CCR4-NOT复合物介导的去腺苷酸化,进而导致mRNA降解[32]。m6A修饰可通过不同分子机制调控mRNA衰变,这一过程主要由m6A阅读蛋白诱导完成。YTHDF2提供了第一个m6A依赖性mRNA衰变途径的直接证据,敲低增加了其靶mRNA的稳定性,延缓了衰变过程[33]。m6A修饰除了加速mRNA的衰变过程,其对mRNA的稳定性还有着更为复杂的调控作用。例如,m6A阅读蛋白IGF2BPs被发现可与RNA稳定因子(如HuR、MATR3和PABPC1)结合,拮抗YTHDF2诱导的衰变,增强mRNA的稳定性。甲基化转移酶和的敲低被证实可导致其靶mRNA的表达上调[34]。以上结果表明,m6A对RNA稳定性的双重作用可能隐含着更深刻的生物学意义。
综上所述,m6A修饰影响着mRNA生命周期的全过程。表1列出了一些重要的m6A作用因子以及它们的生物学作用,这些调控作用通常是密切关联的:m6A写入器和擦除器共同决定靶mRNA的m6A甲基化水平,“编码”mRNA的生命周期;阅读器则特异性识别mRNA上的m6A修饰信号,“解码”其生命周期,募集相应RBP蛋白,调控mRNA成熟、翻译及代谢过程。这一过程精密准确,能够快速有序的进行RNA的代谢调控,从而在细胞分化和发育等过程中满足突然升高的蛋白质代谢需求。
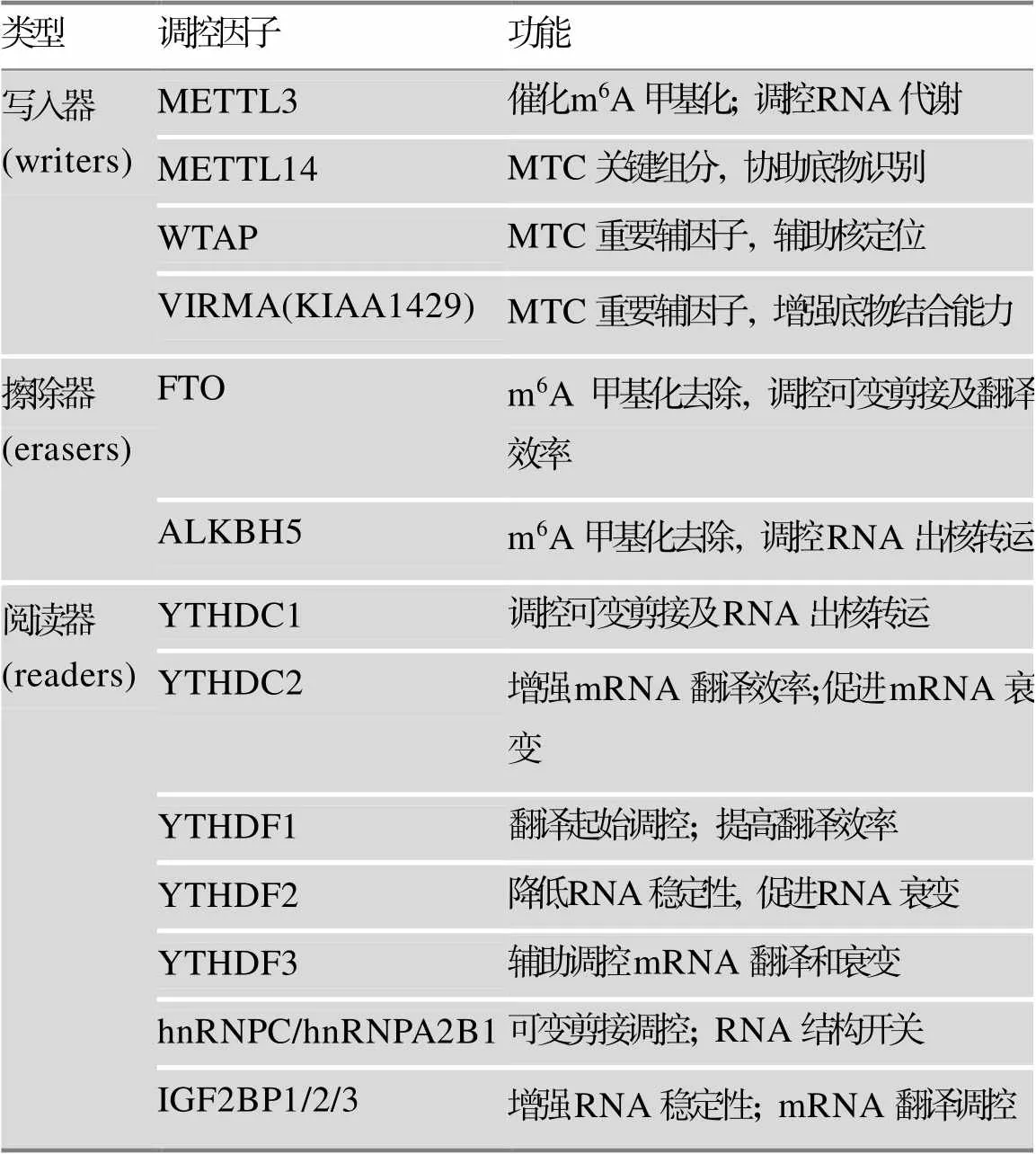
表1 m6A调控因子及功能
3 m6A修饰对女性生殖功能的影响
许多研究已经证实,组织和器官的分化、发育、衰老及病理过程都受到m6A修饰调节。大量证据表明m6A修饰在卵泡发生、卵母细胞成熟、配子结合、胚胎着床和早期胚胎发育中发挥着重要作用[35],但是具体调控机制还未完全阐明。图2展示了m6A修饰在女性生殖过程中的调控作用。
3.1 女性生殖系统的发育和衰老
女性生殖系统由性腺(即卵巢)、生殖道(包括子宫、子宫颈、输卵管和阴道)及外生殖器组成,主要负责产生卵母细胞,并将卵母细胞转运到输卵管和子宫进行受精和胚胎发育[36]。女性生殖系统的发育早在胚胎的5~6周就已经开始,原始生殖细胞形成卵巢,约10周时形成原始卵泡,在12周由促性腺激素、促黄体激素和卵泡刺激素促进发育[37],原始卵泡逐渐过渡到由颗粒细胞(granulosa cell,GC)包围的初级和次级卵母细胞。卵巢的发育在出生前基本停滞,直到青春期由促性激素刺激卵巢生长同时伴随卵母细胞激活[38],以具备排卵能力作为基本成熟的标志。另一方面,由苗勒氏管(Müllerian ducts,MD)分化为女性生殖道。MD的中线融合导致子宫阴道管的形成,该结构头部在9~10周时形成子宫。16周时,子宫阴道管的尾端开始出现宫颈的特征,宫颈腺在18周时出现[39]。类似的,出生后子宫和输卵管发育减缓,需要到青春期在卵巢激素的刺激下继续发育成熟,直到子宫内膜能够随排卵周期规律的脱落即月经形成,预示生殖道基本成熟。已经证实下丘脑-垂体-性腺(hypothalamic–pituitary–gonadal,HPG)轴和BMP信号通路等参与生殖组织发育、分化和生殖细胞形成,而m6A修饰在当中的作用正逐渐被揭示[40~42]。
作为生殖器官,卵巢的衰老速度比其他器官要快得多[43]。相对而言,子宫和输卵管的生殖功能虽然也会受到年龄的影响,但不像卵巢那样明显[44],而更容易由于卵巢功能老化和激素水平变化的影响。卵巢生理性衰老(normal ovarian aging,NOA)被定义为卵母细胞数量和质量随着年龄的增长而逐渐下降。卵巢的衰老从大约38岁开始加速,绝经时卵泡储备从新生儿的约250,000~500,000个减少到少于约100~1000个[45]。此外,在病理条件下卵巢也会出现提前老化或功能缺失。有研究显示,在女性生殖系统的发育和衰老过程中,表观遗传调控是关键的一环[46],其中m6A修饰很可能扮演了重要的角色。
3.2 m6A与卵泡的发育
卵泡的发育与卵母细胞的成熟同步,为卵母细胞建立适当的微环境。卵泡正常发育和卵母细胞的成熟是女性具备生育能力的先决条件[47]。在小鼠()中,m6A水平通常随着卵巢的发育而增加,m6A调节蛋白也表现出类似的趋势。从12.5天到卵巢成熟期间,去甲基化酶减少、甲基转移酶增加,m6A修饰水平逐渐升高,在黄体前达到峰值[48]。在斑马鱼()中,甲基转移酶METTL3在卵巢中的表达高于其他器官,其突变会导致雌性性腺减小和卵泡发育不全。研究发现,缺失会引发整体m6A修饰水平以及性激素合成和促性腺激素信号传导所需关键基因的表达下调,破坏卵母细胞成熟,降低生育能力[49]。甲基转移酶KIAA1429在不同阶段的卵泡细胞和颗粒细胞中也有表达,其缺乏将抑制颗粒细胞增殖并促其凋亡,导致卵泡发育停滞和紊乱[50]。另外,m6A阅读蛋白YTHDC2的缺失会导致睾丸和卵巢显著缩小,而METTL14和YTHDF1被发现与雌性生殖干细胞(female germline stem cell,FGSC)增殖密切相关,这些都提示m6A修饰对于性腺发育过程具有重要影响[51,52]。
在卵母细胞受精后,早期转录的母体mRNA主导卵母细胞和早期胚胎的发育,直到合子基因组激活。以上过程在很大程度上依赖于mRNA的转录后调控,而m6A修饰在其中扮演着重要角色。在小鼠中,敲除会使卵母细胞数量和直径明显减小,并阻碍纺锤体形成和第一极体排出,使得减数分裂过程停滞,这可能是由于减数分裂相关基因的翻译效率下降所致[53]。YTHDC1可与SRSF3等相互作用调控pre-mRNA的可变剪接,其缺失会导致卵母细胞中出现大量异常剪接事件和3′UTR长度的变化[54]。的缺失则会导致SRSF3和YTHDC1在卵母细胞核中的定位失调,扰乱卵子中转录本的正常剪接,导致减数分裂停滞。尽管已有很多文献报道了m6A在卵巢和卵子发育中的关键作用,但只是初步的解释,特别是在人类自身中的功能研究还存在很多空白。
3.3 m6A与早期胚胎发育和着床
卵子在受精后,继续完成减数分裂过程并从输卵管转移到子宫,在子宫内膜上完成着床。在此过程基因水平需要进行母源到合子的转换(maternal- to-zygotic transition,MZT),具体是指母体mRNA降解,合子基因组逐渐控制基因表达[55]。母本mRNA的降解为合子基因组激活和早期胚胎发育奠定了基础[56,57]。m6A修饰对mRNA稳定性的改变是调节MZT的重要机制。YTHDF2是卵母细胞成熟和早期合子发育所必需的。在斑马鱼中,敲低会减缓m6A修饰的母体mRNA的降解并阻碍合子基因组活化,导致合子和早期胚胎发育缺陷[58]。在哺乳动物中观察到更多m6A参与调控的痕迹。在小鼠中,除了发现YTHDF2促进母体mRNA降解以外,敲除也会降低卵母细胞中母体mRNA的整体翻译效率,抑制卵母细胞成熟,并且干扰母体mRNA的降解,阻碍MZT过程[59]。人类MZT过程中关键的调控因子表达模式与小鼠相近,但m6A阅读蛋白与RNA降解的动力学和生物化学机制尚未得到充分的研究[60]。
卵子在输卵管腔中完成受精后开始早期胚胎的发育和分化。受精卵首先发育为囊胚,囊胚植入子宫内膜的过程称为着床。分布于囊胚外层的是滋养层干细胞,滋养层将进一步发育为胎膜和胎盘[61]。胎盘在妊娠过程中起着关键作用,胎盘畸形与早期胚胎死亡和发育异常密切相关。在自然流产患者胎盘滋养层中发现去甲基化酶FTO和白细胞抗原HLA-G显著降低,YTHDF2的靶mRNA表达也降低,表明绒毛细胞FTO导致的异常甲基化改变了母胎界面的免疫耐受和血管生成,进而导致了流产[62]。复发性流产患者绒毛细胞中mRNA的整体m6A修饰水平显著降低,研究发现敲低将导致CYR61 mRNA稳定性降低,抑制怀孕早期母胎界面滋养层细胞的增殖和侵袭[63]。m6A修饰的失调可能导致滋养层的分化和妊娠功能异常,最终导致生育能力减弱,例如流产和先兆子痫(preeclampsia,PE)等。滋养层细胞和肿瘤细胞有许多相似之处,包括侵袭、血管生成和免疫抑制环境,两者都需要免疫微环境支持。肿瘤免疫细胞m6A修饰相关研究可能有助于了解母胎界面基质细胞、滋养层和蜕膜免疫细胞功能障碍的成因及分子机制。
3.4 m6A与卵巢衰老
对于哺乳动物特别是人类,雌性生殖寿命主要取决于卵母细胞的退行,而不是子宫的衰老。对小鼠卵母细胞翻译景观的研究表明,母体mRNA亚群的降解及翻译减弱是卵母细胞衰老的标志[64]。大多数研究显示m6A修饰位点随着衰老过程逐渐增加,m6A修饰异常与器官衰老密切有关。对衰老卵巢的生物信息学分析显示,m6A修饰的基因主要富集在FOXO信号通路、粘附连接和肌动蛋白细胞骨架调节中[65]。
在NOA中,卵巢颗粒细胞的衰老伴随着FTO的下调和m6A修饰的增加。研究发现,去甲基化酶FTO下调增加了FOS-mRNA-3′UTR的m6A甲基化修饰,减缓了FOS-mRNA的降解,进而上调颗粒细胞中的FOS表达,导致卵巢衰老[66]。相对NOA而言,卵巢病理性衰老是指原发性卵巢功能不全(primary ovarian insufficiency,POI)及其发展的卵巢早衰(premature ovarian failure,POF)[67]。POI早期通常表现为不孕症,在发展期间出现隐匿性卵巢功能异常,包括卵巢早老化、卵巢储备降低、卵巢反应低下,临床表现为稀发排卵和月经不规律,最终导致卵巢的衰竭和闭经。2018年首次报道了POI患者和小鼠中的m6A含量明显高于对照组,而基因的mRNA和蛋白表达水平则明显低于对照组,这可能是POI风险增加的原因[68]。近来研究发现,环磷酰胺(cyclophosphamide,CTX)以时间和浓度依赖性的方式提高了大多数甲基转移酶的表达水平,并抑制FTO和m6A结合蛋白的表达[69]。
FTO在NOA和POI中的研究表明m6A在卵巢正常和病理衰老过程可能共用类似的调控机制。此外还发现,补充褪黑素可以通过抑制ROS-YTHDF2- MAPK-NF-κB通路的方式保护卵巢上皮细胞[70]。这些结果说明恢复正常的m6A水平可能延缓卵巢衰老。但是,卵巢衰老中其他m6A致病机制和调控网络并不清楚,有待进一步研究。
4 m6A与生殖系统疾病
表观遗传调控对于女性生殖系统健康具有重要影响。诸多证据表明m6A修饰在女性生殖系统生理和病理过程中发挥着重要作用,与多种生殖系统疾病密切相关,表2汇总了这些过程和调控因子。了解驱动这些过程的潜在机制,对于寻找疾病治疗靶点和保护女性生殖健康有着重要意义。

表2 m6A在女性生殖系统发育、衰老和疾病中的作用
4.1 多囊卵巢综合征
多囊卵巢综合征(polycystic ovary syndrome,PCOS)的病理学特征是颗粒细胞功能障碍导致的排卵障碍和高雄激素血症,其发病率较高,严重影响着女性的生殖和生理健康[71]。研究报道,PCOS患者黄体化颗粒细胞中的m6A水平显著增加,但FOXO3 mRNA的m6A修饰减少。值得注意的是,选择性敲除m6A甲基转移酶或去甲基化酶,仅改变了对照组中FOXO3的表达水平,但PCOS组无明显变化。这些结果表明,在PCOS患者中m6A介导的FOXO3转录后表达调控异常[72]。另一项研究发现,FTO过表达减少了FLOT2转录本的m6A修饰,增强了其在颗粒细胞中的表达,诱发颗粒细胞功能障碍。这可能与肥胖型PCOS的病理生理学有关,因为FLOT2可以调节细胞增殖、凋亡和胰岛素抵抗[73]。
4.2 子宫内膜异位
在子宫内膜异位中则发现m6A含量急剧下降,主要归因于METTL3的减少和ALKBH5的增加。敲低促进子宫内膜基质细胞的迁移和侵袭,激发子宫内膜异位症。生物信息学分析显示,与正常子宫内膜相比,异位子宫内膜中大多数m6A修饰调节因子表达失调[74]。在这些调节因子中,hnRNPA2B1和hnRNPC与子宫内膜异位症的严重程度密切相关,可能与免疫应答有关,可作为诊断子宫内膜异位症的生物标志物[75]。类似的,在子宫肌层功能障碍患者中,同样发现m6A调节因子的表达模式与正常组织不同,其靶基因可能潜在地影响免疫反应和细胞粘附[76]。
4.3 先兆子痫
在先兆子痫中,胎盘滋养层中的表达上调,整体m6A甲基化修饰水平有所增加[77]。研究人员还发现在PE患者的胎盘中,去甲基化酶ALKBH5的表达显著上调,而抑制其表达可减轻妊娠小鼠的PE症状。表达下调可增加PPARG mRNA的m6A修饰水平,使其表达上调;而PPARG可通过抑制ALCAM启动子区域的组氨酸甲基化水平增加后者的表达水平,进而激活Wnt/β-catenin通路,最终减缓PE进展[78]。
4.4 生殖系统肿瘤
m6A修饰已被证实在多种女性生殖系统肿瘤的发生发展中发挥重要作用,包括卵巢癌、子宫内膜癌和宫颈癌等。接受化疗或放疗的年轻女性患者,术后可能会出现性腺损伤、卵巢早衰,严重者可致不孕不育。癌症基因组图谱(The Cancer Genome Atlas,TCGA)和基因型组织表达(Genotype-Tissue Expression,GTEx)数据集的生物信息学分析结果显示,与正常组织相比,m6A修饰相关蛋白因子在癌组织中的异常表达模式各不相同[79]。值得注意的是,在不同肿瘤中或同一肿瘤在不同环境下,m6A调节因子发挥的功能也不尽相同,其可能促进癌症发展,也可能发挥抑癌作用。m6A修饰的功能双重性意味着其调控机制可能比预想的更为复杂,且受到细胞微环境的影响。
5 m6A修饰的检测识别
m6A甲基化在20世纪70年代被初次发现,此后受限于当时的测序技术和生化分析方法,无法对其进行大规模检测和功能鉴定,仅有少数修饰位点被定位在病毒和细胞RNA中。近年来,随着高通量测序技术出现和发展,快速有效地大规模检测m6A修饰成为可能。借助m6A测序技术,研究人员得以更有效地研究表观遗传调控信号变化。在卵巢衰老和生殖系统疾病研究中,m6A测序已经成为筛选m6A蛋白作用靶点的首选方法,极大丰富了对m6A甲基化修饰在生殖系统发育和衰老过程中功能作用的了解,并在修饰异常与临床疾病的关联上取得突破[80]。新的m6A测序和鉴定技术的出现与组合使用有利于减小对稀有样品的需求,降低批次效应,同时更精确地检测修饰位点和定量修饰水平,帮助理解上下游调控机制[81],将基础研究成果转化为临床治疗应用。目前,m6A检测技术在成本、分辨率、定量水平和灵敏度等方面具有不同的优缺点,研究者应根据需要选择合适的检测方法。表3汇总了当前主流m6A检测技术及其特点。
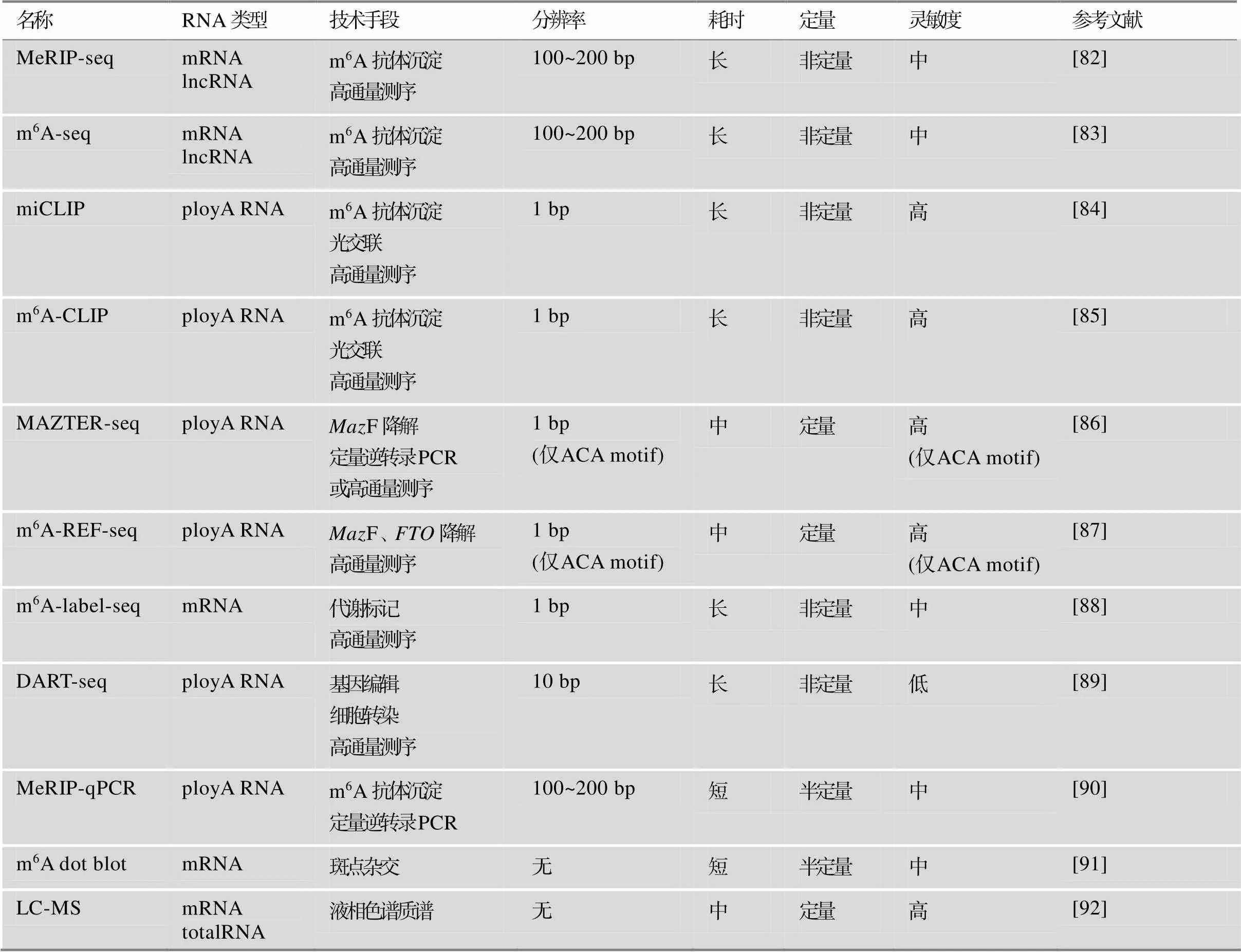
表3 不同m6A检测方法比较
5.1 高通量检测方法
大多数RNA修饰是低丰度的,且由于单链RNA的不稳定性和结构的复杂性,很难直接检测RNA上的化学修饰。在单链RNA反转录成双链cDNA的建库过程中,由于无法根据腺苷酸6位置的甲基化修饰状态对测序片段进行区分,导致m6A修饰相关信息的丢失。因此,鉴定m6A修饰的关键即在于将修饰与未修饰的RNA片段区分开,据此催生出多种基于抗体的高通量检测方法。
2012年,两个研究小组各自独立地开发了基于抗体的m6A检测方法:MeRIP-seq[82]和m6A-seq[83]。这两种方法首次将m6A修饰映射到mRNA上,第一次展现了m6A修饰景观,在人类转录组中发现了超过7000个m6A修饰位点,直到现在二者仍然是检测m6A修饰最常用的测序技术。简单来讲,MeRIP-seq/ m6A-seq需准备两个文库:免疫沉淀文库(immuneprecipitation,IP)和RNA-seq文库(Input)。Input文库直接使用传统方法构建,而IP文库则需先使用特异性抗体对m6A修饰的RNA片段进行免疫沉淀。在m6A修饰位点附近,读段(reads)的出现频率增加,会形成一个约两倍读长的峰(peak),据此可识别甲基化修饰的转录本及其m6A修饰位点的位置。MeRIP-seq/m6A-seq是一个标准化流程,作为开创性的方法无疑是非常成功的,在m6A修饰对生长发育、疾病和癌症的作用研究中被广泛应用。图3A展示了MeRIP-seq的实验流程和基本原理。
为进一步提高m6A修饰位点的检测分辨率,miCLIP[84]和m6A-CLIP[85]方法被开发出来。首先将纯化后的RNA打断,再与抗体共沉淀,并在254纳米的紫外线下进行交联,之后用变性聚丙烯酰胺凝胶电泳和硝酸纤维素(nitrocellulose,NC)膜转移共价结合的m6A抗体-RNA复合物。未交联的RNA片段不会被收集到膜上,这样就减少了背景噪音。随后释放RNA片段进行逆转录和高通量测序。尽管此类方法价格较贵,耗费时间长,但是通过使用不同的抗体有效地区分了m6A与m6Am,并将m6A识别的分辨率提高到了单碱基水平,使得其成为研究m6A的重要工具。
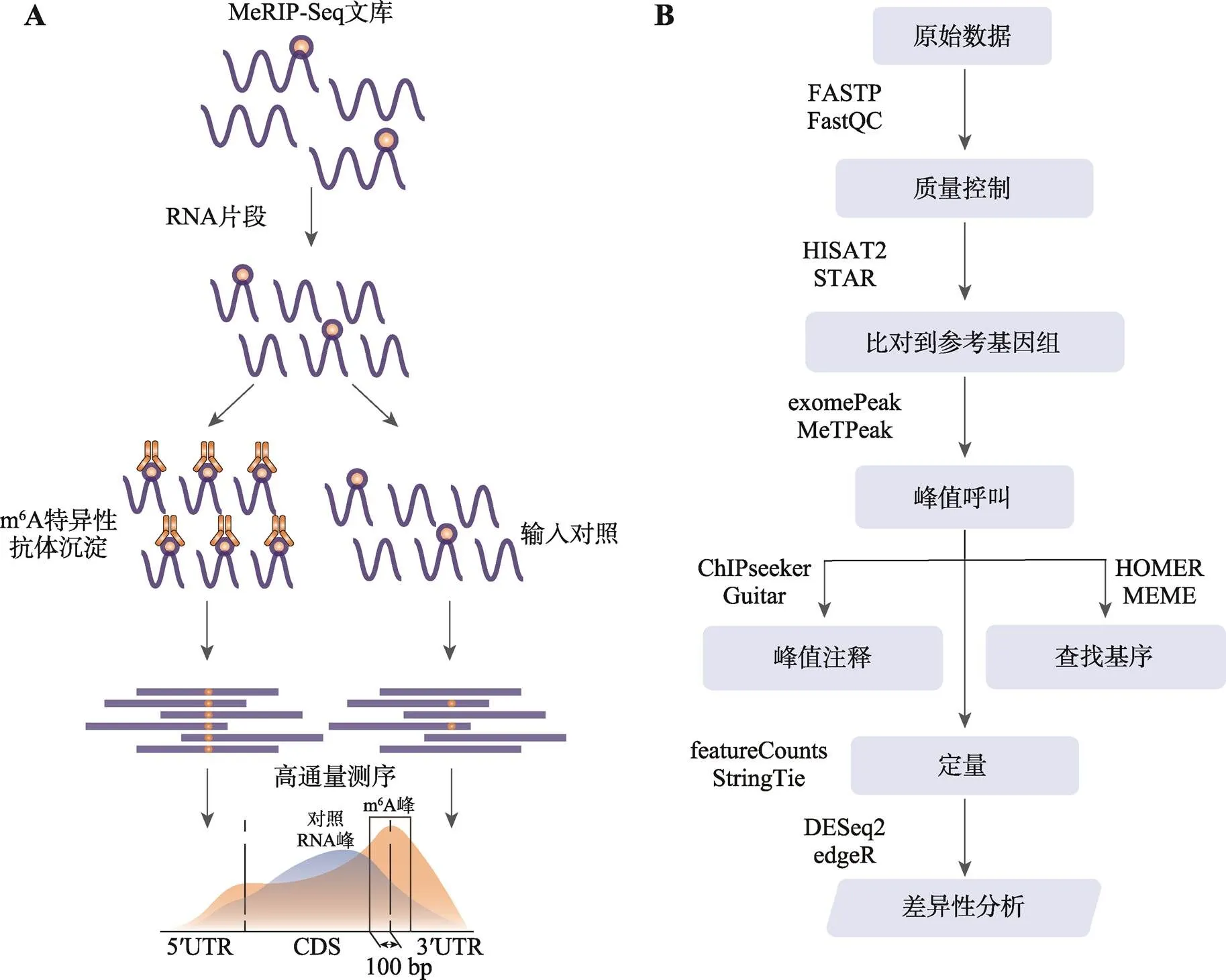
图3 MeRIP-seq实验及其数据分析流程
A:MeRIP-seq测序原理示意图;B:m6A数据分析方法流程图。
抗体介导的方法由于抗体亲和力的变化和批次效应,有着可重复差和分辨率低等问题,而其改进方法基本都需要复杂的建库流程。因此,需要一种简单的、不使用抗体沉淀的方法确定m6A修饰的单核苷酸位置。F是一种可特异性识别ACA基序,并在第一个A的5′端对单链RNA进行切割的限制性核酸内切酶。但如果第一个A携带m6A修饰(m6ACA),则阻断F的识别。联用F和NGS技术,两个小组独立开发了MAZTER-seq[86]和m6A-REF-seq[87]。测序流程只需将纯化的RNA用F水解,然后直接进行高通量测序,通过分析限制性酶切位点位于reads的内部还是末端来鉴定m6A修饰位点。由于F的高灵敏度和特异性,可区分m6A与m6Am。此外,以去甲基化酶处理过的mRNA作为阴性对照,可大大降低检测结果的假阳性。
利用m6A代谢途径,将6甲基基团替换成更容易识别和纯化的其他类型修饰,进而便捷高效地检测m6A修饰,m6A-label-seq技术是此类方法的典型代表[88]。S-腺苷甲硫氨酸(S-adenosyl methionine,SAM)是甲基转移酶的辅助因子,负责将其甲基转移到腺嘌呤。在m6A-label-seq测序中,烯丙基甲硫氨酸被转化为烯丙基-SAM或其硒同源物烯丙基- SeAM。这种烯丙基取代的辅助因子,将在原来的m6A位点生成6-烯丙基腺苷,诱导形成环化的1-6环化腺苷(cyc-A),导致在逆转录过程中碱基不匹配。通过生物信息学分析突变位点,即可得到单碱基分辨率的m6A修饰图谱。
随着基因工程的发展,CRISPR基因编辑技术在测序领域的应用逐渐普及。利用基因编辑技术将胞嘧啶脱氨酶(APOBEC1)和家族基因融合,可使APOBEC1在YTH结构域的帮助下定位到m6A位点,进而将m6A基序RRACH中的C变为U。随后NGS测序可检测到发生在A之后的C-U转化位点,DART-seq[89]即基于此原理检测m6A修饰位点。作为补充方法,其对于直接检测生物体中m6A修饰位点的改变有很好的效果。
5.2 低通量检测技术
高通量的m6A修饰检测方法仍面临一些挑战,例如两个文库所带来的成本增长;m6A 抗体特异性不够显著,使其也可与m6Am等其他位点结合;实验环境对m6A抗体结合灵敏度的影响;以及低分辨率导致修饰位点无法准确定位等问题。对此,研究人员开发了一些低通量的检测手段,以降低成本,提高m6A修饰识别和定量的准确性。
MeRIP-qPCR[90]适用于鉴定特定RNA上的m6A修饰。富集的RNA-抗体复合物首先被蛋白酶去除抗体,然后将RNA进行RT-PCR。这种改进提高了灵敏度,对于难以获取的样本,只需少量样本即可检测m6A修饰并进行半定量。通常MeRIP-seq方法用于较大范围地筛选m6A修饰,然后采用MeRIP- qPCR对结果进行验证,同时研究其对特定转录产物的调控功能。m6A dot plot[91]在m6A分析中主要是在半定量时使用。将含有RNA混合物的样品涂抹在NC膜上形成一个点,嵌入RNA并干燥膜后,用抗体检测生物分子。Dot plot技术明显节省了时间,因为它不需要色谱、凝胶电泳等复杂程序。然而其只能验证m6A的存在或比较不同组间m6A的数量,不能确定m6A的位置和定量。
鉴于m6A具有其特定的物理和化学特性,由此可基于单核苷酸降解和紫外线检测方法对其进行测定。液相色谱质谱技术(liquid chromatograph mass spectrometer,LC-MS)[92]已成为测定RNA整体m6A修饰水平的标准定量方法。RNase T1和RNase A是最常见的RNA降解酶,将RNA切成小的寡聚核苷酸片段或单核苷酸。单个核苷酸可以与正常核苷酸进行比较,以定量m6A的水平。LC-MS操作简单且灵敏度高,但不能确定m6A修饰位点的位置,也不能确定修饰的RNA。因此,其仅可用于捕获m6A修饰水平的整体动态变化。
5.3 生物信息学工具
随着RNA修饰高通量测序技术的出现和发展,利用组学数据破译表观转录组已成为生物信息学领域新兴的重大挑战。基于MeRIP-seq组学数据的m6A修饰分析流程(图3B)主要包含以下步骤:(1)对数据进行质控及预处理,将reads映射到参考基因组;(2)根据IP和Input文库中的reads分布进行m6A峰检测(call peak),对峰值区域进行注释、Motif挖掘;(3)对基因或转录本的m6A修饰水平进行定量及差异表达分析。
在MeRIP-seq数据分析流程中,最关键的步骤是m6A峰值检测。峰值检测基于以下假设:reads随机分布在基因组上,当某个位点被m6A修饰时,在IP样本中这一位点区域会有远高于随机分布的reads富集,即peak区域。将基因组划分为一个个小的序列窗口,将reads与窗口间的序列比对匹配结果看作是伯努利实验。由于reads和窗口数量极多,并且每一个reads匹配到窗口上的概率很小,根据概率论原理,分布就会趋近于泊松分布据此,可以估计窗口上reads的分布频率,利用假设检验给出峰的分布并计算其置信度。
从技术角度来看,MeRIP-seq可以被认为是两种测序技术的结合:ChIP-seq和RNA-seq。起初由于生信工具的匮乏,研究人员通常使用以MACS2[93]为代表的针对ChIP-seq测序数据开发的峰值检测方法。MACS2是用来鉴定转录因子结合位点的工具,即检测蛋白质结合的DNA区域。由于是从DNA片段两条链的5′末端测序,因此真正结合位点周围的标签密度应呈现双峰富集模式。据此,MACS2利用这种双峰模式进行经验建模,以更好地精确识别结合位点区域。然而,MeRIP-seq是对m6A修饰的RNA片段进行测序,其与ChIP-seq有着本质的不同。首先,由于MeRIP-seq从cDNA间接测序RNA,需要考虑RNA发生的可变剪接,否则基于ChIP-seq数据的峰值检测方法会错误检测出多个孤立峰;其次,由于RNA分子是单链的,在ChIP-seq中观察到的双链DNA的双峰特性不适用于RIP-seq;再次,不完全和非特异性的免疫沉淀和更容易导致的噪声或缺失;最后,由于RNA片段转录丰度差异、转录起始位点周围的核小体丢失等因素,MeRIP-seq数据的背景分布偏差较大,需要在免疫沉淀前配对片段化的RNA作为对照样品。总之,针对ChIP-seq的峰值检测算法并未根据MeRIP-seq数据的内在特征进行优化。
随着MeRIP-seq等测序技术在m6A相关研究中的大规模应用,开发更为准确高效的峰值检测及定量分析工具成为亟需解决的问题。目前已有多个针对m6A峰值的计算检测工具出现,其中应用最多的是exomePeak[94]。exomePeak对特定基因外显子而非全基因组集合进行峰检测,它可以自动连接由内含子分隔的外显子区域,并将它们整合为单个peak。该方法可有效解决转录丰度和剪接异构体的问题。连接后,用动态平均函数计算滑动窗口内reads密度,背景参数由窗口内的reads或基因的平均tag密度进行估计。使用基于两个Poisson分布均值的比较方法(C-test)对峰进行假设检验。得到的两个显著性水平用Fisher或Stouffer方法结合以推断最终的富集结果。错误发现率由交换IP和input样本的总富集面积来计算。此外,exomePeak对PCR过程产生的多重比对(duplicates)产物设置过滤函数来降低假阳性率。这些方法提高了峰值检测的准确性,使得exomePeak相比MACS2等工具更适合用于m6A测序数据。与MACS2算法相比,虽然MACS2可以得到更多的峰数,但假阳性率偏高,而且根据exomePeak结果鉴定出的m6A motif更为准确,甲基化位点也更接近峰的中心。
值得一提的是,随着以大数据+深度神经网络为代表的人工智能技术的发展,一些基于深度学习模型预测m6A甲基化位点的方法逐渐出现。此外,随着m6A修饰组学数据的大规模挖掘,一些相应的数据库也逐渐建立起来,大大降低了m6A修饰位点相关信息搜集的时间成本,例如RMBase、m6Avar、m6A2target、RMVar、m6A-Atlas等。公共数据的整合尽可能的减小了批次效应的影响,极大地帮助了探索m6A修饰的潜在功能,并对m6A与全基因组关联性和致病机制的研究有着重要意义。
6 结语与展望
m6A修饰有利于细胞对RNA进行分类管理,帮助细胞进行有序代谢和功能调节,进而更有效地调控基因表达[95]。在女性生殖系统中,m6A修饰调节卵泡发育、卵母细胞成熟和母体到合子的过渡,以及体外女性种系干细胞的自我更新。此外,m6A还与多种女性生殖疾病密切相关。然而,m6A修饰及其调节因子的分子作用机制仍不明确,且缺乏体内实验的验证支持,极大限制了其在临床治疗中的转化应用。最近,借助CRISPR基因工程技术可实现对转录本单个位点的甲基化或去甲基化的编辑[96]。m6A在再生医学和精准医学中表现出巨大的应用潜力,更加深入地了解m6A调节机制将有助于理解女性生殖系统发育和衰老的内在机理,为疾病的治疗策略和人工辅助生殖提供更有效的参考。
[1] Tatone C, Amicarelli F. The aging ovary—the poor granulosa cells.. 2013, 99(1): 12–17.
[2] He MN, Zhang T, Yang Y, Wang C. Mechanisms of oocyte maturation and related epigenetic regulation., 2021, 9: 654028.
[3] An SQ, Huang WX, Huang X, Cun YX, Cheng WS, Sun X, Ren ZJ, Chen YX, Chen WF, Wang JK. Integrative network analysis identifies cell-specific trans regulators of m6A., 2020, 48(4): 1715–1729.
[4] Fu Y, Dominissini D, Rechavi G, He C. Gene expression regulation mediated through reversible m6A RNA methylation., 2014, 15(5): 293–306.
[5] Zhang X, Jia GF. RNA epigenetic modification:6-methyladenosine., 2016, 38(4): 275–288.张笑, 贾桂芳. RNA表观遗传修饰:6-甲基腺嘌呤. 遗传, 2016, 38(4): 275–288.
[6] Zeng CW, Huang WX, Li YQ, Weng HY. Roles of METTL3 in cancer: mechanisms and therapeutic targeting., 2020, 13(1): 117.
[7] Wang X, Feng J, Xue Y, Guan YZ, Zhang DL, Liu Z, Gong Z, Wang Q, Huang JB, Tang C, Zou TT, Yin P. Structural basis of6-adenosine methylation by the METTL3-METTL14 complex., 2016, 534(7608): 575–578.
[8] Zhao BS, Roundtree IA, He C. Post-transcriptional gene regulation by mRNA modifications., 2017, 18(1): 31–42.
[9] Ping XL, Sun BF, Wang L, Xiao W, Yang X, Wang WJ, Adhikari S, Shi Y, Lv Y, Chen YS, Zhao X, Li A, Yang Y, Dahal U, Lou XM, Liu X, Huang J, Yuan WP, Zhu XF, Cheng T, Zhao YL, Wang X, Rendtlew Danielsen JM, Liu F, Yang YG. Mammalian WTAP is a regulatory subunit of the RNA6-methyladenosine methyltransferase., 2014, 24(2): 177–189.
[10] Schwartz S, Mumbach MR, Jovanovic M, Wang T, Maciag K, Bushkin GG, Mertins P, Ter-Ovanesyan D, Habib N, Cacchiarelli D, Sanjana NE, Freinkman E, Pacold ME, Satija R, Mikkelsen TS, Hacohen N, Zhang F, Carr SA, Lander ES, Regev A. Perturbation of m6A writers reveals two distinct classes of mRNA methylation at internal and 5' sites., 2014, 8(1): 284–296.
[11] Wen J, Lv RT, Ma HH, Shen HJ, He CX, Wang JH, Jiao FF, Liu H, Yang PY, Tan L, Lan F, Shi YG, He C, Shi Y, Diao JB. Zc3h13 regulates nuclear RNA m6A methylation and mouse embryonic stem cell self-renewal., 2018, 69(6): 1028–1038.e6.
[12] Patil DP, Chen CK, Pickering BF, Chow A, Jackson C, Guttman M, Jaffrey SR. m6A RNA methylation promotes XIST-mediated transcriptional repression., 2016, 537(7620): 369–373.
[13] Jia GF, Fu Y, Zhao X, Dai Q, Zheng GQ, Yang Y, Yi CQ, Lindahl T, Pan T, Yang YG, He C.6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO., 2011, 7(12): 885–887.
[14] Zheng GQ, Dahl JA, Niu YM, Fedorcsak P, Huang CM, Li CJ, Vågbø CB, Shi Y, Wang WL, Song SH, Lu ZK, Bosmans RPG, Dai Q, Hao YJ, Yang X, Zhao WM, Tong WM, Wang XJ, Bogdan F, Furu K, Fu Y, Jia GF, Zhao X, Liu J, Krokan HE, Klungland A, Yang YG, He C. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility., 2013, 49(1): 18–29.
[15] Xu C, Wang X, Liu K, Roundtree IA, Tempel W, Li YJ, Lu ZK, He C, Min JR. Structural basis for selective binding of m6A RNA by the YTHDC1 YTH domain., 2014, 10(11): 927–929.
[16] Mao YH, Dong LM, Liu XM, Guo JY, Ma HH, Shen B, Qian SB. m6A in mRNA coding regions promotes translation via the RNA helicase-containing YTHDC2., 2019, 10(1): 5332.
[17] Liu T, Wei QL, Jin J, Luo QY, Liu Y, Yang Y, Cheng CM, Li LF, Pi JN, Si YM, Xiao HL, Li L, Rao S, Wang F, Yu JH, Yu J, Zou DL, Yi P. The m6A reader YTHDF1 promotes ovarian cancer progression via augmenting EIF3C translation., 2020, 48(7): 3816– 3831.
[18] Wang X, Zhao BS, Roundtree IA, Lu ZK, Han DL, Ma HH, Weng XC, Chen K, Shi HL, He C.6-methyladenosine modulates messenger RNA translation efficiency., 2015, 161(6): 1388–1399.
[19] Shi HL, Wang X, Lu ZK, Zhao BS, Ma HH, Hsu PJ, Liu C, He C. YTHDF3 facilitates translation and decay of6-methyladenosine-modified RNA., 2017, 27(3): 315–328.
[20] Bi Z, Liu YH, Zhao YL, Yao YX, Wu RF, Liu Q, Wang YZ, Wang XX. A dynamic reversible RNA6-methyladenosine modification: current status and perspectives., 2019, 234(6): 7948–7956.
[21] Huang HL, Weng HY, Sun WJ, Qin X, Shi HL, Wu HZ, Zhao BS, Mesquita A, Liu C, Yuan CL, Hu YC, Hüttelmaier S, Skibbe JR, Su R, Deng XL, Dong L, Sun M, Li CY, Nachtergaele S, Wang YG, Hu C, Ferchen K, Greis KD, Jiang X, Wei MJ, Qu LH, Guan JL, He C, Yang JH, Chen JJ. Recognition of RNA6-methyladenosine by IGF2BP proteins enhances mRNA stability and translation., 2018, 20(3): 285–295.
[22] Liu N, Dai Q, Zheng GQ, He C, Parisien M, Pan T.6-methyladenosine-dependent RNA structural switches regulate RNA-protein interactions., 2015, 518(7540): 560–564.
[23] Dai XX, Pi SB, Zhao LW, Wu YW, Shen JL, Zhang SY, Sha QQ, Fan HY. PABPN1 functions as a hub in the assembly of nuclear poly(A) domains that are essential for mouse oocyte development., 2022, 8(43): eabn9016.
[24] Geula S, Moshitch-Moshkovitz S, Dominissini D, Mansour AA, Kol N, Salmon-Divon M, Hershkovitz V, Peer E, Mor N, Manor YS, Ben-Haim MS, Eyal E, Yunger S, Pinto Y, Jaitin DA, Viukov S, Rais Y, Krupalnik V, Chomsky E, Zerbib M, Maza I, Rechavi Y, Massarwa R, Hanna S, Amit I, Levanon EY, Amariglio N, Stern-Ginossar N, Novershtern N, Rechavi G, Hanna JH. Stem cells. m6A mRNA methylation facilitates resolution of naïve pluripotency toward differentiation., 2015, 347(6225): 1002–1006.
[25] Molinie B, Wang JK, Lim KS, Hillebrand R, Lu ZX, Van Wittenberghe N, Howard BD, Daneshvar K, Mullen AC, Dedon P, Xing Y, Giallourakis CC. m6A-LAIC-seq reveals the census and complexity of the m6A epitranscriptome., 2016, 13(8): 692–698.
[26] Roundtree IA, Luo GZ, Zhang ZJ, Wang X, Zhou T, Cui YQ, Sha JH, Huang XX, Guerrero L, Xie P, He E, Shen B, He C. YTHDC1 mediates nuclear export of6-methyladenosine methylated mRNAs., 2017, 6: e31311.
[27] Jiang JC, Zhang H, Cao LR, Dai XX, Zhao LW, Liu HB, Fan HY. Oocyte meiosis-coupled poly(A) polymerase α phosphorylation and activation trigger maternal mRNA translation in mice., 2021, 49(10): 5867–5880.
[28] Sha QQ, Dai XX, Dang Y, Tang F, Liu JP, Zhang YL, Fan HY. A MAPK cascade couples maternal mRNA translation and degradation to meiotic cell cycle progression in mouse oocytes., 2017, 144(3): 452–463.
[29] Dai XX, Jiang JC, Sha QQ, Jiang Y, Ou XH, Fan HY. A combinatorial code for mRNA 3'-UTR-mediated translational control in the mouse oocyte., 2019, 47(1): 328–340.
[30] Fabian MR, Sonenberg N, Filipowicz W. Regulation of mRNA translation and stability by microRNAs., 2010, 79: 351–379.
[31] Lin SB, Choe J, Du P, Triboulet R, Gregory RI. The m6A methyltransferase METTL3 promotes translation in human cancer cells., 2016, 62(3): 335–345.
[32] Jiang ZY, Fan HY. Five questions toward mRNA degradation in oocytes and preimplantation embryos: when, who, to whom, how, and why?†., 2022, 107(1): 62–75.
[33] Wang X, Lu ZK, Gomez A, Hon GC, Yue YN, Han DL, Fu Y, Parisien M, Dai Q, Jia GF, Ren B, Pan T, He C.6-methyladenosine-dependent regulation of messenger RNA stability., 2014, 505(7481): 117–120.
[34] Liu JZ, Yue YN, Han DL, Wang X, Fu Y, Zhang L, Jia GF, Yu M, Lu ZK, Deng X, Dai Q, Chen WZ, He C. A METTL3-METTL14 complex mediates mammalian nuclear RNA6-adenosine methylation., 2014, 10(2): 93–95.
[35] Pan ZX, Zhang JB, Li QF, Li YX, Shi FX, Xie Z, Liu HL. Current advances in epigenetic modification and alteration during mammalian ovarian folliculogenesis., 2012, 39(3): 111–123.
[36] 边立华, 孟元光. 女性生殖系统发育异常的诊断与治疗. 中国妇产科临床杂志, 2017, 18(2): 182–183.
[37] Overland MR, Li Y, Derpinghaus A, Aksel S, Cao M, Ladwig N, Cunha GR, Himelreich-Perić M, Baskin LS. Development of the human ovary: Fetal through pubertal ovarian morphology, folliculogenesis and expression of cellular differentiation markers., 2023, 129: 37–59.
[38] Yao SJ, Lopez-Tello J, Sferruzzi-Perri AN. Developmental programming of the female reproductive system-a review., 2021, 104(4): 745–770.
[39] Cunha GR, Robboy SJ, Kurita T, Isaacson D, Shen J, Cao M, Baskin LS. Development of the human female reproductive tract., 2018, 103: 46–65.
[40] Wang HQ, Zhang JB, Zheng Y, Zhang WD, Guo HX, Cong S, Ding Y, Yuan B. Comprehensive analysis of differences in6-methyladenosine RNA methylomes in the rat adenohypophysis after GnRH treatment., 2022, 36(3): e22204.
[41] Magro-Lopez E, Muñoz-Fernández MÁ. The role of BMP signaling in female reproductive system development and function., 2021, 22(21): 11927.
[42] Gong YQ, Jiang QS, Liu LJ, Liao QY, Yu J, Xiang Z, Luo XG. METTL3-mediated m6A modification promotes processing and maturation of pri-miRNA-19a to facilitate nasopharyngeal carcinoma cell proliferation and invasion., 2022, 54(9): 337–349.
[43] Tatone C, Amicarelli F, Carbone MC, Monteleone P, Caserta D, Marci R, Artini PG, Piomboni P, Focarelli R. Cellular and molecular aspects of ovarian follicle ageing., 2008, 14(2): 131–142.
[44] Amstislavsky SY, Brusentsev EY, Petrova OM, Naprimerov VA, Levinson AL. Development and aging of the mammalian reproductive system., 2020, 51(1): 45–56.
[45] Gougeon A. Human ovarian follicular development: from activation of resting follicles to preovulatory maturation., 2010, 71(3): 132–143.
[46] Liu CM, Ding LJ, Li JY, Dai JW, Sun HX. Advances in the study of ovarian dysfunction with aging., 2019, 41(9): 816–826.刘传明, 丁利军, 李佳音, 戴建武, 孙海翔. 衰老导致卵巢功能低下研究进展. 遗传, 2019, 41(9): 816–826.
[47] Rimon-Dahari N, Yerushalmi-Heinemann L, Alyagor L, Dekel N. Ovarian folliculogenesis., 2016, 58: 167–190.
[48] Sun XF, Zhang JN, Jia Y, Shen W, Cao HG. Characterization of m6A in mouse ovary and testis., 2020, 10(4): e141.
[49] Xia H, Zhong CR, Wu XX, Chen J, Tao BB, Xia XQ, Shi MJ, Zhu ZY, Trudeau VL, Hu W. Mettl3 mutation disrupts gamete maturation and reduces fertility in zebrafish., 2018, 208(2): 729–743.
[50] Hu Y, Ouyang ZY, Sui XS, Qi MJ, Li MR, He YL, Cao YM, Cao QQ, Lu QN, Zhou S, Liu L, Liu L, Shen B, Shu WJ, Huo R. Oocyte competence is maintained by m6A methyltransferase KIAA1429-mediated RNA metabolism during mouse follicular development., 2020, 27(8): 2468–2483.
[51] Zeng M, Dai X, Liang ZB, Sun RL, Huang S, Luo LP, Li ZX. Critical roles of mRNA m6A modification and YTHDC2 expression for meiotic initiation and progression in female germ cells., 2020, 753: 144810.
[52] Zhao XY, Tian GG, Fang Q, Pei XY, Wang ZX, Wu J. Comparison of RNA m6A and DNA methylation profiles between mouse female germline stem cells and STO cells., 2020, 23: 431–439.
[53] Mu HY, Zhang T, Yang Y, Zhang DR, Gao J, Li JH, Yue L, Gao DF, Shi BB, Han Y, Zhong L, Chen XZ, Wang ZB, Lin Z, Tong MH, Sun QY, Yang YG, Han JY. METTL3- mediated mRNA6-methyladenosine is required for oocyte and follicle development in mice., 2021, 12(11): 989.
[54] Kasowitz SD, Ma J, Anderson SJ, Leu NA, Xu Y, Gregory BD, Schultz RM, Wang PJ. Nuclear m6A reader YTHDC1 regulates alternative polyadenylation and splicing during mouse oocyte development., 2018, 14(5): e1007412.
[55] Tadros W, Lipshitz HD. The maternal-to-zygotic transition: a play in two acts., 2009, 136(18): 3033–3042.
[56] Sha QQ, Zhang J, Fan HY. A story of birth and death: mRNA translation and clearance at the onset of maternal- to-zygotic transition in mammals†., 2019, 101(3): 579–590.
[57] Sha QQ, Zhu YZ, Li S, Jiang Y, Chen L, Sun XH, Shen L, Ou XH, Fan HY. Characterization of zygotic genome activation-dependent maternal mRNA clearance in mouse., 2020, 48(2): 879–894.
[58] Zhao BS, Wang X, Beadell AV, Lu ZK, Shi HL, Kuuspalu A, Ho RK, He C. m6A-dependent maternal mRNA clearance facilitates zebrafish maternal-to-zygotic transition., 2017, 542(7642): 475–478.
[59] Ivanova I, Much C, Di Giacomo M, Azzi C, Morgan M, Moreira PN, Monahan J, Carrieri C, Enright AJ, O'Carroll D. The RNA m6A reader YTHDF2 is essential for the post-transcriptional regulation of the maternal transcriptome and oocyte competence., 2017, 67(6): 1059–1067.e4.
[60] Sha QQ, Zheng W, Wu YW, Li S, Guo L, Zhang SP, Lin G, Ou XH, Fan HY. Dynamics and clinical relevance of maternal mRNA clearance during the oocyte-to-embryo transition in humans., 2020, 11(1): 4917.
[61] West RC, Ming H, Logsdon DM, Sun JW, Rajput SK, Kile RA, Schoolcraft WB, Roberts RM, Krisher RL, Jiang ZL, Yuan Y. Dynamics of trophoblast differentiation in peri-implantation-stage human embryos., 2019, 116(45): 22635–22644.
[62] Qiu WY, Zhou YX, Wu HW, Lv XL, Yang LL, Ren ZX, Tian H, Yu QY, Li J, Lin WX, Zhao L, Luo SP, Gao J. RNA demethylase FTO mediated RNA m6A modification is involved in maintaining maternal-fetal interface in spontaneous abortion., 2021, 9: 617172.
[63] Li XC, Jin F, Wang BY, Yin XJ, Hong W, Tian FJ. The m6A demethylase ALKBH5 controls trophoblast invasion at the maternal-fetal interface by regulating the stability of CYR61 mRNA., 2019, 9(13): 3853–3865.
[64] Wu YW, Li S, Zheng W, Li YC, Chen L, Zhou Y, Deng ZQ, Lin G, Fan HY, Sha QQ. Dynamic mRNA degradome analyses indicate a role of histone H3K4 trimethylation in association with meiosis-coupled mRNA decay in oocyte aging., 2022, 13(1): 3191.
[65] Liu C, Li LS, Yang B, Zhao YQ, Dong XY, Zhu LX, Ren XL, Huang B, Yue J, Jin L, Zhang HW, Wang L. Transcriptome-wide6-methyladenine methylation in granulosa cells of women with decreased ovarian reserve., 2022, 23(1): 240.
[66] Zhang JJ, Chen Q, Du DF, Wu T, Wen JY, Wu M, Zhang Y, Yan W, Zhou S, Li Y, Jin Y, Luo AY, Wang SX. Can ovarian aging be delayed by pharmacological strategies?, 2019, 11(2): 817–832.
[67] Jiang ZX, Wang YN, Li ZY, Dai ZH, He Y, Chu K, Gu JY, Ji YX, Sun NX, Yang F, Li W. The m6A mRNA demethylase FTO in granulosa cells retards FOS-dependent ovarian aging., 2021, 12(8): 744.
[68] Ding CY, Zou QY, Ding J, Ling MF, Wang W, Li HX, Huang B. Increased6-methyladenosine causes infertility is associated with FTO expression., 2018, 233(9): 7055–7066.
[69] Huang BX, Ding CY, Zou QY, Wang W, Li H. Cyclophosphamide regulates6-methyladenosine and m6A RNA enzyme levels in human granulosa cells and in ovaries of a premature ovarian aging mouse model., 2019, 10: 415.
[70] Zhu RG, Ji X, Wu X, Chen JJ, Li XS, Jiang H, Fu HP, Wang H, Lin Z, Tang X, Sun SX, Li QG, Wang BJ, Chen HS. Melatonin antagonizes ovarian aging via YTHDF2- MAPK-NF-κB pathway., 2020, 9(2): 494–509.
[71] Cooney LG, Lee I, Sammel MD, Dokras A. High prevalence of moderate and severe depressive and anxiety symptoms in polycystic ovary syndrome: a systematic review and meta-analysis., 2017, 32(5): 1075–1091.
[72] Zhang S, Deng WL, Liu QY, Wang PY, Yang W, Ni WH. Altered m6 A modification is involved in up-regulated expression of FOXO3 in luteinized granulosa cells of non-obese polycystic ovary syndrome patients., 2020, 24(20): 11874–11882.
[73] Zhou L, Han X, Li W, Wang N, Yao L, Zhao YH, Zhang LQ.6-methyladenosine demethylase FTO induces the dysfunctions of ovarian granulosa cells by upregulating flotillin 2., 2022, 29(4): 1305–1315.
[74] Li XO, Xiong WQ, Long XF, Dai X, Peng Y, Xu Y, Zhang ZB, Zhang L, Liu Y. Inhibition of METTL3/m6A/miR126 promotes the migration and invasion of endometrial stromal cells in endometriosis†., 2021, 105(5): 1221–1233.
[75] Jiang L, Zhang MM, Wu JN, Wang SX, Yang X, Yi MY, Zhang XY, Fang XL. Exploring diagnostic m6A regulators in endometriosis., 2020, 12(24): 25916–25938.
[76] Zhai JY, Li S, Sen S, Opoku-Anane J, Du YZ, Chen ZJ, Giudice LC. m6A RNA methylation regulators contribute to eutopic endometrium and myometrium dysfunction in adenomyosis., 2020, 11: 716.
[77] Gu Y, Chu XD, Morgan JA, Lewis DF, Wang YP. Upregulation of METTL3 expression and m6A RNA methylation in placental trophoblasts in preeclampsia.,2021,103:43–49.
[78] Guo YP, Song WX, Yang YL. Inhibition of ALKBH5- mediated m6A modification of PPARG mRNA alleviates H/R-induced oxidative stress and apoptosis in placenta trophoblast., 2022, 37(4): 910–924.
[79] Chen JY, Fang YW, Xu Y, Sun HT. Role of m6A modification in female infertility and reproductive system diseases., 2022, 18(9): 3592–3604.
[80] Huang H, Wang YN, Kandpal M, Zhao GY, Cardenas H, Ji YR, Chaparala A, Tanner EJ, Chen JJ, Davuluri RV, Matei D. FTO-dependent6-methyladenosine modifications inhibit ovarian cancer stem cell self-renewal by blocking cAMP signaling., 2020, 80(16): 3200–3214.
[81] Wu SQ, Liu KT, Zhou BY, Wu SW.6-methyladenosine modifications in maternal-fetal crosstalk and gestational diseases., 2023, 11: 1164706.
[82] Meyer KD, Saletore Y, Zumbo P, Elemento O, Mason CE, Jaffrey SR. Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons., 2012, 149(7): 1635–1646.
[83] Dominissini D, Moshitch-Moshkovitz S, Schwartz S, Salmon-Divon M, Ungar L, Osenberg S, Cesarkas K, Jacob-Hirsch J, Amariglio N, Kupiec M, Sorek R, Rechavi G. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq., 2012, 485(7397): 201–206.
[84] Linder B, Grozhik AV, Olarerin-George AO, Meydan C, Mason CE, Jaffrey SR. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome., 2015, 12(8): 767–772.
[85] Ke S, Alemu EA, Mertens C, Gantman EC, Fak JJ, Mele A, Haripal B, Zucker-Scharff I, Moore MJ, Park CY, Vågbø CB, Kusśnierczyk A, Klungland A, Darnell JE Jr, Darnell RB. A majority of m6A residues are in the last exons, allowing the potential for 3' UTR regulation., 2015, 29(19): 2037–2053.
[86] Garcia-Campos MA, Edelheit S, Toth U, Safra M, Shachar R, Viukov S, Winkler R, Nir R, Lasman L, Brandis A, Hanna JH, Rossmanith W, Schwartz S. deciphering the "m6A code" via antibody-independent quantitative profiling., 2019, 178(3): 731–747.e16.
[87] Zhang Z, Chen LQ, Zhao YL, Yang CG, Roundtree IA, Zhang ZJ, Ren J, Xie W, He C, Luo GZ. Single-base mapping of m6A by an antibody-independent method., 2019, 5(7): eaax0250.
[88] Shu X, Cao J, Cheng MH, Xiang SY, Gao MS, Li T, Ying XE, Wang FQ, Yue YN, Lu ZK, Dai Q, Cui XL, Ma LJ, Wang YZ, He C, Feng XH, Liu JZ. A metabolic labeling method detects m6A transcriptome-wide at single base resolution., 2020, 16(8): 887–895.
[89] Meyer KD. DART-seq: an antibody-free method for global m6A detection., 2019, 16(12): 1275–1280.
[90] Wang Y, Li Y, Toth JI, Petroski MD, Zhang Z, Zhao JC.6-methyladenosine modification destabilizes developmental regulators in embryonic stem cells., 2014, 16(2): 191–198.
[91] Nagarajan A, Janostiak R, Wajapeyee N. Dot blot analysis for measuring global6-methyladenosine modification of RNA., 2019, 1870: 263–271.
[92] Thüring K, Schmid K, Keller P, Helm M. LC-MS analysis of methylated RNA., 2017, 1562: 3–18.
[93] Feng JX, Liu T, Qin B, Zhang Y, Liu XS. Identifying ChIP-seq enrichment using MACS., 2012, 7(9): 1728–1740.
[94] Meng J, Cui XD, Rao MK, Chen YD, Huang YF. Exome-based analysis for RNA epigenome sequencing data., 2013, 29(12): 1565–1567.
[95] Yang Y, Chen YS, Sun BF, Yang YG. RNA methylation: regulations and mechanisms., 2018, 40(11): 964–976.杨莹, 陈宇晟, 孙宝发, 杨运桂. RNA甲基化修饰调控和规律. 遗传, 2018, 40(11): 964–976.
[96] Li JX, Chen ZJ, Chen F, Xie GY, Ling YY, Peng YX, Lin Y, Luo N, Chiang CM, Wang HS. Targeted mRNA demethylation using an engineered dCas13b-ALKBH5 fusion protein., 2020, 48(10): 5684– 5694.
Effects of6-methyladenosine modification on the function of the female reproductive system
Shan He, Jian Zhao, Xiaofeng Song
In recent years, the rate of female infertility in China has been increasing, posing an urgent challenge to improve fertility. A healthy reproductive system is essential for successful reproduction, and6-methyladenosine (m6A) is the most abundant chemical modification in eukaryotes and plays a critical role in cellular processes. Recent studies have shown that m6A modifications also have a keying effect in various physiological and pathological processes in the female reproductive system, although their regulatory mechanisms and biological functions remain unclear. In this review, we first introduce the reversible regulatory mechanisms of m6A and its functions, discuss the role of m6A in female reproductive function and disorders of the reproductive system, and present recent advances in m6A detection technologies and methods. Our review provides new insights into the biological role of m6A and its potential application in the treatment of female reproductive disorders.
m6A; methylation modification; female reproductive system; development and aging;diseases
2023-03-23;
2023-05-12;
2023-05-17
江苏省重点研发计划项目(编号:BE2022843)和国家自然科学基金项目(编号:62273175, 62003165, 61973155)资助[Supported by the Key R&D Plan Projects in Jiangsu Province (No. BE2022843), and the National Natural Science Foundation of China (Nos. 62273175, 62003165, 61973155)]
何山,在读硕士研究生,专业方向:生物信息学。E-mail: heshan@nuaa.edu.cn
宋晓峰,博士,教授,研究方向:生物信息学。E-mail: xfsong@nuaa.edu.cn
赵健,博士,副研究员,研究方向:生物信息学。E-mail: zhaojian@nuaa.edu.cn
10.16288/j.yczz.23-071
(责任编委: 严冬)
