鸡NRG4基因组及转录本结构分析
2023-05-23高智慧黄佳新罗昊玉徐海冬娄明宁博林邢晓旭牟芳李辉王宁
高智慧,黄佳新,罗昊玉,徐海冬,娄明,宁博林,邢晓旭,牟芳,李辉,王宁
研究报告
鸡基因组及转录本结构分析
高智慧1,2,3,黄佳新1,2,3,罗昊玉1,2,3,徐海冬1,2,3,娄明1,2,3,宁博林1,2,3,邢晓旭1,2,3,牟芳1,2,3,李辉1,2,3,王宁1,2,3
1. 农业农村部鸡遗传育种重点实验室,哈尔滨 150030 2. 黑龙江省普通高等学校动物遗传育种与繁殖重点实验室,哈尔滨 150030 3. 东北农业大学动物科学技术学院,哈尔滨 150030
神经调节蛋白4(neuregulin 4,NRG4)是一个重要的脂肪细胞因子,在维持哺乳动物能量平衡、调节糖脂代谢和预防非酒精性脂肪性肝病中起着非常重要的作用。目前,人基因的基因组结构、转录异构体和蛋白异构体等已有深入的研究。本实验室前期研究显示,鸡脂肪组织也表达,但是目前有关鸡(chicken,)基因的基因组结构、转录异构体和蛋白异构体尚不清楚。为此,本研究采用RACE(rapid-amplification of cDNA ends)和RT-PCR(reverse transcription-PCR)等技术,系统开展了基因的基因组和转录本的结构分析。结果发现,基因编码区很小,但该基因却非常复杂,存在选择性转录起始位点、选择性拼接、内含子滞留、隐匿外显子和选择性多聚腺苷酸化,这导致基因产生4种不同的5ʹUTR异构体(、、和)和6种不同的3ʹUTR异构体(、、、、和)。基因组结构分析发现,基因跨越基因组21,969 bp (Chr.10: 3,490,314~3,512,282),由11个外显子和10个内含子构成。与NCBI数据库中的基因mRNA序列(NM_001030544.4)相比,本研究发现了基因的2个新外显子和1个隐匿外显子。生物信息学分析、RT-PCR、克隆和测序分析发现,基因能编码3种蛋白异构体(cNRG4-1、cNRG4-2和 cNRG4-3)。本研究为进一步开展基因的功能和调控研究奠定了基础。
鸡;NRG4;基因组结构;转录异构体;蛋白异构体
神经调节蛋白4(neuregulin4,NRG4)是一个重要的脂肪细胞因子,属于EGF(epidermal growth factor)家族。哺乳动物的研究发现,基因在棕色脂肪组织中高表达,此外,在白色脂肪组织、胰腺、肺、心脏等多种器官和组织中也有不同程度的表达[1]。NRG4通过其EGF结构域与受体ErbB4(v-erb-b2 avian erythroblastic leukemia viral oncogene homolog 4)的胞外区域相结合,使ErbB4和ErbB3发生二聚化,进而激活下游磷脂酰肌醇(phosphatidyl inositol 3 kinase,PI3K)和丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)等信号通路[2],从而促进细胞增殖,抑制细胞凋亡以及改善细胞能量代谢等。临床研究显示,成人非酒精性脂肪性肝病(nonalcoholic fatty liver disease,NAFLD)的发生率与血清NRG4水平呈负相关[3]。对转基因小鼠()的研究发现,NRG4可降低肝脏的脂肪合成,改善NAFLD和胰岛素抵抗[4]。流行病学调查显示,糖尿病患者血清中的NRG4含量明显高于正常对照组[5]。这些研究提示,NRG4有望成为多种疾病的潜在治疗靶点。
神经调节蛋白(neuregulins,NRGs)家族包括4个成员,分别为NRG1、NRG2、NRG3和NRG4,这4个NRG家族成员均具有多个转录异构体和蛋白异构体[6~9]。人基因包含9个外显子和8个内含子,跨越基因组71,509 bp[8]。人基因能产生、、、和共5种转录异构体,这些转录异构体拥有相同的起始密码子,由于选择性剪接它们的终止密码子位置不同,导致人基因的5种转录异构体编码5种不同的NRG4蛋白异构体(NRG4A1、NRG4A2、NRG4B1、NRG4B2和NRG4B3)[10]。其中NRG4A1和NRG4A2具有完整的EGF结构域和跨膜结构域,定位于细胞膜,经过ADAM/TACE型蛋白酶加工后,两者位于膜外的EGF结构域被释放到细胞外,作为分泌因子参与机体的调控[8]。与NRG4A1和NRG4A2相比,其余3种NRG4异构体(NRG4B1~3)缺失跨膜结构域,EGF结构域也不完整,均定位在细胞质[11]。
与哺乳动物不同,鸡()缺乏棕色脂肪组织。本实验室前期研究显示,鸡腹部脂肪组织表达,并且发现东北农业大学高脂系肉鸡脂肪组织表达量是低脂系肉鸡脂肪组织表达量的2.55倍[12]。目前人们对哺乳动物基因的基因组结构、转录异构体和蛋白异构体都已有了深入研究,但对鸡(chicken,)基因的基因组结构、转录异构体和蛋白异构体还不甚了解。本文采用RACE(rapid-amplification of cDNA ends)和RT-PCR(reverse transcription-PCR)等技术,对基因的基因组和转录本结构进行了分析。
1 材料与方法
1.1 实验材料
克隆载体pEASY-Blunt Simple和pEASY-T1 Simple、菌株Trans1-T1 Phage Resistant Chemically Competent Cell、Trans2K DNA Marker和Trans5K DNA Marker均购自北京全式金生物技术有限公司;AxyPrep DNA凝胶回收试剂盒购自美国Axygen公司;Phanta Max super-fidelity DNA polymerase 购自南京Vazyme公司;RNAiso Plus、SMARTer®RACE 5ʹ/3ʹKit和PrimeScriptTMRT reagent Kit with gDNA Eraser购自宝生物工程(大连)有限公司;FirstChoiceTMRLM-RACE Kit购自美国Thermo Fisher公司;5ʹ/3ʹRACE Kit,2nd Generation购自德国Roche公司。腹部脂肪组织采自4周龄AA白羽肉鸡(东北农业大学阿城实习基地)。
1.2 引物设计与合成
根据NCBI数据库中基因(NM_ 001030544.4)序列,利用Oligo 7软件设计基因的RACE引物和RT-PCR引物(表1),由北京睿博兴科生物技术有限公司合成。
1.3 RNA提取
取AA白羽肉鸡腹部脂肪组织样,置于研钵中,加入液氮充分研磨成粉末,采用RNAiso Plus试剂提取RNA。使用紫外分光光度计测定样品总RNA在260 nm与280 nm的吸光度,根据两者的比值(260/280)评价总RNA的质量,并计算总RNA的浓度,分装冻存于–80℃。
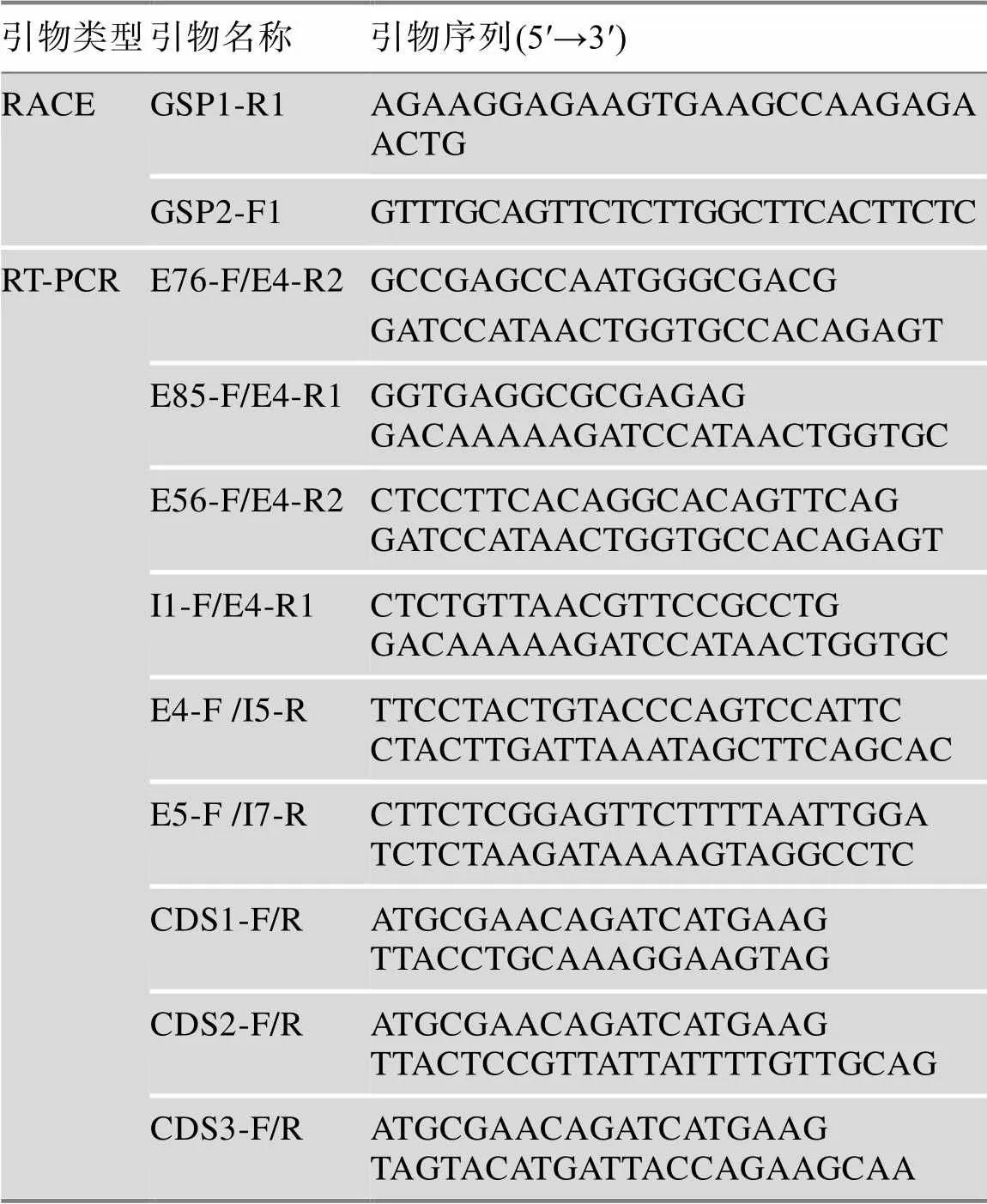
表1 本研究所用引物信息
1.4 cDNA末端快速扩增(RACE)
按照SMARTer®RACE 5ʹ/3ʹKit、FirstChoiceTMRLM-RACE Kit和5ʹ/3ʹRACE Kit,2nd Generation说明书分别扩增基因mRNA的5′和3′端。5′和3′RACE引物序列如表1所示。以鸡腹部脂肪组织提取的混合RNA为模板,分别反转录获得5ʹRACE-Ready cDNA和3ʹRACE-Ready cDNA。利用获得的cDNA分别进行PCR扩增,反应体系均为:PCR-Grade H2O 15.5 µL,2×SeqAmp Buffer 25.0 µL,SeqAmp DNA Polymerase 1.0 µL,5ʹ或者3ʹRACE-Ready cDNA 2.5 µL,10×Universal Primer A Mix(UPM)(由试剂盒提供)5 µL,5ʹ或者3ʹGSP 1 µL,总体积为50 µL。
基因5ʹ和3ʹRACE的PCR反应条件均为:94℃ 5 min;94℃ 30 s,72℃ 3 min,5个循环;94℃ 30 s,70℃ 30 s,72℃ 3 min,5个循环;94℃ 30 s,68℃ 30 s,72℃ 3 min,25个循环;72℃ 5 min。5ʹ和3ʹRACE的PCR产物经过琼脂糖凝胶电泳之后,进行切胶回收,使用AxyPrep DNA凝胶回收试剂盒对RACE-PCR产物进行凝胶纯化。将纯化的5ʹ和3ʹRACE的PCR产物分别与pEASY-T1 Simple克隆载体连接,将连接产物转化Trans1-T1感受态细胞后,涂布氨苄青霉素(Ampicillin,Amp)抗性LB平板,37℃过夜培养,然后随机挑选阳性菌落,由北京睿博兴科生物技术有限公司进行测序。
1.5 RT-PCR检测
RT-PCR用于新发现的外显子和内含子滞留的验证以及CDS区克隆。取鸡腹部脂肪组织总RNA (1 µg),按照PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒说明书,去除基因组DNA并反转录合成cDNA。以获得的cDNA为模板进行PCR扩增,RT-PCR引物序列如表1所示。RT-PCR反应体系为:2×Phanta Max Buffer 25 µL,dNTP Mix(10 mmol/L) 1 µL,上、下游引物(10 µmol/L)各2 µL,Phanta Max Super-Fidelity DNA Polymerase 1 µL,模板1~5 µL,加ddH2O至总体积为50 µL。反应条件:95℃预变性3 min;95℃变性 15 s,60℃退火15 s,72℃延伸30 s,共35个循环;72℃延伸5 min。RT-PCR产物经过琼脂糖凝胶电泳之后,进行切胶回收,使用AxyPrep DNA凝胶回收试剂盒对产物进行凝胶纯化。将RT-PCR产物和pEASY-Blunt Simple克隆载体连接,将连接产物转化Trans1-T1感受态细胞后,涂布Amp抗性LB平板,37℃过夜培养,随机挑选阳性菌落,由北京睿博兴科生物技术有限公司进行测序。
1.6 生物信息学分析
利用UCSC(http://genome.ucsc.edu/)和NCBI(http:// www.ncbi.nlm.nih.gov)数据库查询基因序列;采用Oligo 7软件进行引物设计;采用DNAMAN 6.0进行序列比对;采用Chromas查看测序峰图;采用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测编码蛋白;采用Signal IP-5.0(http://www.cbs. dtu.dk/services/SignalP/)预测分泌信号肽;采用TMHMM2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构;采用SMART(http://smart.embl- heidelberg.de/)分析蛋白序列;使用ITB tools (http://itbtools.ba.itb.cnr.it/utrscan)分析多聚腺苷酸化信号(polyadenylation signal,PAS)。
2 结果与分析
2.1 鸡NRG4基因5ʹ末端克隆和序列分析
为了确定mRNA转录本的5ʹ端序列,取5只AA白羽肉鸡腹部脂肪组织的混合RNA进行5ʹRACE分析。琼脂糖电泳分析显示,基因5ʹRACE扩增产物主要有3条带,大小分别约为600 bp、500 bp和400 bp(图1A)。将5ʹRACE扩增产物进行凝胶回收和克隆,共随机挑选187个重组质粒进行测序分析。测序结果与基因mRNA序列(NM_001030544.4)和基因组序列(NC_052541.1)比对发现,这些5ʹRACE扩增产物序列与基因的外显子3(E3)、E4和E5序列完全相同,但E3上游序列差异较大。根据E3上游序列的不同,将这些基因5ʹRACE扩增产物命名为(19个克隆)、(147个克隆)、(17个克隆)和(4个克隆)(图1B)。与NM_001030544.4相比,本研究发现、和分别存在1个新的外显子,大小分别是76 bp、85 bp和56 bp,按照NM_ 001030544.4的外显子命名,本研究将新发现的这3个基因外显子分别命名为E76、E85和E56。其中E85和E76位于E1的上游,E76的3ʹ端与E85的5ʹ端有38 bp的序列重叠,两者为互斥外显子(mutually exclusive exons,MEE)。E56是一个隐匿外显子(cryptic exon),位于E2与E3之间的内含子区。此外,本研究发现在和都存在内含子1(I1)滞留,大小为194 bp。序列分析显示,、、和的剪接都遵循GT-AG 法则。
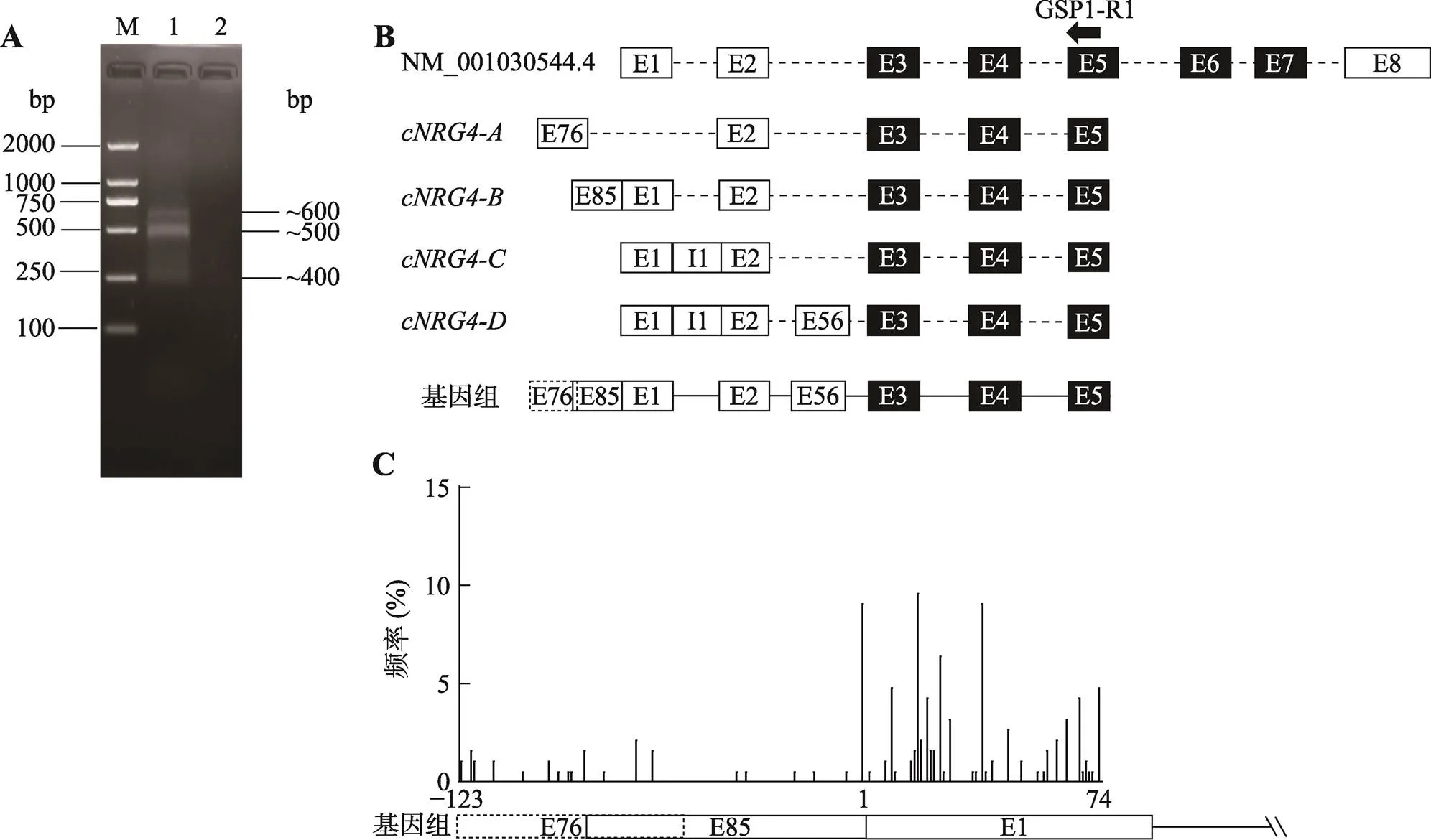
图1 鸡NRG4基因5ʹRACE分析
A:基因5ʹRACE PCR扩增产物的琼脂糖凝胶电泳分析。M: DL2000 DNA marker;泳道1:基因5ʹRACE PCR扩增片段;泳道2:阴性对照。B:基因5ʹRACE产物的外显子分析。上面一排为基因NM_001030544.4外显子分析结果;中间4排分别为4种不同5ʹRACE扩增产物的外显子分析结果;最下面一排为5ʹRACE扩增产物序列在基因组上的位置;GSP1-R1为5ʹRACE的特异性引物。C:基因的转录起始位点分析。基因TSS在-123~+74范围内的分布频率(%)。
测序分析发现具有54个转录起始位点(transcription start site,TSS),如图1C所示,以NM_001030544.4的第一个碱基为+1,的TSS离散地分布在–123~+74(–123/+74)的范围内。其中E76有12个TSS,分布在(–123/–64)范围内,导致E76大小在17~76 bp范围内变化;E85有9个TSS,分布在(–85/0)范围内,导致E85大小在4~85 bp范围内变化;E1有33个TSS,分布在(+1/+74)范围内,导致E1大小在2~75 bp范围内变化。
2.2 鸡NRG4基因3ʹ末端克隆和序列分析
为了获得完整mRNA转录本序列,取5只AA白羽肉鸡腹部脂肪组织的混合RNA进行3ʹRACE分析。琼脂糖电泳分析显示,基因3ʹRACE的PCR扩增产物主要有3条带,大小分别约为2000 bp、1500 bp和600 bp(图2A)。将3ʹRACE扩增产物进行凝胶回收和克隆,共随机挑选36个重组质粒进行测序分析。测序分析结果显示,基因有5种不同的3ʹRACE扩增产物,大小分别是2140 bp、553 bp、1458 bp、1449 bp和2422 bp,命名为(1个克隆)、(15个克隆)、(13个克隆)、(4个克隆)和(3个克隆)。
将3ʹRACE扩增产物序列与基因mRNA序列(NM_001030544.4)和基因组序列(NC_052541.1)进行比对,发现3ʹRACE扩增产物均包含基因E6和E7,且序列与NM_001030544.4的3ʹ端序列完全相同,提示本研究已成功获得基因mRNA 3ʹ端序列,但是这些扩增产物的3ʹ末端序列和长度差异较大,说明基因存在选择多聚腺苷酸化(alternative polyadenylation,APA)。序列分析发现、和存在内含子滞留,内含子分别为I5和I7,大小分别是2068 bp和896 bp;的poly(A)加尾位点位于I5上,和的poly(A)加尾位点位于E8中部,和的poly(A)加尾位点位于E8末端(图2B)。
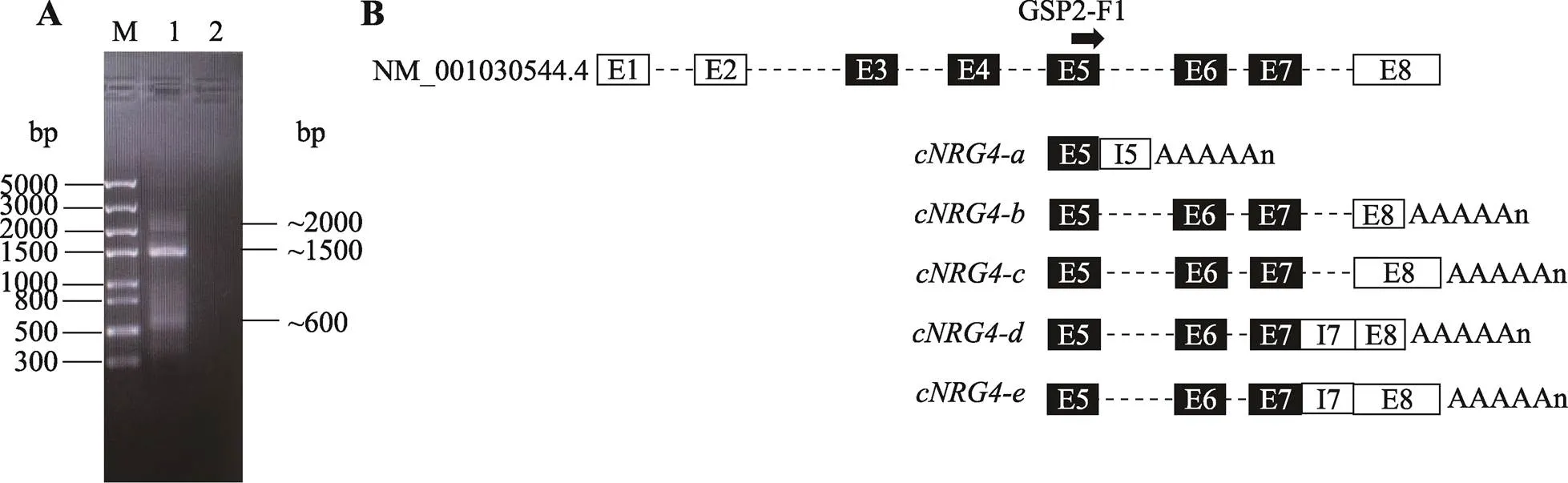
图2 鸡NRG4基因3ʹRACE分析
A:基因3ʹRACE PCR扩增产物的琼脂糖凝胶电泳分析。M:DL5000 DNA marker;泳道1:基因3ʹRACE PCR扩增片段;泳道2:阴性对照。B:基因3ʹRACE产物的外显子分析。最上面一排为基因mRNA序列(NM_001030544.4)的外显子分析结果,下面为5种不同3ʹRACE扩增产物序列的外显子分析结果。GSP2-F1为3ʹRACE特异性引物。
2.3 鸡NRG4基因新外显子和内含子滞留验证
5ʹRACE和3ʹRACE测序分析结果显示,基因的一些转录本存在新外显子(E76、E85和E56)和内含子滞留(I1、I5和I7)。为验证它们的存在,本研究跨内含子设计了这些新外显子和内含子滞留的6对特异性检测引物,引物位置如图3A所示。以鸡腹部脂肪组织总RNA为材料进行RT-PCR验证。RT-PCR扩增、克隆及测序分析显示:E76、E85、E56、I1、I5和I7经RT-PCR扩增,条带大小与预期相符,分别为294 bp、366 bp、123 bp、346 bp、219 bp和269 bp(图3B),测序结果与RACE结果一致,说明基因确实存在外显子E76、E85、E56及3个内含子(I1、I5和I7)的滞留。在E85和I5的RT-PCR验证中,除了扩增出预期大小的条带外,都扩增出了一条额外条带(图3B)。测序分析显示,在E85的RT-PCR验证中扩增出的额外条带为非特异性条带,其序列与基因无关,出现非特异扩增带的主要原因是E85比较小,只有85 bp,难以设计高特异性的上游检测引物。但值得注意的是,在I5的RT-PCR验证中扩增出的额外条带确实为基因转录本的一部分,该扩增产物包含I5,但是其E5外显子的3ʹ端缺失了79 bp序列,本研究将其命名为E5ʹ外显子,将该序列命名为(图3C)。生物信息学分析发现,在EST数据库中有1个基因序列(BU348396),其3ʹ端与完全相同,说明基因确实存在这样一种3ʹ末端序列。
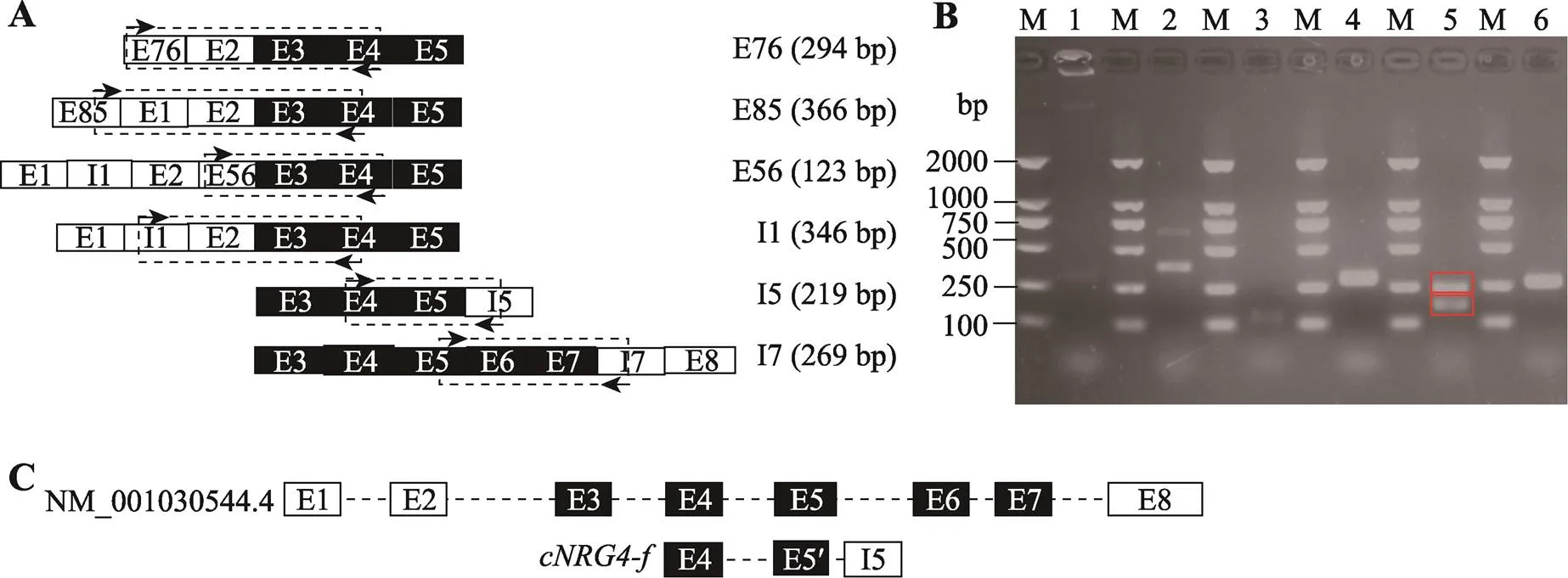
图3 鸡NRG4基因新外显子和内含子滞留的RT-PCR鉴定
A:RT-PCR鉴定新外显子和内含子滞留的引物位置(箭头所示)示意图。由上到下分别鉴定E76、E85、E56、I1、I5和I7;虚线框代表引物预计扩增出的片段。B:RT-PCR鉴定新外显子和内含子滞留的琼脂糖凝胶电泳分析。M:DL2000 DNA marker;泳道1~6依次为RT-PCR扩增E76、E85、E56、I1、I5和I7的琼脂糖电泳图,在鉴定I5的电泳图中分子量大的条带为,分子量小的条带为,两者均用红色方框标记。C:和基因mRNA序列(NM_001030544.4)外显子分析结果。E5序列长度为150 bp,E5ʹ序列长度71 bp。
2.4 鸡NRG4基因转录本结构分析
整合5ʹRACE、3ʹRACE以及新外显子和内含子滞留的验证结果,按照随机组合排列,基因可能产生多达24种转录异构体(图4),本研究进一步利用ORF Finder对这24种潜在转录本的CDS进行了分析。结果提示,这些基因转录异构体可能存在3种不同的CDS(CDS1、CDS2和CDS3),大小分别为258 bp、351 bp和198 bp。将这3个CDS分别编码的蛋白命名为cNRG4-1、cNRG4-2和cNRG4-3。在24种潜在基因转录异构体中,4种潜在异构体拥有CDS1,16种潜在异构体拥有CDS2,4种潜在异构体拥有CDS3。CDS1、CDS2和CDS3的起始密码子ATG均位于E3;CDS1和CDS3的终止密码子位于I5,而CDS2的终止密码子位于E7(图4)。
基因的起始密码子均位于E3,根据5ʹRACE扩增产物序列,基因存在4种5ʹUTR异构体。其中所包含的5ʹUTR由E76、E2和部分E3组成,其5ʹUTR命名为;所包含的5ʹUTR由E85、E1、E2和部分E3组成,其5ʹUTR命名为;所包含的5ʹUTR由E1、I1、E2和部分E3组成,其5ʹUTR命名为;所包含的5ʹUTR由E1、I1、E2、E56和部分E3组成,其5ʹUTR命名为(图1B)。这4种的5ʹUTR的最长和最短序列已提交GenBank,序列号分别为OP893938、OP893939、OP893940、OP893941、OP893942、OP893943、OP893944和OP893945。
根据3ʹRACE克隆测序结果和终止密码子的位置,基因存在6种3ʹUTR异构体,大小分别是2064 bp、384 bp、1289 bp、1280 bp、2184 bp和2045 bp。3ʹRACE扩增产物所包含的3ʹUTR由部分I5组成,其3ʹUTR命名为;所包含的3ʹUTR由部分E7和部分E8组成,其3ʹUTR命名为;所包含的3ʹUTR由部分E7和E8组成,其3ʹUTR命名为,与NM_001030544.4的3ʹUTR序列相同;所包含的3ʹUTR由部分E7、I7和部分E8组成,其3ʹUTR命名为;所包含的3ʹUTR由部分E7、I7和E8组成,其3ʹUTR命名为;所包含的3ʹUTR由部分I5组成,其3ʹUTR命名为(图2B,图3C)。这6个转录异构体的3ʹUTR序列已提交GenBank,序列号分别为OP893946、OP893947、OP893948、OP893949、OP893950和OP893951。
基因转录本具有不同长度和序列的3ʹUTR,表明基因存在APA。与此相一致,ITB tools分析发现,基因具有3个PAS,分别位于I5和E8,其中E8有两个PAS(图4)。根据PAS的位置,PAS分为编码区PAS(coding region PAS,CR-PAS)和非编码区PAS(untranslated region PAS,UTR-PAS)。基因的3个PAS分别为1个CR-PAS和2个UTR-PAS。和的PAS位于I5,导致编码蛋白变小;另外4种3ʹUTR异构体的PAS都位于E8上,均不影响基因的蛋白编码。此外,基因的3个PAS都位于poly(A)加尾位点上游10~30核苷酸处,都具有六聚体AAUAAA或其相近变异体、下游的U/GU-rich元件和临近剪接位点的CA(或UA)序列等。
2.5 鸡NRG4蛋白异构体分析
由于基因转录异构体数量众多(24种),且转录本间序列差异较小,难以利用常规方法逐一加以鉴定。因此,本研究设计引物,以鸡腹部脂肪组织的总RNA为材料,采用RT-PCR验证了基因3种不同CDS。RT-PCR扩增结果显示:利用CDS1特异扩增引物(CDS1-F/R)和CDS2特异扩增引物(CDS2-F/R)分别扩增出一条预期大小的特异条带(258 bp和351 bp);利用CDS3扩增引物(CDS3-F/R)扩增出预期大小的两条带,一条包含CDS1的片段(321 bp)和一条包含CDS3的扩增片段(242 bp) (图5A)。这些扩增产物的测序分析结果与预期完全一致,CDS1、CDS2和CDS3全长分别为258 bp、351 bp和198 bp,表明基因转录异构体确实存在3种不同CDS(CDS1、CDS2和CDS3)。将这3个CDS分别编码的蛋白命名为cNRG4-1、cNRG4-2和cNRG4-3。其中cNRG4-1蛋白异构体包含85个氨基酸,cNRG4-2蛋白异构体包含116个氨基酸,cNRG4-3蛋白异构体包含65个氨基酸(图5B)。cNRG4-2与NCBI数据库中的cNRG4氨基酸序列(NP_001025715.2)完全相同,cNRG4-1和cNRG4-3是两个新发现的cNRG4蛋白异构体。cNRG4-1与人的6个NRG4蛋白异构体(NP_612640.1、CAL35830.1、EAW99228.1、CAL35831.1、CAL35829.1和XP_047288140.1)同源性分别为68%、76%、77%、72%、65%和65%;与小鼠的3个NRG4蛋白异构体(NP_114391.1、AAH34839.1和EDL25847.1)同源性分别为61%、74%和46%。cNRG4-3与人的6个NRG4蛋白异构体同源性分别为73%、76%、77%、72%、73%和73%;与小鼠的3个NRG4蛋白异构体同源性分别为81%、74%和78%。基因3种CDS全长序列已提交GenBank,序列号分别为OP288945、OP288946和OP288947。
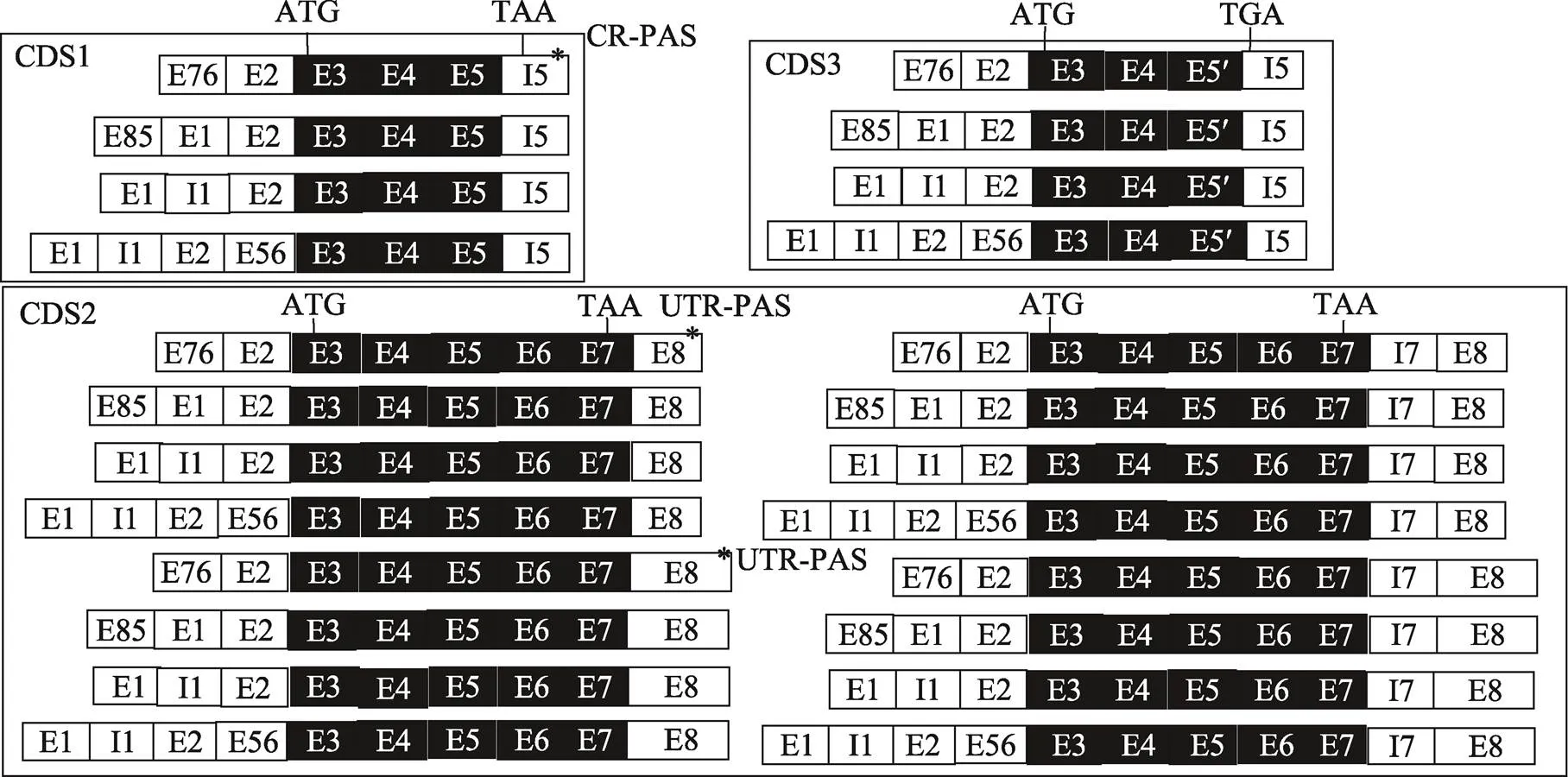
图4 鸡NRG4基因转录本结构分析
基因24种潜在转录异构体的CDS分析。这24种基因潜在转录异构体共存在3种不同的CDS:CDS1、CDS2和CDS3。这3种CDS的起始密码子均位于E3,CDS1和CDS3编码的蛋白终止密码子均位于I5,CDS2编码的蛋白终止密码子位于E7。3个星号(*)为本研究发现的3个PAS,其中1个位于I5,为CR-PAS,另外2个位于E8,均为UTR-PAS。
NRG4是作为前体合成的,前体NRG4通过蛋白酶水解后成为有活性的形式[4]。利用在线软件Signal IP-5.0预测分析显示,这3个cNRG4蛋白异构体缺乏分泌蛋白的经典分泌信号,这与人、小鼠NRG4蛋白分泌信号肽的分析结果一致。利用TMHMM2.0进行跨膜结构分析显示,cNRG4-1和cNRG4-2都存在一个跨膜螺旋结构,cNRG4-3没有跨膜结构。利用SMART进行结构域分析,结果发现cNRG4-1和cNRG4-2都包含1个EGF结构域、1个跨膜结构域和1个与跨膜结构域重叠的低复杂性区域(low-complexity regions,LCRs),其二者的区别在于跨膜结构域后的蛋白长度不同,而cNRG4-3只包含一个EGF结构域,没有跨膜结构域和低复杂性区域。
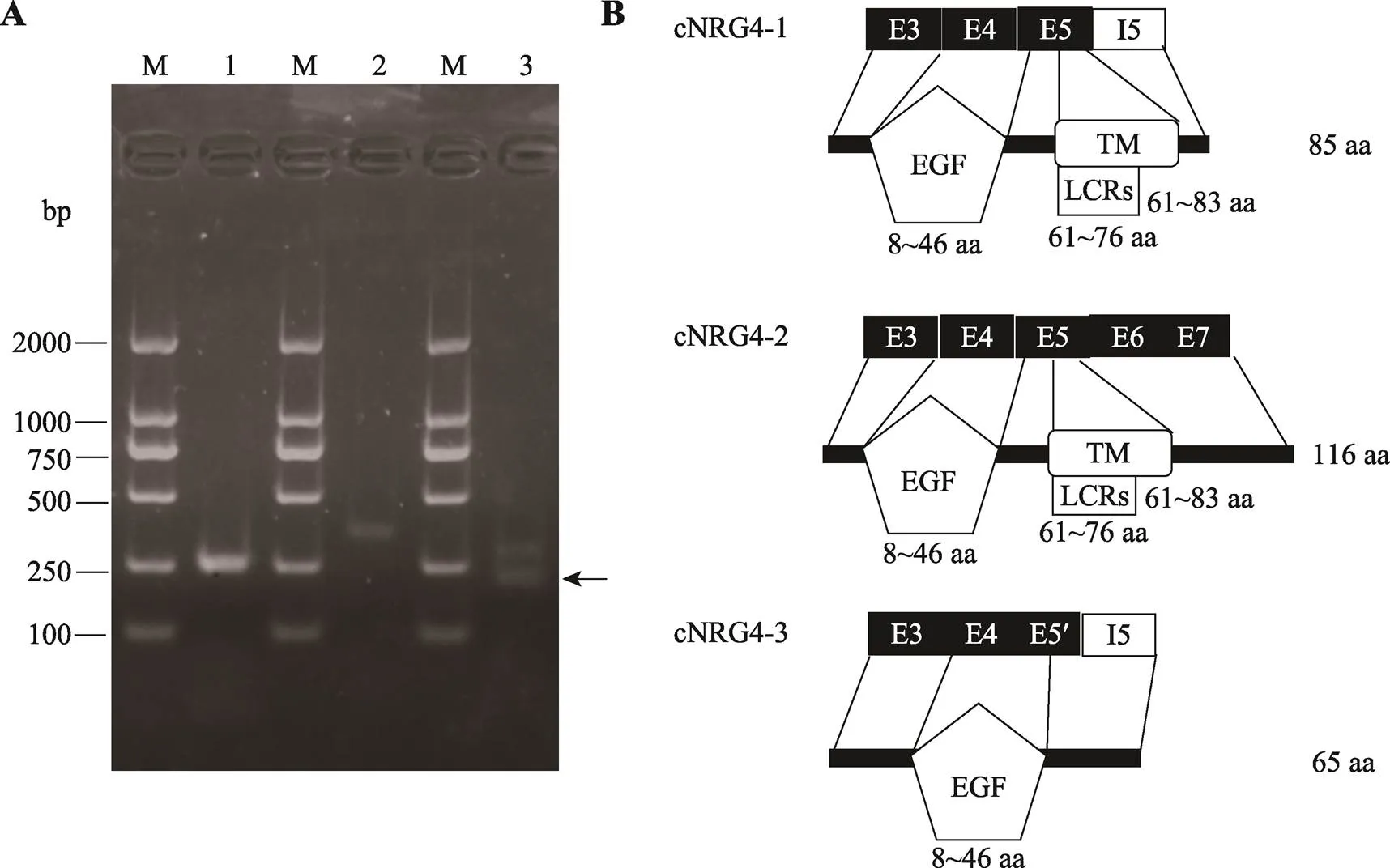
图5 鸡NRG4蛋白质异构体的鉴定与分析
A:基因3种不同CDS的 RT-PCR鉴定分析。M:DL2000 DNA marker;泳道1:CDS1的RT-PCR扩增片段;泳道2:CDS2的RT-PCR扩增片段;泳道3:CDS3的RT-PCR扩增片段。箭头所指的条带为目标条带。B:cNRG4蛋白异构体的结构域分析。LCRs:low-complexity regions,cNRG4-1全长有85个氨基酸(aa),其中8~46 aa为EGF结构域,61~83 aa为跨膜结构域(TM),61~76 aa为低复杂性区域(LCRs)。cNRG4-2全长116 aa,其中8~46 aa为EGF结构域,61~83 aa为跨膜结构域(TM),61~76 aa为低复杂性区域(LCRs)。cNRG4-3全长65 aa,其中8~46 aa为EGF结构域。
3 讨论
本研究发现基因存在多个转录异构体,这些转录异构体编码3个cNRG4蛋白异构体。与本研究结果类似,人基因能产生5种转录异构体和相对应的5种蛋白异构体(NRG4 A1、NRG4 A2、NRG4 B1、NRG4 B2和NRG4 B3)[11]。NRGs家族其他成员也普遍存在多转录异构体和蛋白异构体。例如,人基因由于选择性转录起始位点和选择性剪接能产生33种转录异构体和6种蛋白异构体(NRG1 I-VI)[6];人基因由于选择性剪接能产生至少10种转录异构体和2种蛋白异构体(NRG2-α和NRG2-β)[7];人基因由于选择性剪接能产生15种转录异构体[9]。
在5ʹRACE测序分析中,本研究发现基因存在选择性转录起始位点、互斥外显子、隐匿外显子和内含子滞留现象,其转录本的5ʹ端非常复杂,为了保证结果的准确性和可靠性,本研究对187个5ʹRACE的PCR产物克隆质粒进行了测序。而在3ʹRACE测序分析中发现,基因mRNA的3ʹ端比较简单,只存在内含子滞留和选择性多聚腺苷酸化,因此本研究只对36个3ʹRACE的扩增产物的克隆质粒进行了测序。本研究发现,基因具有选择性转录起始位点、选择性剪接、选择性多聚腺苷酸化以及内含子滞留和隐匿外显子,这导致基因存在多个转录异构体和蛋白异构体。人基因也具有多个转录异构体和蛋白异构体,但其造成的原因是选择性剪接[11]。目前尚无报道其他动物基因存在选择性转录起始位点、隐匿外显子、内含子滞留以及选择性多聚腺苷酸化。此外,生物信息学分析显示,基因5ʹUTR与人和小鼠基因5ʹUTR的序列同源性均低于45%,基因3ʹUTR与人和小鼠基因的3ʹUTR序列的同源性均低于40%。这些结果提示,鸡与人和鼠等其他动物基因的表达调控机制不同。
本研究发现基因可以编码3种蛋白异构体(cNRG4-1、cNRG4-2和cNRG4-3)。研究显示,人NRG4具有5种蛋白异构体(NRG4 A1、NRG4 A2、NRG4 B1、NRG4 B2和NRG4 B3)[11]。其中cNRG4-1和cNRG4-2与人的NRG4 A型相似,具有完整的EGF结构域和跨膜结构域。人NRG4 A蛋白异构体是目前研究最广泛的,其定位于细胞膜,通过蛋白酶加工,释放膜外EGF结构域,分泌入血液,作为经典配体发挥作用[5],本研究推测cNRG4-1和cNRG4-2与人NRG4 A类似,均定位于细胞膜,经过酶切加工后释放EGF结构域,进入血液,通过结合靶细胞受体ErbB4,触发下游信号通路。cNRG4-3蛋白异构体是鸡独有的,其具有完整的EGF样结构域,但缺失跨膜结构域。人NRG4 B蛋白异构体具有2/3的EGF结构域,缺失跨膜结构域,定位于细胞内[11]。由于cNRG4-3没有跨膜结构域,推测其可能定位于细胞质或细胞核而发挥作用[4,13]。
本研究发现基因的基因组(NC_052541.1)大小为21,969 bp(Chr.10: 3,490,314~3,512,282),由11个外显子和10个内含子构成。与基因mRNA序列(NM_001030544.4)相比,本研究发现了基因存在2个新外显子和1个隐匿外显子;基因5ʹ端在基因组的位置向前延伸了123 bp(图6)。
最近的研究表明,人和鼠的大多数基因并非只有1个TSS[14,15],而是具有一系列分布紧密的多个TSS,即选择性转录起始位点,这一区域构成一个TSS簇[16]。有研究将TSS簇根据TSS数目与分布特征分为2种类型:尖峰型,分布集中且具有单一、丰度较高的TSS,通常从一个固定位置开始转录,大多与TATA-box相关[17];宽峰型,具有多个丰度相似的邻近TSS,离散分布于宽泛区域[14,16~18],可从多个位置开始转录,绝大多数与CpG岛相关[17]。本研究发现基因具有多个TSS,离散地分布在基因组(–123/+74)。为了排除由于RNA降解和试剂盒差异等原因导致基因出现多个TSS的可能性,本研究尝试了多个批次的新鲜实验材料,试用了3种不同原理的RACE分析试剂盒,结果都证实基因确实存在选择性TSS,基因TSS簇呈宽峰型。与此相一致,基因启动子区存在一个1206 bp的CpG岛(–1019/+186)[12]。人基因的结构信息是通过生物信息学和RT-PCR扩增获得的[11],人类基因是否存在选择性转录起始位点还不清楚。选择性转录起始点会导致基因转录本的5ʹUTR长度及其所包含调控元件的不同,这些调控元件如上游开放阅读框(upstream open reading fragment,uORF)会影响mRNA稳定性、翻译效率和定位等[18]。推测选择性TSS可能是基因表达的一个重要调控方式。
内含子滞留最初在植物和病毒中被描述,后来发现哺乳动物的基因也存在内含子滞留[19~22]。内含子滞留在基因表达调控中发挥多种作用,不仅可以通过导致无义介导的mRNA降解来降低基因表达,还可以导致产生新的转录异构体和蛋白异构体[21,22]。CDS区的内含子滞留会导致基因产生新的蛋白异构体[23],5ʹUTR区的内含子滞留会影响mRNA稳定性和翻译效率等[24,25];3ʹUTR区的内含子滞留也能影响mRNA的稳定性、翻译效率和定位等[26,27]。本研究首次发现c基因存在内含子滞留(I1、I5和I7),其中I1位于5ʹUTR区,产生新的5ʹUTR异构体(和);I5位于主CDS区,导致产生了新的蛋白异构体cNRG4-1和cNRG4-3;I7位于3ʹUTR区域,产生新的3ʹUTR(和)。I1和I7滞留并未影响蛋白编码,推测它们会引入新的调控元件,从而实现基因表达的精细调控。
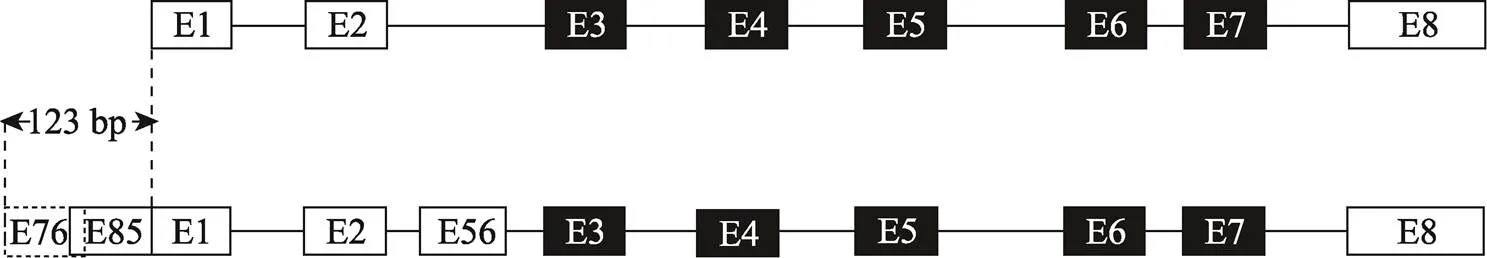
图6 鸡NRG4基因组结构分析
图上方为基因mRNA序列(NM_001030544.4)的基因组结构。下方为本研究所确定的基因组结构,E76、E85和E56为本研究新发现的基因的3个新外显子。
真核生物基因普遍具有多个PAS,APA是一个重要的转录后调控机制[28]。PAS分为CR-PAS和UTR-PAS,其中UTR-PAS最为常见[29],UTR-PAS产生具有不同3ʹUTR长度的转录异构体[30];CR-PAS会导致基因编码不同C端的蛋白异构体,其中一些异构体可能缺乏某些重要的功能域[31]。本研究首次发现基因存在APA,并同时具有UTR-PAS和CR-PAS。有研究报道,鸡生长激素受体()基因和鸡转化生长因子β受体II()基因同时具有CR-PAS和UTR-PAS[32,33]。NRG4在动物体内发挥着多种重要的作用,考虑到APA在转录后调控中的重要性,未来有必要探讨APA在基因表达调控中的作用和机制。
下游开放阅读框(downstream open reading fragment,dORFs)是真核生物基因中广泛存在的一个转录后调节元件,它可以提高mRNA的翻译效率[34~36]。本研究分析发现基因的3ʹUTR()有一个dORF,可以编码一个由233个氨基酸组成的蛋白,其编码区大于基因的编码区。将该dORF的核苷酸序列进行BLAST分析,发现的dORF序列与兔()、白斑兔()、鹌鹑()、鸿雁()、黑天鹅()和红鸭()等物种基因的mRNA同源性在78%以上,提示该dORF可能在基因的转录后调控中发挥重要作用。下一步有必要探究该dORF对基因表达的调控作用。
[1] Yang F, Li XN. Research progress of neuregulin 4 biological function., 2017, 69(3): 351–356.杨帆, 李晓南. 神经调节蛋白4生物学功能的研究进展. 生理学报, 2017, 69(3): 351–356.
[2] Ledonne A, Mercuri NB. On the modulatory roles of neuregulins/ErbB signaling on synaptic plasticity., 2019, 21(1): 275.
[3] Dai YN, Zhu JZ, Fang ZY, Zhao DJ, Wan XY, Zhu HT, Yu CH, Li YM. A case-control study: association between serum neuregulin 4 level and non-alcoholic fatty liver disease., 2015, 64(12): 1667–1673.
[4] Wang GX, Zhao XY, Meng ZX, Kern M, Dietrich A, Chen ZM, Cozacov Z, Zhou DQ, Okunade AL, Su X, Li SM, Blüher M, Lin JD. The brown fat-enriched secreted factor Nrg4 preserves metabolic homeostasis through attenuation of hepatic lipogenesis., 2014, 20(12): 1436– 1443.
[5] Chen LL, Peng MM, Zhang JY, Hu X, Min J, Huang QL, Wan LM. Elevated circulating Neuregulin4 level in patients with diabetes., 2017, 33(4): e2870.
[6] Steinthorsdottir V, Stefansson H, Ghosh S, Birgisdottir B, Bjornsdottir S, Fasquel AC, Olafsson O, Stefansson K, Gulcher JR. Multiple novel transcription initiation sites for NRG1., 2004, 342(1): 97–105.
[7] Rimer M, Prieto AL, Weber JL, Colasante C, Ponomareva O, Fromm L, Schwab MH, Lai C, Burden SJ. Neuregulin-2 is synthesized by motor neurons and terminal Schwann cells and activates acetylcholine receptor transcription in muscle cells expressing ErbB4., 2004, 26(2): 271–281.
[8] Hayes NVL, Gullick WJ. The neuregulin family of genes and their multiple splice variants in breast cancer., 2008, 13(2): 205–214.
[9] Carteron C, Ferrer-Montiel A, Cabedo H. Characterization of a neural-specific splicing form of the human neuregulin 3 gene involved in oligodendrocyte survival., 2006, 119(Pt 5): 898–909.
[10] Hayes NVL, Newsam RJ, Baines AJ, Gullick WJ. Characterization of the cell membrane-associated products of the neuregulin 4 gene., 2008, 27(5): 715–720.
[11] Hayes NVL, Blackburn E, Smart LV, Boyle MM, Russell GA, Frost TM, Morgan BJT, Baines AJ, Gullick WJ. Identification and characterization of novel spliced variants of neuregulin 4 in prostate cancer., 2007, 13(11): 3147–3155.
[12] Guo YQ, Wang WJ, Gao ZH, Mu F, Xu HD, Li H, Wang N. Cloning, expression and promoter analysis of adipokinegene in chicken.,2021, 29(11): 2129–2138.郭亚琦, 王伟佳, 高智慧, 牟芳, 徐海冬, 李辉, 王宁. 鸡脂肪细胞因子NRG4基因的克隆、表达及启动子分析. 农业生物技术学报, 2021, 29(11): 2129–2138.
[13] Pfeifer A. NRG4: an endocrine link between brown adipose tissue and liver., 2015, 21(1): 13–14.
[14] Haberle V, Stark A. Eukaryotic core promoters and the functional basis of transcription initiation., 2018, 19(10): 621–637.
[15] Forutan M, Ross E, Chamberlain AJ, Nguyen L, Mason B, Moore S, Garner JB, Xiang RD, Hayes BJ. Evolution of tissue and developmental specificity of transcription start sites in Bos taurus indicus., 2021, 4(1): 829.
[16] Mejía-Guerra MK, Li W, Galeano NF, Vidal M, Gray J, Doseff AI, Grotewold E. Core promoter plasticity between maize tissues and genotypes contrasts with predominance of sharp transcription initiation sites., 2015, 27(12): 3309–3320.
[17] Carninci P, Sandelin A, Lenhard B, Katayama S, Shimokawa K, Ponjavic J, Semple CAM, Taylor MS, Engström PG, Frith MC, Forrest ARR, Alkema WB, Tan SL, Plessy C, Kodzius R, Ravasi T, Kasukawa T, Fukuda S, Kanamori-Katayama M, Kitazume Y, Kawaji H, Kai C, Nakamura M, Konno H, Nakano K, Mottagui-Tabar S, Arner P, Chesi A, Gustincich S, Persichetti F, Suzuki H, Grimmond SM, Wells CA, Orlando V, Wahlestedt C, Liu ET, Harbers M, Kawai J, Bajic VB, Hume DA, Hayashizaki Y. Genome-wide analysis of mammalian promoter architecture and evolution., 2006, 38(6): 626–635.
[18] Thieffry A, López-Márquez D, Bornholdt J, Malekroudi MG, Bressendorff S, Barghetti A, Sandelin A, Brodersen P. PAMP-triggered genetic reprogramming involves widespread alternative transcription initiation and an immediate transcription factor wave., 2022, 34(7): 2615– 2637.
[19] Braunschweig U, Barbosa-Morais NL, Pan Q, Nachman EN, Alipanahi B, Gonatopoulos-Pournatzis T, Frey B, Irimia M, Blencowe BJ. Widespread intron retention in mammals functionally tunes transcriptomes., 2014, 24(11): 1774–1786.
[20] Hammarskjöld ML. Regulation of retroviral RNA export., 1997, 8(1): 83–90.
[21] Ner-Gaon H, Halachmi R, Savaldi-Goldstein S, Rubin E, Ophir R, Fluhr R. Intron retention is a major phenomenon in alternative splicing in arabidopsis., 2004, 39(6): 877–885.
[22] Rekosh D, Hammarskjold ML. Intron retention in viruses and cellular genes: detention, border controls and passports., 2018, 9(3): e1470.
[23] Marquez Y, Höpfler M, Ayatollahi Z, Barta A, Kalyna M. Unmasking alternative splicing inside protein-coding exons defines exitrons and their role in proteome plasticity., 2015, 25(7): 995–1007.
[24] Tahmasebi S, Jafarnejad SM, Tam IS, Gonatopoulos- Pournatzis T, Matta-Camacho E, Tsukumo Y, Yanagiya A, Li WC, Atlasi Y, Caron M, Braunschweig U, Pearl D, Khoutorsky A, Gkogkas CG, Nadon R, Bourque G, Yang XJ, Tian B, Stunnenberg HG, Yamanaka Y, Blencowe BJ, Giguère V, Sonenberg N. Control of embryonic stem cell self-renewal and differentiation via coordinated alternative splicing and translation of YY2., 2016, 113(44): 12360–12367.
[25] Weatheritt RJ, Sterne-Weiler T, Blencowe BJ. The ribosome-engaged landscape of alternative splicing., 2016, 23(12): 1117–1123.
[26] Sun SY, Zhang Z, Sinha R, Karni R, Krainer AR. SF2/ASF autoregulation involves multiple layers of post- transcriptional and translational control., 2010, 17(3): 306–312.
[27] Thiele A, Nagamine Y, Hauschildt S, Clevers H. AU-rich elements and alternative splicing in the beta-catenin 3'UTR can influence the human beta-catenin mRNA stability., 2006, 312(12): 2367–2378.
[28] Nourse J, Spada S, Danckwardt S. Emerging roles of RNA 3'-end cleavage and polyadenylation in pathogenesis, diagnosis and therapy of human disorders., 2020, 10(6): 915.
[29] Chen W, Jia Q, Song YF, Fu HH, Wei G, Ni T. Alternative polyadenylation: methods, findings, and impacts., 2017, 15(5): 287–300.
[30] Jambhekar A, Derisi JL. Cis-acting determinants of asymmetric, cytoplasmic RNA transport., 2007, 13(5): 625–642.
[31] Tian B, Manley JL. Alternative polyadenylation of mRNA precursors., 2017, 18(1): 18–30.
[32] Lau JS, Yip CW, Law KM, Leung FC. Cloning and characterization of chicken growth hormone binding protein (cGHBP)., 2007, 33(1): 107–121.
[33] Ning BL, Huang JX, Xu HD, Lou YQ, Wang WS, Mu F, Yan XH, Li H, Wang N. Genomic organization, intragenic tandem duplication, and expression analysis of chicken TGFBR2 gene., 2022, 101(12): 102169.
[34] Wu QS, Wright M, Gogol MM, Bradford WD, Zhang N, Bazzini AA. Translation of small downstream ORFs enhances translation of canonical main open reading frames., 2020, 39(17): e104763.
[35] Bazzini AA, Johnstone TG, Christiano R, Mackowiak SD, Obermayer B, Fleming ES, Vejnar CE, Lee MT, Rajewsky N, Walther TC, Giraldez AJ. Identification of small ORFs in vertebrates using ribosome footprinting and evolutionary conservation., 2014, 33(9): 981–993.
[36] Dodbele S, Wilusz JE. Ending on a high note: downstream ORFs enhance mRNA translational output., 2020, 39(17): e105959.
Characterization of the genomic and transcriptional structure of chickengene
Zhihui Gao1,2,3, Jiaxin Huang1,2,3, Haoyu Luo1,2,3, Haidong Xu1,2,3, Ming Lou1,2,3, Bolin Ning1,2,3, Xiaoxu Xing1,2,3, Fang Mu1,2,3, Hui Li1,2,3, Ning Wang1,2,3
Neuregulin 4 (NRG4) is an important adipocytokine, which plays crucial roles in maintaining energy balance, regulating glucose and lipid metabolism, and preventing non-alcoholic fatty liver disease in mammals. At present, the genomic organization, transcript and protein isoforms of humangene have been fully explored. Previous studies in our laboratory have shown that thegene is expressed in chicken adipose tissue, but the chickengenomic structure, transcript and protein isoforms are still unknown. To this end, in this study, the genomic and transcriptional structure of thegene were systematically investigated using rapid amplification of cDNA ends (RACE) and reverse transcription-polymerase chain reaction (RT-PCR). The results showed thatthe coding region (CDS) of thegene was small, but it had a very complextranscriptional structure characterized by multiple transcription start sites, alternative splicing, intron retention, cryptic exons, and alternative polyadenylation, thus leading to production of four 5ʹUTR isoforms (,,, and) and six 3ʹUTR isoforms (,,,,, and)of thegene. Thegene spanned 21,969 bp of genomic DNA (Chr.10:3,490,314~3,512,282) and consisted of 11 exons and 10 introns. Compared withthegene mRNA sequence (NM_001030544.4), two novel exons and one cryptic exon of thegene were identified in this study. Bioinformatics analysis, RT-PCR, cloning and sequencing analysis showed that thegene could encode three protein isoforms (cNRG4-1, cNRG4-2 and cNRG4-3). This studylays a foundation for further research on the function and regulation of thegene.
chicken; NRG4; genomic structure; transcript isoform; protein isoform
2023-01-03;
2023-04-04;
2023-04-19
国家自然科学基金项目(编号:31872346)和国家肉鸡产业技术体系(编号:CARS-41)资助[Supported by the National Natural Science Foundation of China (No. 31872346) and the China Agriculture Research System (No. CARS-41)]
高智慧,在读硕士研究生,专业方向:动物遗传育种与繁殖。E-mail: gaozhihui@neau.edu.cn
王宁,博士,教授,博士生导师,研究方向:动物遗传育种与繁殖。E-mail: wangning@neau.edu.cn
10.16288/j.yczz.23-001
(责任编委: 赵要风)
