基于生物信息学的胰腺导管腺癌核心风险基因筛选和分析
2023-05-10景晓莹谢娜娜吕环环马占兵
景晓莹,彭 亮,谢娜娜,吕环环,党 洁,2,马占兵,2
(1.宁夏医科大学基础医学院,银川 750004;2.宁夏医科大学生育力保持教育部重点实验室,银川 750004)
胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)是一种恶性程度极高并具有高度侵袭性的消化道恶性肿瘤。因其5 年生存率不足7%,是恶性肿瘤中预后最差的[1-2]。PDAC 早期的症状隐匿、不典型[3],约80%的患者被确诊时为中晚期或出现转移,已错失最佳手术根治的窗口和机会。即使成功实施手术干预,术后12 个月内的复发率和转移率仍高达60%[4]。因此,准确有效的生物标记物筛选及其分子机制研究对于PDAC的诊断、治疗和不良预后改善具有十分重要的临床意义和研究价值。
加权基因共表达网络(weighted gene co-expression network analysis,WGCNA)已被广泛用于寻找各种癌症中的枢纽基因。癌症基因组图谱(the cancer genome atlas,TCGA)数据库中缺乏正常组织的PDAC 样本,差异表达数据不完整。因此,本研究整合基因型-组织表达数据库(genotypetissue expression,GTEx)数据库中正常对照组织的表达数据,有效地克服TCGA 数据库对照样本不足的问题。通过差异表达分析,获得PDAC 原发癌全面的转录组表达谱,并使用WGCNA、蛋白质相互作用网络(protein-protein interaction,PPI)网络分析结合表达生存分析,获得核心风险基因,以识别潜在准确的PDAC 生物标记物。
1 材料与方法
1.1 数据获取与处理
PDAC 组织的RNA-seq 数据来自TCGA,匹配的正常组织表达数据来自GTEx[5]。过滤去除基因表达值低于lcpm 剪切阈值80%以上的样本,通过filterByExpr 函数去除表达矩阵中不表达或低表达的基因。最后纳入共312 个样本,包含147 例原发肿瘤组织,165 例正常组织(图1)。
图1 数据下载、处理和分析流程
1.2 差异表达分析
使用edgeR 包筛选差异表达基因(differential expression genes,DEGs),以>1且调整P<0.05 为标准,筛选DEGs 并绘制热图和火山图。
1.3 GO 和KEGG 富集分析
基因本体(gene ontology,GO)包含生物过程(biological process,BP)、细胞组成(cellular component,CC)以及分子功能(molecular function,MF)3 个部分的信息,可用于基因归类注释[6]。京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)是整合了基因组、化学和系统功能信息的通路注释数据库[7]。本研究采用FunRich 软件对PDAC 的DEGs 进行GO 和KEGG富集分析及可视化[8]。
1.4 WGCNA 的构建和分析
通过WGCNA R 包构建加权基因共表达网络[9]。根据无尺度网络拟合指数和平均连接度,选择合适的软阈值,以确保无标度拓扑,使其满足无尺度网络和较好的网络连接性。利用拓扑重叠矩阵(topological overlap matrix,TOM)相似度和相应的不相似度(diss TOM)将邻接度转换为TOM。用动态树切分法将至少30 个高度相关的共表达基因聚集成不同颜色的模块[10]。WGCNA 采用分层聚类方法识别基因模块,并用不同颜色来表示。通过计算模块和临床表型之间的相关性(correlation),筛选出与临床表型显著相关的基因。最后,计算模块内的基因表达与性状的相关性GS 值和某个基因表达与模块内基因主成分表达的相关系数MM 值,设置参数cor.gene Module Membership>0.8 和cor.gene Trait Significance>0.2,从而识别和鉴定出模块基因。
1.5 PPI 的构建和核心基因筛选
获得候选核心风险基因,利用Cytoscape Gene MINIA 插件,构建PPI 网络和MCODE 插件筛选网络核心基因。采用默认参数,聚类算法为MCC。
1.6 核心风险基因的功能分析
使用GEPIA2 在线数据库(http://gepia2.cancer-pku.cn/#index)对筛选出的PDAC 核心风险基因进行表达、生存分析,其生存分析主要包括生存率和风险比(hazard ratio,HR)。P≤0.05 为差异有统计学意义。
1.7 核心风险基因的临床诊断价值评价
使用SPSS 25.0 统计学软件绘制受试者工作特征(receiver operating characteristic curve,ROC)曲线,并计算ROC 曲线下的面积(area under curve,AUC),评估对肿瘤和正常组织的区分能力。
2 结果
2.1 DEGs 的筛选
经选取上调和下调各Top 50 的基因进行无监督垂直聚类分析,结果显示,DEGs 能够显著区分PDAC 组织和正常组织(图2A)。经差异表达分析,共筛选出4 346 个DEGs,其中包含2 284个上调基因和2 062 个下调基因(图2B)。
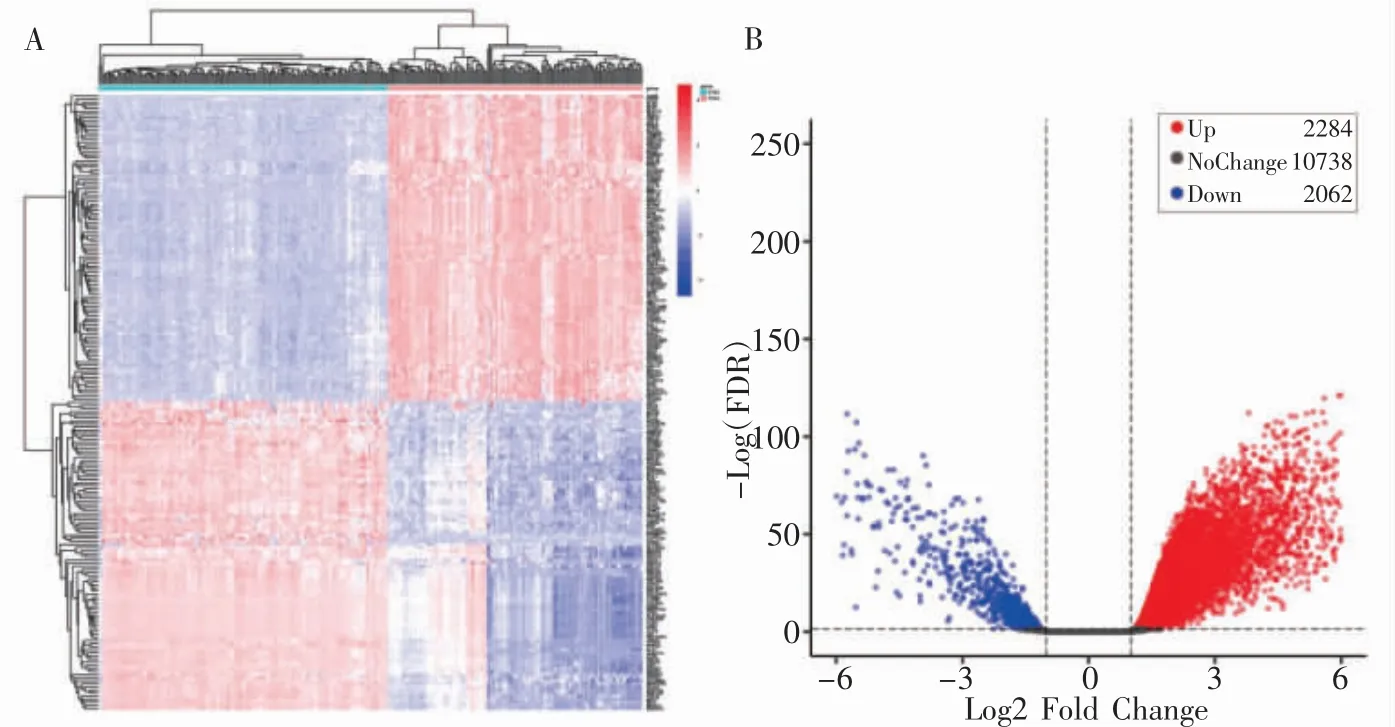
图2 差异表达基因聚类热图和火山图
2.2 DEGs 的GO 功能富集分析和KEGG 通路分析
为进一步分析DEGs 的功能,将DEGs 输入FunRich 进行GO 和KEGG 富集分析。DEGs 中BP 主要包括细胞外和质膜整体等(图3A);CC主要包括细胞通讯和细胞生长等(图3B);MF 主要包括细胞黏附分子活性和受体活性等(图3C)。KEGG 通路富集分析PDAC 中的DEGs 与间充质向上皮细胞转变(EMT)、上皮细胞向间充质转变(MET)和整合素细胞表面相互作用等信号通路密切相关(图3D)。

图3 DEGs 的GO 和KEGG 富集分析
2.3 WGCNA 的构建及分析
通过绘制样本聚类树,设定剪切高度为90,去除异常值(图4A),进行样本聚类和表型关联分析(图4B)。根据无尺度网络拟合指数和平均连接度计算软阈值(图4C),根据基因模块连通性确定软阈值(图4D),选取β=10(无标度R2=0.8,斜率=-2.11)作为网络构建软阈值(图4E),并根据TOM 矩阵构建基因间的分层聚类树(图4F)。表型-模块关联分析结果显示,lightgreen 模块与TNM 分期有相关性(r=0.33,P=0.02),magenta模块与肿瘤大小程度有相关性(r=0.3,P=0.03),grey 模块与组织学分级有相关性(r=0.34,P=0.02)(图5A~图5C),且能够较好区分肿瘤组织和正常对照(图5D)。进一步将lightgreen、magenta 和grey 模块分别作为关键模块进行GS 和MM 分析,lightgreen 模块的GS 与MM 有相关关系(r=0.27,P<0.001)(图6A);magenta 模块的GS与MM 有相关关系(r=0.39,P<0.001)(图6B);grey模块的GS 与MM 有相关关系(r=0.54,P<0.001)(图6C)。对各颜色模块的连通性进行分析,发现lightgreen 模块的基因显著性与连通性有相关关系(r=0.33,P=0.001 3);magenta 模块的基因显著性与连通性有相关关系(r=0.12,P=0.046);grey模块的基因显著性与连通性有相关关系(r=-0.26,P=0.006 8)(图6D)。最后,经过多重检验矫正后P<0.01,剔除非编码基因后,lightgreen 模块筛选出42 个基因,magenta 模块筛选出271 个基因,grey 模块筛选出96 个基因,后结合差异表达分析结果,最终筛选出50 个基因(表1)。
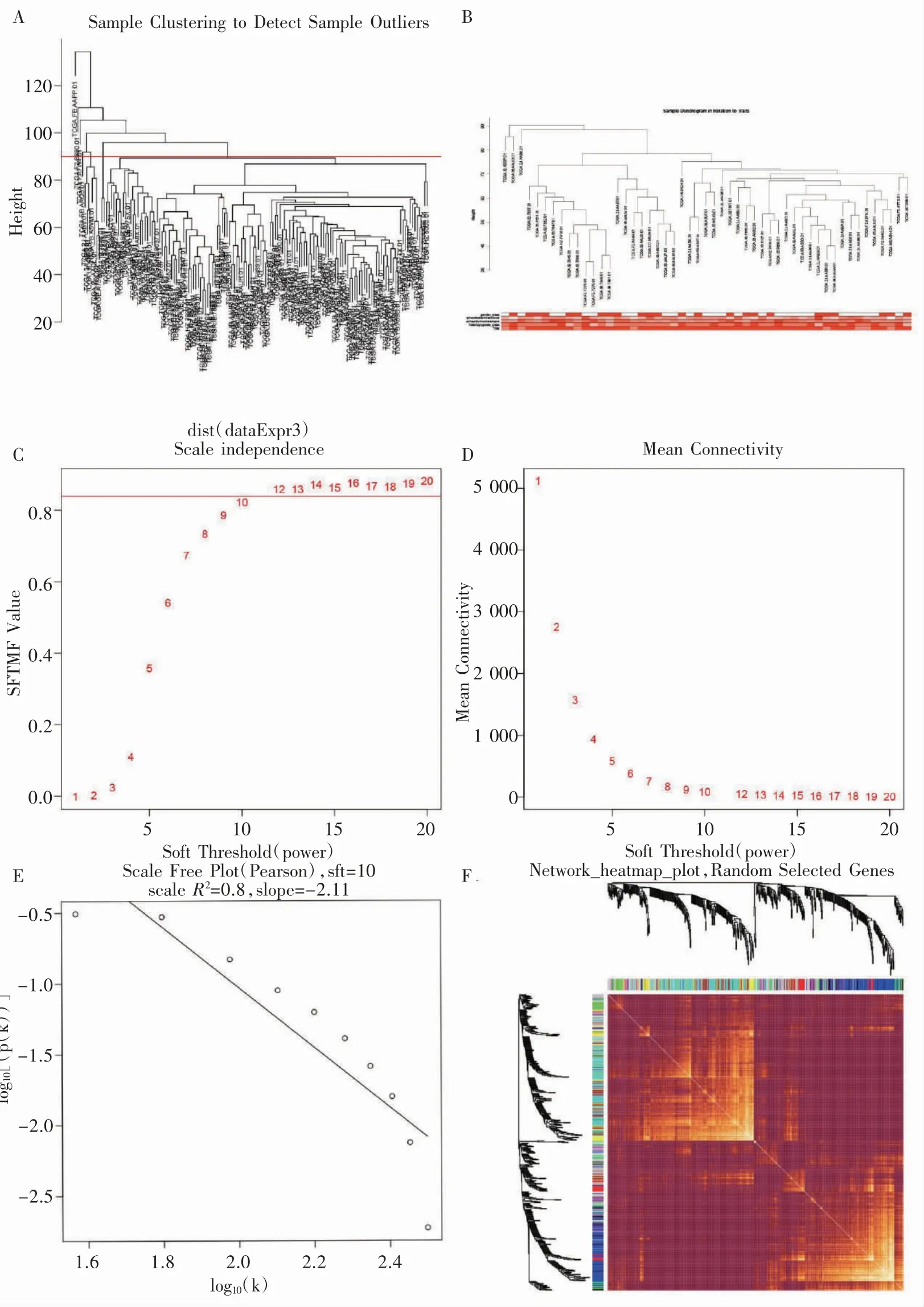
图4 加权基因共表达网络的构建
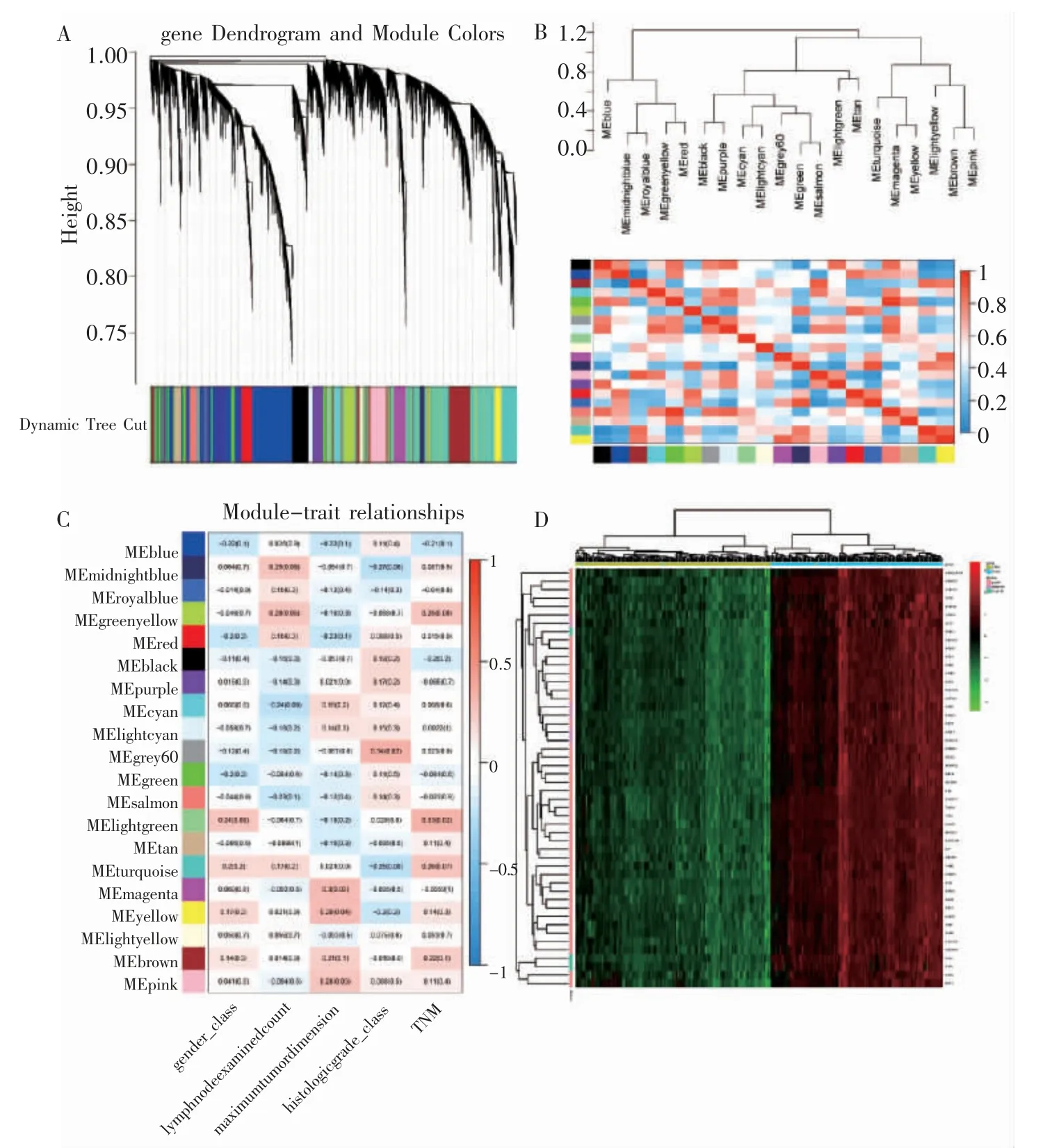
图5 共表达模块与临床表型的相关性分析
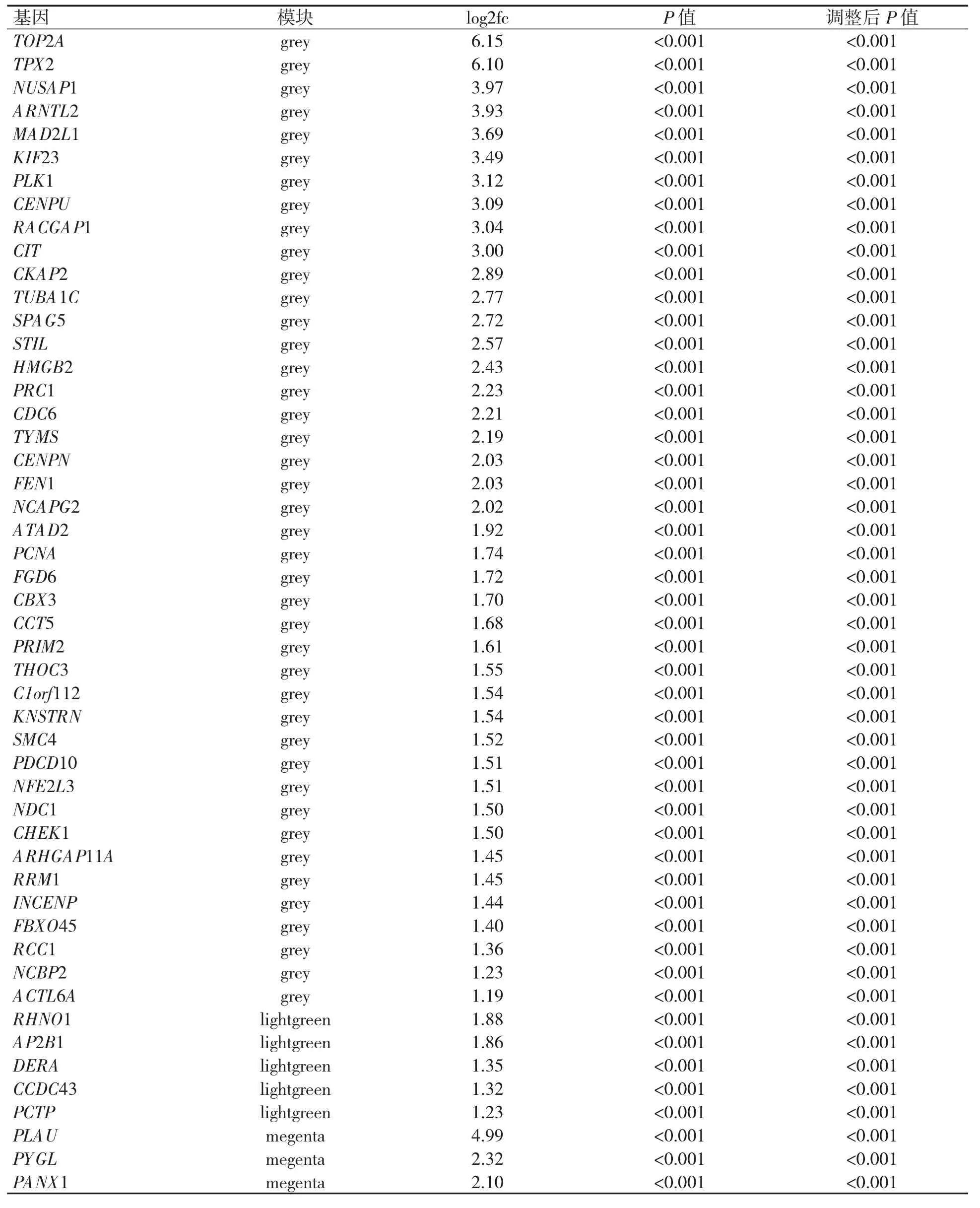
表1 从每个模块中选择的核心基因
2.4 PPI 构建和核心基因筛选
采用Cytoscape 构建WGCNA 模块基因的PPI 网络。设置参数cor.gene Module Membership>0.8 和cor.gene Trait Significance>0.2 用于筛选模块中的核心基因。共获得36 个候选基因,采用聚类分析方法和MCC 算法,最后获得22 个核心基 因(TOP2A、MAD2L1、TPX2、RACGAP1、PRC1、KIF23、NUSAP1、PLK1、SMC4、CHEK1、CENPU、CENPN、TYMS、FEN1、PCNA、CDC6、INCENP、ARHGAP11A、SPAG5、ATAD2、RRM1、NCAPG2)(图7)。PPI 网络中颜色是MCC 分析的度量值映射,圈的大小是PPI 得分映射。
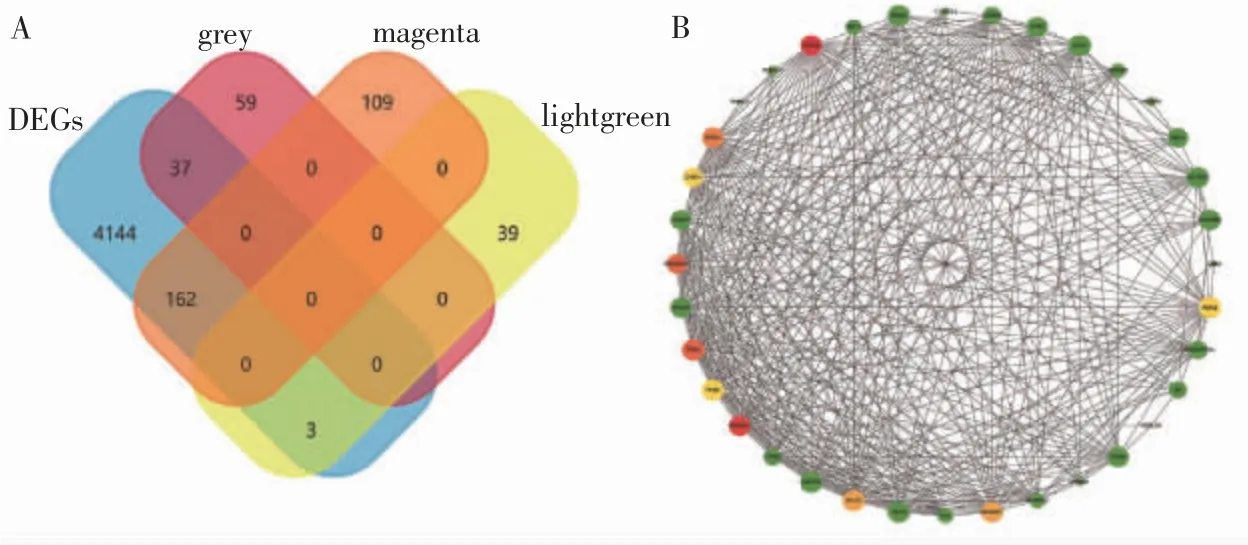
图7 PPI 构建和核心基因筛选
2.5 核心风险基因的表达和生存分析
通过在线网站GEIPA 搜寻PPI 网络得分前10 个的核心风险基因在PDAC 和正常组织中的表达趋势。结果显示,核心基因在PDAC 样本中的表达量均高于正常对照组(P 均>0.05)(图8A~图8J)。生存分析显示,TPX2(HR=2.2,P=0.000 26)、PRC1(HR=2,P=0.001 3)、KIF23(HR=1.9,P=0.002 9)、RACGAP1(HR=1.9,P=0.003 1)和NUSAP1(HR=1.8,P=0.004 6)等核心基因高表达与PDAC不良预后均相关(图9A~图9J)。

图8 前10 个核心基因在肿瘤组织和非肿瘤组织中差异表达的箱线图
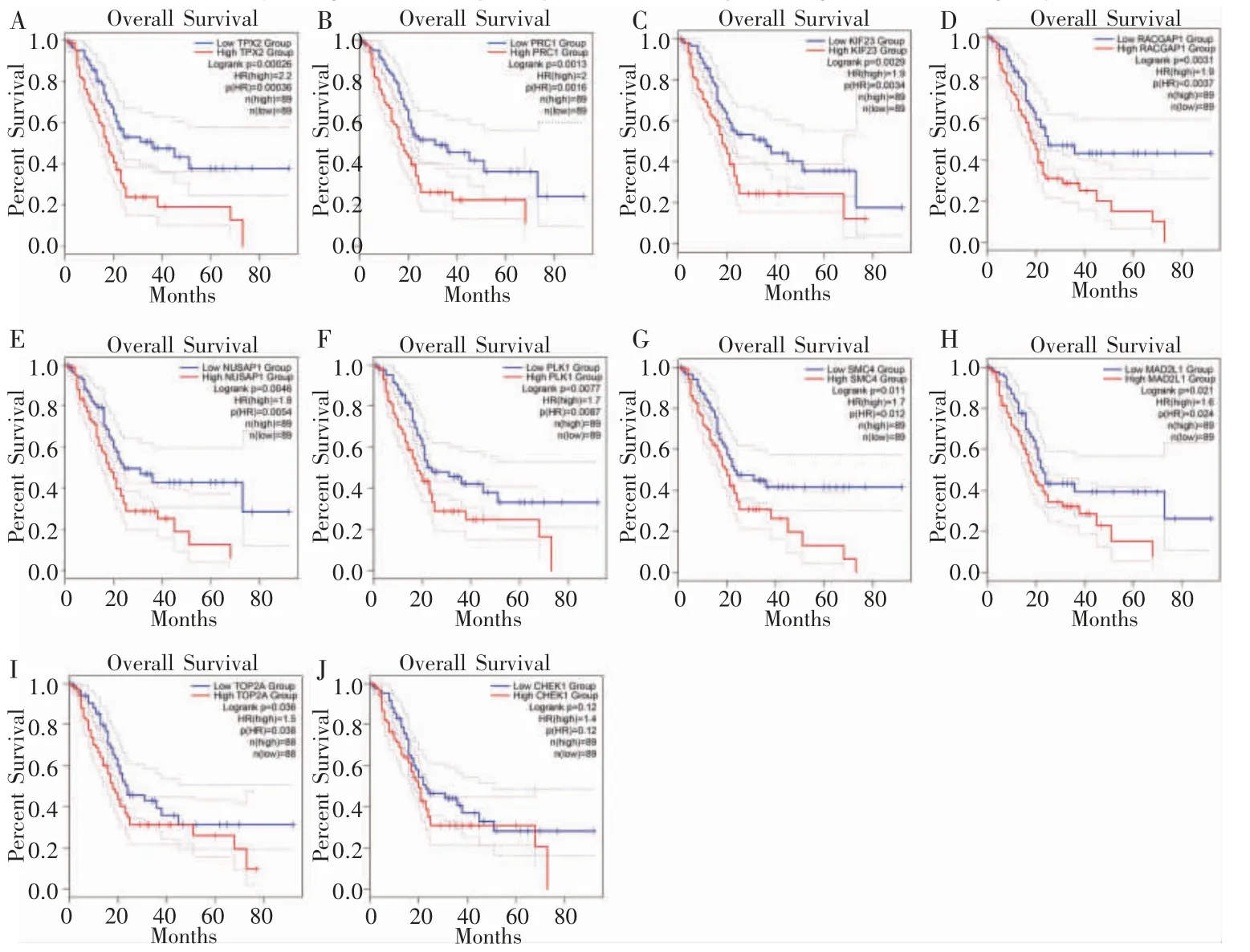
图9 前10 个核心基因的生存分析曲线
2.6 核心风险基因的诊断价值
10 个基 因 的AUC 值(MAD2L:0.999、TPX2:0.998、TOP2A:0.997、RACGGAP1:0.995、KIF23:0.995、NUSAP1:0.995、PLK1:0.992、PRC1:0.989、SMC4:0.986、CHEK1:0.986)均>0.5,表明核心风险基因对肿瘤和正常组织具有良好的区分和诊断能力(表2)。
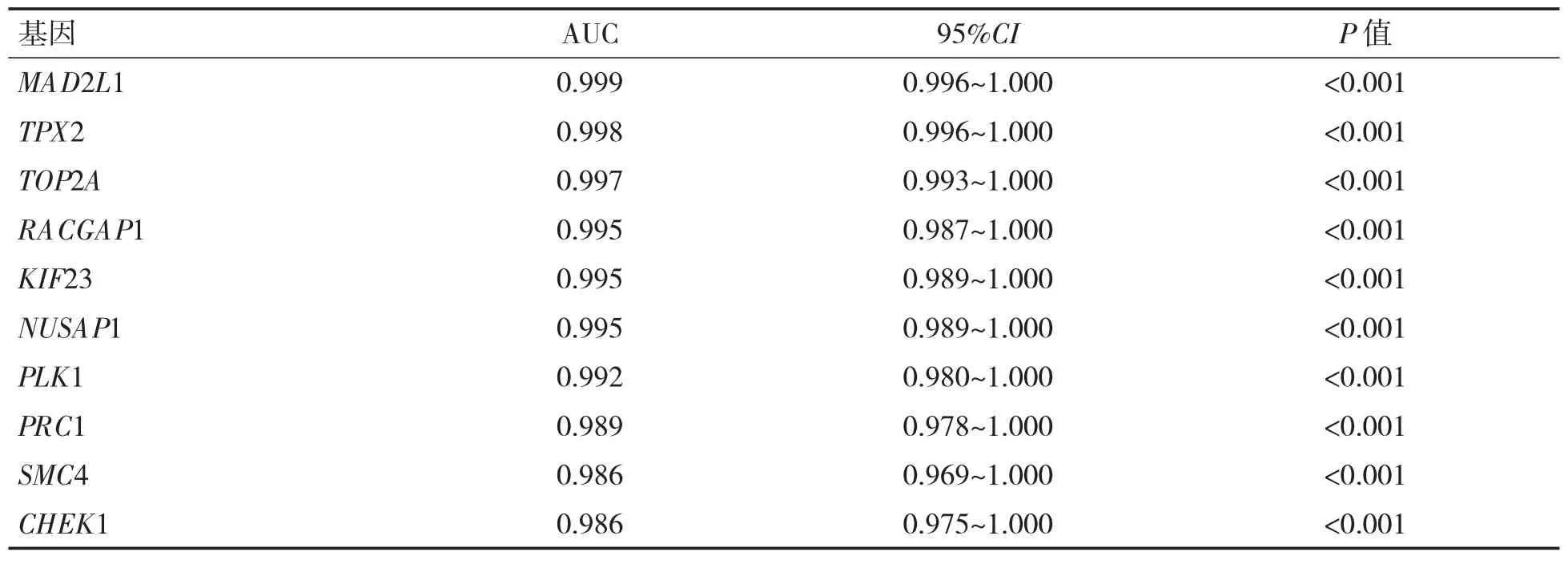
表2 基于不同核心风险基因预测PDAC 的ROC 分析
3 讨论
PDAC 是一种病死率高、诊疗困难的消化道恶性肿瘤,预后极差。对于肿瘤发生潜在机制的研究可能是PDAC 诊断、治疗和延长患者生存时间的关键。高通量测序技术的发展为其分子病理、临床诊断和靶向治疗提供了新的希望[11]。
WGCNA 作为有效的基于表型-基因表达权重关联分析的方法,能够有效提取高维基因表达数据中有效的模块信息,已被广泛用于疾病相关基因的挖掘[12]。在本研究中,通过联合GTEx 中正常组织数据,有效克服TCGA 数据库中PDAC 正常对照缺乏的问题,剔除异常和低表达样本,通过差异表达分析,最终获得了PDAC 全面的转录组表达谱,为PDAC 基因表达和功能研究提供了较好的数据集。
经差异表达分析,本研究共筛选出4 346 个DEGs,其中上调基因2 284 个,下调基因2 062个。对前125 个上调和下调基因聚类分析显示,DEGs 可以显著区分PDAC 组织和正常组织。通过富集分析,模块基因所涉及的BP 主要包括细胞外、质膜整体和质膜;CC 主要包括细胞通讯、细胞生长和信号转导;MF 主要包括细胞黏附分子活性、受体活性和催化活性。利用WGCNA 分析筛选了与PDAC 组织学分级、肿瘤大小和TNM分期密切相关的grey 模块、magenta 模块和lightgreen 模块,进一步区分共表达网络和36 个PPI网络候选基因。通过生物信息学分析鉴定出10个核心基因,包括TOP2A、MAD2L1、TPX2、RACGAP1、PRC1、KIF23、NUSAP1、PLK1、SMC4、CHEK1,最后经过ROC 曲线的验证,与PDAC 的进展和预后密切相关。这些核心基因的表达在PDAC 和正常组织之间差异有统计学意义。同时,它们与PDAC 的组织学分级高度相关,可能是潜在的生物标记物。以上结果可能有助于改善PDAC 患者的治疗决策、风险分层和预后预测。
这10 个核心基因通过对肿瘤细胞周期的调控,参与了肿瘤的发生和增殖。本研究中,筛选获得的NUSAP1、PRC1 和SMC4 基因在PDAC 中研究相对较少。其中,NUSAP1 是一种在多种生物学功能中起着关键作用的微管相关蛋白,包括纺锤体组装、染色体分离、胞质分裂、微管交联、捆绑和附着在染色体上[13]。研究[14]表明,NUSAP1 参与了多种人类恶性肿瘤的生物学行为调控,如胰腺癌、胶质母细胞瘤、肝细胞癌、前列腺癌、胃癌等。PRC1 是有丝分裂早期CDK1(Cdc2/细胞周期蛋白B)磷酸化的细胞质分裂所必需的微管相关蛋白。PRC1 被敲除的细胞通常经历间期、前期和中期;但纺锤体中心区域的结构在后期出现异常,导致细胞因子的异常表达和双核或多核细胞的形成[15],从而促进肿瘤的发生和进展[16]。PRC1的过表达可通过调节Wnt 信号通路的致癌作用,导致早期复发和患者的不良预后。PRC1 的下调也被证明可以显著抑制胃癌细胞的增殖,减少单层集落的形成,并抑制胃癌细胞的侵袭性和转移[16]。PRC1 在PDAC 中异常表达,并显著富集于EMT过程中,提示PRC1 基因可能通过Wnt 信号通路参与PDAC 的EMT 过程,但需要进一步实验研究证明。SMC4 是细胞分裂中的凝缩蛋白,参与细胞分裂过程中的染色体凝集、姐妹染色单体的凝聚、DNA 修复和复制[17]。SMC4 可通过激活宫颈癌中的NF-κB 通路促进宫颈癌的发生[18]。在侵袭性乳腺癌细胞中,SMC4 的mRNA 表达上调。上调的mRNA 可以提高CDK1 在进入有丝分裂时驱动染色质压缩的敏感性,增强癌细胞的侵袭性、增殖活性和去分化能力[18]。SMC4 的高表达可能通过增强TOP2A 的作用而增加双链DNA 断裂,并导致乳腺上皮细胞中的突变、错配和独特的染色体重排[19]。过表达SMC4 可激活JAK2/Stat3和TGFβ/Smad 通路,促进癌细胞的侵袭性[20]。SMC4 与PDAC 的发病机制密切相关,其高表达导致PDAC 的预后差[17,21]。
综上所述,本研究经过不同生物信息学分析方法,发现grey 模块与PDAC 组织学分级高度相关,结合表达和生存分析,从模块中筛选出了TPX2、PRC1、KIF23、RACGAP1、NUSAP1、PLK1、SMC4、MAD2L1、TOP2A、CHEK1 等核 心风险基因。以上基因中,部分已在PDAC 相关研究中报道,而大部分基因在PDAC 中作用机制尚无明确报道。通过注释发现,核心风险基因可能通过调控细胞周期、DNA 复制、EMT 等生物学过程参与PDAC 的发病和预后,但上述基因的功能需要进一步实验证实。总之,本研究通过系统的基因差异表达和WGCNA 分析,进一步结合生存分析和ROC 曲线的验证,发现了一系列重要的PDAC 核心风险基因,为PDAC 未来的临床诊疗与更好的预后干预提供了潜在的分子理论基础。
