组蛋白甲基化转移酶SETD4对鼻咽癌细胞增殖和迁移的影响*
2023-03-10封慕茵郑阿秀白建荣曾玉梅罗泊涛申志华揭伟
封慕茵, 郑阿秀▲, 白建荣, 曾玉梅, 罗泊涛, 申志华△, 揭伟,2,3△
组蛋白甲基化转移酶SETD4对鼻咽癌细胞增殖和迁移的影响*
封慕茵1, 郑阿秀1▲, 白建荣1, 曾玉梅4, 罗泊涛1, 申志华1△, 揭伟1,2,3△
(1广东医科大学基础医学院病理学系,广东 湛江 524023;2海南医学院第一附属医院肿瘤内科,海南 海口 570102;3海南医学院肿瘤研究所,海南 海口 571199;4中山市人民医院病理科,广东 中山 528400)
明确组蛋白甲基化转移酶含SET结构域蛋白4 (SET-domain-containing protein 4, SETD4)在临床鼻咽癌(nasopharyngeal carcinoma, NPC)组织中的表达,分析SETD4对NPC细胞增殖和迁移的影响。采用免疫组织化学法检测86例临床NPC标本与对照组30例鼻咽黏膜慢性炎症(nasopharyngeal chronic inflammation, NPI)组织中SETD4蛋白的表达;基于CRISPR/Cas9技术敲除CNE2细胞中基因,DNA测序结合半定量RT-PCR进行鉴定;以敲除前后的CNE2细胞为研究对象,于倒置显微镜下观察细胞形态,采用CCK-8实验分析细胞增殖活性、Transwell实验观察细胞迁移能力变化、Western blot检测增殖细胞核抗原(proliferating cell nuclear antigen, PCNA)、细胞周期相关蛋白、上皮-间充质转化标志物和组蛋白赖氨酸甲基化的变化,并通过生物信息学富集相关联的功能和信号通路。SETD4蛋白主要定位于NPC细胞核中,NPC组织中SETD4阳性表达率显著低于NPI对照组(<0.01)。基于CRISPR/Cas9技术成功敲除了CNE2细胞的基因,获得基因敲除的纯合子细胞。与野生型(wild-type, WT)细胞株相比,敲除(knockout, KO)细胞株呈现更明显的梭形和多角形,增殖活性和迁移能力均显著增加(<0.01),E-cadherin和p21蛋白表达显著下调(<0.05),而cyclin D1、cyclin E1、N-cadherin和vimentin蛋白表达则显著上调(<0.05)。此外,与WT组相比,KO组细胞中H3K4me2、H3K4me3、H3K9me1、H3K27me3、H3K79me2和H4K20me2水平显著降低(<0.05)。基因集富集结果显示,与细胞周期的功能和信号通路相关。临床NPC组织中SETD4蛋白的表达减弱或缺失;SETD4表达减弱通过上调细胞周期相关蛋白促进NPC细胞增殖,通过诱导上皮-间充质转化而促进细胞迁移;SETD4影响NPC细胞中H3K4、H3K9、H3K27、H3K79和H4K20多个位点的甲基化。
鼻咽癌;组蛋白甲基转移酶;含SET结构域蛋白4;细胞增殖;细胞迁移;基因集富集
鼻咽癌 (nasopharyngeal carcinoma, NPC)是起源于鼻咽黏膜上皮的恶性肿瘤。遗传易感性和肿瘤微环境因素的胁迫与NPC发病和进展有很强的相关性[1-2],而环境胁迫往往带来表观遗传修饰的改变[3-4]。组蛋白甲基化修饰被认为是表观遗传修饰中重要的方式之一[5]。含SETD结构域蛋白4(SET domain-containing protein 4, SETD4)作为组蛋白甲基化的重要成员之一,主要由位于羧基末端的SET结构域和位于氨基末端的底物结合结构域组成。其中SET结构域因其共同存在于果蝇的三个调节因子Su(var)3-9、E(z)、和Trithorax的羧基末端而得名,而含SET结构域的蛋白统称为 SET 家族蛋白[6]。SET家族蛋白普遍具有组蛋白和非组蛋白甲基化转移酶的功能,通过甲基化的修饰作用能够调控基因表达。课题组前期的研究提示对组蛋白H3K4,H4K20的甲基化有影响[7],但迄今对基因的功能研究相对较少,特别是有关与肿瘤关系的报道不多[8-9],本研究首次探讨基因与NPC的关系,旨在基于表观遗传修饰角度为NPC的发病机制提供新的证据。
材料和方法
1 细胞系与临床组织样本
稳定表达Epstein-Barr病毒潜伏膜蛋白1的低分化NPC细胞系CNE2为我室构建。86例NPC和30例鼻咽黏膜慢性炎(chronical nasopharyngeal inflammation,NPI) 患者的石蜡包埋组织来自2017~2019年中山市人民医院病理科保存的标本蜡块。NPC患者组织学类型均为非角化型未分化性癌,患者均为首次就诊且未接受任何治疗。NPC患者平均年龄(46±6)周岁,对照NPI患者平均年龄(45±4)周岁。临床石蜡包埋组织标本使用符合中山市人民医院有关伦理规定。
2 主要试剂及仪器
RPMI-1640培养液(HyClone);胎牛血清(GIBCO);人Cas9过表达质粒(赛业生物科技有限公司);SETD4 Ⅰ抗(Novus biological);β-actin Ⅰ抗(Santa Cruz);PCNA Ⅰ抗(CST);E-cadherin、N-cadherin、vimentin、cyclinA2、cyclinD1、cyclinB1、cyclinE1、细胞周期蛋白依赖性激酶(cyclin-dependent kinases)2、CDK4、CDK6、p21等Ⅰ抗均为武汉三鹰生物技术有限公司产品;组蛋白相关H4K20me1、H4K20me2、H4K20me3、H3K4me2、H3K4me3、H3K9me1、H3K9me2、H3K9me3、H3K27me3、H3K36me1、H3K36me2、H3K36me3、H3K79me1、H3K79me2和H3K79me3等Ⅰ抗均为Abcam产品;H3、H4、H3K4me1、H3K27me1和H3K27me2等Ⅰ抗均为南京Bioword产品。PCR引物(生工生物工程股份有限公司);细胞培养箱(Espec);荧光定量PCR仪(Light Cycler 480 Ⅱ);凝胶成像分析系统( Tanon);倒置显微镜(Micro Star AO)。
3 方法
3.1免疫组织化学染色参照既往方法进行[10]。简言之,临床NPC组织与对照NPI组织标本的石蜡包埋切片后经系列二甲苯脱蜡和乙醇处理,切片置柠檬酸盐缓冲液中行热抗原修复,用3% H2O2阻断内源性过氧化物酶阻断剂,PBS缓冲液洗涤后滴加SETD4 Ⅰ抗(1∶500),置于4 ℃冰箱中过夜孵育,PBS溶液冲洗3 min×3次,滴加适量辣根酶标记的羊抗小鼠/兔IgG聚合物,室温孵育30 min,PBS溶液冲洗2 min×3次,DAB显色;苏木精复染;1%盐酸分化;自来水冲洗返蓝;脱水,封片,镜检。SETD4表达评分如下,首先按阳性细胞数评分:无阳性细胞为0分,<10%为1分,≥10%但<50%为2分,≥50%为3分,≥75%为4分;其次按显色强度评分:无着色为0分,淡黄色为1分,棕黄色为2分,棕褐色为3分。数据分析时,综合两者分数相乘总分≥6分为高表达。
3.2NPC细胞培养及基因敲除CNE2细胞用含10%胎牛血清、RPMI-1640完全培养液重悬,置于37 ℃、体积分数为5%的CO2饱和湿度培养箱内培养。每2 d换液,待贴壁细胞融合率达80%以上后,用0.25%胰酶消化传代,待细胞扩增至对数生长期后用于相关实验。设计针对人基因的sgRNA并排除脱靶现象。CNE2细胞接种于6孔板中,待细胞生长汇合至60%左右,采用电转染法将sgRNA、Cas9过表达质粒共转染至CNE2细胞中。sgRNA信息:sgRNA-#1为5’-TAAAGTCCACGGTGCTCTTTAGG-3’, sgRNA-#2为5’-CATGAGACCCCTCATACTCTTGG-3’。72 h后将细胞胰酶消化后以每孔一个细胞接种于96孔板,常规换液,待细胞长出单克隆后进行基因型鉴定。对初步鉴定为纯合子的2B9和2C10克隆再次进行DNA测序验证。提取2B9和2C10细胞mRNA,再进行mRNA表达的检测。后续细胞学实验均以被敲除的2B9细胞为敲除(knock out,KO)组、以未敲除的CNE2细胞为野生型(wild type,WT)组进行。
3.3CCK-8法检测细胞的增殖活性参考既往方法进行操作[11]。细胞按每孔2×103个/接种于96孔板内,过夜孵育贴壁。按贴壁后培养时间0 h、24 h、48 h及72 h每孔加入10 µL CCK-8试剂,37 ℃继续孵育2 h,酶标仪上读取450 nm 吸光度(),每组5个复孔,实验重复3次。
3.4Transwell实验参考既往方法进行操作[11]。用含0.5%胎牛血清的RMPI-1640培养液重悬细胞,取2×104个细胞接种于Transwell小室上室,总体积200 µL,下室中加含10%胎牛血清的RPMI-1640培养液500 µL;将24孔板置于37 ℃、5% CO2、饱和湿度的培养箱内孵育24 h,弃去培养液,用PBS洗2次,4%多聚甲醛固定20 min,吸走多聚甲醛,用干净的棉签擦拭小室内表面,加入0.1%结晶紫染液染色30 min;用棉签小心擦除小室的上室面细胞,显微镜下观察并拍照。以穿膜的细胞数差异代表细胞运动能力的改变。每张膜选5个固定视野,计算平均细胞数,每组重复3孔。
3.5PCR采用哺乳动物基因组DNA提取试剂盒提取细胞DNA,PCR检测基因的DNA片段以分析细胞的基因型。PCR反应引物F1:5'-CTCACAGGAACAGCTGGAG-3'; R1:5'-ACAAGGCTGAAGCTGGATGT-3';F2:5'-CAGGTTGTGGGTCTTGGAAC-3';R2:5'-CATATGTACACATCTGAGTGCAAG-3';PCR反应Tm值为62 ℃,按F1-R1,F2-R2和F1-R2引物组进行PCR反应。提取细胞总RNA,取1.0 μg总RNA逆转录为cDNA,采用半定量RT-PCR鉴定基因敲除单克隆细胞中mRNA水平。SETD4上游引物为5'-CTGAAGAGCAGAGAGCCCAC-3',下游引物为5'-ACGCTGCTTTTACCTGGACAT-3',产物长度260 bp。β-actin上游引物为5'-GGGAAATCGTGCGTGACATT-3',下游引物为5'-ACAGGACTCCATGCCCAGG-3',产物长度200 bp, Tm值均为60 ℃。采用定量PCR检测PCNA mRNA的水平,PCNA上游引物为5'-TCCCTTACGCAAGTCTCAGC-3',下游引物为5'-GAGTCCATGCTCTGCAGGTT-3',Tm值为60 ℃;内参照β-actin引物同上。
3.6Western blot参考既往方法进行操作[12-13]。提取细胞总蛋白,用BCA法定量,取50.0 μg蛋白用于凝胶电泳;待凝胶中的蛋白转移至PVDF膜后,用5%封闭液封闭1 h后加入Ⅰ抗,抗体稀释比例如下:PCNA(1∶500)、cyclin A2(1∶500)、cyclin B1(1∶500)cyclin D1,(1∶500)、cyclin E1(1∶500)、CDK2(1∶500)、CDK4(1∶500)、CDK6(1∶500)、p21(1∶500)、E-cadherin(1∶500)、N-cadherin(1∶500)、vimentin(1∶500)、H4(1∶800)、H4K20me1(1∶5 000)、H4K20me2(1∶2 000)、H4K20me3,(1∶1 000)、H3(1∶500)、H3K4me1(1∶1 000)、H3K4me2(1∶2 000)、H3K4me3(1∶1 000)、H3K9me1(1∶20 000)、H3K9me2(1∶1 000)、H3K9me3(1∶1 000)、H3K27me1(1∶1 000)、H3K27me2(1∶1 000)、H3K27me3(1∶1 000)、H3K36me1(1∶1 000)、H3K36me2(1∶10 000)、H3K36me3(1∶5 000)、H3K79me1(1∶10 000)、H3K79me2(1∶2 500)、H3K79me3(1∶1 000)、β-actin(1∶500)。条带置于4 ℃条件下过夜孵育,TBST洗涤3遍后加入HRP标记的IgG(1∶2 000)置于摇床上室温孵育1 h,洗涤后经ECL发光液显影,凝胶成像分析系统扫描获得目的蛋白条带。实验重复3次。
3.7基因集富集分析由于TCGA数据库中缺乏鼻咽癌这一组织类型,本研究选取TCGA数据库头颈部鳞癌中患者的表达数据,使用R语言计算与所有基因的相关性,以<0.05和矫正后<0.25为阈值,使用R语言clusterprofiler包(4.1.3版本)进行基因集富集分析,包括GO(gene oncology)功能和KEGG(Kyoto Encyclopedia of Genes and Genomes)信号通路,将排名前20的GO功能和KEGG信号以波浪图展示。
4 数据学处理
采用统计软件GraphPad Prism 8.0进行统计分析。临床标本中SETD4蛋白表达情况的比较采用卡方检验。体外实验各组数据以均数±标准差(mean±SD)表示,两组间比较采用非配对检验,以<0.05为差异有统计学意义。基因集富集分析中应用R语言包将每个基因集的富集分数标准化,通过计算伪发现率(false discovery rate, FDR) 值来控制假阳性率,以<0.05且FDR<0.25为差异有统计学意义。
结果
1 临床NPC组织中SETD4蛋白表达情况
对86例NPC和30例NPI组织进行免疫组化检测,结果如图1所示,SETD4蛋白表达为细胞核、细胞质出现阳性信号,以细胞核表达为主;临床 NPC组织中SETD4强阳性表达率(16/86,18.6%)显著低于NPI对照组(22/30,73.3%),差异有统计学意义(<0.01)。鉴于NPC中SEDT4蛋白表达较对照组明显下降且总体阳性率不高,故本研究中未进一步分析SETD4蛋白与NPC患者临床参数的相关性。

Figure 1. The expression of SETD4 protein in clinical nasopharyngeal carcinoma and control tissues was detected by immunohistochemistry staining. A: representative cases with different levels of SETD4 protein expression [SETD4 protein was mainly localized in the nuclei of the epithelial cells of nasopharyngeal mucosa and nasopharyngeal carcinoma tumor cells; the lower panels (scale bar=50 μm) are the magnified visual field of the boxes in the upper panels (scale bar=100 μm)]; B: number of cases with high and low expression of SETD4 protein in NPC and NPI tissues (Chi square test; data were expressed as the sample's numbers; **P<0.01).
2 成功获得SETD4基因敲除的NPC细胞系
应用CRISPR/Cas9技术对NPC细胞中基因进行编辑后挑选单克隆细胞进行DNA测序和半定量PCR验证,结果显示,成功筛选到2株基因敲除的纯合子细胞(2B9和2C10),选择其中的2B9细胞作为KO组进行后续实验,见图2。
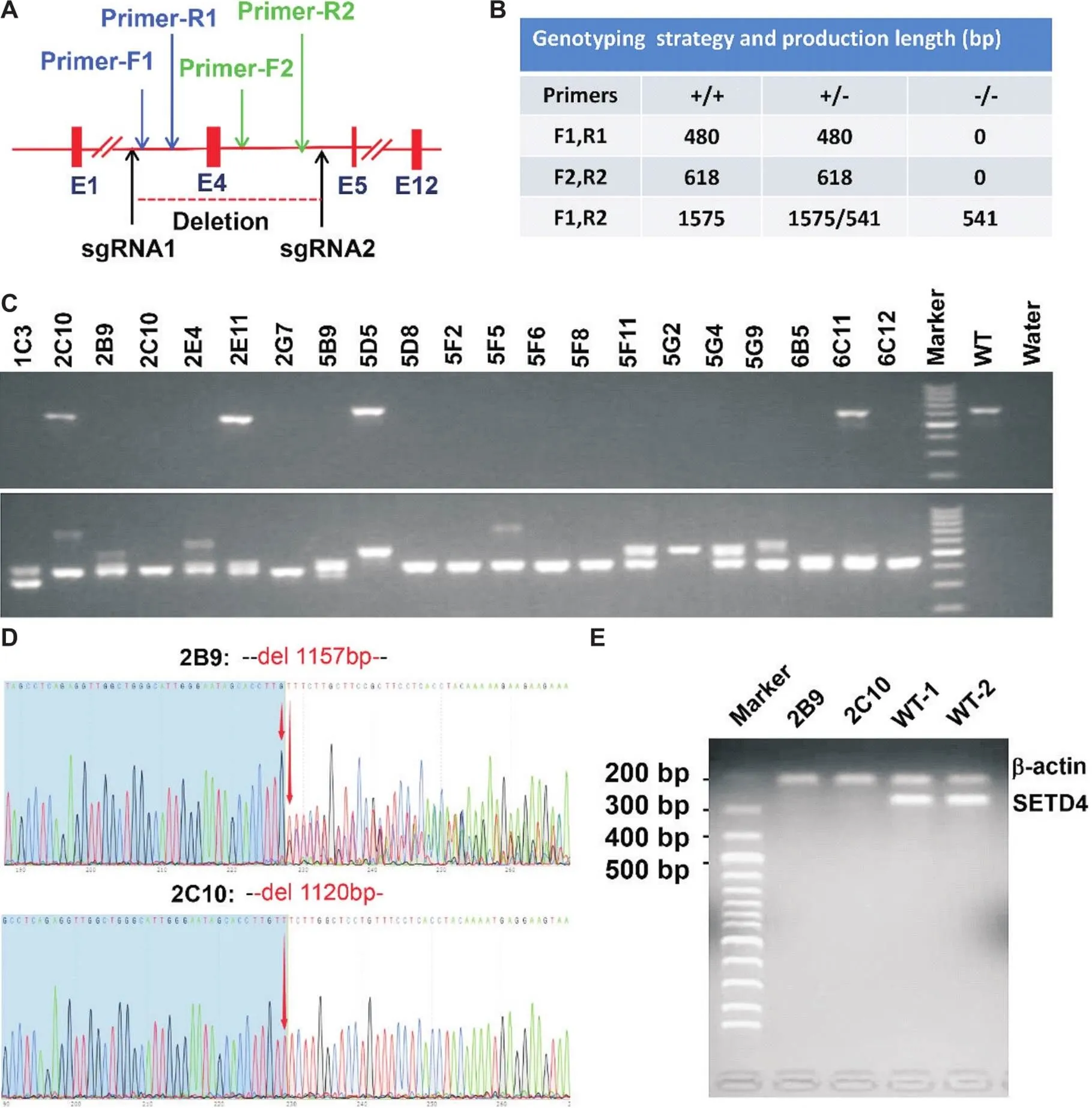
Figure 2. Construction of SETD4 gene knockout NPC cell lines using CRISPR/Cas9 technique. A: schematic diagram of targeted knockout of exon 4 of SETD4 gene and genotype identification; B: genotyping strategy and PCR product size of SETD4 knockout cell lines [the wild type (WT, +/+), heterozygote (+/-) and homozygote (-/-) were determined according to the size of PCR products using different primer groups]; C: agarose electrophoresis image of PCR products for genotype identification of monoclonal cell lines; D: DNA sequencing results of typical SETD4 knockout monoclonal cell lines (the number in red font indicates the length of the deleted DNA fragment of SETD4 gene in these two monoclonal cell lines); E: semi-quantitative RT-PCR detection of SETD4 mRNA levels in SETD4 knockout monoclonal cell lines and wild-type cells (no target bands in PCR product indicates the SETD4 knockout monoclonal cells).
3 SETD4缺失增加NPC细胞的增殖活性
CCK-8实验结果表明,KO组细胞的增殖活性除第24 h较WT组有所减缓外,随着培养时间的延长,KO组细胞增殖活性显著高于WT组(<0.01)。进一步通过定量PCR检测了细胞增殖标志物PCNA的表达,结果提示KO组PCNA mRNA水平高于WT组(<0.01)。通过Western blot检测PCNA和细胞周期相关蛋白的表达,结果显示KO组PCNA、cyclin D1和cyclin E1水平较WT组显著增加(<0.05),p21水平下降(<0.05),见图3。

Figure 3. Knockout of SETD4 gene in NPC cells promoted cell proliferation. A: the cell proliferation in SETD4 KO group and WT group was detected by CCK-8 assay; B: quantitative RT-PCR was used to detect the level of PCNA mRNA in WT and SETD4 KO cell lines; C: the expression levels of cell cycle-associated proteins were detected by Western blot. Mean±SD. n=3. *P<0.05, **P<0.01 vs WT group.
4 SETD4缺失对NPC细胞上皮-间充质转化的影响
KO组细胞的形态较WT组出现更明显的多角形和短梭形(图4A)。通过Western blot检测上皮-间充质转化相关蛋白的表达,观察到KO后显著增加了N-cadherin和vimentin蛋白的表达(<0.05),显著减少了E-cadherin蛋白的表达(<0.05)。功能上,KO细胞较WT组细胞迁移能力显著增加(<0.01),见图4。

Figure 4. SETD4 gene knockout induced epithelial-mesenchymal transition in NPC cells. A: representative morphological images of WT group and SETD4 knockout group (compared with WT cells, SETD4 knockout cells showed more obviously spindle and polygonal morphology); B: Western blot detection of protein levels of epithelial-mesenchymal transition-related markers including E-cadherin, N-cadherin and vimentin; C: representative images and quantification analysis for cell migration in WT and SETD4 knockout cells. Mean±SD. n=3. *P<0.05, **P<0.01 vs WT group.
5 SETD4表达改变对组蛋白赖氨酸甲基化的影响
采用Western blot对常见组蛋白H3和H4的某些位点的赖氨酸甲基化水平进行了检测,结果表明,表达缺失显著减弱了NPC细胞中H3K4me2、H3K4me3、H3K9me1、H3K27me3、H3K79me2和H4K20me2蛋白水平,而对其他位点如H3K39me1、H3K39me2、H3K39me3、H3K4me1、H3K9me2、H3K9me3、H3K27me1、H3K39me2、H3K79me1、H3K79me3、H4K20me1和H4K20me3蛋白表达无明显影响,见图5。
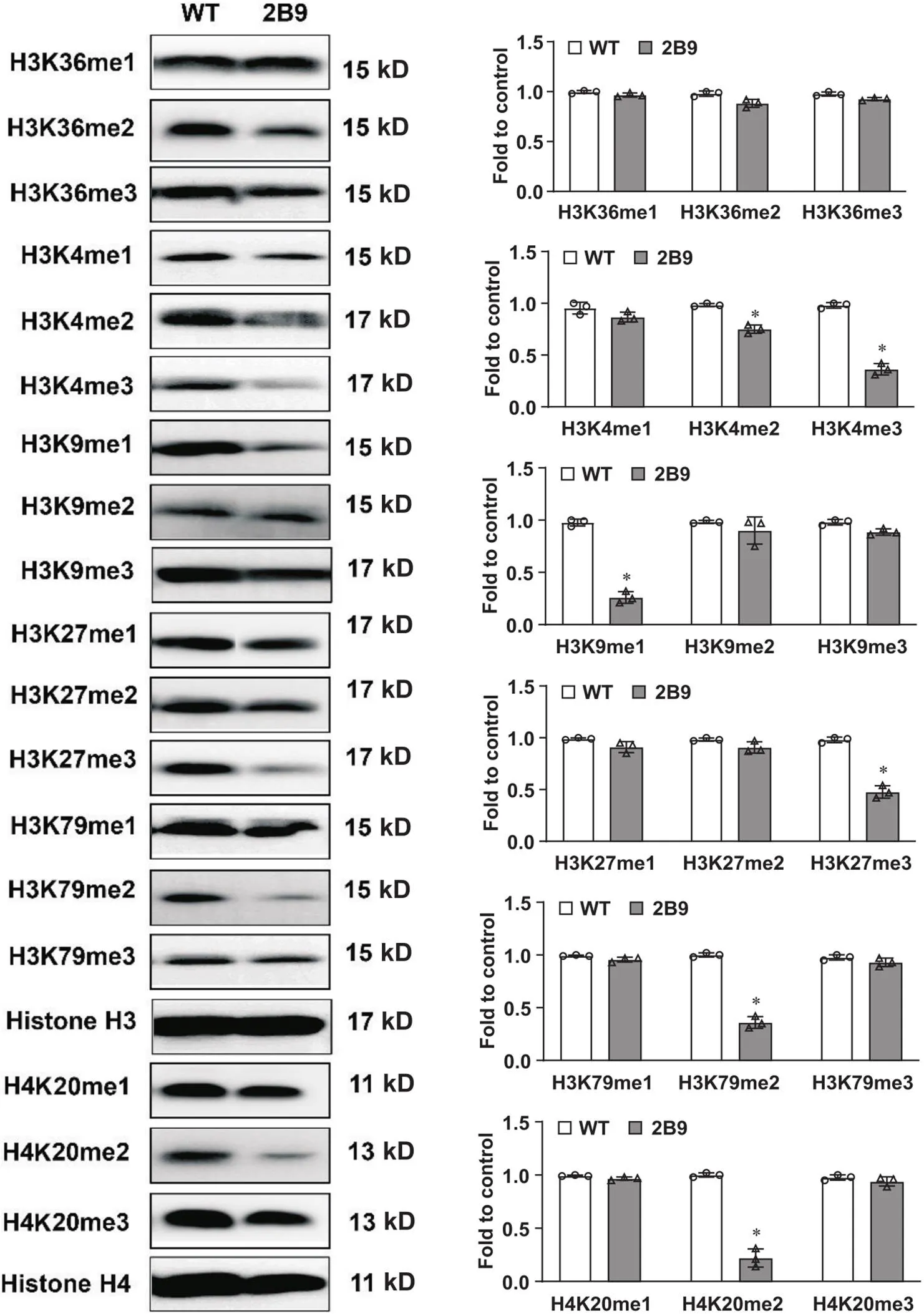
Figure 5. The effect of SETD4 knockout on methylation of histone lysine sites in NPC cells detected by Western blot. Mean±SD. n=3. *P<0.05 vs WT group.
6 与SETD4关联的功能和信号通路
与关联的GO功能主要有DNA复制(DNA replication)、姐妹染色体分离(sister chromatid segregation)、细胞周期检查点信号(cell cycle checkpoint signaling)、有丝分裂细胞周期(mitotic cell cycle)、有丝分裂细胞周期G2/M转换(G2/M transition of mitotic cell cycle)和有丝分裂细胞周期G1/S转换(G1/S transition of mitotic cell cycle)等。KEGG信号主要涉及错配修复(mismatch repair)、细胞周期(cell cycle)、同源重组(homologous recombination)、泛素化介导的蛋白降解(ubiquitin mediated proteolysis)等,见图6。
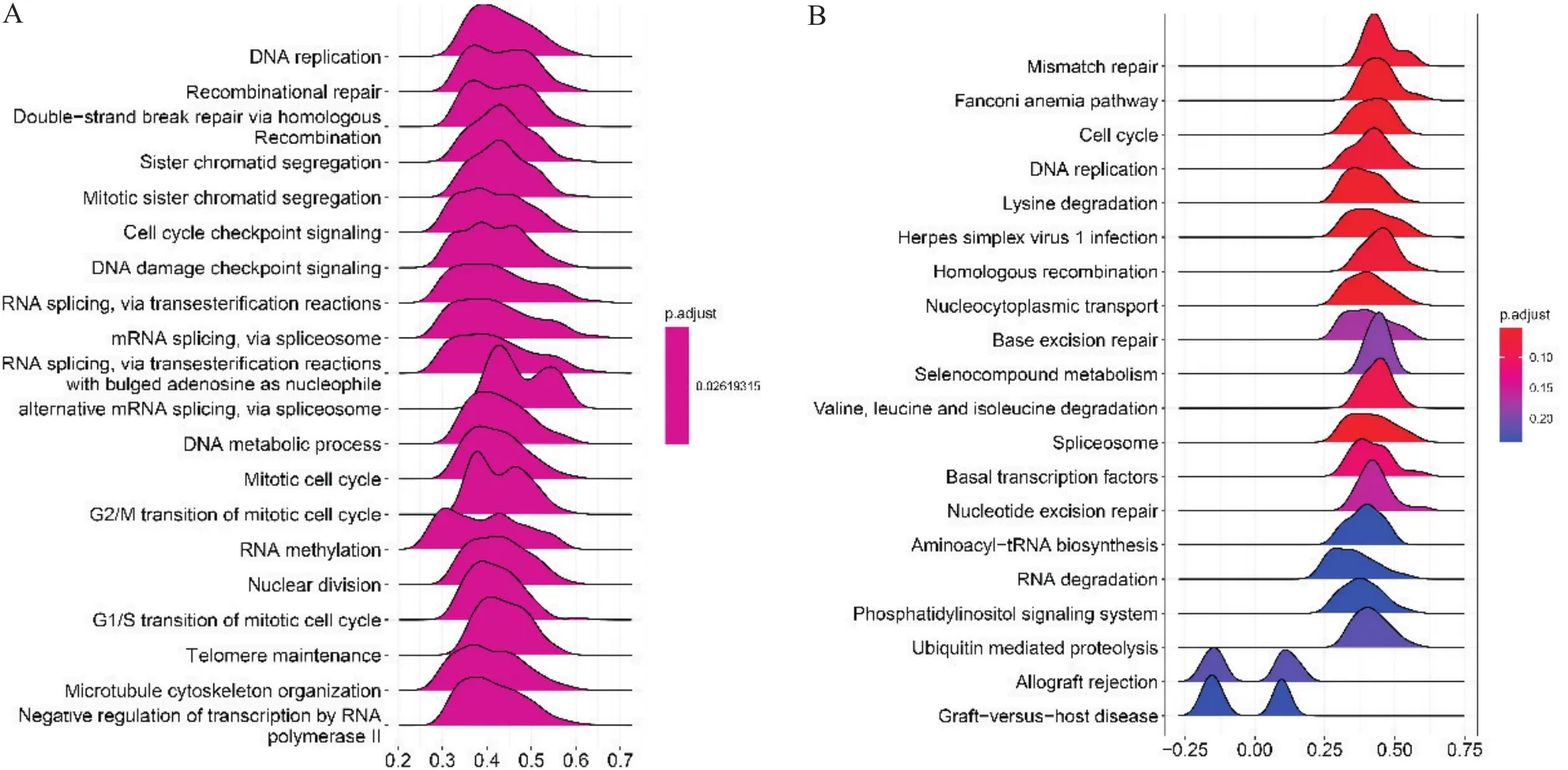
Figure 6. SETD4-related GO annotations and KEGG signaling pathways in head and neck squamous cell carcinoma (HNSC). Datasets of HSNC patients in TCGA database were selected, the correlation between SETD4 and genes were calculated using R language cluster profiler package (version 4.13), and those genes with P<0.05 were extracted for GSEA enrichment analysis of GO annotations (A) and KEGG signaling pathways (B), and the top 20 results based on p.adjust values and normalize enrichment scores were displayed.
讨论
基因位于第21号染色体q22.12上,它编码的SETD4蛋白为全细胞分布,以胞核和胞质为主,且SETD4蛋白在多种细胞中均有表达,因其种属及组织来源的不同表达特征有所差别[14],可通过调节多种基因的表达影响细胞功能并参与多种疾病的发生发展[15]。总体上,目前对基因的功能研究甚少,有研究揭示了其在卤虫滞育形成期间发挥着重要作用[16]。而在哺乳动物体内,研究人员观察到通过上调炎症细胞因子的产生,在宿主抵抗感染的先天免疫反应中发挥重要作用[17]。有关与肿瘤关系的报道不多。有报道显示表达异常与乳腺癌有关[8-9],最近研究表明,成年小鼠的缺失延长了辐射诱导的T淋巴瘤的生存期[18]。截止当前尚未见有关与NPC关系的研究。因此,阐明表达异常与NPC的关系,可能为基于表观遗传角度NPC的发病机制提供新的实验依据。
本研究首次显示,SETD4蛋白在临床NPI标本中普遍呈高表达,阳性信号以胞核定位为主。相反,NPC组织中SETD4蛋白则呈低水平表达或不表达,这一结果提示NPC中SETD4蛋白表达丢失可能在NPC病理生理过程中发挥重要作用。从蛋白的表达定位来看,SETD4主要定位于胞核,这与其组蛋白甲基化调控功能所需的细胞核定位是一致的。为进一步明确在NPC中的生物学功能,应用课题组前期已经掌握的CRISPR/Cas9基因组编辑技术[19],我们对CNE2细胞中的基因进行了敲除。CRISPR/Cas9 基因编辑系统可以靶向剪切DNA双链从而获得特定基因敲除的模式细胞或动物,也可进行基因特异位点的敲入,从而获得特异基因过表达模式细胞或动物[20]。实验结果证实成功获得了基因敲除的NPC细胞,且未发现其他的脱靶效应(未发表资料),这为后续研究奠定了细胞模型基础。
实验从多个角度对基因敲除后的NPC细胞的增殖活性进行了探讨。首先,基于CCK-8实验检测到敲除后有利于细胞的增殖活性增加;其次从增殖细胞的代表性标志基因的表达上看,基因敲除从转录和翻译水平上均上调了mRNA和蛋白的表达;再次,考虑到细胞周期与细胞增殖的密切关系,本研究中检测了细胞周期相关蛋白的表达,结果显示基因敲除增加了NPC细胞中cyclinD1和cyclinE1并下调了P21蛋白的表达;因此,表达缺失可以通过调节细胞周期相关因子的表达参与细胞增殖的调控。2013年Faria等[9]首先报道在乳腺癌细胞中SETD4蛋白表达可以通过调控cyclinD1的表达而介导细胞的增殖;然而有研究指出,分化成熟的乳腺癌细胞中SETD4蛋白表达水平低,而静止期的乳腺癌干细胞中SETD4蛋白表达水平显著增加[8]。也有报道进一步提示,基因敲除有利于骨髓干细胞的增殖从而恢复放射损伤后的骨髓功能[21],这些前期报道提示表达增加与干细胞的增殖活性增强有关。我们当前的研究支持表达缺失促进NPC细胞的增殖的现象,但我们猜测与NPC干细胞也存在某种联系。因此,很可能调节细胞增殖的功能存在细胞类型或微环境的差异。
侵袭和转移是恶性肿瘤的基本特征[22],而上皮-间充质转化是上皮细胞恶性肿瘤侵袭和转移的早期事件。本研究表明,基因敲除后赋予了NPC细胞更明显的间充质细胞形态;从基因表达上显示敲除有利间充质表型基因的上调表达;从功能上看,基因敲除增强了NPC的迁移能力。因此,NPC细胞中表达缺陷可能通过诱导肿瘤细胞出现上皮-间充质转化而促进其侵袭和转移。但相关机制目前尚不清楚,很可能组蛋白的甲基化修饰程度的差异在基因的转录调控上发挥了重要的作用。
组蛋白甲基转移酶可将甲基转移到组蛋白的赖氨酸残基上,被添加的甲基基团主要位于H3K4、H3K9、H3K27、H3K36、H3K79及H4K20上。本研究显示,基因敲除后伴随H3K4me2、H3K4me3、H3K9me1、H3K27me3、H3K79me2和H4K20me2的表达水平的显著下降,提示NPC中参与了H3K4、H3K9、H3K36、H3K79及H4K20等多个赖氨酸位点的甲基化修饰,而这些组蛋白赖氨酸甲基化同时涉及了单甲基化、二甲基化和三甲基化。有关SETD4调控H4K20和H3K4的甲基化程度已有报道[9, 16-17],本研究结果提示可能参与调控除H4K20和H3K4外其他多个位点的组蛋白赖氨酸的甲基化,拓展了SETD4靶点的研究。有观点认为,H3K27、H3K9、H4K20、H3K79和H2BK5的单甲基化都与基因激活有关,而H3K27、H3K9和H3K79的三甲基化与基因抑制有关[23-24]。因此,NPC细胞中基因敲除后对基因组表达调控非常复杂,具体基因的表达调控后期可以通过ChIP测序实验来进行筛选。
最后,进一步采用TCGA数据库,实验分析了可能涉及的功能和信号通路。由于TCGA数据库缺乏鼻咽癌这一组织类型,我们以头颈部鳞癌为例进行了探讨,结果显示,关联的功能大多数与细胞有丝分裂、细胞周期转换有关,且在信号通路中也富集到细胞周期这一信号,这些结果充分提示与细胞增殖活性存在一定的关系。考虑到NPC是头颈部鳞癌的一种特殊类型,因此我们推断,表达异常可通过干扰细胞周期来影响NPC的增殖与活性。
总之,本研究首次观察到,相对于良性对照组织,临床NPC组织中SEDT4蛋白表达呈明显的减弱或缺失状态,而SETD4蛋白的表达不足显著促进了NPC细胞增殖和迁移。表达不足影响NPC细胞基因表达可能与其影响多个组蛋白赖氨酸甲基化修饰有关,并可能重点影响了细胞的周期进程。后期还需要优化体外细胞学实验和增加动物实验来进一步验证。
[1] Chen YP, Chan ATC, Le QT, et al. Nasopharyngeal carcinoma[J]. Lancet, 2019, 394(10192):64-80.
[2] Wong KCW, Hui EP, Lo KW, et al. Nasopharyngeal carcinoma: an evolving paradigm[J]. Nat Rev Clin Oncol, 2021, 18(11):679-695.
[3] García-Giménez JL, Garcés C, Romá-Mateo C, et al. Oxidative stress-mediated alterations in histone post-translational modifications[J]. Free Radic Biol Med, 2021, 170:6-18.
[4] Kubota T. Epigenetic alterations induced by environmental stress associated with metabolic and neurodevelopmental disorders[J]. Environ Epigenet, 2016, 2(3):dvw017.
[5] Zhang Y, Sun Z, Jia J, et al. Overview of histone modification[J]. Adv Exp Med Biol, 2021, 1283:1-16.
[6]邢敬慈, 揭伟. 甲基转移酶SET结构域家族及其在心血管发育和疾病中的作用[J]. 浙江大学学报(医学版), 2022, 51(2):251-260.
Xing JC, Jie W. Methyltransferase SET domain family and its relationship with cardiovascular development and diseases[J]. J Zhejiang Univ (Med Sci), 2022, 51(2):251-260.
[7] Liao X, Wu C, Shao Z, et al. SETD4 in the proliferation, migration, angiogenesis, myogenic differentiation and genomic methylation of bone marrow mesenchymal stem cells[J]. Stem Cell Rev Rep, 2021, 17(4):1374-1389.
[8] Ye S, Ding YF, Jia WH, et al. SET domain-containing protein 4 epigenetically controls breast cancer stem cell quiescence[J]. Cancer Res, 2019, 79(18):4729-4743.
[9] Faria JA, Corrêa NC, de Andrade C, et al. SET domain-containing protein 4 (SETD4) is a newly identified cytosolic and nuclear lysine methyltransferase involved in breast cancer cell proliferation[J]. J Cancer Sci Ther, 2013, 5(2):58-65.
[10] Wu Y, Shen Z, Wang K, et al. High FMNL3 expression promotes nasopharyngeal carcinoma cell metastasis: role in TGF-β1-induced epithelia-to-mesenchymal transition[J]. Sci Rep, 2017, 7:42507.
[11] Shen Z, Liao X, Shao Z, et al. Short-term stimulation with histone deacetylase inhibitor trichostatin a induces epithelial-mesenchymal transition in nasopharyngeal carcinoma cells without increasing cell invasion ability[J]. BMC Cancer, 2019, 19(1):262.
[12]袁建玲, 邵钟铭, 邹园, 等. 血清反应因子N端片段对鼻咽癌细胞增殖及迁移能力的影响[J]. 中国病理生理杂志, 2022, 38(3):535-542.
Yuan JL, Sao ZM, Zou Y, et al. Effects of N-terminal fragment of serum response factor on proliferation and migration of nasopharyngeal carcinoma cells[J]. Chin J Pathophysiol, 2022, 38(3):535-542.
[13] 邵钟铭, 王可可, 廖晓敏, 等. 低氧诱导c-Kit+骨髓间充质干细胞增殖及旁分泌中ROCK信号的作用[J]. 中国组织工程研究, 2019, 23(21):3281-3288.
Shao ZM, Wang KK, Liao XM, et al. Roles of ROCK signaling in proliferation and paracrine action of hypoxia-induced c-Kit+bone marrowmesenchymal stem cells[J]. Chin J Tis Eng Res, 2019, 23(21):3281-3288.
[14] 叶萍, 蔡军伟, 付晓霞, 等. p38信号通路对SETD4在细胞中的表达和定位的影响[J]. 中国病理生理杂志, 2012, 28(5):878-883.
Ye P, Cai JW, Fu XX, et al. Effects of p38 MAPK on SETD4 expression and location in p38+/+and p38-/-cells treated with sodium arsenite[J]. Chin J Pathophysiol 2012, 28(5):878-883.
[15] Tran K, Jethmalani Y, Jaiswal D, et al. Set4 is a chromatin-associated protein, promotes survival during oxidative stress, and regulates stress response genes in yeast[J]. J Biol Chem, 2018, 293(37):14429-14443.
[16] Dai L, Ye S, Li HW, et al. SETD4 regulates cell quie-scence and catalyzes the trimethylation of H4K20 during diapause formation in artemia[J]. Mol Cell Biol, 2017, 37(7):e00453-16.
[17] Zhong Y, Ye P, Mei Z, et al. The novel methyltransfe-rase SETD4 regulates TLR agonist-induced expression of cytokines through methylation of lysine 4 at histone 3 in macrophages[J]. Mol Immunol, 2019, 14:179-188.
[18] Feng X, Lu H, Yue J, et al. Loss of Setd4 delays radiation-induced thymic lymphoma in mice[J]. DNA Repair (Amst), 2020, 86:102754.
[19] 王思思, 廖晓敏, 邵钟铭, 等. 基于CRISPR/Cas9技术构建SETD2基因敲除鼻咽癌细胞株并分析其增殖特性[J]. 中国病理生理杂志, 2018, 34(12):2172-2179.
Wang SS, Liao XM, Shao ZZ, et al. Construction ofgene knockout nasopharyngeal carcinoma cell strains using CRISPR/Cas9 technique and analysis of their proliferation property[J]. Chin J Pathophysiol, 2018, 34(12):2172-2179.
[20] Knott GJ, Doudna JA. CRISPR-Cas guides the future of genetic engineering[J]. Science, 2018, 361(6405):866-869.
[21] Feng X, Lu H, Yue J, et al. Deletion of mouse Setd4 promotes the recovery of hematopoietic failure[J]. Int J Radiat Oncol Biol Phys, 2020, 107(4):779-792.
[22] Hanahan D. Hallmarks of cancer: new dimensions[J]. Cancer Discov, 2022, 12(1):31-46.
[23] Barski A, Cuddapah S, Cui K, et al. High-resolution profiling of histone methylations in the human genome[J]. Cell, 2007, 129(4):823-837.
[24] Kimura H. Histone modifications for human epigenome analysis[J]. J Hum Genet, 2013, 58(7):439-445.
Effect of histone methyltransferase SETD4 on proliferation and migration of nasopharyngeal carcinoma cells
FENG Muyin1, ZHENG Axiu1▲, BAI Jianrong1, ZENG Yumei4, LUO Botao1, SHEN Zhihua1△, JIE Wei1,2,3△
(1,,,524023,;2,,570102,;3,571199,;4,,528400,)
To clarify the expression of histone methyltransferase SET-domain-containing protein 4 (SETD4) in clinical nasopharyngeal carcinoma (NPC) tissues, and to analyze the effect of SETD4 on the proliferation and migration of NPC cells.The protein levels of SETD4 in clinical 86 cases of NPC and 30 cases of control nasopharyngeal chronic inflammation (NPI) tissues was detected by immunohistochemistry. Thegene in CNE2 cells was knocked out based on CRISPR/Cas9 technology. The genotypes were identified by DNA sequencing and semi-quantitative RT-PCR. The morphology of cells was observed by inverted microscope, the proliferation activity of cells was analyzed by CCK-8 assay, and the changes in cell migration ability were assessed by Transwell assay. Proliferating cell nuclear antigen (PCNA),cell cycle-related proteins, markers of epithelial-mesenchymal transition and histone lysine methylationwere detected by Western blot. Finally, SETD4-associated functions and signaling pathways were enriched by bioinformatics.SETD4 protein was mainly localized in the nuclei of NPC cells, and the positive expression rate of SETD4 protein in NPC tissue was significantly lower than that in NPI control group (<0.01). Based on CRISPR/Cas9 technology, thegene in CNE2 was successfully knocked out, and homozygous cell lines were obtained. Compared with wild type (WT) cells,knock out (KO)cells showed more obvious spindle and polygonal shapes, and the proliferation and migration ability were significantly increased (<0.01), the expression of E-cadherin and p21 protein were significantly down-regulated (<0.05), while cyclin D1, cyclin E1, N-cadherin, and vimentin protein expressions were significantly up-regulated (<0.05). In addition, the levels of H3K4me2,H3K4me3, H3K9me1,H3K27me3, H3K79me2 and H4K20me2 were decreased in KO group compared with WT group (<0.05).Gene sets enrichment results showed thatwas associated with functions and signaling pathways related to cell cycle.The expression of SETD4 in clinical NPC tissues is generally attenuated or absent. The SETD4 mainly promotes NPC cell proliferation by mediating cell cycle-related proteins, and provokes cell migration by inducing epithelial-mesenchymal transition. The SETD4mainly affects the methylation of H3K4, H3K9, H3K27, H3K79 and H4K20 in NPC cells.
nasopharyngeal carcinoma; histone methyltransferase; SET domain-containing protein 4; cell proliferation; cell migration; gene set enrichment
R363.2; R739.6
A
10.3969/j.issn.1000-4718.2023.02.008
1000-4718(2023)02-0259-10
2022-07-15
2022-11-29
[基金项目]广东省扬帆计划高层次人才项目(No. 4YF16007G)
申志华 Tel: 0759-2388587; E-mail: szh75@126.com; 揭伟 Tel: 0898-66968217; E-mail: wei_jie@hainmc.edu.cn
▲并列共同第一作者:第一作者
(责任编辑:余小慧,李淑媛)
