小麦转录因子TaMYB5-3B与株高和千粒重相关
2023-02-27李超男毛新国郝晨阳王景一常建忠景蕊莲
朱 治 李 龙 李超男 毛新国 郝晨阳 朱 婷 王景一,* 常建忠 景蕊莲
小麦转录因子TaMYB5-3B与株高和千粒重相关
朱 治1,2李 龙2李超男2毛新国2郝晨阳2朱 婷2王景一2,*常建忠1,*景蕊莲2
1山西农业大学山西有机旱作农业研究院 / 有机旱作山西省重点实验室 / 省部共建有机旱作国家重点实验室(筹), 山西太原 030031;2中国农业科学院作物科学研究所, 北京 100081
MYB转录因子在植物生长发育过程中发挥着重要作用。本研究克隆了小麦3B染色体上的-基因, 基因组序列全长3005 bp, 其中编码区上游为2112 bp, 编码区为893 bp, 包含2个外显子和1个内含子, 编码一个R2R3-MYB蛋白。序列多态性分析表明, 在-的–2048、–1632、–1178、–1156、–504、–461、–433和61 bp处各有1个SNP位点, 分别是G/A转换、G/A转换、G/A转换、T/C转换、C/T转换、A缺失、T缺失和T/A颠换, 这8个SNP位点连锁。基于启动子区SNP-1632的变异开发分子标记, 检测小麦自然群体的基因型, 与表型性状进行关联分析, 结果显示-与株高、穗下节长和千粒重显著相关。-在群体中有2种单倍型-3B-1和-3B-2, 其中-3B-2是植株较矮、千粒重较高的优异单倍型。在我国的小麦育种历程中-3B-2受到了正向选择, 在育成品种中的频率逐步增加, 但仍然有进一步的应用潜力。研究结果为深入探讨小麦株高和产量的形成机制提供参考, 也为小麦株型和产量分子育种提供了基因资源与选择标记。
分子标记; 株高; 千粒重; 单倍型; 关联分析; 小麦
小麦是世界的主要粮食作物, 在我国乃至全球的粮食安全中具有重要作用。随着人口的快速增长、耕地面积减小, 小麦供不应求[1]。为解决这一问题, 从小麦丰富的种质资源中发掘优异基因, 借助分子标记培育高产稳产广适新品种, 是提高小麦生产力保障粮食安全的有效途径。MYB超家族广泛存在于各种植物中, 是一类功能丰富的转录因子, 其成员特征为包含高度保守的DNA结合结构域(MYB结构域), 基于结构域的不完全重复序列(R)数量, MYB家族划分为1R-MYB、R2R3-MYB、3R-MYB和4R-MYB四类[2]。在植物中发现的第1个MYB转录因子是[3], 其参与玉米籽粒花青素的合成,随后在拟南芥、玉米、水稻、小麦等植物中相继报道了MYB转录因子[2]。迄今为止, 在拟南芥、水稻、玉米等植物中已有大量的相关研究, 但是关于小麦MYB转录因子的研究相对较少。
MYB转录因子参与植物逆境响应和生长发育等许多重要过程[4-5], 在水稻、大豆、拟南芥和葡萄等植物中MYB转录因子参与各种生长发育过程, 例如大豆过表达能够提早开花和成熟, 并通过促进基因的表达增加株高[6]。过表达能够改变细胞周期进程抑制根生长发育[7]。MYB73/MYB77能够与UV-B光感受器UVR8互作, 调节生长素反应和侧根发育[8]。拟南芥在干旱胁迫下通过整合脱落酸-生长素信号激活侧根分生组织[9]。水稻编码的MYB蛋白能够调节花器官和小穗分生组织发育[10]。百合和基因可决定花色素苷的器官和组织特异性积累[11]。MYB转录因子在籽粒发育过程中也发挥重要作用, 如水稻MYB转录因子通过调节的表达控制籽粒形成, 从而控制粒数和穗结构发育, 穗长的有利等位基因RGN1有助于穗结构的改造[12]。MYB蛋白AH2能够控制水稻谷壳和籽粒的发育[13]。MYB基因过表达导致水稻株高和籽粒产量的增加, 敲除基因导致相反的表型[14]。MYB转录因子还决定果皮的颜色, 例如的等位变异导致葡萄果皮颜色改变[15], 促进花青素积累的等位基因有助于果皮颜色的改造[16]。
MYB转录因子在植株生长发育中发挥重要作用, 然而与小麦重要农艺性状相关的MYB优异等位基因的发掘与相关标记的开发鲜见报道。研究表明, MYB转录因子TaPHR3-A1控制小麦的生物量、粒数和小穗数, 优异等位基因--可提高小麦粒数[17]。发掘优异基因并开发分子标记, 将有效提高优良基因聚合效率, 加速作物育种进程[18-20]。本研究克隆了小麦-基因, 基于其序列多态性位点开发功能分子标记, 明确了该基因的优异等位变异, 以及其在我国育种历史中的应用情况, 为小麦遗传改良提供了基因资源和分子标记。
1 材料与方法
1.1 试验材料及表型鉴定
本研究以小麦品种旱选10号为材料克隆目标基因; 用32份高多态性的小麦材料(表1)分析基因核苷酸序列多态性, 该群体是用209对SSR引物对262份小麦材料进行多态性分析后筛选所得[21]; 用3个小麦群体进行基因单倍型分析和表型-基因型关联分析。群体1是由323份小麦材料组成的自然群体[22], 包括12份地方品种、36份改良品系和275份育成品种, 用于表型-基因型的关联分析; 群体2由157份地方品种组成, 群体3由348份现代育成品种组成[23], 这两个群体的材料均来自我国的10个小麦生态区。群体1在3个生长季(2014—2015、2015—2016、2016—2017)种植于北京顺义(40°23'N, 116°56'E)和昌平(40°13'N, 116°13'E), 设干旱(drought stress, DS)和灌溉(well-watered, WW) 2种水分处理, 以及热处理(heat stress, HS)[22]。DS为全生长期雨养, 3个生长季的总降水量分别为161、173和143 mm; WW分别在越冬前、开花期和灌浆期进行灌溉, 灌溉量为750 m3hm–2(75 mm)。HS是在花后1周用聚乙烯塑料膜覆盖的温室模拟高温。群体2和群体3于2002、2005年种植于河南洛阳(34°61'N, 112°45'E), 2010年种植于北京顺义。每份材料种4行, 小区行距30 cm, 行长2 m, 每行点播40粒种子。成熟期从小区中间2行随机取5株考察株高、穗长、穗下节长、单株产量、每穗小穗数和千粒重等农艺性状。
表1 32份高多态性小麦材料的信息
1.2 引物设计及数据分析
从NCBI数据库中获得-参考序列, 用Primer Premier 5.0软件设计引物; 利用dCAPS Finder 2.0网站(http://helix.wustl.edu/dcaps/)开发分子标记; SeqMan 7.1软件进行基因序列的比对拼接; 利用DNAMAN 6软件进行蛋白序列比对; TraesCS3B02G290600蛋白序列为参考, 在NCBI中BLASTP分析, 检索其他物种的直系同源蛋白, 利用MEGA 7.0软件进行系统进化树构建; TASSEL 5.0和SPSS 16.0软件进行关联分析和显著性分析。本研究所用引物见表2。
1.3 TaMYB5-3B的克隆与序列多态性分析
以小麦品种旱选10号为材料克隆基因, 用高保真DNA聚合酶TransStart FastPfu进行PCR扩增。PCR反应体系为20 μL, 包括Pfu buffer (5×) 4 μL、dNTP (2.5 μmol L–1) 1.6 μL、Fast Pfu DNA酶0.4 μL、TaMYB5-3B-cDNA-F/R引物(10 μmol L–1)各0.5 μL、模板cDNA (100 ng μL–1) 1 μL, 以及ddH2O 12 μL。PCR扩增程序为95℃ 2 min; 95℃ 30 s, 60℃ 30 s, 72℃ 30 s, 40个循环; 72℃ 10 min。

表2 本研究所用引物
以32份高多态性小麦材料的基因组DNA为模板, 用高保真DNA聚合酶TransStart FastPfu进行PCR扩增。PCR反应体系为20 μL, 包括Pfu buffer (5×) 4 μL、dNTP (2.5 μmol L–1) 1.6 μL、TaMYB5-3B- F/R引物(10 μmol L–1)各0.5 μL、Fast Pfu DNA酶0.4 μL、模板DNA (50 ng μL–1) 1 μL, 以及ddH2O 12 μL。PCR扩增程序为95℃ 2 min; 95℃ 30 s, 60℃ 30 s, 72℃ 2 min, 35个循环; 72℃ 10 min。利用1.2%琼脂糖凝胶电泳分离PCR产物, 回收纯化目标片段, 测序。利用SeqMan7.1软件比对拼接测序序列, 分析-基因的序列多态性。
1.4 分子标记开发
基于-启动子区−1632 bp的SNP (G/A)开发--dCAPS标记, 通过错配碱基(T到C)产生II限制性酶切位点以区分G/A基因型。通过基因组特异性引物--F/--R获得一轮PCR产物, 产物稀释50倍后作为二轮PCR的模板。二轮PCR产物用限制性内切酶II在37℃酶切2 h, 之后利用4%的琼脂糖凝胶电泳分离目的片段。
1.5 关联分析
利用分子标记dCAPS-1632检测群体1材料的基因型。采用TASSEL 5.0软件的一般线性模型(GLM), 将群体结构(Q)作为协变量[22], 对-的基因型和农艺性状进行关联分析, 以< 0.05作为相关显著性阈值。用SPSS 16.0软件进行显著性分析, 检测不同单倍型对农艺性状的效应。
2 结果与分析
2.1 小麦TaMYB5-3B基因的克隆与蛋白序列分析
以旱选10号为材料克隆候选基因的基因组序列。结果表明, 基因序列全长3005 bp, 包含编码区上游2112 bp和编码区893 bp, 其编码区含2个外显子和1个内含子, 编码272个氨基酸, 含有R2R3-MYB基因家族成员的典型结构, 即R2-HTH和R3-HTH的MYB结构域(图1-A), 这2个结构域组成MYB的DNA结合结构域。序列比对表明TraesCS3B02G290600蛋白序列与MYB家族成员具有较高同源性。进化分析显示TaMYB与其他物种中的MYB5亲缘关系较近, 因此命名为TaMYB5-3B (图1-B)。

(图1)
A: TaMYB5氨基酸序列比对图。黑线指示R2和R3结构域; B: TaMYB5蛋白进化树。TaMYB5-3B用红点标注, Ta: 小麦; Td: 野生二粒小麦; Hv: 大麦; Bd: 二穗短柄草; Sb: 高粱; Zm: 玉米; Si: 谷子; Ph: 哈氏黍; Pv: 柳枝稷; Ob: 短花药野生稻; Os: 水稻。
A: TaMYB5 sequence alignment. The R2 and R3 structure domains are marked with black underlines; B: phylogenetic tree of TaMYB5 proteins. TaMYB5-3B is marked with a red dot. Ta:; Td:; Hv:; Bd:; Sb:; Zm:; Si:; Ph:; Pv:; Ob:; Os:.
2.2 序列多态性分析与分子标记开发
利用32份高多态性小麦材料分析-序列多态性, 在–2048、–1632、–1178、–1156、–504、–461、–433和61 bp处各检测到1个SNP位点, 分别是G/A转换、G/A转换、G/A转换、T/C转换、C/T转换、A缺失、T缺失和T/A颠换, 编码区61 bp处的碱基变异引起氨基酸变异(图2-A)。对小麦群体中的-多态性分析显示, 8个SNP位点连锁, 组成2种单倍型-3B-1和-3B-2。基于启动子区SNP-1632的碱基差异, 通过dCAPS Finder2.0网站设计分子标记dCAPS-1632, 引入1个错配碱基C, 形成II酶切位点(A▼GATCT)以区分2种单倍型(图2-B)。经过两轮PCR反应的产物用II酶切后电泳检测, 若–1632 bp位点为G时, PCR产物不被酶切, 片段长度为237 bp; 若–1632 bp位点为A时, PCR产物被酶切, 产生2个片段216 bp和21 bp (图2-C), 利用dCAPS-1632标记可以区分-的2种单倍型。
2.3 分子标记与农艺性状的关联分析
用-分子标记dCAPS-1632检测小麦群体1中323份材料的基因型, 然后与16种环境(年份×地点×处理组合)的农艺性状进行关联分析。结果显示, 该标记与株高和千粒重显著关联, 在15种环境条件下与株高显著关联, 在12种环境条件下与穗下节长显著关联, 在11种环境条件下与千粒重显著关联(表3)。
2.4 TaMYB5-3B两种单倍型的农艺性状比对
对小麦群体1基因型的分析显示, 323份材料中单倍型-3B-1、-3B-2的占比分别为53.56%和46.44%。在16种环境条件下, 具有单倍型- 3B-2的材料, 其株高均显著低于-3B-1 (图3-A), 平均降低13.11%;-3B-2的穗下节长均显著低于-3B-1 (图3-B), 平均降低10.17%; 与之相反, 16种环境条件下, 具有-3B-2的材料, 其千粒重均显著高于-3B-1 (图3-C),-3B-2千粒重平均较-3B-1高2.49 g。同时, 我们也分析了来自于我国十大麦区的地方品种(群体2)和育成品种(群体3)的组合群体, 得到了相似的结果。在3种环境条件下,具有-3B-2的材料, 其株高均显著低于-3B- 1,千粒重均显著高于-3B-1 (图3-D)。以上结果表明, 单倍型-3B-2是矮秆、高千粒重的优异单倍型。

图2 TaMYB5-3B的核苷酸多态性和分子标记开发
A:-的结构示意图与多态性位点, 红色字母表示设计分子标记的SNP位点; B: 分子标记dCAPS-1632的开发, 红框和红点代表通过碱基T错配为C引入II酶切位点, 红色字母代表2种单倍型碱基差异; C: PCR产物用II酶切的结果。M: 100 bp DNA ladder。
A: the schematic diagram and polymorphism sites of-. Red letter indicates the SNP that is designed for molecular marker. B: the development of the molecular marker dCAPS-1632, red rectangle and dot represent an introduction of theII restriction site by a base mismatched (T to C), and red letters represent two haplotype base differences. C: PCR products were digested byII enzyme. M: 100 bp DNA ladder.
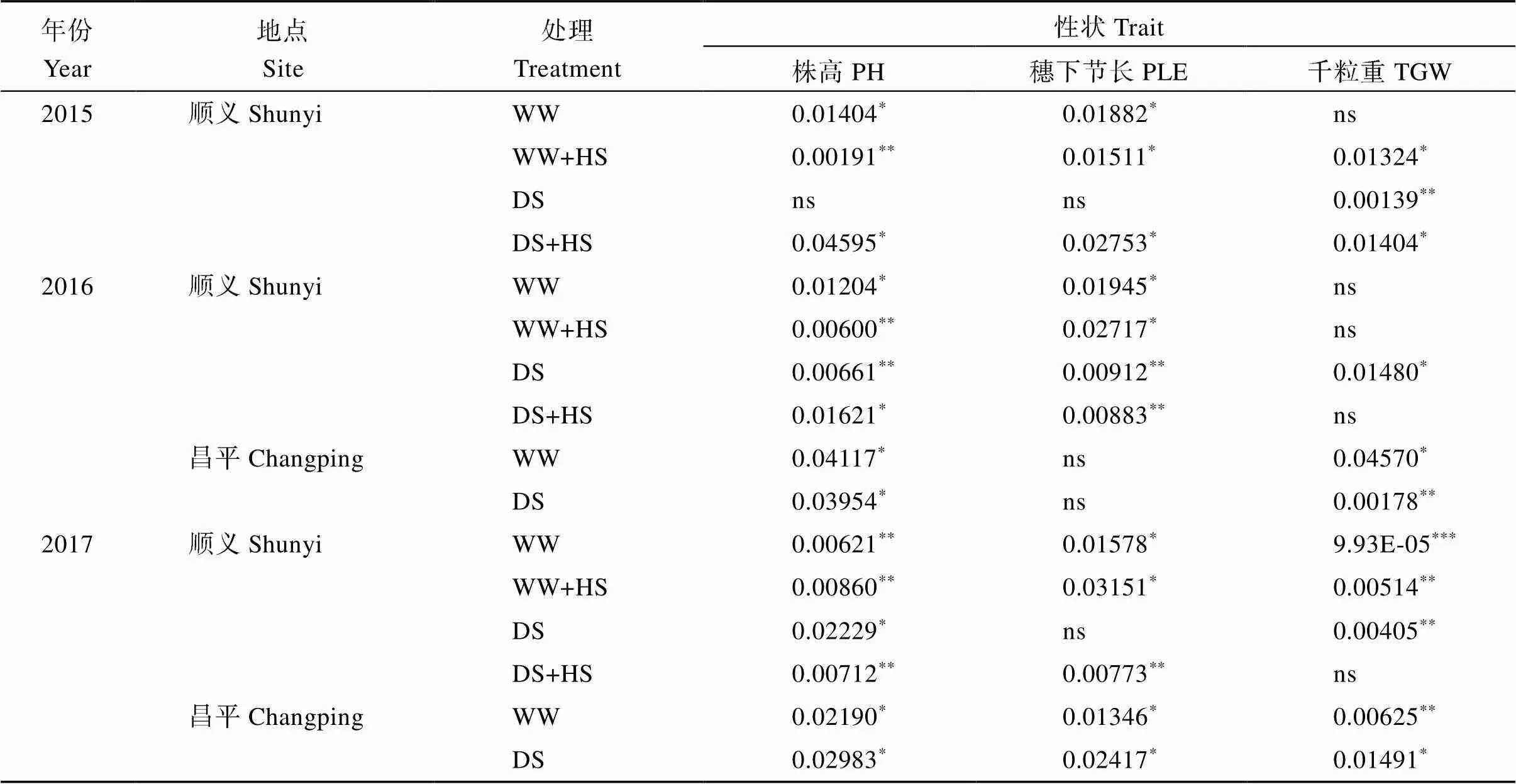
表3 小麦dCAPS-1632标记与农艺性状关联分析
WW: 灌溉; DS: 干旱胁迫; HS: 热胁迫; PH: 株高; PLE: 穗下节长; TGW: 千粒重;*:< 0.05;**:< 0.01;***:< 0.001; ns: 差异不显著。
WW: well-watered; DS: drought stress; HS: heat stress; PH: plant height; PLE: peduncle length; TGW: 1000-grain weight.*:< 0.05;**:< 0.01;***:< 0.001; ns: not significant.
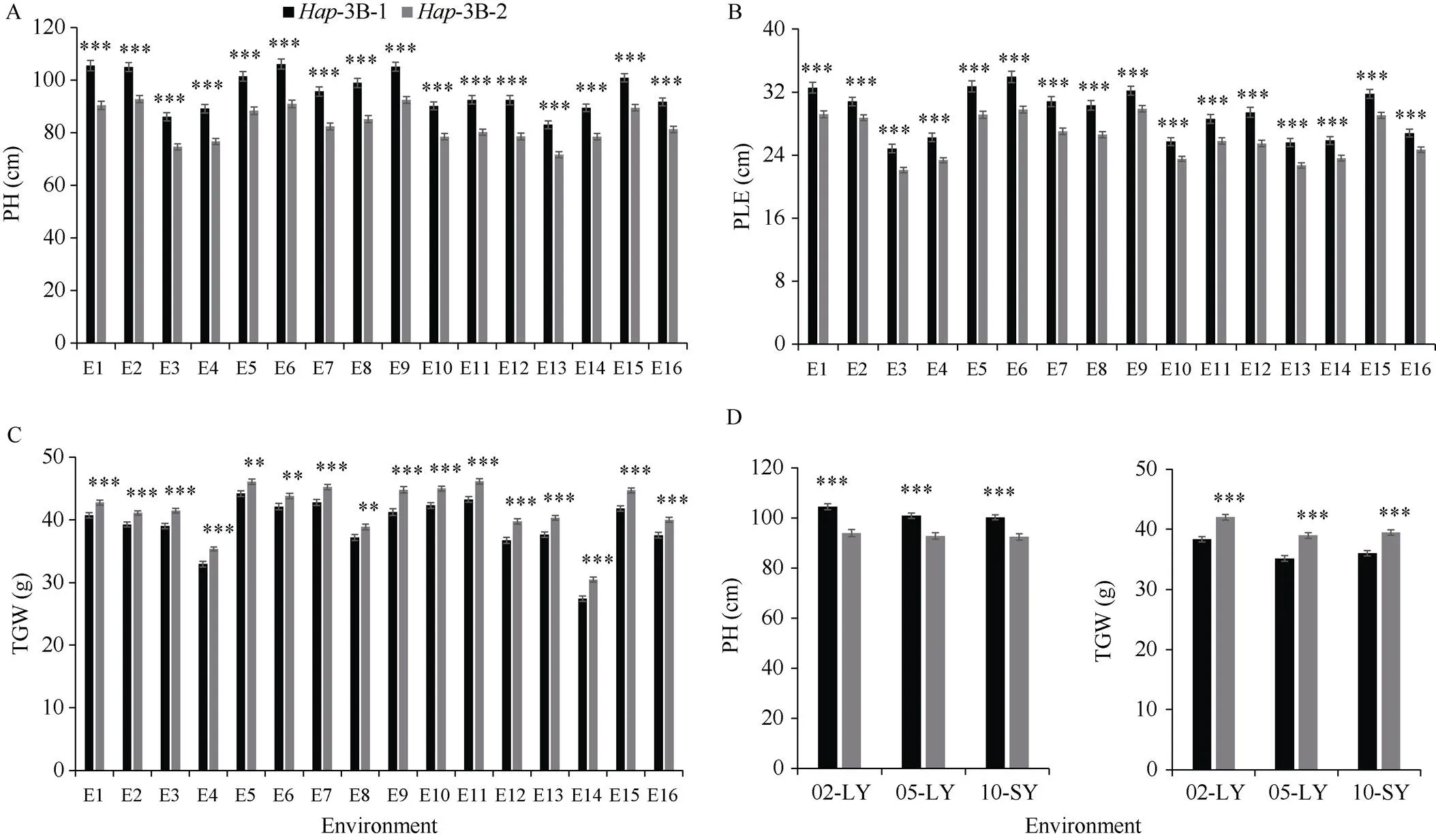
图3 TaMYB5-3B两种单倍型的农艺性状对比
A~C:-的2种单倍型在16种环境中株高(A)、穗下节长(B)和千粒重(C)的比较; D: 2种单倍型在3种环境中株高和千粒重的比较。E: 环境, E1: 15-SY-WW; E2: 15-SY-WW-HS; E3: 15-SY-DS; E4: 15-SY-DS-HS; E5: 16-SY-WW; E6: 16-SY-WW-DS; E7: 16-SY-DS; E8: 16-SY-DS-HS; E9: 16-CP-WW; E10: 16-CP-DS; E11: 17-SY-WW; E12: 17-SY-WW-HS; E13: 17-SY-DS; E14: 17-SY-DS-HS; E15: 17-CP-WW; E16: 17-CP-DS。15: 2015; 16: 2016; 17: 2017; 02: 2002; 05: 2005; 10: 2010。SY: 顺义; CP: 昌平; LY: 洛阳。WW: 水分充足; DS: 干旱胁迫; HS: 高温胁迫。数据显著性采用检验。**< 0.01; ***< 0.001。误差值: ±SE。
A–C: PH (A), PLE (B), and TGW (C) comparisons of two-haplotypes in 16 environments; D: PH and TGW comparisons of two-haplotypes in three environments. E: environment; E1: 15-SY-WW; E2: 15-SY-WW-HS; E3: 15-SY-DS; E4: 15-SY-DS-HS; E5: 16-SY-WW; E6: 16-SY-WW-DS; E7: 16-SY-DS; E8: 16-SY-DS-HS; E9: 16-CP-WW; E10: 16-CP-DS; E11: 17-SY-WW; E12: 17-SY-WW-HS; E13: 17-SY-DS; E14: 17-SY-DS-HS; E15: 17-CP-WW; E16: 17-CP-DS. 15: 2015; 16: 2016; 17: 2017; 02: 2002; 05: 2005; 10: 2010. SY: Shunyi; CP: Changping; LY: Luoyang. WW: well-watered; DS: drought stress; HS: heat stress. Significance of data is tested by Student’s-test. **:< 0.01; ***:< 0.001. Error bar: ±SE.
2.5 TaMYB5-3B2种单倍型的频率与地理分布
为了研究-单倍型在小麦育种进程中受选择的情况, 我们分析了小麦群体2和群体3中-2种单倍型在我国十大麦区的分布频率。在群体2的地方品种中, 具有单倍型-3B-2的材料占22.88%, 其中新疆冬春麦区(X)-3B-2的比例较高, 达66.67%, 其余9个麦区中-3B-2的比例均低于-3B-1, 尤其是青藏冬春麦区(IX), 所有的材料均为-3B-1 (图4-A)。在群体3的现代育成品种中, 具有单倍型-3B-2的材料占53.37%,与地方品种相比,-3B-2的频率大幅提高, 特别是在小麦主产区, 黄淮冬麦区(II)、长江中下游麦区(III)、西南冬麦区(IV), 以及东北春麦区(VI)和青藏冬春麦区(IX),-3B-2占比均超过-3B-1 (图4-B)。可见, 从地方品种到现代育成品种, 除麦区X外, 其余麦区单倍型-3B-2的频率均大幅增加(图4)。这表明在我国的小麦育种历史中矮秆、粒重的优异单倍型-3B-2受到了育种家的正向选择。

图4 TaMYB5-3B两种单倍型的频率与分布
A~B:-两种单倍型在中国10个小麦产区的157个地方品种(A)和348个现代育成品种(B)中的分布。I: 北方冬麦区; II: 黄淮冬麦区; III: 长江中下游麦区; IV: 西南冬麦区; V: 华南冬麦区; VI: 东北春麦区; VII: 北部春麦区; VIII: 西北春麦区; IX: 青藏春冬麦区; X: 新疆冬春麦区。
A–B: the distribution of two-haplotypes in 157 landraces (A) and 348 modern cultivars (B) from 10 Chinese wheat production zones. I: Northern Winter Wheat Zone; II: Yellow-Huai River Valleys Facultative Wheat Zone; III: Middle and Low Yangtze Valleys Autumn-Sown Spring Wheat Zone; IV: Southwestern Autumn-Sown Spring Wheat Zone; V: Southern Autumn-Sown Spring Wheat Zone; VI: Northeastern Spring Wheat Zone; VII: Northern Spring Wheat Zone; VIII: Northwestern Spring Wheat Zone; IX: Qinghai-Tibetan Plateau Spring-Winter Wheat Zone; X: Xinjiang Winter-Spring Wheat Zone.
2.6 TaMYB5-3B两种单倍型在不同年代品种中的分布频率
根据育成年代将群体3的348个现代育成品种分为6组, 每10年为一组, 以观察随着育种年代推移-单倍型的变化趋势。从1950年以前到1990s年代, 我国育成品种的平均株高从110.51 cm降低到80.62 cm, 千粒重从34.29 g增加到43.07 g, 与此同时, 矮秆、粒重优异单倍型-3B-2的比例从22.22%逐步增加到57.14%, 而高秆小粒单倍型-3B-1的比例从77.78%下降到42.86% (图5), 这表明在我国小麦育种中-3B-2受到了育种家的正向选择。尽管如此, 现代育成品种中优异单倍型-3B-2的频率仍然较低, 还有进一步的育种应用潜力。
3 讨论
MYB转录因子是植物中功能强大的超家族转录因子, 近年的研究表明, MYB转录因子在植物生长发育过程中发挥着重要的调控作用[2,4]。株高和籽粒大小是影响作物产量的关键性状[24-26], 但是对于小麦MYB转录因子调控株高和千粒重的研究较少, 尤其是关于其优异等位基因发掘与标记开发的研究鲜有报道。本文通过关联分析发现-与株高、穗下节长和千粒重显著或极显著关联, 表明-参与了对小麦株高和粒重性状的调控。其他植物中也有类似研究报道, 例如拟南芥过表达导致多效性表型, 包括植株矮化, 花器官发育缺陷等[27]。棉花在拟南芥中异源表达, 明显减少叶片表皮毛, 使花器官发育异常, 并产生严重的矮化现象[28]。大豆过表达改变植株结构, 表现为侧枝增加和株高降低[29]。另外两个大豆MYB基因[6]和[30]通过赤霉素途径调控株高, 其中受赤霉素诱导上调表达, 促进表达, 增加株高;则直接或间接地提高GA合成基因和GA反应相关基因的表达水平来调节株高。水稻MYB转录因子能够调控多个细胞伸长和细胞壁合成基因的表达, 增加株高和籽粒产量[14]。小麦TaMYB70通过与基因-1B启动子区MYB识别序列结合提高其表达水平, 进而提高抗旱性及千粒重[31]。
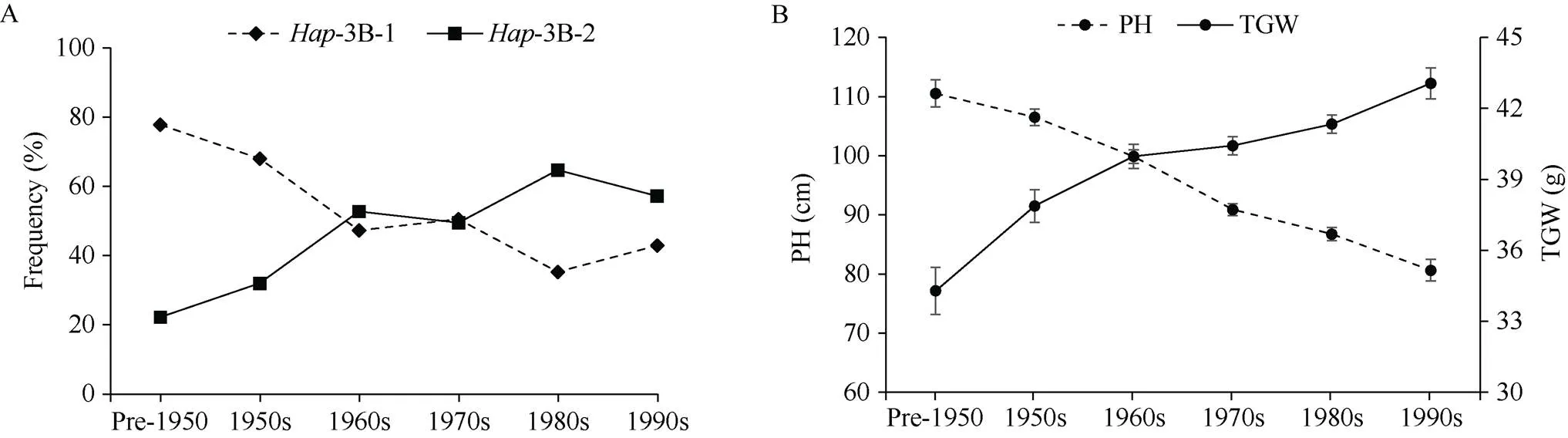
图5 我国348个育成品种中TaMYB5-3B单倍型频率以及PH和TGW变化
A: 随年代推进两个单倍型-3B-1和-3B-2在群体3中的频率变化; B: 随年代推进PH和TGW在群体3中的变化。误差值: ±SE。PH和TGW的数据来自Hao等[23]。
A: the frequency variation of-3B-1 and-3B-2 in Population 3 over decades; B: the changes of PH and TGW in Population 3 over decades. Error bar: ±SE. PH and TGW data are obtained from Hao et al.[23]
株高和千粒重是影响作物产量的关键性状。研究者通过QTL (quantitative trait locus)遗传定位和GWAS (genome-wide association study)分析, 已经在小麦染色体3B上发现了与株高或千粒重相关的基因组区间。例如, Carter等[32]在染色体3B上定位到3个调控株高的相邻QTL,-、-和-。Liu等[33]利用重组自交(RIL)群体在3B上定位到3个调控株高的, 1个调控千粒重的。Cui等[34]利用RIL群体定位到3个调控千粒重的QTL, 均位于3B染色体长臂上, 其中是解释表型变异大于10%的大效应稳定QTL。此外, Janice等[35]在3B染色体短臂和长臂上各检测到一个调控千粒重的QTL, 分别为和。本文研究发现与株高和千粒重显著关联, 并位于3B染色体长臂上, 推测可能与前人定位到的这些性状的QTL区间相关。另外, 我们分析了小麦基因组联合变异数据库(http:// wheat.cau.edu.cn/WheatUnion/)的重测序数据[36-37], 虽然此数据库中没有检测到的InDel变异, 但另外6个SNP变异与本研究结果相吻合, 且这6个变异位点连锁, 小麦材料也相应地分为2种单倍型。
根据前人研究, 碱基序列变异导致表型差异的原因至少有2种, 一种是基因启动子区的变异导致表型差异, 另外一种是编码区的非同义突变引起的表型差异。在启动子区变异导致表型差异方面, 例如小麦-有2种单倍型, 其中-4A-2是高千粒重的优异单倍型, 就是由于启动子区TaERF3结合位点的突变导致了两者的表型差异[38]; 小麦-两种单倍型-2A-1和-2A-2的株高和穗下节长表现显著差异, 是由于启动子区ARFs结合位点的变异导致的[39]。本研究在小麦种质资源中检测到-的2种单倍型-3B-1和-3B-2, 其中-3B-2是与矮秆、粒重相关的优异单倍型, 在2种单倍型的启动子区发现了7个SNP差异, 推测可能是2种单倍型启动子区相关元件出现突变, 并引起表型的差异。关于编码区非同义突变引起表型变异, 也有许多研究报道, 例如苹果MdCIPK22可通过磷酸化MdSUT2.2 Ser381位点促进糖分积累、降低丙二醛含量进而提高植株抗旱性[40]。稻瘟病菌MoSom1 Ser227的磷酸化对其致病力至关重要, Ser227的突变导致孢子形成和真菌致病的缺陷[41]。本研究的TaMYB5-3B为MYB转录因子, 其编码区的SNP 61位点变异导致第21位的丝氨酸转变为苏氨酸, 推测该变化可能引起激酶特异性磷酸化位点的更改, 导致上游蛋白激酶对TaMYB5-3B的磷酸化作用发生改变, 最终导致表型差异。-启动子区和编码区的变异都可能导致2种单倍型对应表型的差异, 后期仍需进一步深入研究以探明其作用机制。
在作物育种过程中, 优异等位变异或单倍型会受到育种家有意或无意的正向选择, 使其在现代育成品种中的频率不断提高。例如小麦的单倍型-6D-3是穗长和每穗小穗数高的最优单倍型, 在我国小麦育种进程中受到了正向选择[21]。-与小麦的根深和根角度、株高和千粒重相关, 在我国小麦育种历程中育种家根据生产需求, 不断选择了矮秆粒重的优异单倍型-4A-2[42]。小麦SBP-Box基因-同时调控株高和籽粒发育, 其矮秆粒重的优异单倍型III虽然也受到了选择, 但是在近年育成的品种中其最高频率仍然不足20%, 表明其在今后的育种工作中具有很大的提升空间[43]。本研究比较了地方品种和育成品种中-优异单倍型-3B-2的频率, 在我国大部分麦区其频率均大幅增加; 且随着育种年代的推进育成品种中优异单倍型-3B-2的频率不断增加, 从1950年之前的22.22%增加到1990s年代的57.14%, 表明优异单倍型-3B-2在育种过程中受到了正向选择。尽管如此,-3B-2在现代育成品种中所占比例仍然较低, 还有较大的应用潜力。
4 结论
本研究克隆了小麦3B染色体上的-基因, 在其编码区和启动子区共检测到8个SNP位点, 小麦自然群体中存在2种单倍型-3B-1和-3B-2。根据基因启动子区SNP-1632位点开发的分子标记dCAPS-1632与株高、穗下节长和千粒重显著相关,-3B-2是矮秆粒重的优异单倍型, 在我国小麦育种进程中受到正向选择, 即随着育种年代推进优异单倍型的频率逐步增加, 但仍然具有较大的应用潜力。
[1] Ray D K, Mueller N D, West P C, Foley J A, Hart J P. Yield trends are insufficient to double global crop production by 2050., 2013, 8: e66428.
[2] Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L. MYB transcription factors in., 2010, 15: 573–581.
[3] Paz-Ares J, Ghosal D, Wienand U, Peterson P A, Saedler H. The regulatorylocus ofencodes a protein with homology to MYB-related proto-oncogene products and with structural similarities to transcriptional activators., 1987, 6: 3553–3558.
[4] Li J L, Han G L, Sun C F, Sui N. Research advances of MYB transcription factors in plant stress resistance and breeding., 2019, 14: 1–9.
[5] 陈清, 汤浩茹, 董晓莉, 侯艳霞, 罗娅, 蒋艳, 黄琼瑶. 植物MYB转录因子的研究进展. 基因组学与应用生物学, 2009, 28: 365–372.
Chen Q, Tang H R, Dong X L, Hou Y X, Luo Y, Jiang Y, Huang Q Y. Progress in the study of plant MYB transcription factors., 2009, 28: 365–372 (in Chinese with English abstract).
[6] Yang X, Li X, Shan J M, Li Y H, Zhang Y T, Wang Y H, Li W B, Zhao L. Overexpression ofaccelerates the transition to flowering and increases plant height in soybean., 2021, 12: 667242.
[7] Mu R L, Cao Y R, Liu Y F, Lei G, Zou H F, Liao Y, Wang H W, Zhang W K, Ma B, Du J Z. An R2R3-type transcription factor generegulates root growth and cell cycle progression in., 2009, 19: 1291–1304.
[8] Yang Y, Zhang L B, Chen P, Liang T, Li X, Liu H T. UV-B photoreceptor UVR8 interacts with MYB73/MYB77 to regulate auxin responses and lateral root development., 2020, 39: e101928.
[9] Seo P J, Xiang F, Qiao M, Park J Y, Park C M. The MYB96 transcription factor mediates abscisic acid signaling during drought stress response in., 2009, 151: 275–289.
[10] Li Y F, Zeng X Q, Li Y, Wang L, Zhuang H, Wang Y, Tang J, Wang H L, Xiong M, Yang F Y. MULTI-FLORET SPIKELET 2, a MYB transcription factor, determines spikelet meristem fate and floral organ identity in rice., 2020, 184: 988–1003.
[11] Yamagishi M, Shimoyamada Y, Nakatsuka T, Masuda K. Two R2R3-MYB genes, homologs of petunia, regulate anthocyanin bio-syntheses in flower tepals, tepal spots and leaves of Asiatic hybrid lily., 2010, 51: 463–474.
[12] Li G L, Xu B X, Zhang Y P, Xu Y W, Khan N U, Xie J Y, Sun X M, Guo H F, Wu Z Y, Wang X Q, Zhang H L, Li J J, Xu J L, Wang W S, Zhang Z Y, Li Z C.controls grain number and shapes panicle architecture in rice., 2021, 20: 158–167.
[13] Ren D Y, Cui Y J, Hu H T, Xu Q K, Rao Y C, Yu X Q, Zhang Y, Wang Y X, Peng Y L, Zeng D L, Hu J, Zhang G H, Gao Z Y, Zhu L, Chen G, Shen L, Zhang Q, Guo L B, Qian Q.encodes a MYB domain protein that determines hull fate and affects grain yield and quality in rice., 2019, 100: 813–824.
[14] Zhang Y X, Yu C S, Lin J Z, Liu J, Liu B, Wang J, Huang A, Li H Y, Zha T.regulates plant height and improves grain yield in rice., 2017, 12: e0180825.
[15] Azuma A, Udo Y, Sato A, Mitani N, Kono A, Ban Y, Yakushiji H, Koshita Y, Kobayashi S. Haplotype composition at the color locus is a major genetic determinant of skin color variation ingrapes., 2011, 122: 1427–1438.
[16] Jiu S T, Guan L, Leng X P, Zhang K K, Haider M S, Yu X, Zhu X D, Zheng T, Ge M Q, Wang C, Jia H F, Shang-Guan L F, Zhang C X, Tang X P, Abdullah M, Javed H U, Han J, Dong Z G, Fang J G. The role ofandalleles of thelocus in the regulation of anthocyanin biosynthesis for molecular breeding of grape (spp.) skin coloration., 2021, 19: 1216–1239.
[17] Zheng X W, Liu C, Qiao L, Zhao J J, Han R, Wang X L, Ge C, Zhang W Y, Zhang S W, Qiao L Y, Zheng J, Hao C Y. The MYB transcription factor TaPHR3-A1 is involved in phosphate signaling and governs yield-related traits in bread wheat., 2020, 71: 5808–5822.
[18] Liu Y N, He Z H, Appels R, Xia X C. Functional markers in wheat: current status and future prospects., 2012, 125: 1–10.
[19] Kage U, Kumar A, Dhokane D, Karre S, Kushalappa A C. Functional molecular markers for crop improvement., 2016, 36: 917–930.
[20] Salgotra R K, Stewart C N Jr. Functional markers for precision plant breeding., 2020, 21: 4792.
[21] 张宏娟, 李玉莹, 苗丽丽, 王景一, 李超男, 杨德龙, 毛新国, 景蕊莲. 小麦转录因子基因参与调控穗长和每穗小穗数. 作物学报, 2019, 45: 1615–1627.
Zhang H J, Li Y Y, Miao L L, Wang J Y, Li C N, Yang D L, Mao X G, Jing R L. Transcription factor geneinvolved in regulation spike length and spikelet number per spike in common wheat., 2019, 45: 1615–1627 (in Chinese with English abstract).
[22] Li L, Mao X G, Wang J Y, Chang X P, Reynolds M, Jing R L. Genetic dissection of drought and heat-responsive agronomic traits in wheat., 2019, 42: 2540–2553.
[23] Hao C Y, Wang L F, Ge H M, Dong Y C, Zhang X Y. Genetic diversity and linkage disequilibrium in Chinese bread wheat (L.) revealed by SSR markers., 2011, 6: e17279.
[24] Cao S H, Xu D G, Hanif M, Xia X C, He Z H. Genetic architecture underpinning yield component traits in wheat., 2020, 133: 1811–1823.
[25] Khobra R, Sareen S, Meena B K, Kumar A, Tiwari V, Singh G P. Exploring the traits for lodging tolerance in wheat genotypes: a review., 2019, 25: 589–600.
[26] Foulkes M, Slafer G, Davies W, Berry P, Sylvester-Bradley R, Martre P, Calderini D F, Griffiths S, Reynolds M P. Raising yield potential of wheat: III. Optimizing partitioning to grain while maintaining lodging resistance., 2011, 62: 469–486.
[27] Yang X Y, Li J G, Pei M, Gu H, Chen Z L, Qu L J. Over-expression of a flower-specific transcription factor genecauses aberrant anther development., 2007, 26: 219–228.
[28] 王诺菡, 于霁雯, 吴嫚, 马启峰, 李兴丽, 裴文锋, 李海晶, 黄双领, 张金发, 喻树迅. 棉花基因的克隆、表达分析及功能鉴定. 作物学报, 2014, 40: 1540–1548.
Wang N H, Yu J W, Wu M, Ma Q F, Li X L, Pei W F, Li H J, Huang S L, Zhang J F, Yu S X. Cloning, expression, and functional analysis ofgene from cotton (L.)., 2014, 40: 1540–1548 (in Chinese with English abstract).
[29] Yang H, Xue Q, Zhang Z Z, Du J Y, Yu D Y, Fang H. GmMYB181, a soybean R2R3-MYB protein, increases branch number in transgenic., 2018, 9: 1027.
[30] Cheng Q, Dong L D, Su T, Li T Y, Gan Z R, Nan H Y, Lu S J, Fang C, Kong L P, Li H Y, Hou Z H, Kou K, Tang Y, Lin X Y, Zhao X H, Chen L Y, Liu B H, Kong F J. CRISPR/Cas9-mediated targeted mutagenesis ofgenes alters plant height and internode length in soybean., 2019, 19: 562.
[31] Mao H D, Jian C, Cheng X X, Chen B, Mei F M, Li F F, Zhang Y F, Li S M, Du L Y, Li T, Hao C Y, Wang X J, Zhang X Y, Kang Z S. The wheat ABA receptor genecontributes to drought tolerance and grain yield by increasing water-use efficiency., 2022, 20: 846–861.
[32] Carter A H, Garland-Campbell K, Kidwell K K. Genetic mapping of quantitative trait loci associated with important agronomic traits in the spring wheat (L.) cross ‘Louise’ × ‘Penawawa’., 2011, 51: 84–95.
[33] Liu J, Wu B H, Singh R P, Velu G. QTL mapping for micronutrients concentration and yield component traits in a hexaploid wheat mapping population., 2019, 88: 57–64.
[34] Cui F, Zhao C H, Ding A M, Li J, Wang L, Li X F, Bao Y G, Li J M, Wang H G. Construction of an integrative linkage map and QTL mapping of grain yield-related traits using three related wheat RIL populations., 2014, 127: 659–675.
[35] Cuthbert J L, Somers D J, Brule-Babel A L, Brown P D, Crow G H. Molecular mapping of quantitative trait loci for yield and yield components in spring wheat (L.)., 2008, 117: 595–608.
[36] Hao C Y, Jiao C Z, Hou J, Li T, Liu H X, Wang Y Q, Zheng J, Liu H, Bi Z H, Xu F F, Zhao J, Ma L, Wang Y M, Majeed U, Liu X, Appels R, Maccaferri M, Tuberosa R, Lu H F, Zhang X Y. Resequencing of 145 landmark cultivars reveals asymmetric sub-genome selection and strong founder genotype effects on wheat breeding in China., 2020, 13: 1733–1751.
[37] Guo W L, Xin M M, Wang Z H, Yao Y Y, Hu Z R, Song W J, Yu K H, Chen Y M, Wang X B, Guan P F, Appels R, Peng H R, Ni Z F, Sun Q X. Origin and adaptation to high altitude of Tibetan semi-wild wheat., 2020, 11: 5085.
[38] Wang J Y, Wang R T, Mao X G, Zhang J L, Liu Y N, Xie Q, Yang X Y, Chang X P, Li C N, Zhang X Y, Jing R L. RING finger ubiquitin E3 ligase genecontributes to determination of grain size in common wheat., 2020, 71: 5377–5388.
[39] Xue Y H, Wang J Y, Mao X G, Li C N, Li L, Yang X, Hao C Y, Chang X P, Li R Z, Jing R L. Association analysis revealed thatgenes are linked to plant growth related traits in multiple environments., 2021, 12: 641087.
[40] Ma Q J, Sun M H, Lu J, Kang H, You C X, Hao Y J. An apple sucrose transporter MdSUT2.2 is a phosphorylation target for protein kinase MdCIPK22 in response to drought., 2019, 17: 625–637.
[41] Deng S Z, Xu L, Xu Z, Lv W Y, Chen Z X, Yang N, Talbot N J, Wang Z Y. A putative PKA phosphorylation site S227 in MoSom1 is essential for infection-related morphogenesis and pathogenicity in., 2021, 23: e13370.
[42] Zhuang M J, Li C N, Wang J Y, Mao X G, Li L, Yin J, Du Y, Wang X, Jing R L. The wheatgenecontrols root length in an auxin-dependent pathway., 2021, 72: 6977–6989.
[43] Zhang B, Xu W N, Liu X, Mao X G, Li A, Wang J Y, Chang X P, Zhang X Y, Jing R L. Functional conservation and divergence among homoeologs ofand, two SBP-Box genes governing yield-related traits in hexaploid wheat., 2017, 174: 1177–1191.
Transcription factor TaMYB5-3B is associated with plant height and 1000- grain weight in wheat
ZHU Zhi1,2, LI Long2, LI Chao-Nan2, MAO Xin-Guo2, HAO Chen-Yang2, ZHU Ting2, WANG Jing-Yi2,*, CHANG Jian-Zhong1,*, and JING Rui-Lian2
1Shanxi Institute of Organic Dryland Farming, Shanxi Key Laboratory of Organic Dry Farming, State Key Laboratory of Integrative Sustainable Dryland Agriculture (in Preparation), Shanxi Agricultural University, Taiyuan 030031, Shanxi, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
MYB transcription factor plays an important role in plant growth and development. In this study, we cloned-gene on chromosome 3B in wheat. The full-length genome sequence is 3005 bp, including 2112 bp promoter region and 893 bp coding region.-coding region consists of two exons and one intron, which encodes a R2R3-MYB protein. The polymorphism of-was analyzed by sequencing 32 wheat accessions with wide variations. A total of eight SNPs were detected at –2048, –1632, –1178, –1156, –504, –461, –433, and 61 bp, respectively. They were eight SNPs linked by G/A conversion, G/A conversion, G/A conversion, T/C conversion, C/T conversion, A deletion, T deletion and T/A inversion, respectively. A pair of molecular markers were developed based on the promoter region SNP-1632 to detect the genotypes of wheat natural population. The association analysis of genotype and phenotypic traits showed that-was significantly associated with plant height (PH), peduncle length (PLE), and 1000-grain weight (TGW). Two haplotypes (-3B-1 and-3B-2) were detected in the population, in which-3B-2 was an excellent haplotype with short PH and high TGW.-3B-2 had been positively selected in the breeding, and its frequency in modern cultivars gradually increased with the advance of breeding years in China. Therefore,-could be used to further understand the mechanism of wheat plant height and grain yield formation, and its molecular markers may contribute to ideal plant architecture and grain yield breeding of wheat.
molecular markers; plant height; 1000-grain weight; haplotype; association analysis; wheat
10.3724/SP.J.1006.2023.21029
本研究由山西农业大学省部共建有机旱作农业国家重点实验室自主研发项目(202105D121008-2-7)和财政部和农业农村部国家现代农业产业技术体系建设专项(小麦, CARS-03)资助。
This study was supported by the State Key Laboratory of Integrative Sustainable Dryland Agriculture, the Shanxi Agricultural University (202105D121008-2-7), and the China Agriculture Research System of MOF and MARA (Wheat, CARS-03).
常建忠, E-mail: cjzyfx@163.com; 王景一, E-mail: wangjingyi@caas.cn
E-mail: zz1752782610@163.com
2022-04-21;
2022-07-21;
2022-08-29.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220826.1455.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
