橡胶树不同粒径橡胶粒子结合凝集相关蛋白的分析
2023-01-15吴绍华夏志辉史敏晶
丁 欢,吴绍华,夏志辉,黄 惜,史敏晶,*
橡胶树不同粒径橡胶粒子结合凝集相关蛋白的分析
丁 欢1,吴绍华2,夏志辉1,黄 惜1,史敏晶1,2*
1. 海南大学热带作物学院,海南海口 570228;2. 中国热带农业科学院橡胶研究所/农业农村部橡胶树生物学与遗传资源利用重点实验室/省部共建国家重点实验室培育基地-海南省热带作物栽培生理学重点实验室,海南海口 571101
橡胶树是重要的产胶植物,其树皮组织中的次生乳管是合成和贮存天然橡胶的主要组织。天然橡胶是从乳管中的胶乳中提炼而成,由特殊的细胞器-橡胶粒子合成。橡胶粒子为半个单位膜的球状结构,内部贮存合成的天然橡胶,外周膜上结合有多种蛋白质,这些蛋白与其执行的功能密切相关。目前对天然橡胶合成相关蛋白的研究较多,对与橡胶粒子凝集相关的蛋白研究甚少。以‘RY7-33-97’胶乳为材料,利用不同的离心速度,分级分离获取不同粒径的橡胶粒子,并进行不同程度的清洗,以Tricine-SDS-PAGE以及Western-blotting技术对橡胶粒子上结合的蛋白进行比较分析。研究结果表明,不同粒径的橡胶粒子上结合的蛋白含量存在差异。小橡胶粒子膜蛋白(SRPP)随着粒径的减小含量明显增加;而橡胶延伸因子蛋白(REF)的含量基本维持稳定,与粒径大小关系不大。存在于C-乳清中的Hev b7胶乳过敏原蛋白和3-磷酸甘油醛脱氢酶(GAPDH)与橡胶粒子有明显的结合;存在于黄色体B-乳清中的β-1,3-葡聚糖酶(Glu)、橡胶素(Hev)和几丁质酶(Chit)均能与橡胶粒子结合,但几丁质酶与之的结合能力最弱。橡胶粒子经清洗后,作为橡胶粒子的主要膜蛋白,SRPP含量随着清洗次数增加明显降低,而REF蛋白含量变化不明显,可见SRPP与橡胶粒子膜的结合紧密程度不如REF;来自C-乳清和B-乳清的凝集相关蛋白随清洗次数增加也明显降低含量。体外孵育小橡胶粒子与不同的蛋白样品,结果表明,橡胶粒子可体外结合多种不同的蛋白,其中多种蛋白都与橡胶粒子的凝集有关。本研究对不同粒径橡胶粒子上结合的凝集相关蛋白进行了初步解析,旨在为阐明橡胶树排胶机制奠定基础。
橡胶树;橡胶粒子;凝集相关蛋白;蛋白免疫印迹
橡胶树是当前世界上最重要的人工栽培产胶植物,其胶乳主要是由橡胶树树干中的次生乳管合成和贮藏[1]。次生乳管是由维管形成层中的纺锤状原始细胞分裂形成,与形成层呈同心圆状排列,其数量的多少与胶乳的产量密切相关[2-3]。在次生乳管发育过程中,同列(或同层)的乳管细胞之间形成互相融合的接合管,最终使得同一层的乳管细胞形成彼此连通的网状结构[1]。生产中通过割胶,即机械割破树皮的乳管使胶乳流出,从而收集胶乳制备天然橡胶。
橡胶树中的天然橡胶是一种顺式-聚异戊二烯次生代谢物,由乳管中的特殊细胞器——橡胶粒子合成[4-5]。橡胶粒子是一种半个单位膜构成的球状结构,其大小分布范围为0.04~6.00 μm,膜组分主要是脂类和蛋白质以及其他非橡胶成分组成的亲水性层,而合成的天然橡胶烃则贮存在粒子内部[1, 6-7]。天然橡胶的合成是一个复杂的过程,多年来,学者们一直致力于研究橡胶粒子的天然橡胶合成机制[8-11],对橡胶粒子的结构以及其上的膜蛋白进行了大量的研究,发现了小橡胶粒子膜蛋白(SRPP)[12]和橡胶延伸因子蛋白(REF)[13-15]2个高丰度蛋白,以及橡胶转移酶[16-17]等其他多种蛋白[18-19]。随着研究的深入,天然橡胶的合成途径被逐渐剖析,大量与橡胶合成相关的蛋白质也被发现[19]。
作为胶乳中最主要的、含量最高的细胞器,除了橡胶生物合成的功能外,橡胶粒子在胶乳的停排中也起到了重要的作用。人们最早认为橡胶粒子彼此之间进行膜融合形成橡胶凝块,从而堵塞了乳管伤口,导致胶乳排胶终止,由此提出了“胶盖-胶塞模型”[20-21]。胶乳中的黄色体被认为是橡胶粒子的促凝固系统,其内含物中的多种蛋白被发现具有促进胶乳凝固的作用[21],据此,提出了多个有关橡胶粒子凝固的假说[22-26],其中的“凝集素”假说得到较为广泛的认同[25-26]。“凝集素”假说认为来自黄色体中的凝集素类蛋白质与橡胶粒子上的一种约22 kDa蛋白结合,导致了橡胶粒子的凝集,而这种22 kDa蛋白被证明为SRPP[27]。我国郝秉中等[28-29]研究发现,胶乳停排时,橡胶粒子仍然保持膜结构的完整性,并未出现凝固现象。SHI等[30]对橡胶粒子的凝集进行研究,发现C-乳清中的3-磷酸甘油醛脱氢酶(GAPDH)和Hev b7这2种蛋白也参与了橡胶粒子的凝集,并提出了以黄色体主要蛋白构成的“蛋白质网”为核心结合橡胶粒子的乳管伤口堵塞机制[28, 31]。由此可见,橡胶粒子不仅是天然橡胶合成的细胞器,也是排胶堵塞的主要参与者,它与黄色体构成了乳管伤口堵塞物的核心组分。橡胶粒子上除了SRPP外,是否还有其他蛋白参与橡胶粒子的凝集,不同粒径的橡胶粒子在蛋白凝集互作上是否存在差异,目前对这些问题的研究都很少。本研究以‘RY7-33-97’为研究材料,采集胶乳后进行不同粒径的橡胶粒子的分级分离,利用电泳以及蛋白质免疫印迹对不同处理的橡胶粒子上的蛋白进行分析,旨在为最终阐述橡胶粒子的凝集原理和橡胶树乳管排胶机制奠定基础。
1 材料与方法
1.1 材料
1.1.1 供试材料 选取种植于中国热带农业科学院儋州试验场九队(海南儋州宝岛新村)开割7年的‘RY7-33-97’成龄健康橡胶树,冰上收集正常排胶第5~30 min的胶乳,迅速带回实验室,低温高速分级离心,收集不同粒径大小的橡胶粒子组分,低温保存备用。
1.1.2 试剂与仪器 Tricine、Glycine、SDS、Tris购自AMRESCO(美国);丙烯酰胺(Acrylamide)购自NOVON(美国);N-N甲叉双丙烯酰胺(Bis-Acrylamide)、过硫酸铵(AP)、TEMED购自Sigma(西格玛科技有限公司,美国);β-巯基乙醇、甘露醇购自BBI公司(中国);甲醇、乙醇、冰乙酸、盐酸等均为国产分析纯。分子量标准蛋白(10、17、26、34、43、55、72、95、130、180 kDa,共10条带)为Fermentas产品。带碱性磷酸酶标记的羊抗兔二抗和羊抗鼠二抗为PIERCE(皮尔斯,美国)产品,碱性磷酸酶标记的兔抗鸡二抗购自Sigma。带辣根过氧化物酶标记的羊抗兔、抗鸡和抗鼠二抗购自Thermo Fisher Scientific (美国)。BCIP/NBT底物显色试剂盒购自天根生化科技(北京)有限公司;PVDF膜(0.2 μm)购自Bio-Rad(伯乐)。橡胶素一抗体由厦门博欣生物技术有限公司(Bambio)制备,几丁质酶一抗由宝赛生物生物技术有限公司合成,β-1,3-葡聚糖酶(Glu)一抗为北京华大基因研究中心制备;小橡胶粒子膜蛋白(SRPP)、橡胶延伸因子(REF)、HEV b7、3-磷酸甘油醛脱氢酶(GAPDH)抗体均为兔抗,由厦门博欣生物技术有限公司(Bambio)制备。
高速离心机购自Thermo fisher Scientific(美国);DYY-Ⅲ-7B转移电泳仪、DYY-12C型,电泳仪及小型电泳槽购自北京六一仪器厂;凝胶成像仪ImageQuant LAS 4000购自GE(美国)有限公司;LA-960激光粒度仪购自HORIBA(日本)公司。
1.2 方法
1.2.1 不同大小粒径橡胶粒子的制备 冰上保存的胶乳于高速离心机中以10 000 r/min,4℃条件下离心30 min,收集上层橡胶膏保存备用,余下的胶乳溶液依次以13 000、15 000、18 000 r/min 4℃条件下离心30 min,每次分离均收集上层橡胶膏备用,最后将余下的主要为小橡胶粒子的悬液于18 000 r/m, 4℃,离心60 min,收集上层半透明状的橡胶膏备用。
1.2.2 不同清洗强度橡胶粒子的制备 将分级离心后收集的不同粒径大小的橡胶粒子按照1 g分散悬浮到3.5 mL等渗缓冲液(0.05 mol/L Tris- HCl+0.4 mol/L甘露醇,pH 7.2)中清洗,然后同1.2.1中对应的离心速度离心收集橡胶粒子,每种样品均清洗3次,然后收集清洗完毕的橡胶粒子制备电泳样品。
1.2.3 橡胶粒子粒径测定 取不同离心速度收集的橡胶粒子,分散到等渗缓冲液中,LA-960激光粒度仪测定,具体方法按照LA-960湿法操作手册进行。
1.2.4 C-乳清和B-乳清等不同蛋白样品与小橡胶粒子的结合实验 胶乳于18 000 r/min,4℃条件下离心120 min,收集底部黄色体以及中间清液C-乳清;黄色体经反复冻融后离心收集清液即B-乳清;硫酸铵分级沉淀,分别收集B-乳清和C-乳清的85%硫酸铵沉淀收集的样品;柱层析纯化β-1,3-葡聚糖酶。取平均粒径为0.16 μm的小橡胶粒子按照1 g分散悬浮到3.5 mL等渗缓冲液(0.05 mol/L Tris-HCl+0.4 mol/L甘露醇,pH 7.2)中,然后取100 μL与前各种蛋白样品100 μL于25℃孵育30 min,加入10 μL的3%醋酸终止反应,7000 r/min,25℃条件下离心10 min,收集上层橡胶膏,制备电泳样品。
1.2.5 Tricine-SDS-Page Tricine-SDS-PAGE参照SCHAGGER等[32]和史敏晶等[33]的方法,简化成两层胶。不同的橡胶粒子膏状物均按照0.01 g分散到70 μL超纯水中,然后加入等体积的SDS上样缓冲液,煮沸5 min,离心后取上清电泳。每泳道上样量为10 μL(等体积上样)。电泳于浓缩胶中电压为30 V,样品完全到达分离胶界面后,升至100 V恒压电泳至结束。
1.2.6 Western Blotting 参照TOWBIN等[34]的方法将Tricine-SDS-PAGE电泳后凝胶中的蛋白质转移到0.2 μm PVDF膜上,50 mA低温转移12 h。转移电极缓冲液含20 mmol/L Tris碱,150 mmol/L甘氨酸,20%甲醇(/)。转移后漂洗,TBS溶液中过渡5 min,加入0.2%(/)戊二醛的TBS溶液中固定铰链45 min,漂洗,于10%脱脂奶粉的TBS封闭液中4℃封闭过夜。橡胶素、几丁质酶、β-1,3-葡聚糖酶、SRPP、REF、HEV b7、GAPDH一抗稀释倍数均为1∶3000;二抗则为1∶5000稀释倍数;抗体在37℃孵育90 min,BCIP/NBT显色5~10 min;化学发光1 h内显色,LAS4000成像。
2 结果与分析
2.1 不同粒径橡胶粒子膜结合蛋白分析
对不同离心速度分离获取的橡胶粒子组分进行粒径大小测定,结果表明,随着离心速度的增加,分离出来的橡胶粒子平均粒径越小,并且峰形也由双峰变为单峰(图1)。首次离心分离速度为10 000 r/min,平均粒径为0.45 μm,出现典型的双峰,其中粒径小于0.45 μm的橡胶粒子(即小橡胶粒子)占比为58.06%,余下的直径为0.45~ 1.73 μm的大橡胶粒子占比为41.94%,可见,即使是较低的离心速度,仍然能收集到小橡胶粒子(图1A);13 000 r/min离心后,平均粒径为0.37 μm,仍然表现为双峰,但是大粒径组分的峰值明显降低,粒径小于0.45 μm的小橡胶粒子具体占比升高至68.61%,大橡胶粒子占比则降至31.39%(图1B);15 000 r/min离心后,双峰基本消失,平均粒径为0.22 μm,小橡胶粒子占比高达91.34%,而大橡胶粒子占比仅8.66%,且其中最大粒径仅为0.88 μm,可见,此时离心收集到的橡胶粒子基本上是小橡胶粒子(图1C);18 000 r/min离心后,峰形为单峰,平均粒径为0.16 μm,小橡胶粒子占比达100.00%,大于0.45 μm的橡胶粒子完全消失(图1D);对保留在C-乳清中仍然残存的小橡胶粒子进行延时离心,收集后测定粒径,结果表明,这些橡胶粒子的平均粒径更小,约为0.14 μm,最小的粒径为0.07 μm,最大的也仅为0.39 μm(图1E),可见,随着离心强度增大,橡胶粒子组分的粒径逐渐减小。
对收集的不同组分橡胶粒子的膜上蛋白组分进行分析,电泳结果表明(图2A),不同样品的蛋白含量存在差异,尤其在20 kDa左右的中低分子量蛋白条带,差异明显。通过免疫印迹(WB)对这些蛋白进行鉴定分析,结果表明,免疫印迹化学发光显示,存在于C-乳清中的蛋白3-磷酸甘油醛脱氢酶(GAPDH)(图2B)和过敏原蛋白Hev b7(图2C)都被清晰地检测到;黄色体中的高丰度蛋白β-1,3-葡聚糖酶(Glu)(图2D)和橡胶素(Hev)尤其是其前体(图2E)也能被检测到,但几丁质酶(Chit)只能检测到极弱的条带(图2F),其中,β-1,3-葡聚糖酶(Glu)与粒径较大的橡胶粒子的结合能力明显强于粒径小的橡胶粒子;作为橡胶粒子上公认的主要蛋白SRPP和REF在橡胶粒子上含量极其丰富,通过灵敏度弱于化学发光的BCIP/NBT就可以显示,并且随着粒径减小,SRPP表现出明显的增加趋势(图2G),但REF的含量保持稳定,并未随粒径大小有明显变化(图2H)。由此可见,橡胶粒子上不仅结合大量的高丰度蛋白SRPP和REF,还可以结合胞质中与橡胶粒子凝集相关的3-磷酸甘油醛脱氢酶(GAPDH)和过敏原蛋白Hev b7,同时还能结合黄色体破裂后释放的内含物中的主要蛋白β-1,3-葡聚糖酶(Glu)和橡胶素(Hev),这些非橡胶粒子自身拥有的蛋白与之结合应该与橡胶粒子执行其生物功能有关。
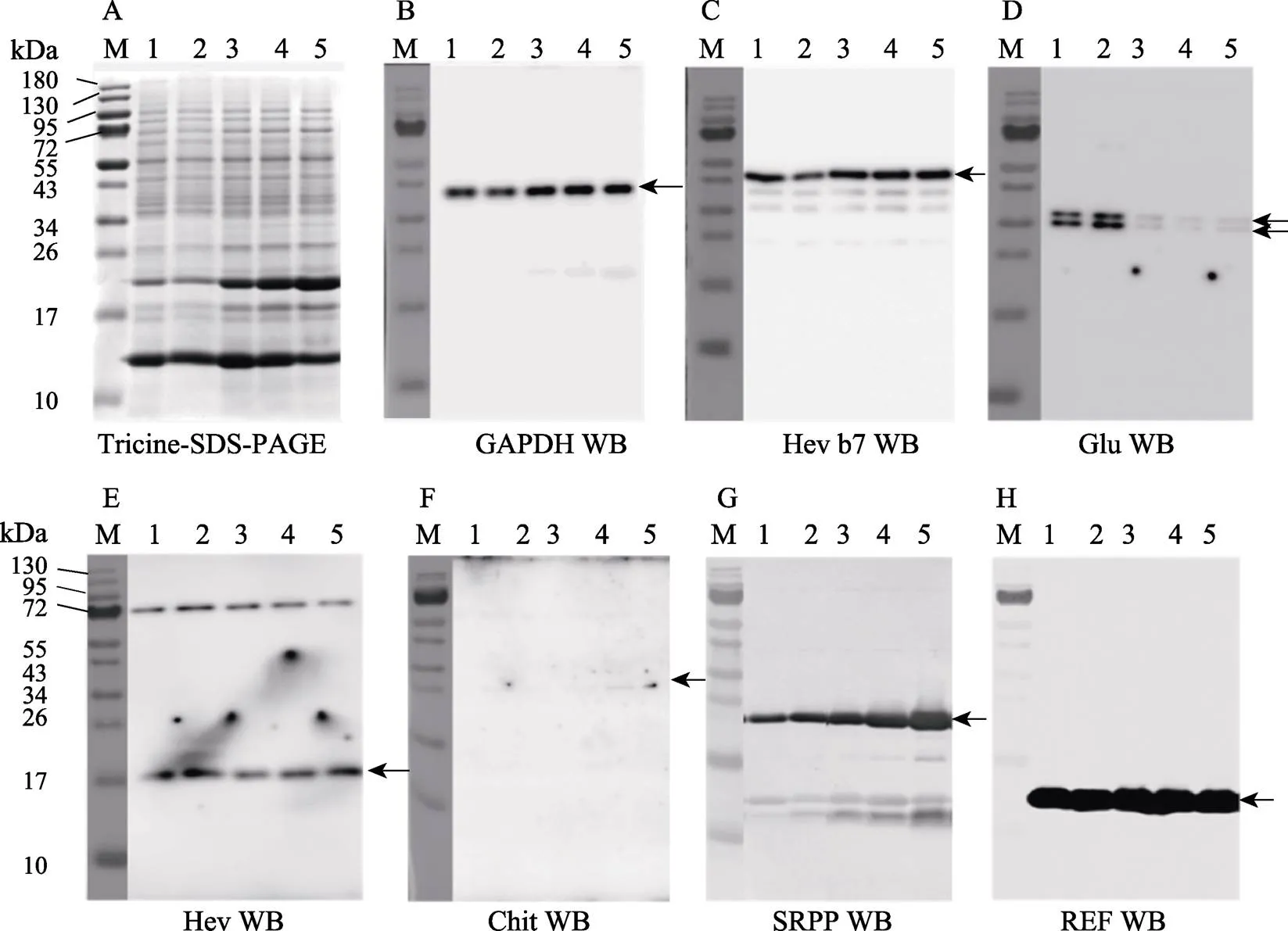
1~5:平均粒径分别为0.45、0.37、0.22、0.16、0.14 μm的橡胶粒子样品;M:蛋白质标样;箭头为目标蛋白带。
2.2 不同粒径橡胶粒子清洗后结合蛋白分析
选取平均粒径0.45(a)、0.22(b)、0.14(c) μm的橡胶粒子新鲜样品分散到等渗缓冲液中清洗3次,电泳检测结果表明(图3A),大部分蛋白条带在第一次清洗后明显减弱,且随着清洗次数增加进一步减弱,其中分子量大约20 kDa的主要蛋白带减弱尤其明显。通过免疫印迹对这些蛋白进行鉴定分析,化学发光显示结果表明,C-乳清中的GAPDH(图3B)和Hev b7(图3C)在未清洗前,在不同粒径的橡胶粒子上均能被清晰地检测到,但清洗1次后,GAPDH蛋白基本消失,仅在平均粒径0.45 μm的橡胶粒子组分中还有微弱的条带,而第二次清洗后,该蛋白未能在样品中检测到,表明进一步被洗脱;Hev b7条带明显强于GAPDH,第一次清洗后,在3种不同粒径的样品中都能检测到,但第二次之后同样未检测到。该结果表明这2个蛋白与橡胶粒子的结合不是很稳定,容易被洗脱,相对小橡胶粒子而言,这2个蛋白可能与大橡胶粒子的结合更紧密一些。黄色体中的高丰度蛋白β-1,3-葡聚糖酶(Glu)与橡胶粒子尤其是与大粒径橡胶粒子的结合能力明显更强(图3D),在0.45、0.22 μm的橡胶粒子组分中经3次洗脱后仍能检测到该蛋白。橡胶素(Hev)则主要是其前体被检测到,并且该蛋白极容易被洗脱(图3E),几丁质酶未能检测到明显的条带,表明结合能力较弱(图3F)。橡胶粒子的主蛋白SRPP和REF通过BCIP/NBT显示,结果表明,随着清洗次数的增加,不同粒径样品上的SRPP均明显减少(图3G),可见,该蛋白与橡胶粒子的结合并不是特别稳定。REF的含量虽然清洗后也有减少的趋势,但总体保持稳定,且粒径越小结合越稳定(图3H)。
2.3 小橡胶粒子体外孵育不同蛋白组分的结合分析
收集平均粒径0.16 μm的小橡胶粒子分散到等渗缓冲液中后与含不同蛋白组分的样品孵育,然后分析小橡胶粒子上的结合蛋白,结果表明,小橡胶粒子与不同蛋白组分孵育后,蛋白条带发生了明显的变化(图4A,图4B),根据条带的粗细可知,这些蛋白的含量较高,因此,可以统一通过BCIP/NBT来显示免疫印迹鉴定的结果(图4C~图4I)。GAPDH和Hev b7仅存在于C-乳清以及85%硫酸铵沉淀的C-乳清样品中,尤其是GAPDH在85%硫酸铵沉淀样本中得到了高度的浓缩,即丰度明显增加,在这2个蛋白样品与小橡胶粒子孵育后,相比对照的微弱结合,这2个样品中GAPDH结合到小橡胶粒子上的蛋白量明显增强(图4C);Hev b7蛋白同样大量结合到小橡胶粒子上(图4D),并且根据85%硫酸铵沉淀样品中Hev b7的丰度远低于GAPDH(图4A),可以推测,Hev b7与小橡胶粒子的结合能力高于GAPDH。黄色体中的高丰度蛋白β-1,3-葡聚糖酶(Glu)与橡胶粒子的结合明显(图4E);橡胶素(Hev)及其前体均被检测到,表明二者都与小橡胶粒子有结合,鉴于橡胶素在样品中的丰度极高,但结合量并不太大,可以认为橡胶素与小橡胶粒子的结合能力并不是很强(图4F)。几丁质酶也大量存在于B-乳清中,在这样的混合蛋白样品中,该蛋白与小橡胶粒子有较好的结合(图4G)。橡胶粒子的主蛋白SRPP和REF都有明显的高丰度条带(图4H,图4I),但在与B-乳清孵育的样品中,SRPP明显减弱(图4I),是否存在该蛋白被其他蛋白竞争而脱离橡胶粒子膜这一现象,值得进一步研究。
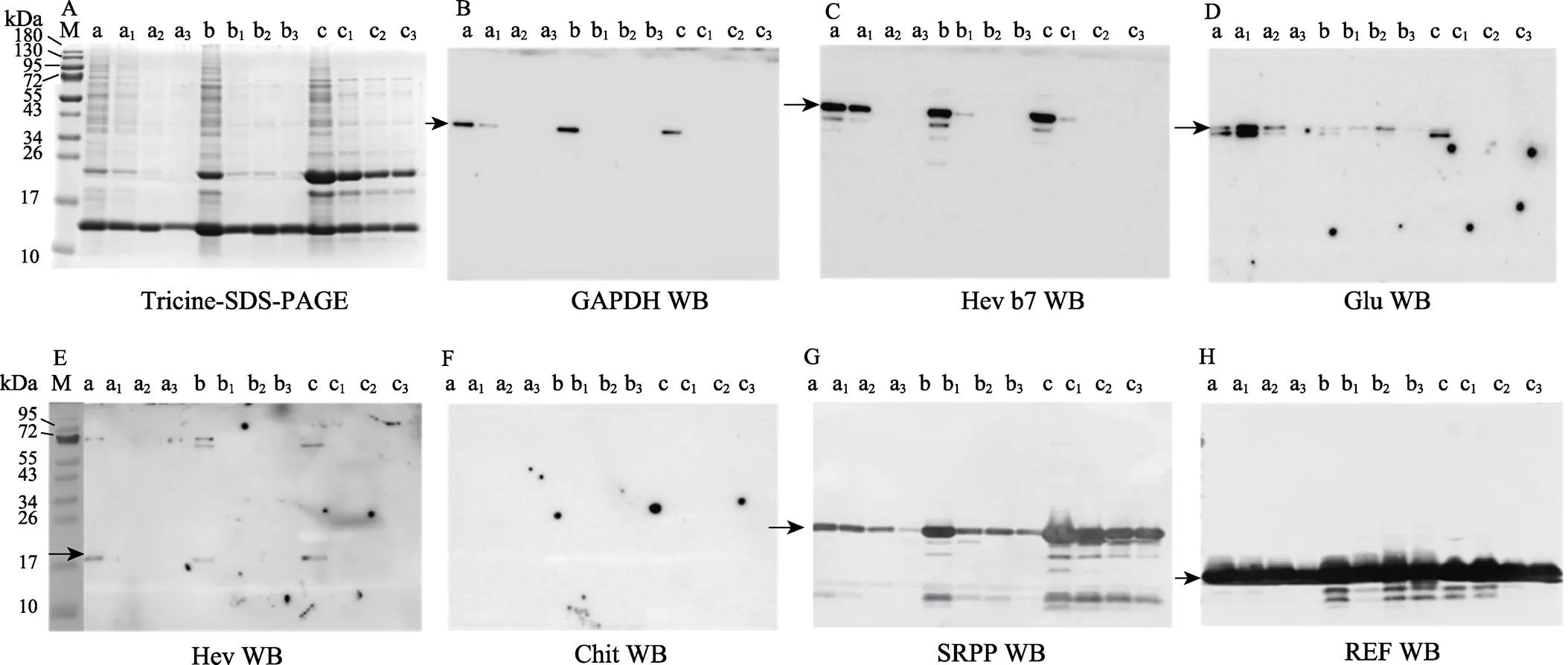
图3 清洗对不同粒径橡胶粒子结合蛋白的影响
3 讨论
橡胶粒子作为橡胶树胶乳中占比最大的细胞器,是天然橡胶合成和贮存的场所,同时也是乳管伤口堵塞物形成的主要参与者。无论是国外学者认为乳管伤口的堵塞是橡胶粒子本身的凝固造成的[21],还是我国学者比较认同的伤口堵塞的根本原因是伤口末端形成“蛋白质网”堵塞物[29, 31],其中都离不开橡胶粒子的参与。橡胶粒子具有不同的粒径,主要分布在0.08~2.00 μm范围内[19],极少数高达5~6 μm。通常以0.45 μm为界限,分为大、小橡胶粒子两大部分[1, 35],目前,普遍认为数量巨大的小橡胶粒子主要执行合成天然橡胶的功能,同时在乳管伤口堵塞中起重要作用[1, 26-27]。
橡胶粒子执行生物功能离不开其表面的结合蛋白,目前研究橡胶粒子上的蛋白主要集中在天然橡胶的生物合成上[8-9, 13, 36-38],对参与胶乳凝集、乳管堵塞方面蛋白的研究较少。本研究分级分离了不同粒径的橡胶粒子,并对不同粒径的橡胶粒子上结合的凝集相关蛋白进行了分析,结果表明无论粒径大小,橡胶粒子都能结合C-乳清中的Hev b7与GAPDH蛋白,根据结合量的高低,可以认为橡胶粒子结合Hev b7的能力强于GAPDH。B-乳清中的3个高丰度蛋白中,β-1,3-葡聚糖酶(Glu)与橡胶粒子的结合能力最强,橡胶素和几丁质酶结合能力则明显弱于β-1,3-葡聚糖酶,可见,在与橡胶粒子的结合中β-1,3-葡聚糖酶(Glu)起到更核心的作用,这一结果与SHI等[31]利用表面等离子共振技术(SPR)分析的这几个蛋白之间的互作能力的结果一致。Hev b7、GAPDH、β-1,3-葡聚糖酶(Glu)、橡胶素和几丁质酶作为分别存在于胞质和黄色体内部的蛋白质,不是橡胶粒子本身的蛋白,只能是通过蛋白互作等方式结合到橡胶粒子上,通过清洗的方式,本研究发现这些蛋白可以在温和的条件下被洗脱,表明这些蛋白与橡胶粒子的结合相对松散。根据清洗的次数和残留的蛋白量,可以推测出Hev b7和β-1,3-葡聚糖酶(Glu)与橡胶粒子具有更好的结合能力,而橡胶素和几丁质酶与之结合能力较弱,已有研究表明橡胶素和几丁质酶这2个蛋白和β-1,3-葡聚糖酶之间存在互作[31],那么,它们是否是通过β-1,3-葡聚糖酶间接结合到橡胶粒子上而非直接结合到橡胶粒子上值得进一步分析研究。
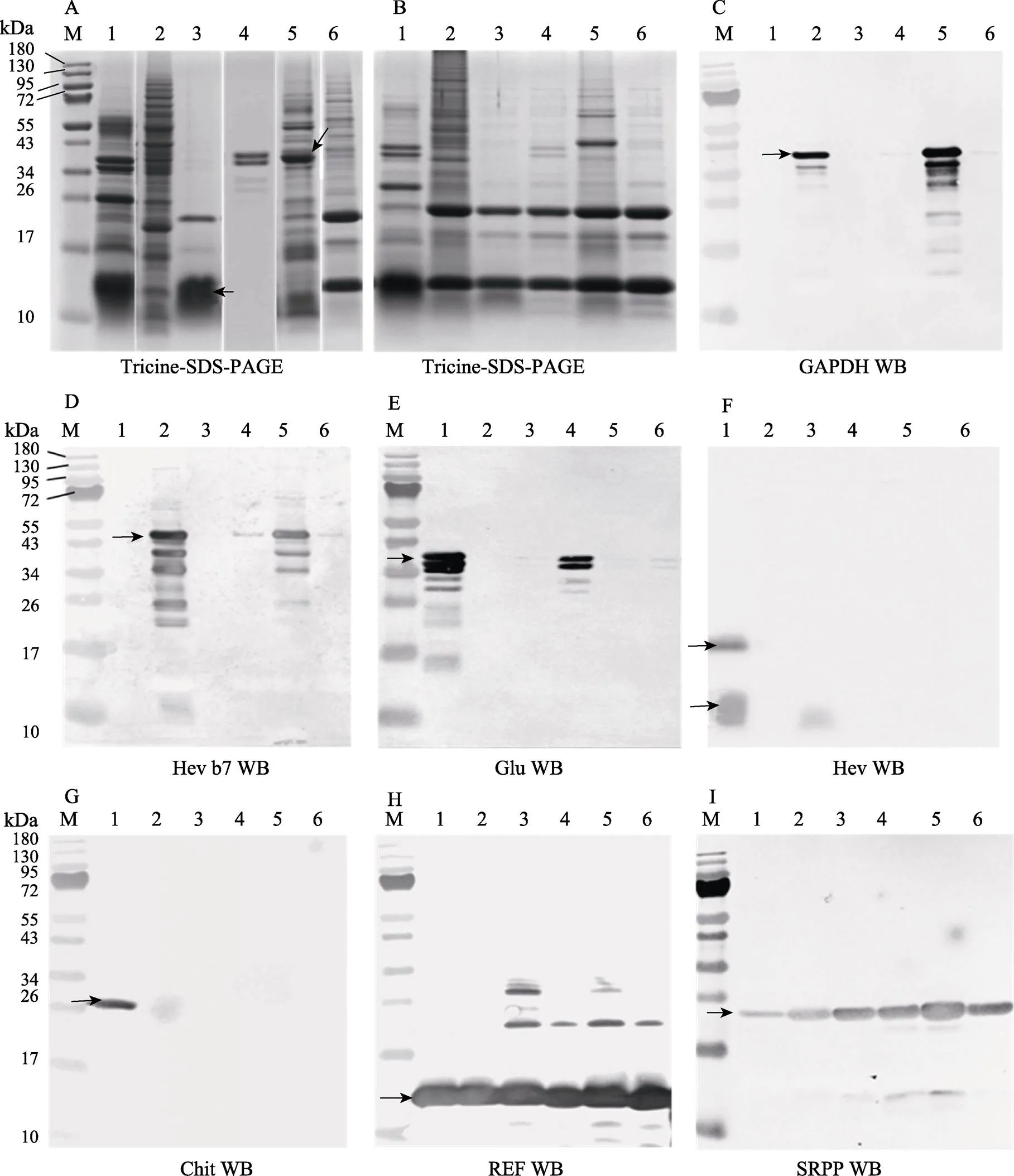
1~6:依次为黄色体B-乳清、胞质C-乳清、B-乳清85%硫酸铵沉淀蛋白组分、纯化的β-1,3-葡聚糖酶(Glu)、C-乳清85%硫酸铵沉淀蛋白组分以及小橡胶粒子样品;6,对照,小橡胶粒子与缓冲液孵育。M:蛋白质标样;A图中泳道3的短箭头表示高丰度的橡胶素蛋白,泳道5中的长箭表示被浓缩的37 kDa的蛋白,其余箭头表示免疫印迹的目标蛋白带。
SRPP和REF作为橡胶粒子自身的标志性蛋白[39-41],在大、小橡胶粒子上的含量存在差别,以前的观点认为SRPP在小橡胶粒子上占优势,而REF在大橡胶粒子上占优势[27],本研究表明,REF在大、小橡胶粒子上的含量差异不明显,发生明显变化的是SRPP,该蛋白主要分布在小橡胶粒子上,胶乳中随着小橡胶粒子比例的加大,SRPP的含量是增加的。SRPP和REF与橡胶粒子膜结合的牢固程度明显不同,REF不容易洗脱,而SRPP极易被洗脱,因此,可以认为SRPP在小橡胶粒子上的结合相对松散,容易脱落。小橡胶粒子成长为大橡胶粒子后,膜表面的SRPP基本消失,执行功能可能由合成天然橡胶转化为贮存天然橡胶,SRPP脱落后是否会重新结合到小橡胶粒子表面从而循环使用也是一个值得研究的问题。
鉴于大、小橡胶粒子都能结合胞质C-乳清和黄色体B-乳清中的凝集相关的蛋白,本研究推测排胶过程中不同大小的橡胶粒子可能都参与了乳管的堵塞,目前研究认为SRPP是橡胶粒子凝集过程中与其他蛋白结合的主要位点[27],但大橡胶粒子上该蛋白含量极少,是否还有其他蛋白作为锚定位点参与橡胶粒子的凝集值得深入研究。
[1] 田维敏, 史敏晶, 谭海燕, 吴继林, 郝秉中. 橡胶树树皮结构与发育[M]. 北京: 科学出版社, 2015.
TIAN W M, SHI M J, TAN H Y, WU J L, HAO B Z. Bark structure and development of[M]. Beijing: Science Press, 2015. (in Chinese)
[2] D’AUZAC J, JACOB J L, CHRESTIN H. Physiology of rubber tree latex[M]. Boca Raton: CRC Press, 1989.
[3] GOMEZ J B. Anatomy ofand its influence on latex production[A]. Kuala Lumper: Malaysia Rubber Research and Development Board, 1982.
[4] KUSH A. Isoprenoid biosynthesis: thefactory[J]. Plant Physiology and Biochemistry, 1994, 32(8): 761-767.
[5] CORNISH K. Similarities and differences in rubber biochemistry among plant species[J]. Phytochemistry, 2001, 57: 1123-1134.
[6] CORNISH K, WOOD D F, WINDLE J J. Rubber particles from four different species, examined by transmission electron microscopy and electron-paramagnetic-resonance spin labeling, are found to consist of a homogeneous rubber core enclosed by a contiguous, monolayer biomembrane[J]. Planta, 1999, 210: 85-96.
[7] WOOD D F, CORNISH K. Microstructure of purified rubber particles[J]. International Journal of Plant Sciences, 2000, 161: 435-45.
[8] CHOW K S, WAN K L, ISA M N, BAHARI A, TAN S H, HARIKRISHNA K, YEANG H Y. Insights into rubber biosynthesis from transcriptome analysis oflatex[J]. Journal of Experimental Botany. 2007, 58: 2429- 2440.
[9] TANG C R, YANG M, FANG Y J, LUO Y F, GAO S H, XIAO X H, AN Z W, ZHOU B H, ZHANG B, TAN X Y, YEANG H Y, QIN Y X, YANG J H , LIN Q, MEI H L, MONTORO P, LONG X Y, QI J Y, HUA Y W, HE Z L, SUN M, LI W J, ZENG X, CHENG H, LIU Y, YANG J, TIAN W M, ZUANG N S, ZENG R Z, LI D J, HE P, LI Z, ZOU Z, LI S L, LI C J, WANG J X, WEI D, LAI C Q, LUO W, YU J, HU S N, HUANG H S. The rubber tree genome reveals new insights into rubber production and species adaptation[J]. Nature Plants, 2016, 2: 16073
[10] LUO Z, IAFALDANO B J, ZHUANG X, FRESNEDO- RAMIREZ J, CORNISH K. Analysis of the firsttranscriptome reveals potential rubber yield related SNPs[J]. Science Reports, 2017, 7: 9939.
[11] LIN T, XU X, RUAN J, LIU S, WU S, SHAO X, WANG X. Genome analysis ofRodin provides new insights into rubber biosynthesis[J]. National Science Review, 2017, 5: 78-87.
[12] OH S K, KANG H, SHIN D H, YANG J, CHOW K S, YEANG H Y, WAGNER B. Isolation, characterization, and functional analysis of a novel cDNA clone encoding a small rubber particle protein from[J]. Journal of Biological Chemistry, 1999, 274: 17132-17138.
[13] DENNIS, M S, LIGHT D R. Rubber elongation factor from. Identification, characterization, and role in rubber biosynthesis[J]. Journal of Biological Chemistry, 1989, 264: 18608-18617.
[14] PRIYA P, VENKATACHALAM P, THULASEEDHARAN A. Molecular cloning and characterization of the rubber elongation factor gene and its promoter sequence from rubber tree (): a gene involved in rubber biosynthesis[J]. Plant Science, 2006, 171: 470-480.
[15] AOKI Y, TAKAHASHI S, TAKAYAMA D, OGATAC Y, SAKURAI N, SUZUKI H, ASAWATRERATAN-AKUL K. Identification of laticifer-specific genes and their promoter regions from a natural rubber producing plant[J]. Plant Science, 2014, 225: 1-8.
[16] CORNISH K. The separate roles of plant cis and trans prenyl transferases in cis-1,4-polyisoprene biosynthesis[J]. European Journal of Biochemistry, 1993, 218: 267-271.
[17] CORNISH K, SOTT D J, XIE W, MAU C D J, ZHENG Y F, LIU X H, PRESTWICH G D. Unusual subunits are directly involved in binding substrates for natural rubber biosynthesis in multiple plant species[J]. Phytochemistry, 2018, 156: 55- 72.
[18] DAI L J, KANG G J, LI Y, NIE Z Y, DUAN C F, ZENG R Z. In-depth proteome analysis of the rubber particle of(para rubber tree)[J]. Plant Molecular Biology, 2013, 82: 155-168.
[19] CHERIAN S, RYU S B, CORNISH K. Natural rubber biosynthesis in plants, the rubber transferase complex, and metabolic engineering progress and prospects[J]. Plant Biotechnology Journal, 2019, 17: 2041-2061.
[20] SOUTHORN W A. Latex flow studies I. Electron microscopy ofin the region of the tapping cut[J]. Journal of Rubber Research Institute of Malaysia, 1968, 20(4): 176-186.
[21] SOUTHORN W A. Physiology of(latex flow)[J]. Journal of Rubber Research Institute of Malaysia, 1969, 21(4): 494-521.
[22] WOO C H. Rubber coagulation by enzymes oflatex[J]. Journal of Rubber Research Institute of Malaysia, 1973, 23(5): 323-332.
[23] WOO C H. “Coagulase” from the fraction oflatex[J]. Journal of Rubber Research Institute of Malaysia, 1976, 24(4): 227-232.
[24] BROEKAERT W, LEE H I, KUSH A, CHUA N H, RAIHEL N. Wound-induced accumulation of mRNA containing a hevein sequence in laticifers of rubber tree ()[J]. Proceeding of the National Academy of Sciences of the USA, 1990, 87(19): 7633-7637.
[25] GIDROL X, CHRESTIN H, TAN H L. Hevein, a lectin-like protein from(rubber tree) is involved in the coagulation of latex[J]. Journal of Biological Chemistry, 1994, 269(12): 9278-9283.
[26] WITITSUWANNAKUL R, PASITUL P, JEWTRAGOON P.latex lectin binding protein in C-serum as an anti- latex coagulating factor and its role in a proposed new model for latex coagulation[J]. Phytochemistry, 2008, 69(3): 656- 662.
[27] WITITSUWANNAKUL R, RUKSEREE K, KANOKWI ROON. A rubber particle protein specific forlatex lectin binding involved in latex coagulation[J]. Phytochemistry, 2008b, 69(5): 1111-1118.
[28] 郝秉中, 吴继林, 谭海燕. 橡胶树乳管切割后的堵塞研究[J]. 热带作物学报, 1996, 17(1): 1-6.
HAO B Z, WU J L, TAN H Y. Laticifer plugging ofafter severing[J]. Chinese Journal of Tropical Crops, 1996, 17(1): 1-6. (in Chinese)
[29] HAO B Z, WU J L, MENG C X. Laticifer wound plugging in: the role of protein-network with rubber particle aggregations in stopping latex flow and protecting wounded laticifers [J]. Journal of Rubber Research, 2004, 7(4): 281-299.
[30] SHI M J, CAI F G, TIAN W M. Ethrel-stimulated prolongation of latex flow in the rubber tree (Muell. Arg.): a Hev b 7-like protein acts as a universal antagonist of rubber particle aggregating factors from lutoids and C-serum[J]. Journal of Biochemistry, 2016, 159(2): 209-216.
[31] SHI M J, LI Y, DENG S N, WANG D D, CHEN Y Y, YANG S G, WU J L, TIAN W M. The formation and accumulation of protein-networks by physical interactions in the rapid occlusion of laticifer cells in rubber tree undergoing successive mechanical wounding[J]. BMC Plant Biology, 2019, 19(4): 1-11.
[32] SCHAGGER H, JAGOW G V. Tricine-sodium dodecyl sulfate-polyacrylamid gel electroporesis for the separation of proteins in the range from 1 to 100 kDa[J]. Analytical Biochemistry, 1987, 166: 368-379.
[33] 史敏晶, 陈月异, 田维敏. 橡胶树黄色体B-乳清可溶性蛋白质电泳分离技术[J]. 热带作物学报, 2009, 30(2): 121-125.
SHI M J, CHEN Y Y, TIAN W M. An improved electrophoresis method for separation of B-serum soluble proteins from lutoids inMuell. Arg[J]. Chinese Journal of Tropical Crops, 2009, 30(2): 121-125. (in chinese)
[34] TOWBIN H, STAEHELIN T, GORDON J. Electrophoretic transfer of protein from polyacrylamide gels to nitrocellulose sheets: procedure and some applications[J]. Proceedings of the National Academy of Sciences of the United States America, 1979, 76: 4350-4354.
[35] MOIR G F J. Ultracentrifugation and staining oflatex[J]. Nature, 1959, 184: 1626-1628.
[36] WANG X C, WANG D, SUN Y, YANG Q, CHANG L L, WANG L, MENG X. Comprehensive proteomics analysis of laticifer latex reveals new insights into ethylene stimulation of natural rubber production[J]. Science Reports, 2015, 5: 13778.
[37] YAMASHITA S, YAMAGUCHI H, WAKI T, AOKI Y, MIZUNO M. Identification and reconstitution of the rubber biosynthetic machinery on rubber particles from[J]. eLife, 2016, 5: e19022
[38] YANASHITA S, TAKAHASHI S. Molecular mechanisms of natural rubber biosynthesis[J]. Annual Review of Biochemistry, 2020, 89(1): 821-851.
[39] BROWN D, FEENEY M, AHMADI M, LONOCE C, SAJARI R, COLA A D, FRIGERIO L. Subcellular localization and interactions among rubber particle proteins from[J]. Journal of Experimental Botany, 2017, 68(18): 5045-5055.
[40] XIANG Q, XIA K, DAI L, KANG G, LI Y, NIE Z, DUAN C, ZENG R. Proteome analysis of the large and the small rubber particles ofusing 2D-DIGE[J]. Plant Physiology and Biochemistry, 2012, 60: 207-213.
[41] DAI L, NIE Z, KANG G, LI L, ZENG R. Identification and subcellular localization analysis of two rubber elongation factor isoforms onrubber particles[J]. Plant Physiology and Biochemistry, 2017, 111: 96-106.
Aggregation-related Proteins Binding to Rubber Particles with Different Diameter in Rubber Tree
DING Huan1, WU Shaohua2, XIA Zhihui1, HUANG Xi1, SHI Minjing1,2*
1. College of Tropical Crops, Hainan University, Haikou, Hainan 570228, China; 2. Rubber Research Institute, Chinese Academy of Tropical Agricultural Sciences / Key Laboratory of Biology and Genetic Resources of Rubber Tree, Ministry of Agriculture and Rural Affairs / State Key Laboratory Incubation Base for Cultivation and Physiology of Tropical Crops, Haikou, Hainan 571101, China
Rubber tree (Muell.Arg.) is an important rubber-producing plant, and thesecondary laticifer in the trunk bark is the major tissue for the synthesis and storage of natural rubber. Natural rubber is extracted from the latex in laticifer by tapping (mechanical wounding) and synthesized by a special organelle, named the rubber particle. Rubber particle is a spherical structure surrounded by a lipid monolayer and membrane-bound proteins, in which the natural rubber is stored. The peripheral membrane is bound to a variety of proteins that are closely related to the functions of rubber particles. At present, there are many studies on proteins related to natural rubber synthesis, but the proteins related to rubber particle aggregation still remains unclear. Using the latex collected from the clone ‘RY7-33-97’ as the material, rubber particles with different particle sizes were obtained by fractionation at different centrifugal speeds. Subsequently, the rubber particle samples were cleaned by buffer for three times and collected for electrophoresis, respectively. The binding proteins on rubber particles were compared and analyzed by Tricine- SDS-PAGE and Western-blotting techniques. There were obvious differences in the content of bound proteins on rubber particles with different particle size. Accompanied by the decrease of rubber particle size, the content of small rubber particle membrane protein (SRPP) increased significantly, while the content of rubber elongation factor protein (REF) remained stable and had little relationship with the rubber particle size. Hevb7 latex allergen protein (Hevb7) and glyceraldehyde 3-phosphate dehydrogenase (GAPDH) in C-serum could bind to the rubber particles, and β-1,3-glucanase (Glu), hevein (Hev) and chitinase (Chit) in B-serum could also bind to rubber particles, but in which the binding ability of chitinase to rubber particles was the weakest. After cleaning, as the main membrane protein of rubber particles, the content of SRPP decreased significantly with the times of cleaning, but the content of REF protein did not change obviously, indicating that the binding ability of SRPP to rubber particle membrane is not as strong as that of REF. The content of agglutination-related proteins from C-serum and B-serum decreased significantly with the times of cleaning, usually, the proteins could not be detected after the second cleaning. Small rubber particles with about 0.16 μm mean diameter were incubated with different protein samples, the results showed that rubber particles could bind a variety of different proteins, in which some proteins were related to the aggregation of rubber particles. In this study, the aggregation-related proteins bound to rubber particles with different particle sizes were preliminarily analyzed, the results would lay a foundation for elucidating the mechanism of latex flow of rubber trees.
Muell.Arg.; rubber particle; aggregation-related protein; western blotting
S794.1
A
10.3969/j.issn.1000-2561.2022.12.003
2022-03-09;
2022-04-22
国家自然科学基金项目(No. 31870590);海南省基金创新团队项目(No. 320CXTD442);国家重点研发计划项目(No. 2018YFD1000502)。
丁 欢(1995—),女,硕士研究生,研究方向:农艺与种业。*通信作者(Corresponding author):史敏晶(SHI Minjiing),E-mail:pzbsmjzifeng@163.com。
