铜死亡相关基因PDHA1与乳腺癌的预后关系及列线图的构建
2022-12-12杨秋怡易嘉宁郭妙兰范培芝喻洁曾杰
杨秋怡,易嘉宁,郭妙兰,范培芝,喻洁,曾杰
(湖南师范大学附属第一医院/湖南省人民医院 乳甲外科,湖南 长沙 410005)
乳腺癌(breast cancer)已超过肺癌成为女性最常见的恶性肿瘤[1],是全球女性癌症死亡的主要原因[2]。随着早期筛查的进步以及抗癌策略的不断发展,乳腺癌的治疗有了很大的改善。然而,乳腺癌的复发及转移率仍然很高。由于肿瘤的异质性和复杂性,现有的生物标志物在预测乳腺癌预后方面受到限制[3]。因此,为了提高预后并指导个体化精准治疗,探索新的生物标志物作为临床研究领域的预后指标成为迫切需要。
即使在氧气存在的情况下,肿瘤细胞的增殖也以增加糖酵解代谢为特征,称为有氧糖酵解,即Warburg 效应[4]。有氧糖酵解已被证实可促进肿瘤的发生、发展[5-6]。丙酮酸脱氢酶复合物(pyruvate dehydrogenase complex,PDHC)是位于线粒体基质的多酶复合物,发挥催化作用可以使丙酮酸氧化脱羧转化成乙酰辅酶A,从而将糖酵解、三羧酸循环以及氧化磷酸化联系起来[7-8]。PDHC 由丙酮酸脱氢酶(E1)、二氢硫辛酰胺转乙酰酶(E2)和二氢硫辛酸脱氢酶(E3)组成,其中丙酮酸脱氢酶(E1)由α 和β 亚单位组成,α 亚单位即PDHA1,是PDHC 的活性调控位点[9]。最近,Tsvetkov 等[10]研究发现一种新的细胞死亡途径,即铜离子载体诱导的细胞死亡,称为铜死亡(cuproptosis),并已证明铜直接与三羧酸循环中的脂酰化成分结合,导致蛋白质毒性应激,最终导致细胞死亡。并且该研究为确定介导铜死亡的特定代谢途径,进行了全基因组CRISPR/Cas9 功能缺失筛选,获得了10 个铜死亡相关基因:FDX1、LIAS、LIPT1、DLD、DLAT、PDHA1、PDHB、MTF1、GLS、CDKN2A,PDHA1 作为铜死亡的相关基因,参与铜死亡过程的调控。研究发现,PDHA1 的表达与多种肿瘤的预后相关,如胃癌[11]、子宫颈鳞状细胞癌[12]、肝癌[13]、头颈鳞状细胞癌[14]、肾细胞癌[15]、卵巢癌[16]等。而乳腺癌中PDHA1 表达与预后关系却少有研究。本研究旨在运用生物信息学方法探讨PDHA1 与乳腺癌预后关系并构建预后相关的列线图模型。
1 资料与方法
1.1 数据来源
癌症基因组图谱数据库(the cancer genome atlas,TCGA)(https//portal.gdc.cancer.gov)是目前最大的癌症基因信息数据库,保存了包括基因表达数据、拷贝数变异等数据[17]。从TCGA 数据库中并下载乳腺癌患者的基因信息和临床病理数据。利用R 语言整理其临床资料,选择TNM 分期、病理分期、人种、年龄、组织学类型、ER、PR 和HER-2状态、PAM50 亚型、绝经状态、放疗情况、生存时间、生存状态等信息完整的患者共1 065 例,将患者分为PDHA1 高表达者533 例,低表达者532 例,比较其临床参数。
1.2 PDHA1在乳腺癌组织中的表达水平
肿瘤免疫评估资源(TIMER)数据库(https://cistrome.shinyapps.io/timer)是一个准确且方便地分析肿瘤免疫的工具,包含32 种癌症类型,涵盖10 897 个来自TCGA 数据库中的样本[18],本研究利用TIMER 数据库差异表达模块分析PDHA1 基因在多种癌症中的表达情况。利用TCGA 数据库对乳腺癌中PDHA1 mRNA 的表达进行分析。UALCAN 数据库(http://ualcan.path.uab.edu)是一个基于TCGA 数据库中相关癌症数据进行在线分析和挖掘的网站工具[19]。本研究利用该数据库分析比较PDHA1 在人正常乳腺组织和乳腺癌组织中的蛋白表达。HPA 数据库(The Human Protein Atlas)(https://www.pro-teinatlas.org)是2003 年由瑞典科学家发起的项目,旨在利用不同的组学技术绘制细胞、组织和器官中所有的人类蛋白质图谱。利用该数据库分析PDHA1 免疫组织化学结果,进一步验证PDHA1的表达水平。
1.3 构建蛋白质-蛋白质互作网络(PPI),基因-基因互作网络
STRING 数据库(https://cn.string-db.org)是专门用于生物体范围蛋白质关联网络的几个在线资源之一[20]。利用该数据库中蛋白质互作信息对PDHA1 构建蛋白质互作网络(PPI)。Gene MANIA数据库可对基因数据集进行分析和预测,以分子网络的形式显示这些基因之间的关系(http://genemania.org)[21]。利用该数据库绘制PDHA1 的基因-基因互作关系网络图
1.4 生存分析
Kaplan-Meier Plotter 数据库(http://kmplot.com/annalyss)包括基因表达数据和临床数据[22]。利用该数据库在线分析上述数据中PDHA1 的表达对乳腺癌患者总生存期(overall survival,OS)、无复发生存期(relapse free survival,RFS)、进展后生存期(post progression survival,PPS)、无转移生存期(distant metastasis free survival,DMFS)的影响。并在TCGA 数据库中进一步验证PDHA1 的表达对乳腺癌患者OS 的影响。运用单因素和多因素Cox 回归分析研究PDHA1 在乳腺癌中的预后价值。将TNM 分期、Stage 分期、人种、年龄、组织学类型、ER、PR 和HER-2 状态、PAM50 亚型、绝经状态、放疗情况等进行分层分析各亚组中PDHA1 的表达与乳腺癌患者OS 的关系。P<0.05 为差异有统计学意义。
1.5 构建基因集富集分析(GSEA)
通过GEPIA 数据库(http://gepia.cancer-pku.cn)[23]筛选出PDHA1 的共表达基因,使用R 包“clusterProfiler”对其进行GSEA,使用校正的P值和归一化的富集分数(NES)表征富集分析的结果。用“ggplot2”包使GSEA 富集分析可视化。
1.6 基因-化合物相互作用分析
GeneCards 数据库(www.genecards.org)是一个全面的、权威的人类基因注释信息纲要,提供了所有已知和预测的人类基因的简明基因组、蛋白质组、转录组、疾病和功能数据[24],本研究利用该数据库寻找与PDHA1 基因有关的潜在相互作用的化合物。
1.7 列线图的构建与校准
运用R 语言survival 和rms 软件包(survival package R 3.6.3、rms package R 3.6.3)构建乳腺癌预后因素预测列线图模型,将基于单因素和多因素Cox 回归分析中筛选出来的影响乳腺癌预后的独立危险因素及PDHA1 的表达纳入列线图中。并绘制校准曲线以评估PDHA1 列线图的预测和实际观察之间的一致性。
2 结 果
2.1 PDHA1表达与临床特征的关系
采用χ2检验分析PDHA1 表达与乳腺癌临床病理特征之间的关系。PDHA1 基因低表达组和高表达组在T 分期(P<0.001)、病理分期(P=0.031)、人种(P<0.001)、组织学类型(P<0.001)、PR 状态(P<0.001)、ER 状态(P<0.001)、PAM50 亚型(P<0.001)方面比较,差异具有统计学意义(表1)。
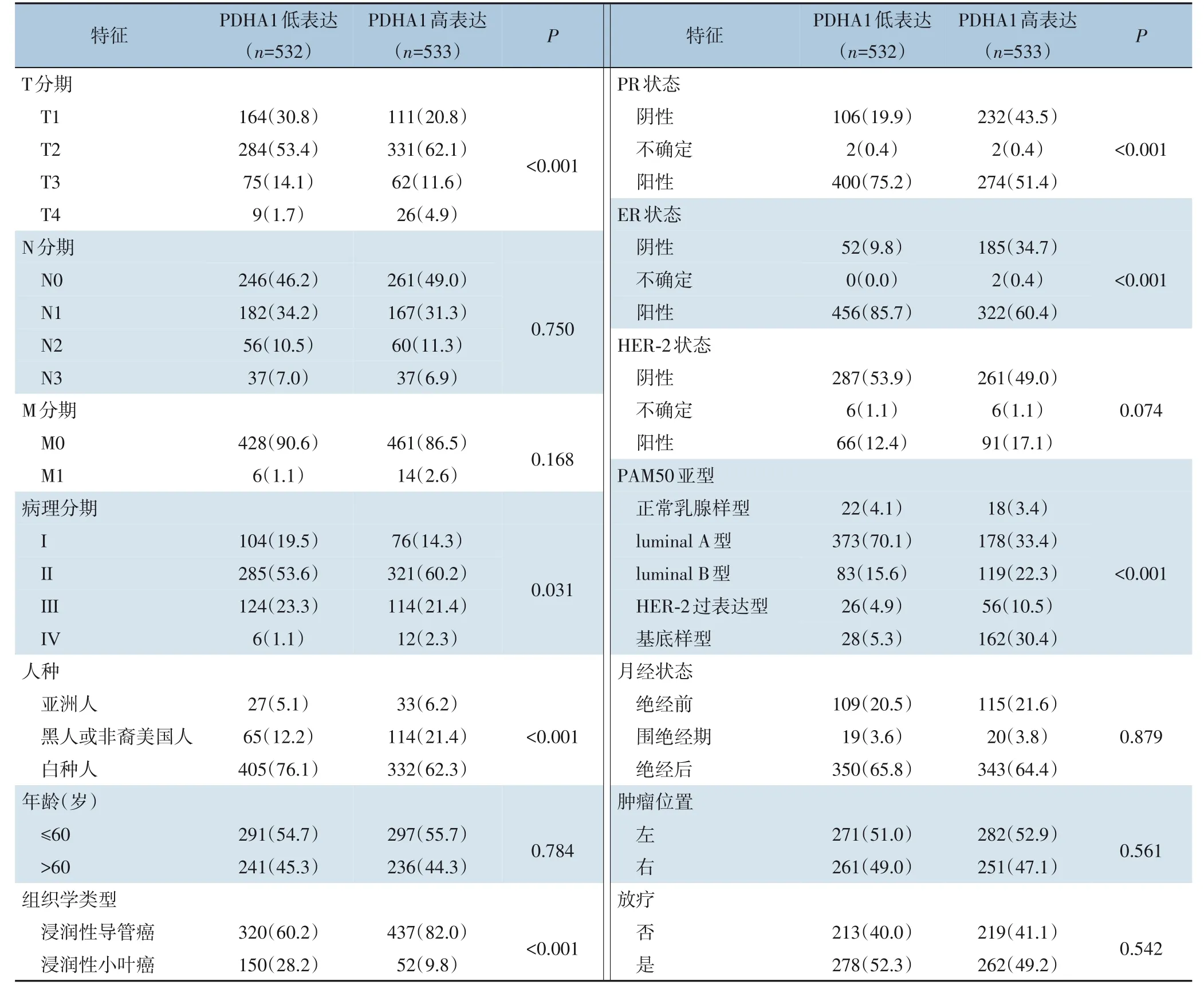
表1 来自TCGA数据库的乳腺癌样本中PDHA1表达与临床病理学特征之间的关联[n(%)]Table 1 Association between PDHA1 expression and clinicopathologic characteristics in breast cancer samples from the TCGA database [n (%)]
2.2 PDHA1在乳腺癌中的表达水平
TIMER 数据库分析显示PDHA1 在乳腺癌中低表达(P<0.001),另外,肾上腺皮质癌(ACC)、食管癌(ESCA)、肾透明细胞癌(KIRC)、急性髓性白血病(LAML)等肿瘤中PDHA1 也明显呈低表达(P<0.001)(图1A);TCGA 数据库分析显示乳腺癌患者PDHA1 mRNA 的表达水平显著低于正常乳腺组织(P<0.001)(图1B);利用UALCAN 数据库分析PDHA1 蛋白在正常与乳腺癌组织中的表达情况,结果(图1C)显示PDHA1 蛋白在乳腺癌组织中低表达(P<0.001),HPA 数据库显示乳腺正常组织中PDHA1 染色为高水平,而在乳腺癌组织中PDHA1 染色为低水平,进一步验证PDHA1 蛋白在乳腺癌组织中低表达这一结果(图1D)。
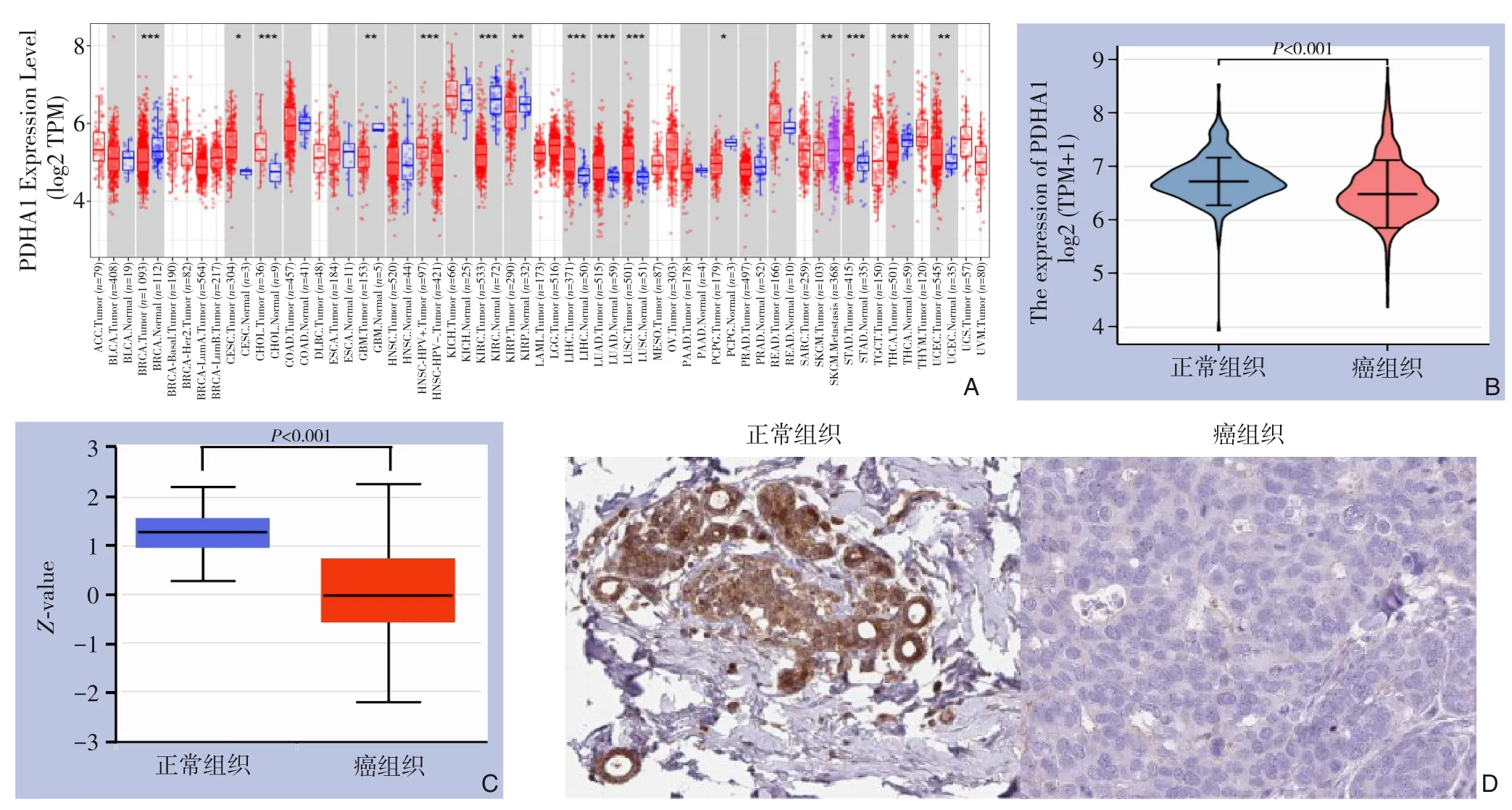
图1 PDHA1 的表达分析 A:PDHA1 在不同肿瘤中的表达;B:乳腺癌组织及正常组织中PDHA1 的表达情况;C:UALCAN数据库中PDHA1蛋白在乳腺癌和正常组织中的表达情况;D:HPA数据库中PDHA1蛋白在乳腺癌和正常组织中的表达情况Figure 1 Analysis of PDHA1 expression A: Expressions of PDHA1 in different tumors;B: Expressions of PDHA1 in breast cancer and normal tissue;C: Expressions of PDHA1 protein in breast cancer and normal tissues in UALCAN database;D: Expressions of PDHA1 protein in breast cancer and normal tissues in HPA database
2.3 构建蛋白PPI网络
通过构建PPI 蛋白互作网络,与PDHA1 相关互作蛋白有15 个,包括:PDHB、DLD、PDHX、IDH2、DLAT、IDH3A、IDH3G、SUCLA2、PDP1、PDK4、PDK1、PDK3、IDH3B、DLST、OGDH(图2)。

图2 DHA1相互作用蛋白网络分析Figure 2 Analysis of PDHA1 interaction protein network
2.4 构建基因-基因互作网络
通过构建基因-基因互作网络,与PDHA1 相关互作基因有20 个,即PDK1、PDK2、PDK3、DLAT、PDHX、PDHA2、PDHB、CS、HSD17B10、DLD、PDPR、PDP1、TRIM2、TRIM3、OGDH、ACO11462.1、DHTKD1、OGDHL、ABAT、OAT(图3)。

图3 PDHA1基因-基因互作网络Figure3 PDHA1 gene-gene interaction network
2.5 PDHA1在乳腺癌中表达的预后价值
乳腺癌患者中PDHA1 的表达与OS、RFS、PPS、DMFS 预后结果的关系如图4 所示:PDHA1基因高表达组的OS(HR=1.26,95%CI=1.02~1.54,P=0.029)、RFS(HR=1.18,95%CI=1.05~1.32,P=0.005 1)、PPS(HR=1.41,95%CI=1.07~1.86,P=0.015)、DMFS(HR=1.29,95%CI=1.1~1.52,P=0.002 3)均低于低表达组。在TCGA 数据库中进一步得到验证PDHA1 基因高表达组的OS 低于低表达组(HR=1.47,95%CI=1.06~2.05,P=0.021)。单因素Cox 回归分析结果提示TNM 分期、病理分期、年龄、分子分型、绝经状态、放射治疗和PDHA1 表达均为影响患者预后的因素(P<0.05)。多因素Cox回归分析显示PDHA1 表达不是乳腺癌患者预后的独立危险因素(表2)。另外,在年龄>60 岁、T2 期、N3 期、M0 期、病理Ⅱ期、绝经后、左乳乳腺癌患者的OS 中,PDHA1 高表达组低于PDHA1 低表达组(图5)(P<0.05)。

表2 PDHA1单因素和多因素Cox回归分析Table 2 Univariate Cox regression analysis and multivariate Cox regression analysis of PDHA1

图5 PDHA1表达与乳腺癌各亚组预后的关系Figure 5 Relationship between PDHA1 expression and prognosis of breast cancer subgroups
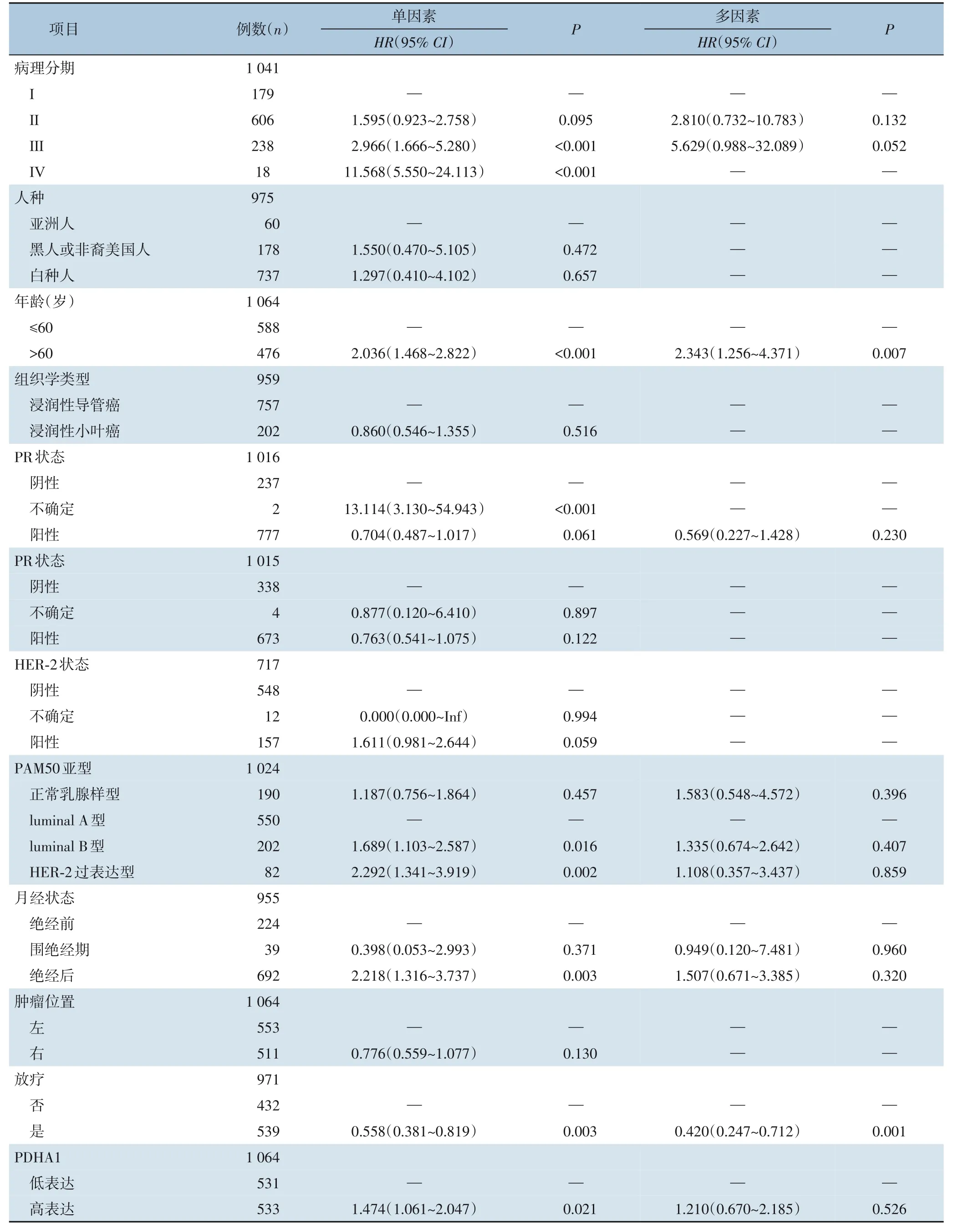
表2 PDHA1单因素和多因素Cox回归分析(续)Table 2 Univariate Cox regression analysis and multivariate Cox regression analysis of PDHA1 (continued)

图4 PDHA1表达与乳腺癌的预后的关系Figure 4 Relationship between PDHA1 expression and prognosis of breast cancer
2.6 共表达基因的基因集富集分析(GSEA)
通过GEPIA 数据库进行相关性分析,筛选得到PDHA1 的共表达基因,为了进一步探索PDHA1共表达基因相关的信号通路,对其进行GSEA 富集分析,并绘制山峦图(图6A-B)。结果显示与乳腺癌中PDHA1 共表达基因显著相关的6 条通路包括:S 期、姐妹染色单体分离、细胞周期检查点、细胞外基质组织、核心基质、三羧酸循环通路。

图6 集富集分析 A:PDHA1共表达基因基因集富集分析;B:PDHA1共表达基因基因集富集分析山峦图Figure 6 Enrichment analysis A: Gene set enrichment analysis of PDHA1 co-expressed gene;B: Mountains map for enrichment analysis of PDHA1 co-expressed gene set
2.7 化合物预测分析s
从GeneCards 数据库中得到与PDHA1 有关的化合物共10 种:辅酶A、CPI-613、硫辛酰胺、辅酶、α-酮异戊酸酯、2-氧代丁酸、丙酮酸、辅羧酶、二氧化碳、还原型烟酰胺腺嘌呤二核苷酸(图7)。其中已批准的化合物是:丙酮酸、辅羧酶、二氧化碳、还原型烟酰胺腺嘌呤二核苷酸;尚在实验中的是:硫辛酰胺、辅酶、α-酮异戊酸酯、2-氧代丁酸;尚在研究中的是:辅酶A、CPI-613。

图7 PDHA1相关化合物Figure 7 PDHA1 related compounds
2.8 构建预后列线图模型
建立一种临床适用的预测乳腺癌患者预后的列线图来预测1、3、5 年生存率(图8A)。5 个预后参数:PDHA1 表达、年龄、放疗、N 分期和M 分期被纳入预测模型。校准图显示列线图预测和实际观察之间有极好的一致性(图8B)。

图8 列线图预测模型 A:乳腺癌预后模型列线图的构建;B:乳腺癌预后列线图模型校准图Figure 8 Nomogram prediction model A: Construction of nomograms for prognosis model of breast cancer;B: Calibration curves of breast cancer prognostic nomogram model
3 讨 论
PDHA1 作为丙酮酸脱氢酶复合物的关键调控位点,发挥着连接糖酵解和三羧酸循环的重要作用,对癌症代谢转变具有重要意义。最新研究[10]发现了一种细胞体内依赖铜离子并且受到调控的新的死亡方式,即铜死亡,PDHA1 是铜死亡的相关基因,参与铜死亡过程的调控。
Sun 等[13]研究发现:肝癌中PDHA1 表达低于正常肝脏组织,其表达与肝癌的肿瘤大小、肿瘤分化和肿瘤分期相关;Song 等[11]研究发现胃癌中PDHA1 的表达低于正常胃黏膜组织,其表达与胃癌的浸润深度、淋巴结转移、TNM 分期和神经浸润显著相关;吴雅珣等研究[12]发现:子宫颈鳞状细胞癌组织中PDHA1 表达显著高于癌旁鳞状上皮组织,其表达与患者年龄、肿瘤浸润深度、盆腔淋巴结转移、FIGO 分期有关。本研究发现:与癌旁乳腺组织相比,PDHA1 在乳腺癌组织中表达下调,其表达与T 分期(P<0.001)、病理分期(P=0.031)、人种(P<0.001)、组织学类型(P<0.001)、PR 状态(P<0.001)、ER 状态(P<0.001)、PAM50亚型(P<0.001)相关。Sun 等[13]报道肝癌中PDHA1表达水平降低与患者OS 低有关;Song 等[11]报道胃癌中PDHA1 的低表达与患者较差的总体生存率显著相关。然而吴雅珣等[12]研究报道子宫颈鳞状细胞癌PDHA1 高表达患者的OS 较低表达患者的长;Chen 等[14]发现头颈鳞状细胞癌中较高的PDHA1 表达似乎与患者5 年内较差的OS 相关,而在长期(>6 年)环境中,PDHA1 表达较高的头颈鳞状细胞癌患者却表现出稳定的生存趋势;Jiang 等[25]研究发现:与正常组织相比,乳腺癌中PDHA1 的表达下调,而PDHA1 水平升高的乳腺癌患者OS、RFS均较差,这与本研究结果一致。本研究发现:乳腺癌中PDHA1 低表达组患者的OS(P=0.029)、RFS(P=0.005 1)、PPS(P=0.015)、DMFS(P=0.002 3)均较PDHA1 高表达组患者的长。另外进一步分层分析中:年龄>60 岁、T2 期、N3 期、M0 期、病理Ⅱ期、绝经后患者的OS PDHA1 高表达组低于PDHA1低表达组(P<0.05)。值得关注的是,PDHA1 在乳腺癌组织中的表达低于正常乳腺组织,但PDHA1低表达乳腺癌患者却有较好的预后,提示PDHA1可能在乳腺癌发生发展过程中不仅发挥单一的抑癌或促癌作用。
为分析PDHA1 在乳腺癌发生发展中的生物学功能及分子调控机制,本研究利用Gene MANIA 数据库绘制PDHA1 基因-基因互作网络,结果显示与PDHA1 相关互作基因有20 个,其中DLD,DLAT,PDHB 是参与铜死亡过程的基因;另外GSEA 结果显示PDHA1 共表达基因显著相关的通路共6 条,包括与铜死亡密切相关的三羧酸循环通路。Tsvetkov 等[10]研究证明铜死亡主要取决于细胞内铜的积累。一项Meta 分析[26]显示:与良性乳腺疾病和健康对照组相比,乳腺癌患者的血清铜水平显著升高。由此,PDHA1 可能通过调节铜死亡在乳腺癌中发挥关键作用。而曹静等[27]研究发现PDHA1 表达及活性下降可导致Warburg 效应增强,肿瘤细胞加速分裂,从而导致肿瘤的侵袭性增加。Liu 等[28]研究显示乙肝病毒X 蛋白结合蛋白(HBXIP)调节糖代谢重编程并下调乳腺癌中的细胞色素c 氧化酶2(SCO2)和PDHA1,SCO2和PDHA1 的下调导致乳酸产生和细胞内葡萄糖的增加,以及活性氧(ROS)的减少,促进乳腺癌细胞的生长。因此,笔者推测PDHA1 可能通过调控Warburg 效应与铜死亡过程促进乳腺癌的发生发展进而影响患者预后,这需后续实验进一步研究。
列线图对肿瘤预后的评估具有重大价值。宋张骏等[29]研究利用SEER 数据库中的临床病理资料构建的列线图预后模型可以准确预测初诊乳腺癌肝转移患者预后状态。Sha 等[30]研究将年龄、肿瘤分期和铜死亡相关基因评分(CRG-score)作为参数所构建的列线图用于预测三阴性乳腺癌患者的总生存率具有较高的准确性。本研究首次将PDHA1 的表达、年龄、放疗、N 分期和M 分期纳入预测模型构建乳腺癌患者的预后预测列线图,校准图结果显示列线图预测和实际观察之间有极好的一致性。本研究构建的列线图可为临床医生对乳腺癌患者预后的评估以及为乳腺癌患者制定个体化治疗方案提供依据。基于PDHA1 对乳腺癌患者预后具有良好的预测作用,本研究利用GeneCards 数据库寻找与PDHA1 有关的潜在相互作用的化合物,得到辅酶A、CPI-613、硫辛酰胺、辅酶等10 种可能使PDHA1 的表达发生逆转,有望成为治疗乳腺癌的化合物。
但本研究存在一定的局限:仅在数据库对乳腺癌PDHA1 的表达及预后进行了研究,缺乏进一步的体内和体外实验验证;对于PDHA1 如何调控铜死亡及Warburg 效应进而影响乳腺癌发生发展缺乏了解,后期我们将设计具体实验进一步探究。
综上所述,PDHA1 基因高表达组患者的OS、RFS、PPS、DMFS 较PDHA1 低表达组患者的短,PDHA1 表达水平是影响乳腺癌患者预后的相关因素。本研究所构建的列线图可为临床医生更准确地判断乳腺癌患者的预后,有望为乳腺癌患者制定个体化治疗方案提供依据。
利益冲突:所有作者均声明不存在利益冲突。
