高变八聚体寡核苷酸指纹基因分型法在石家庄地区布鲁氏菌分子流行病学研究中的应用
2022-11-17高会霞田岳飏李砚峰郑立恒李雅楠
张 志 高会霞 田岳飏 李砚峰 郑立恒 李雅楠
1.石家庄市第五医院检验科,河北石家庄 050021;2.河北省荣军医院检验科,河北保定 071000;3.河北省胸科医院检验科,河北石家庄 050000;4.石家庄平安医院实验诊断学部,河北石家庄 050000
布鲁氏菌病(以下简称“布病”)是由病原体布鲁氏菌感染引起的人兽共患性传染病,属自然疫源性疾病。该病在全世界范围内广泛分布,国外主要流行于地中海、中东、西亚、非洲及拉丁美洲等地区[1-6],国内多见于内蒙古、西北、东北等半牧或纯牧区[7-10]。在石家庄地区,布病多发于每年的3 月至7 月,发病人群以牛羊养殖户和农民为主[11]。近年来,随着畜牧业的发展,布病的发病率有所上升,成为重要的公共卫生问题之一[12]。
高变八聚体寡核苷酸指纹(hypervariable octameric oligonucleotide finger-prints,HOOF-Prints)基因分型法是以八个多态性可变数目串联重复序列(variable number tandem repeats,VNTR)位点为基础对布鲁氏菌进行分子流行病学研究的方法[13],能够提供菌株的亲缘关系,有助于判断新暴发或流行菌株的来源及在传播进程中的生物表型变异。本研究拟通过HOOFPrints 基因分型法[13-14]对石家庄市第五医院收治布病确诊患者的分离菌株进行分子流行病学研究,以期了解本地主要流行菌株,为本地布病防控提供理论依据。
1 资料与方法
1.1 一般资料
选取2016 年1 月至2018 年12 月石家庄市第五医院(以下简称“我院”)就诊的145 例布病确诊患者为研究对象。其中男110 例,女35 例;年龄15~85 岁,平均(48.6±15.9)岁。所有病例均为散发,病例间无明确的流行病学关联。本研究经我院伦理委员会批准(20201013)。
1.2 纳入及排除标准
纳入标准:①符合《布鲁氏菌病诊疗指南(试行)》[15]的诊断标准;②持续或间歇性发热超过7 d;③进行布鲁氏菌菌种检测,阳性结果经河北省疾控中心复核;④签署知情同意书。排除标准:①来诊前经抗菌治疗;②非石家庄市常住人口。
1.3 研究方法
1.3.1 样本提取 采集布病患者的血液、骨髓、其他体液或排泄物等标本,经分离培养获得布鲁氏菌菌株。每一菌株对应一个独立的患者。
1.3.2 PCR 使用Qiagen 公司的MericonTMDNA Bacteria Kit 按照试剂说明书进行操作。用无菌接种环刮取培养基上生长良好的菌落,置于含200 μl 细菌裂解液的2 ml 螺旋帽离心管中,拧紧螺帽,于振荡器中充分振荡5 min。100℃金属浴10 min。待离心管恢复到室温后,以13 000 g 离心5 min(离心半径为10 cm),取上清液备用。参照文献[13]进行,针对8 个VNTR 位点分别设计8 对特异性引物(表1),由北京奥科鼎盛生物科技有限公司合成。反应体系共20 μl:DNA 2 μl,正向引物1 μl,反向引物1 μl,PCR mix 10 μl,ddH2O 6 μl。94℃预变性5 min,94℃变性30 s,55℃退火40 s,72℃延伸30 s,35 个循环;72℃充分延伸10 min。PCR产物送北京奥科鼎盛生物科技有限公司测序。
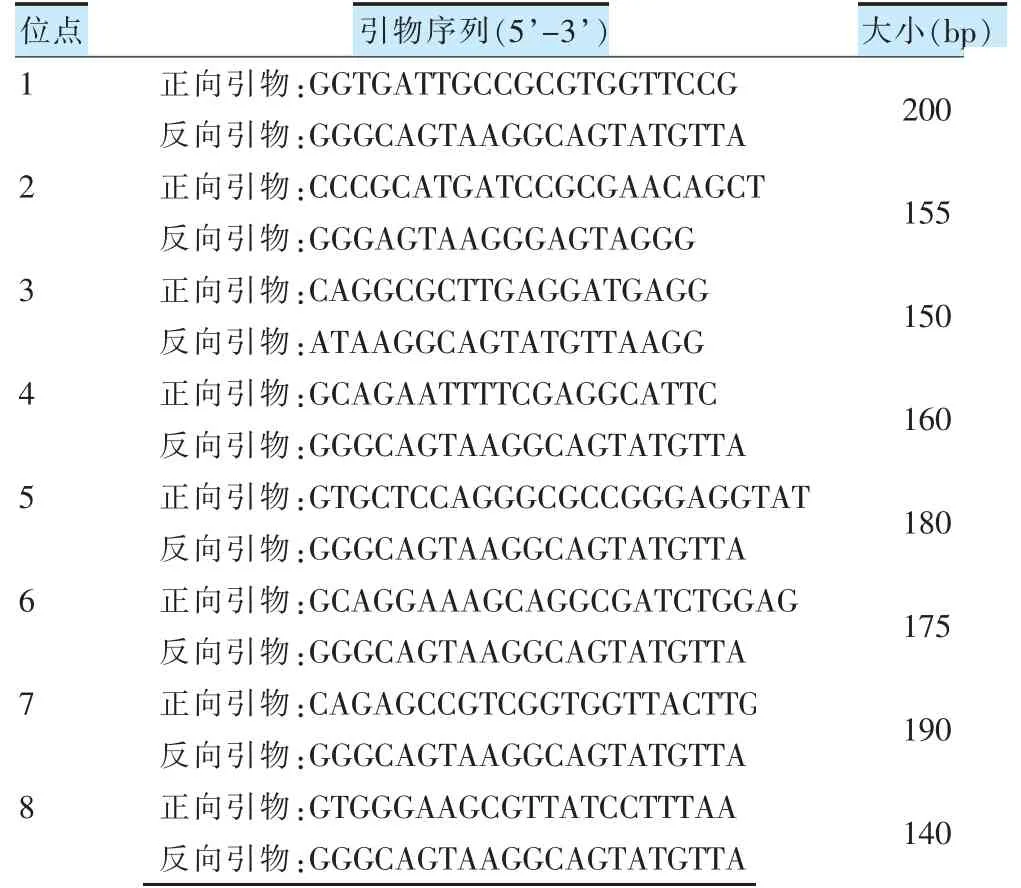
表1 8 个VNTR 位点正反向引物
1.4 构建系统树图
使用BioNumerics 8.0 软件中非加权组平均法生成系统树图。
2 结果
2.1 145 株布鲁氏菌测序结果
145 株布鲁氏菌每个位点重复次数不同,基本序列为AGGCAGT,在第一位和第二位碱基位点存在个体变异,如gGGCAGT、AaGCAGT,重复次数不等。见图1。
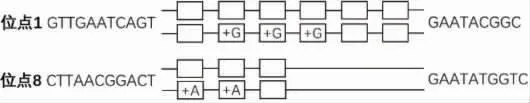
图1 部分VNTR 测序结果
2.2 145 株布鲁氏菌的系统树图
本研究未发现石家庄地区有其他生物型别的布鲁氏菌分布。从系统树图可以看出,同一生物种的布鲁氏菌成簇聚集分布。145 株布鲁氏菌分布在两个大的分枝上,一枝以羊种(B.melitensis biover)为主,一枝以牛种(B.abortus biover)为主。羊种分枝占比最大,为90.34%(131/145)。其中,110 株与羊3 型进化关系最近,占全部菌株的75.86%(110/145),其余21 株与羊2 型进化关系最近,占全部菌株的14.48%(21/145)。牛种分枝占比小,为9.66%(14/145),其中牛1、2、4、6型分别占0.69%(1/145)、0.69%(1/145)、5.52%(8/145)和2.76%(4/145)。见图2。
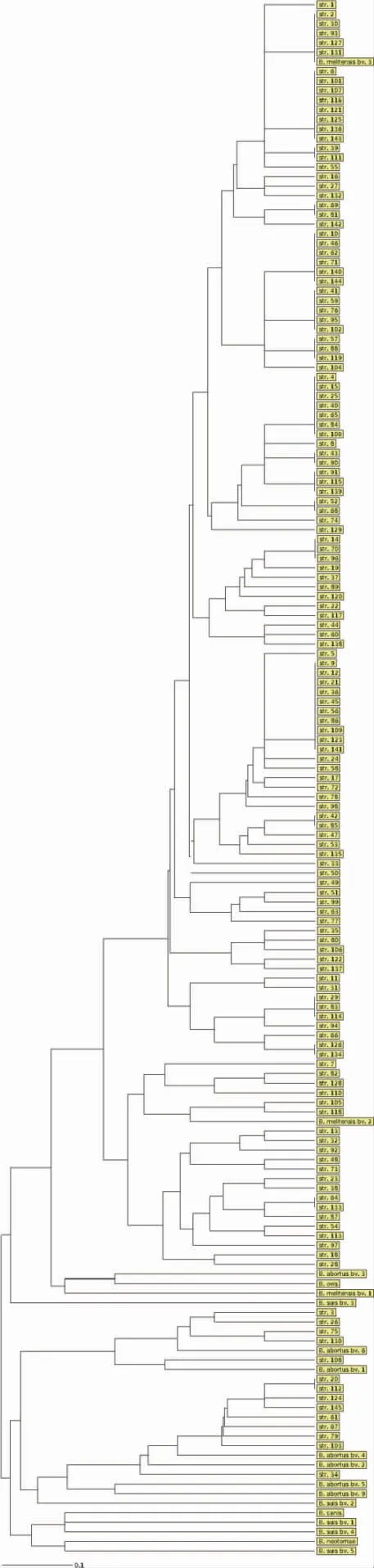
图2 145 株布鲁氏菌的系统树图
3 讨论
布病起病隐匿,可以模拟多系统疾病,表现出广泛的临床多态性,为及时的临床诊断带来诸多困难,往往是排除各系统疾病后,由布病的细菌学和免疫学指标提供诊断依据[16-19]。因此,进行布病的流行病学研究能够有助于了解本地布病的流行季节、人群分布、疾病特征等。分子流行病学能够从基因水平阐明布病的进化规律和流行趋势,为布病的溯源和防控提供理论依据。
布鲁氏菌常用的分型方法包括血清学检测和生化检测等方法。前者存在一定的假阴性或假阳性结果;后者操作繁琐,只能在特殊检测机构进行检测。HOOF-Prints 基因分型法是以多态性VNTR 位点为基础对布鲁氏菌进行分子流行病学研究的方法[13],能够提供菌株的亲缘关系,有助于判断新暴发或流行菌株的来源,以及在传播进程中的生物表型变异,为科学有效地控制布病的流行提供科学依据。随着分子生物学的发展,基于VNTR 基因分型研究被广泛应用于多种病原微生物的分型[20-22]。由于其高度的遗传多态性,在分子流行病学研究中发挥着重要作用。目前,国内对布鲁氏菌分子流行病学的研究尚不多见,采用HOOF-Prints 基因分型法对石家庄地区布鲁氏菌进行基因分型的研究也尚未见报道,故本研究对石家庄地区145 株布鲁氏菌进行分子流行病学研究,以期了解本地的主要流行菌株及进化关系。
布鲁氏菌分为6 个生物种,19 个生物型(biover),分别为羊种(B.melitensis biover 1~3),牛种(B.abortus biover 1~7、9),猪种(B.suis biover 1~5),绵羊附睾种(B.ovis),沙林鼠种(B.neotomae)和犬种(B.canis)。在临床上以羊、牛、猪种最为常见,其中羊种的致病力最强[23-26]。从系统树图看,石家庄地区布鲁氏菌的流行生物种以羊种和牛种为主,羊种所占比例最大。其中,羊3 型和羊2 型为主要流行菌株,个别菌株呈聚集分布,牛种以牛4 型和牛6 型最常见,均为散在分布,这与我国布病疫情监测结果相一致[27]。从菌株的进化关系来看,石家庄地区布病呈散发流行,虽有个别菌株呈聚集分布,但在时空分布上并无关联,提示羊型可能是石家庄地区的优势流行菌株,应重点监测其变化趋势。菌株在进化过程中存在一定的基因变异,但进化距离相差不大,提示石家庄地区布鲁氏菌呈多态性分布,但进化进程缓慢,仍处于可防可控的范围内。
HOOF-Prints 基因分型法对布鲁氏菌的分辨率较高,能够将不同的菌株进行区分,同时又能将同一生物型别菌株进行聚类,在布病分子流行病学研究中具有优势。但是,本研究也存在一定的缺陷。第一,研究样本较小,且仅为横断面研究,无法深入挖掘布鲁氏菌的传染来源和进化过程;第二,本研究未将分子流行病学特征与患者的临床特征、实验室结果、耐药情况等进行相关分析。下一步,将进行纵向研究,将分子流行病学与临床相结合,为石家庄地区布病疫情防控提供理论依据。
