卡拉胶酶的来源、性质、结构与应用研究进展
2022-10-24郭子龙唐天城徐寅啸朱本伟宁利敏
郭子龙,唐天城,徐寅啸,朱本伟,姚 忠,宁利敏
(1.南京工业大学 食品与轻工学院,江苏 南京 211800)(2.南京中医药大学 医学院·整合医学院,江苏 南京 210023)
卡拉胶是红藻(如Eucheumamuricata和Gracilarialemaneiformis)细胞外基质的重要组成部分,是一种由D-半乳糖通过交替的α-1,3和β-1,4糖苷键连接而成的线性硫酸多糖[1]。天然的卡拉胶往往不是均一的多糖,而是多种构型的混合物。为满足卡拉胶这种复杂结构的研究需要,Knutsen等[2]提出了以大写字母代替特定基团的命名方法(图1),根据硫酸基团的数量和位置以及α-1,4-连接的-半乳糖残基中3,6-内醚-α-D-吡喃半乳糖(3,6-AG)的存在,将其分为κ-、ι-和λ-型3种卡拉胶,所对应的重复二糖结构分别为G4S-DA、G4S-DA2S和G2S-D2S,6S[3]。由于卡拉胶具有良好的成胶能力、化学稳定性和多种生理活性,卡拉胶在食品、饮料和化妆品等行业都得到了广泛的应用,还可作为食品增稠剂、澄清剂和凝胶剂等[4-6]。然而,这种硫酸多糖的溶解性差、生物利用度低,其应用也受到了限制[7]。卡拉胶寡糖是卡拉胶的降解产物,具有多种生物活性,如抗炎[8]、抗凝血[9]和抗肿瘤等活性[10-11],此外,它们还具有良好的溶解性和生物利用度[12]。因此,卡拉胶寡糖的制备方法引起了人们越来越多的关注[13],其中,化学水解和酶降解是较为成熟的制备方法。由于化学水解反应过程往往过于剧烈,在水解过程中会造成产物结构的破坏,因此其应用受到了极大的限制[14]。而利用卡拉胶酶进行特异性降解的方式由于具有反应条件温和、产物特异性好等优点,所以被认为是一种具有广阔前景的方法[15]。
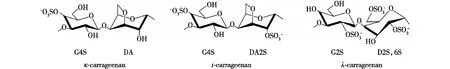
图1 不同构型的卡拉胶结构示意[2]Fig.1 Schematic diagram of carrageenans with different structures[2]
卡拉胶酶属于糖苷水解酶,目前已发现的卡拉胶酶在作用模式上均属于内切酶,该酶作用于卡拉胶分子内的β-1,4糖苷链,产生一系列聚合度为偶数的卡拉胶寡糖。多种海洋细菌均具备产生卡拉胶酶来利用这种多糖的能力,这对海洋中的碳循环至关重要。本课题组从海洋细菌PedobacterhainanensisNJ-02中克隆、异源表达和表征了编码新κ-卡拉胶酶的基因(cgkA),该酶可将卡拉胶内切为κ-卡拉胶四糖与六糖,这对于生产高聚合度(Dps)值的卡拉胶寡糖是一种具有潜在价值的酶工具[16]。近年来,海洋微生物降解和利用卡拉胶的研究主要集中在卡拉胶酶的性质、结构功能分析和生化途径的阐明等方面。本文总结了不同来源的卡拉胶酶的分类和性质,还研究了其结构与功能之间的关系,为推动卡拉胶酶的深入研究和开发应用奠定坚实的理论基础。
1 卡拉胶酶来源、种类、性质与降解模式
卡拉胶酶主要从海洋细菌和海洋动物的消化液中分离得到。Skea等[17]首次成功地从海洋软体动物中纯化并表征了卡拉胶酶。迄今为止,所有报道的卡拉胶酶都具有严格的底物专一性,仅能降解一种构型的卡拉胶。因此,可将卡拉胶酶分为κ-卡拉胶酶(EC 3.2.1.83)、ι-卡拉胶酶(EC 3.2.1.157)和λ-卡拉胶酶(EC 3.2.1.162)。表1列出了不同来源卡拉胶酶及其酶学性质。由表1可知:能够产生卡拉胶酶的细菌包括Pseudoalteromonas、Pseudomonas、Shewanella、Vibrio、Cellulophaga、Cytophaga、Tamlana、Microbulbifer、Alteromonas、Pedobacter和Zobellia等,其中,κ-卡拉胶酶分布最为广泛,属于糖苷水解酶16家族。目前,已报道有多种菌株可以产生κ-卡拉胶酶,如Shewanellasp.Kz7[14]、Pseudoalteromonassp. ZDY3[18]、Pseudoalteromonassp.Q203[19-20]、Pseudoalteromonassp.Q202[21]、P.aeruginosaZSL-2[22]、P.carrageenovoraHLX250[23]、P.elongataMTCC 5261[24]、P.porphyraeLL-1[25]、Pseudoalteromonassp.WZUC10[26]、P.carrageenovoraNCMB 302[27]、Pseudoalteromonassp.AJ5-13[28]、P.tetraodonisJAM-K142[29]、Vibriosp.SY01[30]、Vibriosp.NJ-2[31]、Vibriosp.CA-1004[32]、Tamlanasp.HC4[33]、P.hainanensis13-Q[34]和A.macleodiiKS62[35]等。海洋黄杆菌Z.galactanivorans可以降解琼脂、褐藻胶和卡拉胶等多种多糖[36]。它能够通过产生κ-卡拉胶酶来降解和利用红藻多糖[37-39]。菌株CellulophagalyticaN5-2也可以通过产生κ-卡拉胶酶降解多糖[9,40]。Cytophagastrains MCA-2和Ik-C783也被报道可以产生κ-卡拉胶酶[41-42]。Kalitnik等[43]从红藻Tichocarpuscrinitus中筛选出28株具有降解κ-卡拉胶能力的菌株,且它们均属于Bacteroidetes和Proteobacteria。Kang等[44]通过构建琼脂酶、κ-卡拉胶酶和新琼脂二糖酶的混合体系来降解红藻类底物,发现其还原糖的产量是单酶还原糖产量的3.6倍。Li等[45]从一种温泉细菌BacillusBI-19中获得了一段编码α-淀粉酶的基因amy19,异源表达后发现重组Amy19具有降解κ-卡拉胶的活性。同样地,Liu等[46]在海洋弧菌Vibrioalginolyticus63中也克隆得到了一个具有κ-卡拉胶降解活性的α-淀粉酶。Wu[47]采用商品化的α-淀粉酶来降解卡拉胶制备卡拉胶寡糖也证明了一些α-淀粉酶具有卡拉胶酶的活性。此外,一些革兰氏阳性菌如芽孢杆菌可以降解琼脂和卡拉胶。Bacillussp. SYR4通过利用其产生的琼脂酶和卡拉胶酶(主要是κ-卡拉胶酶)的活性来降解利用红藻废料[48],Bacillussp. HT19也可以通过产生κ-卡拉胶酶利用卡拉胶多糖[49]。Yu等[50]通过利用双启动子、共表达伴侣蛋白和转录因子增强了重组κ-卡拉胶酶在Pichiapastoris中的表达。Wang等[51]表征了一种深海细菌Bacillussp. N1-1的全基因组,并发现其可以降解κ-硒代卡拉胶。在海洋无脊椎动物Turbocornutus的肠道中可以分离出一种能降解琼脂、藻酸盐和κ-卡拉胶的MicrobulbiferagarilyticusGP101,同时该菌株的基因组测序结果被公布[52]。在Cellulosimicrobium培养体系中也检测到κ-卡拉胶酶活性[53-54]。
对于ι-卡拉胶酶的报道较少,仅有Altertermonasfortis[55]、Cellulophaga[56-57]、Microbulbifer[58]、Flavobacterium[59]和Wenyingzhuangia[60]等少数菌株。ι-卡拉胶酶首次从Altertermonasfortis中被提取并表征[55]。更重要的是,该酶的三维结构和催化作用机制也都得以阐明[61]。Xu等[62]开发了一种新的整合载体(pBCGA),用于在Brevibacilluschoshinensis中高效表达优化的ι-卡拉胶酶基因(CGIOP),并纯化表征了该基因的重组酶rCgiA。Hatada等[58]在深海细菌MicrobulbiferthermacoleansJAMBA94T中发现了一种新的ι-卡拉胶酶,并通过定点突变来阐释了其催化机制。Liu等[46]从海洋细菌Flavobacteriumsp.YS-80-122中克隆并表征了一种温度稳定性较高的ι-卡拉胶酶。Ma等[56-57]从Celluphagasp. QY3中鉴定了两种新的ι-卡拉胶酶。Shen等[60]从Wenyingzhuangiafucanilytica中克隆了一种编码GH82家族新的ι-卡拉胶的基因并对重组酶的性质进行了研究。Muzyed等[63]通过单因素试验对Cellulophagabaltica生产ι-卡拉胶酶的工艺进行优化,结果发现优化后酶的比酶活比未优化时的比酶活高26倍。
关于λ-卡拉胶酶的报道很少,更缺少相关的基因序列信息,因此它构成了与κ-卡拉胶酶和ι-卡拉胶酶不相关的新GH家族[64]。目前,λ-卡拉胶酶的相关报道仅有为数不多的几种,包括一种热泉细菌Bacillussp. Lc50-1,它可以生产一种温度稳定性较高的λ-卡拉胶酶[65]。Naik等[66]分离出3株具有生产琼脂酶、λ-卡拉胶酶、淀粉酶、纤维素酶和蛋白酶能力的Ulvalactuca类细菌。另外,有2种λ-卡拉胶酶的基因已经被克隆,一种来自海藻细菌Pseudoalteromonascarrageenovora[67],另一种来自深海细菌Pseudoalteromonassp. CL19[68]。这些λ-卡拉胶酶基因的序列高度相似(同源性98%),序列信息的缺乏导致了至今仍没有将其定义为CAZy的家族成员。随着微生物基因组学不断发展,越来越多具有降解多糖能力的海洋微生物被基因测序[36-37],因此,研究者通过对CAZy数据库中的基因数据分析,阐明了水解酶降解多糖的原理并发现了一些菌株中与编码其水解功能有关的关键基因[69]。因此,发现具有优良特性且具有工业应用潜力的新型卡拉胶酶显得尤为重要。
不同来源的卡拉胶酶有着不同的分子量,其大小分布较广(3.0×104~1.28×105)。如,来自Cytophagasp.MCA-2的CgkMCA-2[41]和来自Pseudoalteromonassp.ASY5的CgkASY[70]的分子量都是3.0×104。来自Pseudoalteromonassp. QY203的CgkP[19]、来自Pseudoalteromonassp. QY203的CgkP203[20]、来自Vibriosp. NJ-2的CgkA[31]、来自Vibriosp.的CgkCA[71]、来自Pseudoalteromonascarrageenovora的CgkPc[72]和来自Pseudoalteromonassp. AJ5-13的CgkPAJ5[28]都有着相近的分子量(约为3.5×104)。被表征的卡拉胶酶大多分子量为4.0×104~6.0×104,如来自PseudoalteromonasporphyraeLL-1的CgkP(4.0×104)[25]、来自PseudoalteromonastetraodonisJAM-K142的CgkA(4.3×104)[29]、来自Pseudoalteromonassp. QY203的CgkX(4.4×104)[19]、来自Shewanellasp. Kz7的CgkS(4.5×104)[14]、来自Zobelliasp. ZM-2的CgkZM-2(4.5×104)[73]和来自Pseudoalteromonassp. WZUC10的CgkP(4.5×104)[26]等。同时,来自CellulophagalyticaN5-2的Cly-κ-CAR(5.5×104)[40]、来自Zobelliagalactanivorans的CgkA(5.7×104)[37]、来自Tamlanasp. HC4的CgkT(6.64×104)[33]、来自Pedobacterhainanensis的CgkP(6.5×104)[34]、来自Cellulophagasp. QY3的CgiA(5.58×104)[56]、来自Cellulophagasp. QY3的CgiB(5.06×104)[57]、来自MicrobulbiferthermotoleransJAMB-A94T的CgiM(5.5×104)[58]则呈现出较大的分子量。另外,来自Cytophagasp. lk-C783的两种κ-卡拉胶酶(Cglk)[42]、来自Pseudomonaselongata的CgkPe[24]、来自Pseudoalteromonascarrageenovora的两种λ-卡拉胶酶(CglPc)[64]、来自Pseudoalteromonassp. CL19的CglCL19[68]则呈现出更大的分子量(>1.0×105)。绝大多数表征过的κ-卡拉胶酶的最适温度在30~40 ℃。但是,来自PseudoalteromonasporphyraeLL1的CgkP[25]、来自Pseudoalteromonassp. QY203的CgkX[19]、来自Pseudoalteromonassp. AJ5-13的CgkAJ5[28]和来自PseudoalteromonastetraodonisJAM-K142的CgkA[29]则都在55 ℃时展现出最大酶活性。另外,来自Pseudoalteromonassp. ASY5的CgkASY最适温度是60 ℃[70]。有趣的是,来自Cytophagasp. lk-C783的Cgklk在25 ℃环境下酶活性最高,这主要是由酶的分离环境所决定[43]。从温泉细菌Bacillussp. Lc50-1中分离的λ-卡拉胶酶最适反应温度是75 ℃,展现出工业应用的巨大潜力[7]。几乎所有表征过的酶的最适pH为7.0~8.0,但是来自Pseudomonaselongata的CgkPe最适pH为5.6~7.7[24]。对于卡拉胶酶促动力学,不同的酶相应的Km也会有所不同。来自Shewanellasp. Kz7的CgkS(0.15 mg/mL)[14]和来自Zobelliasp. ZM-2的CgkZM-2(0.84 mg/mL)[77]的Km比其他卡拉胶酶的低,表明这两种酶对于卡拉胶底物的亲和度相比较于其他酶更高。其他的κ-卡拉胶酶,如来自PseudoalteromonasporphyraeLL1(4.4 mg/mL)[25]、Vibriosp. CA-1004 (3.3 mg/mL)[32]、Tamlanasp. HC4(7.63 mg/mL)[33]、Pseudoalteromonassp. AJ5-13 (9.8 mg/mL)[28]和Pseudoalteromonaselongata(6.6 mg/mL)[24]的卡拉胶酶则显示出对卡拉胶底物较低的亲和力。
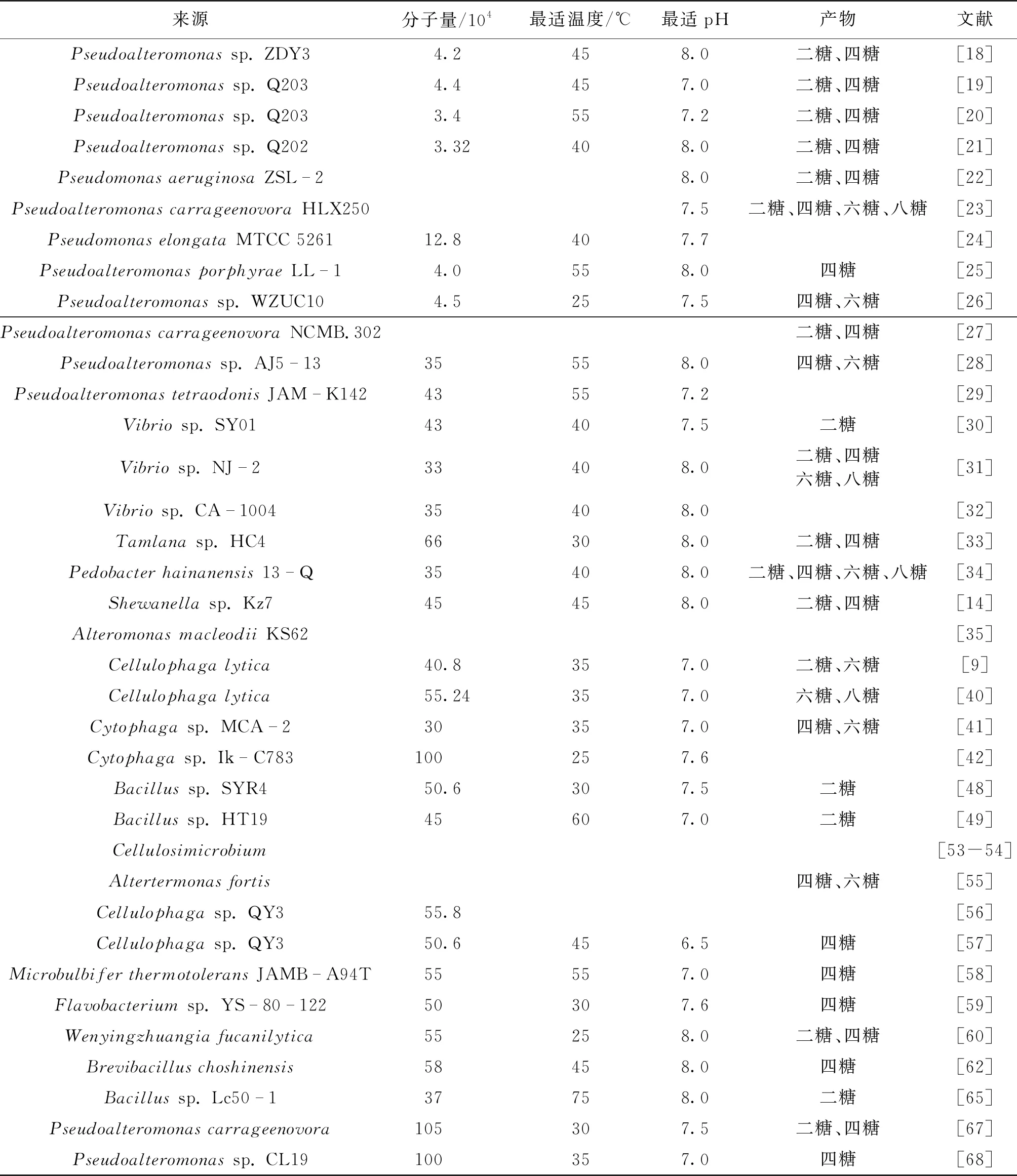
表1 不同来源卡拉胶酶的酶学性质
不同的金属离子和化学试剂对不同细菌来源的卡拉胶酶活性有不同的影响,这在学术界已经达成了共识。大多数情况下,Na+、K+、Ca2+和Mg2+可以激活酶活性。特别是高浓度Na+(达到500 mmol/L)可显著提高酶活性[28]。据推测应该是高浓度NaCl会改变卡拉胶的物理性质,从而影响酶的反应。然而,K+对来自Pseudomonaselongata的CgkPe[24]、来自Tamlanasp. HC4的CgkT[33]和来自Brevibacilluschoshinensis的rCgiA[62]均有抑制作用。几乎所有的卡拉胶酶都被Co2+、Zn2+、Cu2+、Pb2+和Hg2+等重金属所抑制,可能是金属离子与活性中心的催化残基相互作用,从而改变了酶的构象。所有的卡拉胶酶都能将卡拉胶水解成偶数寡糖,这说明它们的作用模式为顺向内切。κ-和ι-卡拉胶酶沿着多糖链依次水解所有糖苷键,得到聚合度(DP)分别为2和4的寡糖[72,74]。而λ-卡拉胶酶则随机破坏糖苷键,产生大量聚合度为6的寡糖[64]。偶数聚合度寡糖的产生,主要取决于卡拉胶的重复二糖单元和相应的水解机制。据推测,含有4个残基的四糖链起到了与底物结合和降解的作用[38]。综上所述,这3种卡拉胶酶都是内切型水解酶,它们可以破坏内部糖苷键,而不是水解还原端和非还原端单元。
2 不同种类的卡拉胶酶序列分析
到目前为止,已经10余个κ-卡拉胶酶的基因被克隆和鉴定[14,18,28,32,37,40,50]。然而,来自P.porphyraeLL-1的CgkP和来自P.tetraodonisJAM-K142的CgkA与来自P.carrageenovora的酶具有很高的同源性(分别为92%和98%)[24,28]。因此,这3种酶被认为是同一种卡拉胶酶。对这3种酶进行多序列比对,结果如图2所示。

图2 不同来源的κ-卡拉胶酶的多序列对比Fig.2 Multi-sequence alignment of κ-carrageenases from different sources
由图2可发现,3个高度保守的区域,即“GVC/SPS/AFW”“YSEIDVVEL”和“TLSL/QGLR”[74]。更重要的是,参与底物结合和活性催化所必需的几个残基在这些序列中都高度保守,如Glu171、Asp173、Glu176、Asp194、Leu197、Ser271和Leu274,其中3个残基(Glu171、Asp173和Glu176)参与底物的结合[72]。根据CAZy数据库的分类,κ-卡拉胶酶属于GH16水解家族,这个家族包括了许多具有不同底物特异性的水解酶,如β-琼脂酶、地衣酶、海带酶和β-半乳甘露聚糖酶。这些水解酶拥有类似的基因序列,因此推断是从共同的祖先进化而来。值得注意的是,κ-卡拉胶酶已从β-琼脂水解酶分支中分离出来[74],但考虑到κ-卡拉胶和琼脂的结构相似性,将他们的2种水解酶都归类于GH-B族,并猜测其具有相同的底物降解机制。
目前,已经有8个ι-卡拉胶酶的基因被克隆分析。首先是从Alteromonasfortis中克隆了第一个ι-卡拉胶酶基因,并由此定义了GH82家族[55]。Ma等[56-57]从Cellulophagasp.QY3中克隆并鉴定了2个ι-卡拉胶酶基因。Hatada等[58]发现了来自MicrobulbiferthermotoleransJAMB-A94T的一种新的ι-卡拉胶酶基因。Li等[59]从细菌Flavobacteriumsp.YS-80-122中克隆了一种新的耐冷热的ι-卡拉胶酶基因。Shen等[60]从Wenyingzhuangiafucanilytica中克隆了一种新的ι-卡拉胶酶的基因。Xu等[62]从Brevibacilluschoshinensis中克隆表达了一种ι-卡拉胶酶基因。图3展示了ι-卡拉胶酶的多序列对比结果。由图3可知,对催化活性来说,关键的残基(即Glu364和His406)在序列中高度保守。此外,参与底物结合的残基(如Gly336、Tyr337、Gly338、Gln341和Arg362)在序列中也呈现保守的状态[74]。目前为止,只发现了2种λ-卡拉胶酶的基因,它们的同源性达到98%。

图3 不同来源的ι-卡拉胶酶的多序列对比Fig.3 Multi-sequence alignment of ι-carrageenases
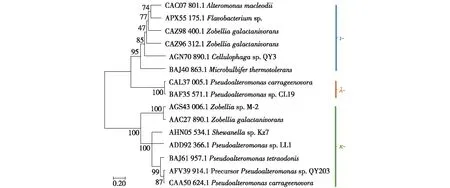
图4 不同家族的卡拉胶酶的系统进化分析Fig.4 Phylogenetic analysis of carrageenases from different families
为了阐明3个不同类群之间的进化关系,在序列比对的基础上构建了系统发育树,如图4所示。由图4可知:3种卡拉胶酶聚集成了3个小的分支,说明与κ-卡拉胶酶相比,λ-卡拉胶酶比ι-卡拉胶酶具有更高的同源性。由于缺乏λ-卡拉胶酶的序列信息,这一结论未得到证实[75]。虽然这3种卡拉胶酶对结构相关的底物具有特异性,但是它们在序列上的同源性较低。这些酶主要通过识别3种卡拉胶中的半乳糖重复单元的硫酸化模式来区分它们的底物。在结构上,κ-卡拉胶由中性糖残基和负电荷糖残基组成,而κ-卡拉胶酶则通过与一些保守残基(Glu171、Asp173和Glu176)的相互作用来识别底物[75]。ι-卡拉胶由带负电荷的二糖单元组成,其反应机制可能是底物与保守的精氨酸残基反应结合在一起[76]。
3 卡拉胶酶的结构和催化机制
卡拉胶酶在结构层面上的表征和研究还不够深入,但是随着结构生物学技术的发展,越来越多的卡拉胶酶的结构得以解析。到目前为止,有4种卡拉胶酶的晶体结构被完全表征,即来自Pseudoalteromonascarrageenovora的κ-卡拉胶酶(CgkA)[74]、来自ZobelliagalactanivoransDsiJT的ZgCgkA[37]、来自Alteromonasfortis的ι-卡拉胶酶(CgiA)[61]以及来自Bacteroidesovatus的外切卡拉胶酶BovGH167[77]。CgkA和ZgCgkA都属于β果冻卷折叠,主要由2个紧密包裹的弯曲反平行β板构成(图5),形成了一个隧道活动场所,暗示着一种过程作用模式。根据二级分析,该折叠由3张小的反平行β板和一条α-螺旋组成。ZgCgkA与CgkA在结构上没有显著性差异[38],而其中一些细微的结构差异可以通过结构叠加来加以强调,包括催化通道的末端、附加的手指、存在于ZgCgkA顶部的一个螺旋等。这2种酶的机制是双取代机制,涉及2个催化羧基,相应的亲核成分和酸碱催化残基分别为E163(亲核)和E168(酸碱)。E168只与4种溶剂分子进行氢键连接,它们是催化进入水分子的候选分子。相反,E163与Y161定向地通过氢键牢固连接,存在于目前所表征的2种κ-卡拉胶酶中[37]。
图5(c)为CgiA晶体结构,它采用右旋β-螺旋折叠,与2个κ-卡拉胶酶的β果冻卷折叠无关。与CgkA和ZgCgkA相比,CgiA在C末端区具有2个附加的结构域((a)和(b))。结构域(a)类似于DNA/RNA结合蛋白并显示了一个α/β-折叠,而结构域(b)由具有2个二硫键组成的环构成。与底物络合的CgiA的结构表明其中有几个残基参与底物结合, 即Asn123、Arg125、Lys163、Lys394、Arg243、Arg303、Gly423、Gln424、Tyr341、Arg321和Arg353。当底物与这些残基结合时,结构域(a)经历剧烈的构象变化,向β-螺旋裂移动,因此形成一个含有四糖底物的隧道。同时,二聚体结合在N末端区,一个开放的构象到一个封闭的隧道类似于一个启动开关,这就从一个合理的角度解释了这种高效的行动模式[61]。
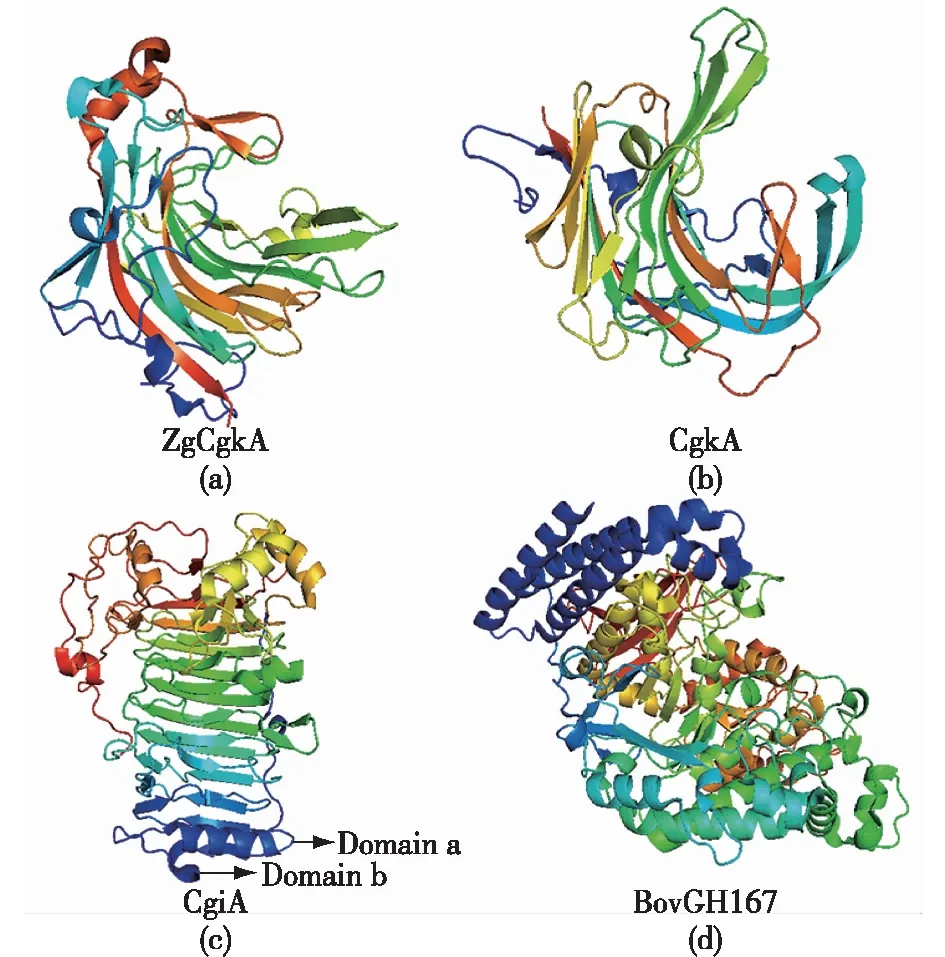
图5 不同家族的卡拉胶酶的结构Fig.5 Structures of carrageenases from different families
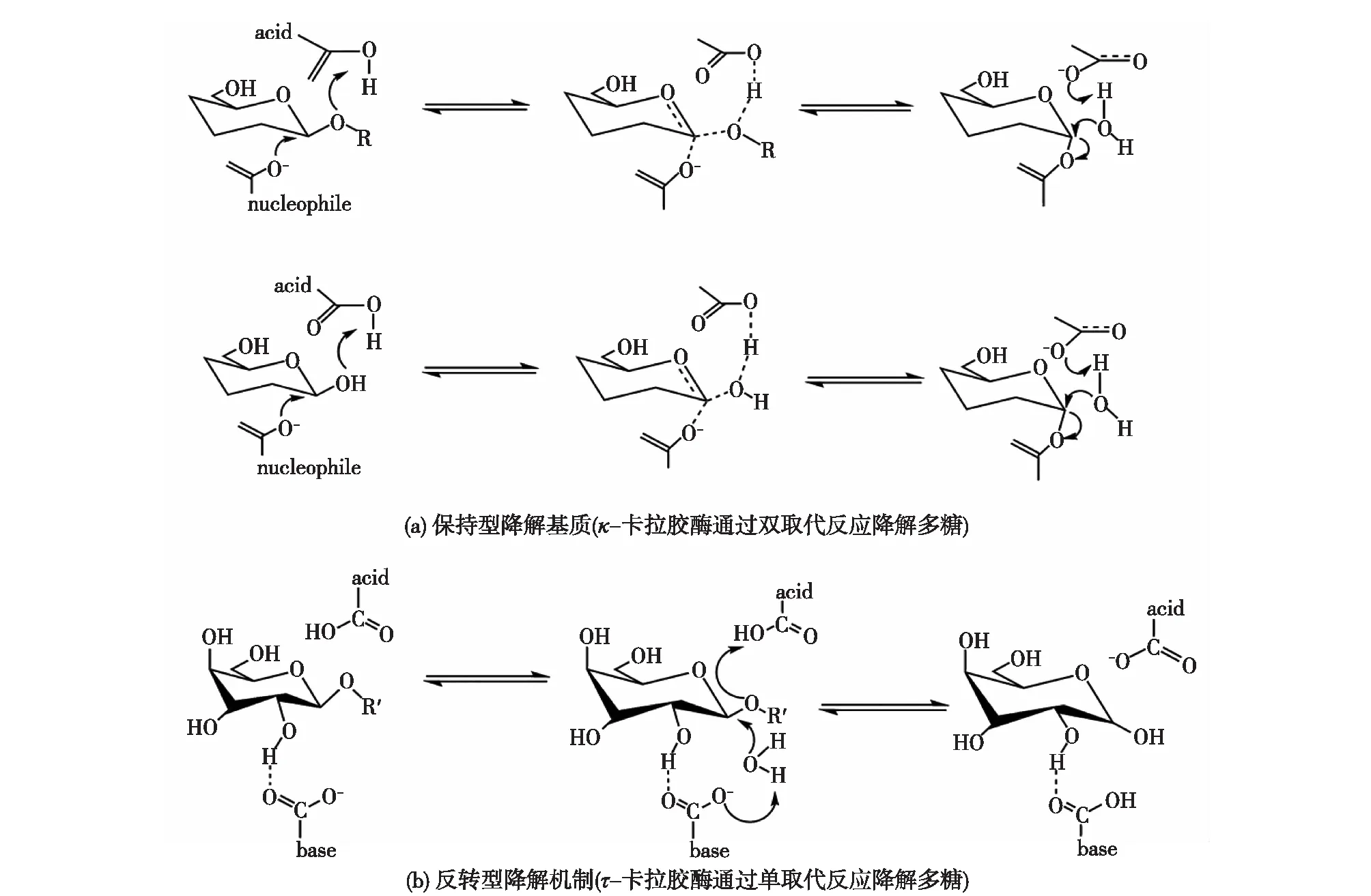
图6 卡拉胶的降解机制Fig.6 Presentation of retaining mechanisms for the degradation of carrageenan
图6为卡拉胶酶的反应机制。Glu245在CgiA中充当催化质子供体的作用,而Asp247是激活催化水分子的通用碱。另外,3个残基间接参与酶活性的激活:Gln222在连接催化H2O和Cl-的同时发挥构建活性位点中水网络的基本作用;His281参与ι-卡拉胶的结合并可能参与Glu245的质子交换;而Glu310则起到了稳定底物中间构象的作用。但是,由于缺乏对卡拉胶序列的认知,卡拉胶的结构信息仍然不足。微生物基因组学的发展为发现新的卡拉胶提供了有效的途径。随着高通量测序技术和生物信息学的进展,可以更高效地获得定制酶的序列信息。此外,通过结合高通量筛选和最新的结构生物学技术,如低温电子显微术,使得深入了解这些酶的结构和反应机制成为可能。总之,新技术的发展与应用无疑将有利于研究卡拉胶的结构信息,为其工业应用奠定坚实的基础。
4 卡拉胶酶的生产、纯化及活性测定方法研究
现有多种方法从复杂的发酵系统中提取卡拉胶酶以对其进行更加深入的研究。为了从野生菌株中提取卡拉胶酶并以此构建重组工程菌株,规模化培养是现在采用最多的方式。Dyrset等[78]构建了卡拉胶酶的发酵系统,最终最大酶活达到84 000 U/L;此外,他们用等量的谷氨酸取代了酪氨酸,得到的最高酶活比先前报道的酶活高出17倍。Ziayoddin等[22]采用正交试验法对PseudomonasaeruginosaZSL-2的培养条件进行了优化,结果发现生产的卡拉胶酶的最大酶活(257 U/mL)在对数后期出现,随后卡拉胶酶的产量出现了下降的趋势。Khambhaty等[79]对Pseudomonaselongata生产的κ-卡拉胶酶培养基组分进行了统计优化后发现,与文献[80]报道的培养基相比,κ-卡拉胶酶的产量增加了32倍(32.08 U/mg)。Guo等[81]采用Box-Behnken实验设计并运用响应面法,优化了Thalassospirasp. Fjfst-332生产卡拉胶酶的培养条件,结果发现Thalassospirasp. Fjfst-332的酶活最高为267 U/mL,是目前为止发现产κ-卡拉胶酶最具活力的野生细菌;同时在磁性Fe3O4-壳聚糖载体上对该菌株进行了固定化,以制备κ-卡拉胶酶和κ-卡拉胶寡糖,与游离菌相比,固定化菌株能在温度更高、pH范围更广的情况下生产κ-卡拉胶酶。更重要的是,产生的κ-卡拉胶酶活性至少可以持续7个循环[82]。
为了扩大卡拉胶酶的生产规模,可采取克隆它们的基因,然后在工程菌株中进行异源表达的方法。Liu等[83]利用野生型信号序列增加κ-卡拉胶酶在Escherichiacoli的胞外生产,结果发现,生产的51%的卡拉胶酶被分泌到培养基中,33%的卡拉胶酶积累到周质空间中。这种重组κ-卡拉胶酶的高水平胞外分泌在卡拉胶酶的生产中有着广泛的前景。Zhao等[84]在从Brevibacilluschoshinensis中生产ι-卡拉胶酶时建立了一个高水平的胞外表达系统,胞外ι-卡拉胶酶的最大酶活达到(51.5±1.2) U/mL。Yu等[85]将κ-卡拉胶酶基因插入毕赤酵母中,摇瓶发酵120 h后,重组酶的酶活达到4.68 U/mL。为了提高卡拉胶酶在工业应用中的稳定性,已经利用酶的固定化方法。Xu等[86]以戊二醛为偶联剂,将来自Pseudoalteromonascarrageenovora的κ-卡拉胶酶固定在羧基功能化的磁铁纳米粒子中,测定固定化酶的比酶活和回收率分别为326.0 U/g和46.9%。随后,Xiao等[87]制备了磁性氧化金属纳米粒子,并在纳米粒子上固定了来自Pseudoalteromonassp. ASY5 的κ-卡拉胶酶,结果发现,固定化后的κ-卡拉胶酶具有更低的热稳定性和更好的pH稳定性和储存稳定性;此外,固定化的κ-卡拉胶酶在使用4次后依旧保持了原活性的43.5%,但是对底物的亲和力有所降低。从复杂的蛋白质混合物中纯化卡拉胶酶的过程通常由几个连续的操作组成。(NH4)2SO4沉淀通常作为从野生型菌株发酵系统中纯化卡拉胶酶的初始步骤[9,20-21,23-24,26,28,33-34,69]。然后,采用离子交换色谱从粗酶制剂中提取酶。通常,阳离子交换剂可以将卡拉胶酶与碱性pI结合[25,28,33,42,72],而阴离子交换柱与低pI的卡拉胶酶表现出特定的相互作用[21,23,26,59]。此外,凝胶过滤也被引入净化程序中[9,24,31]。少有的几种卡拉胶酶,如来自Pseudoalteromonassp.QY203的CgkP、来自Pseudoalteromonassp.CL19的CglCL19已经通过疏水层析成功纯化[20,68]。另一方面,因为重组卡拉胶酶含有组氨酸标记,通常用亲和层析法进行纯化[14,19,29,40,56-58,60,87]。
现在已有多种方法对卡拉胶酶的酶活进行检测。如,在类水解酶活性的检测中,随着底物被生长的菌落生产的酶水解后,观察到不透明介质的液化或消耗,因此,在培养板上通过利用0.5%的氯化十六烷基吡啶筛选生产卡拉胶酶的微生物也是一种检测卡拉胶酶活性的方法[88]。Smith等[89]利用酶谱法来测定卡拉胶酶活,原理为卡拉胶酶(以前用PAGE分离)可在覆盖凝胶中降解其底物,而寡糖被洗涤后,剩下的底物被阿尔新蓝染色,所以卡拉胶酶的活性在覆盖凝胶上的区域清晰显示出来。对于进一步的定量测定,通常采用二硝基水杨酸法测定卡拉胶水解过程中还原糖的增加量[90]。一个酶活单位被定义为每分钟释放1 μmol还原糖(D-半乳糖当量)所需要的酶量。此外,卡拉胶酶的酶活也可通过黏度法进行测定[91]。在适当的条件下,利用水浴中的黏度计测量黏度,通过绘制黏度随时间变化的反比值可以得到一条直线。此时,一单位的酶活被定义为单位时间内使斜率上升0.000 1所需要的酶量[91]。
5 卡拉胶酶的应用
卡拉胶酶应用广泛,其筛选与表征已经成为海洋酶工程相关领域的焦点。卡拉胶酶有作为生产寡糖的生物工具的巨大潜力,而生产的卡拉胶寡糖有许多功能活性。如,Yuan等[92]发现低分子量的卡拉胶寡糖具有抗肿瘤的作用。Aliste等[4]发现,卡拉胶寡糖具有抗血栓以及抑制ECV304细胞增殖、迁移的作用。另外,寡糖还显示出抗氧化、抗病毒和增强免疫刺激活性的作用[14,93-97]。卡拉胶寡糖的活性与其结构、分子量、组成成分和硫酸基团的位置紧密相关[98]。卡拉胶寡糖的酶法制备具有反应条件温和、过程可控等优点。酶法制备卡拉胶寡糖具有广泛的应用前景[9,40,58,60]。
卡拉胶酶还用于分离和制备与其他酶结合的藻类原生质体(如纤维素酶和酶)。Zablackis等[98]通过使用卡拉胶酶和纤维素酶从Kappaphycusalvarziivar.tambalang(Rhodophyta)分离原生质体。Gross等[99]通过使用来自Pseudomonascarrageenovora的κ-卡拉胶酶从Gigartinacorymbifera中制备原生质体。Gall等[100]使用ι-卡拉胶酶和κ-卡拉胶酶制备原生质体。
因为酶类对不同结构的底物具有严格的底物特异性,所以卡拉胶酶被广泛用于阐明卡拉胶的精细结构以及研究其在海洋细菌中的代谢过程[101]。Guibet等[102]用来自Pseudoalteromonassp. CL19的λ-卡拉胶酶研究了来自Gigartinaskottsbergii的λ-卡拉胶的精细结构。Antonopoulos等[103]通过重组ι-卡拉胶酶降解ι-卡拉胶并用高效液相色谱法结合蒸发光散射测定其寡糖的结构。此外,因为卡拉胶酶具有优异的降解卡拉胶的能力,所以它也被广泛用于纺织工业,特别是在印刷纺织品中用作增稠剂[104]。近年来,卡拉胶酶在海藻废物处理中的应用引起了越来越多的关注[47]。因为海藻加工企业在生产加工时产生了大量含有卡拉胶多糖的废料,造成了一定的环境问题,但是对于卡拉胶酶的进一步研究是降解卡拉胶废料、保护生态环境的关键。
6 结论与展望
近年来,卡拉胶酶以其广泛的应用引起了人们的关注,大量研究已经证明了卡拉胶酶在生物工业领域中有极大的潜在应用可能。本文总结了3种主要的卡拉胶酶的来源、性质、结构、机制及应用等方面的研究进展,为推动卡拉胶酶的深入研究和开发应用奠定了理论基础。但是所发现并报道的酶仍不能很好地满足工业应用的要求,一部分原因在于相应基因信息的缺乏、过低的活性和较差的稳定性。因此,迫切需要获得具有全基因信息、高活性、高稳定性的卡拉胶酶以在工业领域中得到全面的应用。
