lncRNA介导的ceRNA网络在肌肉减少症中作用机制的研究进展*
2022-10-13傅泽铤李伦宇官孝天刘青松刘颖丁海丽
傅泽铤, 李伦宇, 官孝天, 刘青松, 刘颖, 丁海丽
lncRNA介导的ceRNA网络在肌肉减少症中作用机制的研究进展*
傅泽铤, 李伦宇, 官孝天, 刘青松, 刘颖, 丁海丽△
(成都体育学院,四川 成都 610041)
肌肉减少症;长链非编码RNA;竞争性内源RNA;衰老
肌肉减少症(sarcopenia;简称肌少症)是一种进行性和全身性骨骼肌疾病,会降低老年人生活质量,引起机体功能障碍,增加老年人跌倒,引发骨折甚至死亡的风险。报道称,65岁及以上老年人肌少症患病率达14%~33%,80岁及以上老年人的患病率则高达50%~60%[1]。亚洲肌少症工作组2019年报告,亚洲人的肌少症患病率范围为5.5%~25.7%,其中男性患病率达5.1%~21.0%,高于女性的4.1%~16.3%[2]。我国作为全球老年人口最多的国家之一,随着人口老龄化的加剧,肌少症正成为社会关注的热点。非编码RNA(non-coding RNA, ncRNA)于20世纪70年代被首次发现,近年来越来越多的研究表明,ncRNA不同程度上参与了包括细胞分化、凋亡、代谢等机体生理功能或病理过程的调控[3]。长链非编码RNA(long non-coding RNA, lncRNA)与神经、肌肉、皮肤等的分化过程紧密相关[4],既可正向调节骨骼肌生成、分化,也可诱导肌萎缩的发生[5-6]。同时,lncRNA与微小RNA(microRNA, miRNA)、mRNA、蛋白质等分子相互作用并相互调节。基于此,本文拟对lncRNA介导的竞争性内源RNA(competing endogenous RNA, ceRNA)网络在肌少症中的作用及机制进行综述。
1 肌少症的发病机制
1.1肌少症概述肌少症的概念最先由Irwin在1989年提出[7],直到2016年肌少症才被正式纳入国际疾病分类ICD-10疾病编码中,编码为M62.84。2018年欧洲肌少症工作组最新发布的报告将肌少症定义为:肌肉数量减少、肌肉力量下降和(或)躯体功能减退,以及肌肉质量下降[8-9]。亚洲肌少症工作组针对亚洲人在体型、生活习惯以及社会环境与欧美人的差异,在此基础上提出新的诊断参数[10]。肌少症作为一种老年综合征,其病因包括多方面,主要可分为原发性肌肉减少和继发性肌肉减少。原发性肌肉减少是指年龄增长引起的肌肉减少;继发性肌肉减少包括活动减少相关(卧床、静态生活方式、零重力状态)、疾病相关(各器官衰竭、恶性肿瘤、内分泌疾病)和营养相关(能量或蛋白质的摄入减少、营养吸收障碍、胃肠功能失调、药物性厌食)等的肌肉减少[8]。
1.2肌少症发生的信号调控网络正常成年人从30岁开始,肌肉质量开始以每年约1%的速度下降,70岁以后肌肉减少的速度加快。65~70岁的人中肌肉减少症的患病率为14%,并且随着年龄的增长,在80岁以上的人群中达到53%[11]。老年人肌肉质量和力量的降低是由于进行性生理变化破坏了肌肉稳态平衡造成的。当蛋白质降解超过蛋白质合成时会发生肌肉萎缩,进而引起自噬、泛素化失衡,并导致骨骼肌质量发生不同程度的丢失[12-14]。衰老引起的全身慢性低水平炎症也与肌少症的患病相关[15],老年人存在的高水平血清炎症因子可诱发骨骼肌质量及肌力降低[16]。与年龄相关的肌肉减少症是由多种信号通路诱导的,包括由转化生长因子β(transforming growth factor-β, TGF-β)、骨形态发生蛋白(bone morphogenetic protein, BMP)和胰岛素样生长因子1(insulin-like growth factor 1, IGF-1)介导的信号通路[17]。
1.2.1TGF-β信号通路TGF-β信号通路抑制骨骼肌生长并诱导肌肉萎缩,TGF-β家族的几个成员在调节肌肉生理机能中起关键作用。例如TGF-β1、肌肉生长抑制素(myostatin)和生长分化因子11(growth differentiation factor 11,GDF11)是骨骼肌生长发育的负调节因子[18]。TGF-β与其受体结合后,磷酸化Smad 2/3,从而诱导叉头蛋白转录因子(Forkhead box transcription factor O, FoxO)的易位。这些转录因子促进肌萎缩Fbox-1蛋白(muscle atrophy F-box protein,MAFbx,也称作Atrogin-1)和肌环指蛋白1(muscle-specific RING finger protein 1,MuRF-1)的表达,导致蛋白质降解和肌肉萎缩[19-20]。
1.2.2BMP信号通路BMP通路是骨骼肌质量的正向调节剂[21],BMP 7与BMP受体结合并激活Smad 1/5/8的磷酸化,后者与Smad 4形成转录复合物并抑制如MuRF-1、Atrogin-1和F-box30等的转录,从而减轻肌肉萎缩[22, 23]。研究表明TGF-β/BMP信号轴平衡的打破影响骨骼肌的分化和再生[24]。
1.2.3IGF-1信号通路肌细胞释放的IGF-1与IGF结合蛋白结合后可以经由胰岛素受体底物1(insulin receptor substrate,IRS-1)和IRS-2激活蛋白激酶B(protein kinase B,PKB也称作Akt),后者通过抑制FoxO和激活雷帕霉素(mammalian target of rapamycin,mTOR)来增加肌肉质量和力量,从而促进蛋白质合成[25];此外,IGF-1通过Akt/mTOR通路抑制自噬,预防肌肉萎缩[26]。
1.3与肌少症相关的关键蛋白分子
1.3.1炎症相关因子炎性因子白细胞介素6(interleukin 6,IL-6)可以增加蛋白质水解,减弱蛋白质合成代谢和能量稳态[27],但IL-6的单独作用不足以诱导骨骼肌萎缩,它的分解代谢作用取决于与介导炎症反应的其他因子例如肿瘤坏死因子α(tumor necrosis factorα,TNF-α)的协同相互作用[28],TNF-α可激活金属蛋白酶调控细胞凋亡, 通过FoxO激活溶酶体自噬途径[16]。衰老的肌肉会产生大量的TGF-β1,不仅能诱导骨骼肌纤维化,而且还是削弱肌卫星细胞再生能力的关键因子[29],TGF-β1与下游受体结合会刺激泛素连接酶蛋白分解代谢途径,抑制合成代谢途径。衰老过程中骨骼肌的神经肌肉接头附近也表现出TGF-β1表达增加,这表明TGF-β1也可能在伴随衰老发生的神经肌肉接头降解和碎裂中发挥作用[30]。
1.3.2E3泛素连接酶Atrogin-1和MuRF-1是两种E3泛素连接酶,通过26S蛋白酶体进行蛋白水解,是骨骼肌中泛素介导的蛋白质降解的重要调节剂[19]。它们响应于myostatin/TGF-β信号传导而被激活;FoxO转录因子家族也是Atrogin-1和MuRF-1基因表达的重要调节因子[19]。而Akt可以磷酸化FoxO蛋白,使其无法进入细胞核以促进转录[20],这为IGF-1信号传导以抑制泛素介导的蛋白水解提供了一种途径。
2 与肌少症相关的转录组调控
有证据表明,在复杂的生物体发育过程中,RNA包含一个隐藏的调控信息层。它不仅充当DNA和蛋白质之间的信使,而且在基因表达的调节中发挥作用[31]。人类基因组报告表明,人类大约80%的DNA被转录成RNA,其中只有2%被翻译成蛋白质[32],其余大部分被归类为ncRNA。根据长度,ncRNA可进一步分为较小的ncRNA(如miRNA和iwi-interacting RNA)和较长的ncRNA[如lncRNA和环状RNA(circRNA)][33]。诸多研究已证明mRNA与miRNA在肌少症病程中扮演重要角色。
2.1与肌少症相关的mRNA有研究[34]基于临床样本和GTEX数据库对ceRNA网络中各元素进行相关性分析后,提示SEPP1这种mRNA可能是评估肌少症的关键调节因子。硒蛋白P1(selenoprotein P1,SEPP1)抑制能量代谢中关键介质的氧化磷酸化[35],这可能与肌少症的发病机制有关。研究人员提出SEPP1的高表达可能导致硒缺乏,硒缺乏会导致肌肉萎缩症[36]。糖尿病患者糖代谢异常也会导致SEPP1表达上调[35],而SEPP1的高表达可能是糖尿病引起肌少症的机制之一。同时,SEPP1与TNF-α水平呈正相关关系,TNF-α高表达是原发性肌肉疾病的特征[37]。
2.2与肌少症相关的miRNA骨骼肌纤维化随着年龄的增长而增加,miR-34a可以刺激多种细胞和组织类型的纤维化。沉默调节蛋白1(recombinant sirtuin 1,Sirt1)是miR-34a的公认靶标,Sirt1在肌肉中随着年龄的增长而下降,miR-34a对Sirt1的抑制可加速衰老。此外,骨骼肌中p53随年龄增长而增加,p53通过活性氧(reactive oxygen species,ROS)激活miR-34a,miR-34a靶向Sirt1以促进TGF-β1的表达,诱导骨骼肌衰老[38, 39];研究发现,较低水平的miR-133b和miR-206与营养不良和肌少症密切相关[40];miR-434-3p在衰老小鼠的骨骼肌中显着下调,miR-434-3p可抑制细胞凋亡,阻止包括 caspase-3和caspase-9在内的促细胞凋亡蛋白的激活,并通过靶向真核翻译起始因子5A1提高线粒体跨膜电位[41]。miR-21是一种炎症标志物,在肌肉衰老过程中可能由肌卫星细胞和肌纤维中升高的TNF-α和IL-6激活。已发现抑制老年小鼠肌卫星细胞中的miR-21对肌生成有促进作用,并可改善体外肌管直径的减小[42]。衰老过程中miR-181a的下调会导致线粒体功能障碍并激活自噬相关蛋白,恢复miR-181a表达可改善线粒体动力学和骨骼肌生理功能[43]。
2.3lncRNA转录调控肌少症lncRNA是一类长度大于200 nt的RNA转录本,不具有传统的蛋白质编码能力,lncRNA已被证明正在成为细胞生物学的核心参与者,其数量可能超过蛋白质编码转录本,其中一小部分已被证明参与基因表达的调节[44],包括表观遗传、转录、转录后和翻译调控[45]。lncRNA在三个方面发挥上述作用[46],在表观遗传水平上,lncRNA介导染色质重塑和修饰,发挥表观遗传调控作用;lncRNA可以与转录因子相互作用以影响转录调控;此外,lncRNAs还可以与mRNAs结合形成双链,特异性调节mRNAs的各种转录后过程,包括剪接、转运、翻译和降解。许多lncRNA与骨骼肌功能障碍和肌肉疾病有关,尤其是肌少症[47],lncRNA的差异表达通过各种信号通路加剧或减轻肌少症的发展进程。
以往对肌少症的研究多围绕蛋白分子、mRNA以及miRNA展开,传统观点认为lncRNAs是不编码蛋白质的,但最新研究发现一部分lncRNAs是具有编码功能蛋白质的能力的;因此我们将目光聚焦在lncRNA,探究其在肌少症中的作用机制,为探索肌少症的治疗靶点提供理论依据。
3 lncRNA在肌少症中的作用机制
3.1lncRNA与衰老的关系大多数研究已使用全基因组分析(例如微阵列或下一代测序)确定了衰老骨骼肌中差异表达的ncRNA。Zhang等[48]利用lncRNA微阵列比较C57BL/6J成年小鼠(6月龄)和老年小鼠(24月龄)在腓肠肌中有差异性表达的lncRNA。结果发现1945个在衰老骨骼肌组织中异常表达的lncRNAs。其中,894个lncRNAs表达下降,1051个lncRNAs表达上升。De等[49]对久坐不动的老人以及长期进行耐力、抗阻训练的同龄人的股外侧肌进行了转录组分析,结果显示242个lncRNAs在久坐及长期锻炼的骨骼肌中表达具有差异,并且lncRNAs在抗阻组与有氧组之间无表达差异。也有研究[50]对老年人和年轻人的骨骼肌样本进行lncRNA测序,并且与DEG数据结合,鉴定出152个DElncRNAs,其中76个在衰老骨骼肌中表达上调,76个表达下调。Chai的团队[51]将6月龄雌性大鼠分为去卵巢组和假手术组,12周后成功建立老年肌少症联合骨质疏松模型,通过RNA-seq分析股四头肌以及股骨中lncRNA的差异表达,结果发现分别有13个lncRNAs(8个上调,5个下调)、17个lncRNAs(9个上调,8个下调)发生差异性表达,并且在两种检测样本中均发现了LNC_004549发生上调,表明这种lncRNA同时参与骨骼肌和骨骼的分化。
据报道,在迄今为止的研究中,小鼠和大鼠的衰老骨骼肌中没有一种lncRNA受到相同的调节,这可能是物种之间的差异或研究样本过少的原因。
3.2lncRNA在骨骼肌生成中的作用肌分化因子(myogenic differentiation antigen, myoD)在肌生成过程中发挥着重要作用,越来越多的研究表明lncRNA可以通过MyoD促进肌生成。linc-RNA肌生成激活剂(linc-RNA activator of myogenesis, linc-RAM)可以直接与MyoD相互作用并且增强MyoD的转录活性[52];机械卸载诱导的肌肉萎缩相关lncRNA(mechanical unloading-induced muscle atrophy-related lncRNA, lncMUMA)通过充当miR-762分子海绵调节MyoD从而促进肌生成[53];MyoD上游非编码RNA(MyoD upstream noncoding RNA, MUNC)是MyoD的上游调节因子,可以通过MyoD表达促进肌生成[54];MyoD激活的lncRNA 肌肉生长促进因子(lncRNA muscle growth promoting factor, lncMGPF)在细胞分化过程中从细胞核转移到细胞质,主要通过转录后调控肌生成,lncMGPF作为miR-135a-5p的海绵削弱其对肌细胞增强因子2C(myocyte enhancer factor 2C, MEF2C)的抑制作用,增加MEF2C的表达,lncMGPF还可通过增强人抗原R(human antigen R, HuR)介导的MyoD和肌细胞生成素(myogenin, MyoG) mRNA的稳定性促进肌源性分化[55]。MyoD不仅可以作为lncRNA的靶点蛋白接受调控,同样也可作为上游调节因子调控lncRNA。lncMyoD可以在成肌细胞分化过程中被MyoD直接激活,进而负调控IGF2 mRNA结合蛋白2(IGF2 mRNA-binding protein 2, IMP2)介导的增殖基因的翻译从而抑制增殖,促进分化[56];发育多能性相关2上游结合肌肉lncRNA [developmental pluripotency-associated 2 (Dppa2) upstream binding muscle lncRNA, Dum]与肌卫星细胞的增殖、活化以及自我更新相关,研究表明MyoD转录起始位点上游26 bp处与Dum存在一个结合位点,推测Dum受MyoD调节在肌肉再生中发挥作用[57]。
肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1, MALAT1)是一种在骨骼肌中随着年龄增长表达下降的lncRNA,在肌细胞分化的后期和肌管肥大过程中高度表达。p53激活miR-34a的同时抑制MALAT1水平,MALAT1表达降低通过miR-34a靶向抑制Sirt1以及增加TGF-β1的表达,从而加速骨骼肌萎缩[38]。
H19是最早被鉴定为在动物骨骼肌中高表达的lncRNA之一[46]。BMP信号和DNA复制因子在成肌细胞分化过程中表达下调。miR-675-3p通过靶向Smad1和Smad5抑制BMP通路,而miR-675-5p抑制DNA复制起始因子Cdc6。-/-小鼠的卫星细胞分化减少,通过重新引入miR-675-3p和miR-675-5p,一定程度上恢复卫星细胞的分化[58]。此外,成肌细胞抑制基因Sirt1和FoxO1会抑制肌分化,H19通过抑制Sirt1/FoxO1信号通路促进成肌细胞分化[59]。
lncIRS1从两个途径发挥它在骨骼肌中的作用,lncIRS1可以通过海绵化miR-15家族发挥ceRNA的作用,来激活IRS1并调节IGF1-PI3K/AKT通路促进肌生成;lncIRS1还参与调控Atrogin-1和MuRF1的表达从而抑制肌萎缩[60]。
研究发现,Sirt1 AS通过与miR-34a竞争来调控肌肉生成,优先与Sirt1 mRNA相互作用,以促进成肌细胞增殖并抑制分化[61]。
3.3lncRNA调控肌萎缩的机制lnc Chronos是一种与年龄相关的lncRNA,随着年龄增长表达上调,在衰老小鼠骨骼肌中作为一种诱导肌肉肥大的负调节剂促进肌萎缩[62]。在体内、体外抑制lnc Chronos的表达可以上调BMP7,进而激活Smad1/5正向调节骨骼肌肥大。衰老引起的lnc Chronos表达增加会减少肌纤维横截面积,并通过使BMP7信号失活加剧肌萎缩或肌少症的发生。
萎缩相关的lncRNA-1 (atrophy-related lncRNA-1,Atrolnc-1)是一种与恶病质相关的lncRNA,抑制Atrolnc-1可以降低蛋白质降解速率从而改善慢性肾脏病小鼠骨骼肌萎缩[63]。Atrolnc-1在体外的过表达可升高肌细胞中蛋白质水解速率从而减小肌管直径,其机制是Atrolnc-1通过阻碍A20结合NF-κB活化抑制蛋白(A20 binging inhibitor of NF-κB activation, ABIN)的抑制能力,激活核转录因子NF-κB进而促进MuRF-1的表达,MuRF-1能够增加肌球蛋白的蛋白水解速率来诱导肌肉萎缩[20],这说明Atrolnc-1可以通过泛素-蛋白酶体系统诱导肌萎缩。
Smad泛素调节因子2上游lncRNA[Smad ubiquitin regulatory factor 2 (SMURF2) upstream lncRNA,SMUL]是新发现的一种lncRNA[64],SMUL过表达可抑制琥珀酸脱氢酶SDH的活性,增强乳酸脱氢酶的活性,促进了骨骼肌的糖酵解能力并诱导慢肌向快肌转化。SMURF2是TGF-β/Smad通路的上游调控基因,SMUL抑制SMURF2以激活TGF-β/Smad通路诱导肌萎缩,这说明SMUL通过激活蛋白酶体降解和自噬来促进骨骼肌萎缩。
lncRNA浆细胞瘤变体易位1(plasmacytoma variant translocation 1,Pvt1)已在癌症中进行了广泛研究,最近的研究表明在肌萎缩过程中Pvt1可以影响与线粒体相关的自噬、细胞凋亡[65]。Pvt1表达下调后通过与c-Myc相互作用可以上调Bcl-2的表达,Bcl-2能够与beclin-1相互作用阻止自噬体结构的形成;Bax是Bcl-2的同源物,Bcl-2表达下调时能促进Bax表达升高,通过破坏线粒体的完整性来促进细胞凋亡,并通过与Bcl-2结合来减弱Bcl-2的抗细胞凋亡作用[66]。众所周知,线粒体功能障碍与肌少症相关[67],这表明Pvt1可以通过影响线粒体诱导肌萎缩。
长链非编码RNA淋巴细胞白血病缺失基因2 (deleted in lymphocytic leukemia 2,DLEU2)是一种在肌少症患者中高度表达的lncRNA,miR-181a则相反,它增加MyoD和MyoG的表达,促进成肌分化和肌生成标志的表达,但DLEU2作为miR-181a的海绵,与miR-181a相互作用上调转录后SEPP1的表达水平抑制了这种进程,推测是与TNF-α相互作用促进了肌萎缩的发生[34]。
GO富集分析提示免疫与炎症相关基因ITK参与了“细胞活化”、“免疫系统过程”、“免疫系统过程调节”、“免疫系统过程正向调节”等过程,提示蛋白激酶 CGMP依赖性1-反义1(protein kinase CGMP-dependent 1-antisense 1,PRKG1-AS1)可能在一定程度上通过靶向ITK调节骨骼肌衰老中的免疫炎症反应[50]。
3.4lncRNA与肌少症防治在骨骼肌中已检测到数千个lncRNA,但只有一小部分在肌少症中具有差异性表达。可以明确的是,这些lncRNA表达水平的变化会影响多种信号通路并改变其靶基因表达(表1),从而影响肌少症的进程。
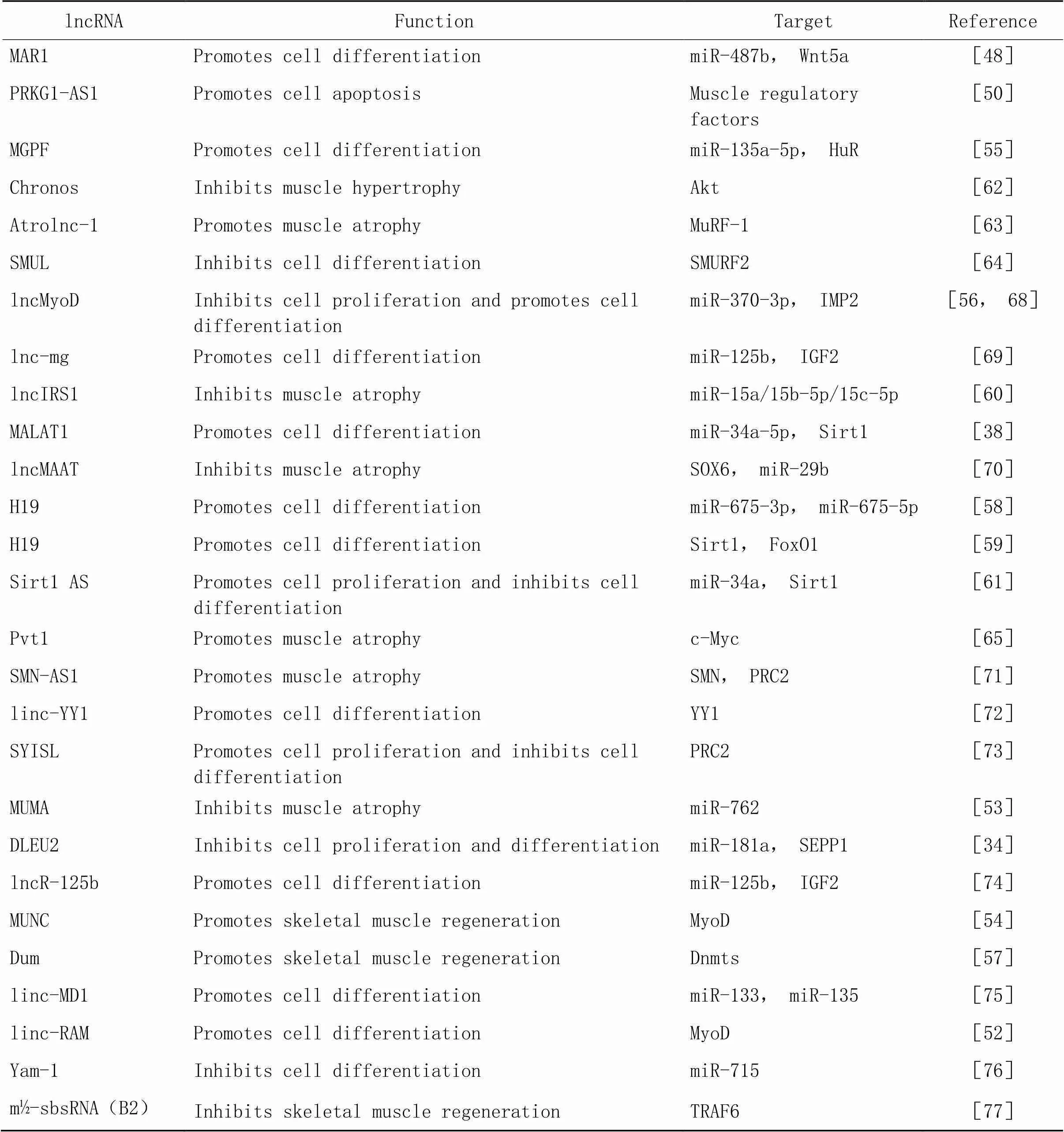
表1 在肌少症中发挥功能的lncRNA
4 总结与展望
近年来对于lncRNA-miRNA-mRNA调节网络的研究逐渐成为热点。大量研究表明,miRNA和lncRNA在癌症或其他疾病中发挥着重要作用。在癌症中,miRNA与lncRNA存在协同调节、相互调节。在骨骼肌疾病中,lncRNA可作为miRNA的分子海绵,通过竞争一个或多个miRNA的结合位点来影响其对mRNA的调控,抑制后者翻译或者导致mRNA降解,从而实现转录后调控基因表达的功能。在心血管疾病以及糖尿病中,lncRNA通过多种途径参与疾病的发生和发展[78-79]。我们仍处于研究骨骼肌中lncRNA的“婴儿阶段”,lncRNA作为一类新兴的调节因子,对骨骼肌的发育及其相关疾病具有重要调控作用,在体内调节复杂的生理过程,包括维持骨骼肌质量及结构,参与自噬、泛素化、全身慢性炎症反应等过程。通过全身性或组织特异性敲除lncRNA动物模型,对lncRNA与骨骼肌生理功能有了一定探索,但是也面临诸多问题亟需解决。目前,大多数关于肌少症中lncRNA分析的研究都以老年小鼠模型为主,仅有为数不多的以人类作为样本的研究。由于lncRNA的序列保守性不高,不同物种之间lncRNA作用机制相互借鉴的意义不大。此外,在同一个实验模型中使用不同方法分析鉴定出的lncRNA并不完全一致,这也对后续研究的深入造成了一定的局限性。
lncRNA作为骨骼肌疾病和肌生成的重要调节因子之一,明确lncRNA维持骨骼肌稳态的调节机制,有助于治疗肌肉萎缩。如lncIRS1可以促进IGF-1的表达进而抑制蛋白质降解;调节Pvt1表达以维持线粒体形态的动态平衡可以防止肌肉萎缩;通过MALAT1保持蛋白合成/分解速率稳态保持肌肉质量,以期为寻找肌少症的治疗靶点提供依据。
未来研究中,仍有许多问题有待进一步探索和证实,如虽然已鉴定出许多lncRNA,但它们的功能和靶标尚未得到认识;数据分析工具的不断创新加速了lncRNA的研究和鉴定,但数据库包括不同转录本之间的信息仍不够完善;几项研究已经确定了肌肉生长抑制素和Akt/mTOR 轴之间存在交叉作用,lncRNA是否靶向分子同时调节这两种信号通路,从而影响肌肉减少症的表型,仍然是个未知数;运动是治疗肌少症最有效的手段,识别调节性lncRNA及其靶标并阐明运动后逆转肌少症进程的分子机制,可为实现针对肌肉萎缩和肌肉功能障碍的治疗提供新的策略。
[1] Chen LK, Liu LK, Woo J, et al. Sarcopenia in Asia: consensus report of the Asian Working Group for Sarcopenia [J]. J Am Med Dir Assoc, 2014, 15(2):95-101.
[2]刘娟, 丁清清, 周白瑜, 等. 中国老年人肌少症诊疗专家共识(2021)[J]. 中华老年医学杂志, 2021, 40(8):943-952.
Liu J, Ding QQ, Zhou BY, et al. Chinese expert consensus on diagnosis and treatment for elderly with sarcopenia(2021)[J]. Chin J Geriatr, 2021, 40(8):943-952.
[3] ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome[J]. Nature, 2012, 489(7414):57-74.
[4] Flynn RA, Chang HY. Long noncoding RNAs in cell-fate programming and reprogramming [J]. Cell Stem Cell, 2014, 14(6):752-761.
[5] Abdelmohsen K, Panda A, Kang MJ, et al. Senescence-associated lncRNAs: senescence-associated long noncoding RNAs[J]. Aging Cell, 2013, 12(5):890-900.
[6]陈晓敏, 张栋栋, 骆健俊, 等. 长非编码RNA研究进展[J]. 生物化学与生物物理进展, 2014, 41(10):997-1009.
Chen XM, Zhang DD, Luo JJ, et al. Research progress on long noncoding RNAs[J]. Prog Biochem Biophys, 2014, 41(10):997-1009.
[7] Epidemiologic and methodologic problems in determining nutritional status of older persons. Proceedings of a conference. Albuquerque, New Mexico, October 19-21, 1988[J]. Am J Clin Nutr, 1989, 50(5 Suppl):1121-1235.
[8] Cruz-Jentoft A J, Baeyens JP, Bauer JM, et al. Sarcopenia: European consensus on definition and diagnosis: report of the European Working Group on Sarcopenia in Older People[J]. Age Ageing, 2010, 39(4):412-423.
[9] Cruz-Jentoft AJ, Bahat G, Bauer J, et al. Sarcopenia: revised European consensus on definition and diagnosis[J]. Age Ageing, 2019, 48(1):16-31.
[10] Chen LK, Woo J, Assantachai P, et al. Asian Working group for sarcopenia: 2019 consensus update on sarcopenia diagnosis and treatment[J]. J Am Med Dir Assoc, 2020, 21(3):300-307.e302.
[11] Dodds RM, Roberts HC, Cooper C, et al. The epidemiology of sarcopenia[J]. J Clin Densitom, 2015, 18(4):461-466.
[12] Bonaldo P, Sandri M. Cellular and molecular mechanisms of muscle atrophy[J]. Dis Model Mech, 2013, 6(1):25-39.
[13] Sandri M. Protein breakdown in muscle wasting: role of autophagy-lysosome and ubiquitin-proteasome[J]. Int J Biochem Cell Biol, 2013, 45(10):2121-2129.
[14] Schiaffino S, Dyar KA, Ciciliot S, et al. Mechanisms regulating skeletal muscle growth and atrophy[J]. FEBS J, 2013, 280(17):4294-4314.
[15] Tyrrell DJ, Goldstein DR. Ageing and atherosclerosis: vascular intrinsic and extrinsic factors and potential role of IL-6[J]. Nat Rev Cardiol, 2021, 18(1):58-68.
[16] 周永战, 陈佩杰, 肖卫华. 老年人肌少症发生机制 [J]. 生理学报, 2018, 70(4):445-454.
Zhou YZ, Chen PJ, Xiao WH. Mechanism of the occurrence of sarcopenia in the elderly[J]. Acta physiol Sin, 2018, 70(4):445-454.
[17] Lee J, Kang H. Role of microRNAs and long non-coding RNAs in sarcopenia[J]. Cells, 2022, 11(2):187-201.
[18] Delaney K, Kasprzycka P, Ciemerych MA, et al. The role of TGF-β1 during skeletal muscle regeneration[J]. Cell Biol Int, 2017, 41(7):706-715.
[19] Gumucio JP, Mendias CL. Atrogin-1, MuRF-1, and sarcopenia[J]. Endocrine, 2013, 43(1):12-21.
[20] Egerman MA, Glass DJ. Signaling pathways controlling skeletal muscle mass[J]. Crit Rev Biochem Mol Biol, 2014, 49(1):59-68.
[21] Winbanks CE, Chen JL, Qian H, et al. The bone morphogenetic protein axis is a positive regulator of skeletal muscle mass[J]. J Cell Biol, 2013, 203(2):345-357.
[22] Sartori R, Schirwis E, Blaauw B, et al. BMP signaling controls muscle mass[J]. Nat Genet, 2013, 45(11):1309-1318.
[23] Edström E, Altun M, Hägglund M, et al. Atrogin-1/MAFbx and MuRF1 are downregulated in aging-related loss of skeletal muscle[J]. J Gerontol A Biol Sci Med Sci, 2006, 61(7):663-674.
[24] Scimeca M, Piccirilli E, Mastrangeli F, et al. Bone morphogenetic proteins and myostatin pathways: key mediator of human sarcopenia[J]. J Transl Med, 2017, 15(1):34-43.
[25] Yoshida T, Delafontaine P. Mechanisms of IGF-1-mediated regulation of skeletal muscle hypertrophy and atrophy [J]. Cells, 2020, 9(9):1970-1994.
[26] Wang S, Gu K. Insulin-like growth factor 1 inhibits autophagy of human colorectal carcinoma drug-resistant cells via the protein kinase B/mammalian target of rapamycin signaling pathway[J]. Mol Med Rep, 2018, 17(2):2952-2956.
[27] Carson JA, Baltgalvis KA. Interleukin 6 as a key regulator of muscle mass during cachexia[J]. Exerc Sport Sci Rev, 2010, 38(4):168-176.
[28] Tuttle CSL, Thang LAN, Maier AB. Markers of inflammation and their association with muscle strength and mass: a systematic review and meta-analysis[J]. Ageing Res Rev, 2020, 64:101185.
[29] Carlson ME, Hsu M, Conboy IM. Imbalance between pSmad3 and Notch induces CDK inhibitors in old muscle stem cells[J]. Nature, 2008, 454(7203):528-532.
[30] Blasco A, Gras S, Mòdol-Caballero G, et al. Motoneuron deafferentation and gliosis occur in association with neuromuscular regressive changes during ageing in mice[J]. J Cachexia Sarcopenia Muscle, 2020, 11(6):1628-1660.
[31] Morris KV, Mattick JS. The rise of regulatory RNA[J]. Nat Rev Genet, 2014, 15(6):423-437.
[32] Cabili MN, Trapnell C, Goff L, et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses[J]. Genes Dev, 2011, 25(18):1915-1927.
[33] Liu Q, Deng J, Qiu Y, et al. Non-coding RNA basis of muscle atrophy[J]. Mol Ther Nucleic Acids, 2021, 26:1066-1078.
[34] Wang Y, Zhao ZJ, Kang XR, et al. lncRNA DLEU2 acts as a miR-181a sponge to regulate SEPP1 and inhibit skeletal muscle differentiation and regeneration[J]. Aging (Albany NY), 2020, 12(23):24033-24056.
[35] Steinbrenner H. Interference of selenium and selenoproteins with the insulin-regulated carbohydrate and lipid metabolism[J]. Free Radic Biol Med, 2013, 65:1538-1547.
[36] Yao H, Zhao W, Zhao X, et al. Selenium deficiency mainly influences the gene expressions of antioxidative selenoproteins in chicken muscles[J]. Biol Trace Elem Res, 2014, 161(3):318-327.
[37] Meyer SU, Krebs S, Thirion C, et al. Tumor necrosis factor alpha and insulin-like growth factor 1 induced modifications of the gene expression kinetics of differentiating skeletal muscle cells[J]. PLoS One, 2015, 10(10):e0139520.
[38] Ruan L, Mendhe B, Parker E, et al. Long Non-coding RNA MALAT1 is depleted with age in skeletal muscleand MALAT1 silencing increases expression of TGF-β1[J]. Front Physiol, 2021, 12:742004-742010.
[39] Myers MJ, Shepherd DL, Durr AJ, et al. The role of SIRT1 in skeletal muscle function and repair of older mice[J]. J Cachexia Sarcopenia Muscle, 2019, 10(4):929-949.
[40] Iannone F, Montesanto A, Cione E, et al. Expression patterns of muscle-specific miR-133b and miR-206 correlate with nutritional status and sarcopenia[J]. Nutrients, 2020, 12(2):297-311.
[41] Pardo PS, Hajira A, Boriek AM, et al. MicroRNA-434-3p regulates age-related apoptosis through eIF5A1 in the skeletal muscle[J]. Aging (Albany NY), 2017, 9(3):1012-1029.
[42] Borja-Gonzalez M, Casas-Martinez JC, McDonagh B, et al. Inflamma-miR-21 negatively regulates myogenesis during ageing[J]. Antioxidants (Basel), 2020, 9(4):345-363.
[43] Goljanek-Whysall K, Soriano-Arroquia A, McCormick R, et al. miR-181a regulates p62/SQSTM1, parkin, and protein DJ-1 promoting mitochondrial dynamics in skeletal muscle aging[J]. Aging Cell, 2020, 19(4):e13140-e13155.
[44] Derrien T, Johnson R, Bussotti G, et al. The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression[J]. Genome Res, 2012, 22(9):1775-1789.
[45] Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions[J]. Nat Rev Genet, 2009, 10(3):155-159.
[46] Zhao Y, Chen M, Lian D, et al. Non-coding RNA regulates the myogenesis of skeletal muscle satellite cells, injury repair and diseases[J]. Cells, 2019, 8(9):988-1002.
[47] Li Y, Meng X, Li G, et al. Noncoding RNAs in muscle atrophy[J]. Adv Exp Med Biol, 2018, 1088:249-266.
[48] Zhang ZK, Li J, Guan D, et al. A newly identified lncRNA MAR1 acts as a miR-487b sponge to promote skeletal muscle differentiation and regeneration [J]. J Cachexia Sarcopenia Muscle, 2018, 9(3):613-626.
[49] De Sanctis P, Filardo G, Abruzzo PM, et al. Non-coding RNAs in the transcriptional network that differentiates skeletal muscles of sedentary from long-term endurance- and resistance-trained elderly[J]. Int J Mol Sci, 2021, 22(4):1539-1564.
[50] Zheng Y, Liu T, Li Q, et al. Integrated analysis of long non-coding RNAs (lncRNAs) and mRNA expression profiles identifies lncRNA PRKG1-AS1 playing important roles in skeletal muscle aging[J]. Aging (Albany NY), 2021, 13(11):15044-15060.
[51] Chai S, Wan L, Wang JL, et al. Systematic analysis of long non-coding RNA and mRNA profiling using RNA sequencing in the femur and muscle of ovariectomized rats [J]. J Musculoskelet Neuronal Interact, 2019, 19(4):422-434.
[52] Yu X, Zhang Y, Li T, et al. Long non-coding RNA Linc-RAM enhances myogenic differentiation by interacting with MyoD[J]. Nat Commun, 2017, 8:14016.
[53] Zhang ZK, Li J, Guan D, et al. Long noncoding RNA lncMUMA reverses established skeletal muscle atrophy following mechanical unloading[J]. Mol Ther, 2018, 26(11):2669-2680.
[54] Mueller AC, Cichewicz MA, Dey BK, et al. MUNC, a long noncoding RNA that facilitates the function of MyoD in skeletal myogenesis[J]. Mol Cell Biol, 2015, 35(3):498-513.
[55] Lv W, Jin J, Xu Z, et al. lncMGPF is a novel positive regulator of muscle growth and regeneration[J]. J Cachexia Sarcopenia Muscle, 2020, 11(6):1723-1746.
[56] Gong C, Li Z, Ramanujan K, et al. A long non-coding RNA, LncMyoD, regulates skeletal muscle differentiation by blocking IMP2-mediated mRNA translation[J]. Dev Cell, 2015, 34(2):181-191.
[57] Wang L, Zhao Y, Bao X, et al. LncRNA Dum interacts with Dnmts to regulate Dppa2 expression during myogenic differentiation and muscle regeneration[J]. Cell Res, 2015, 25(3):335-350.
[58] Dey BK, Pfeifer K, Dutta A. The H19 long noncoding RNA gives rise to microRNAs miR-675-3p and miR-675-5p to promote skeletal muscle differentiation and regeneration[J]. Genes Dev, 2014, 28(5):491-501.
[59] Xu X, Ji S, Li W, et al. LncRNA H19 promotes the differentiation of bovine skeletal muscle satellite cells by suppressing Sirt1/FoxO1[J]. Cell Mol Biol Lett, 2017, 22:10-19.
[60] Li Z, Cai B, Abdalla BA, et al. LncIRS1 controls muscle atrophy via sponging miR-15 family to activate IGF1-PI3K/AKT pathway[J]. J Cachexia Sarcopenia Muscle, 2019, 10(2):391-410.
[61] Wang GQ, Wang Y, Xiong Y, et al. Sirt1 AS lncRNA interacts with its mRNA to inhibit muscle formation by attenuating function of miR-34a[J]. Sci Rep, 2016, 6:21865.
[62] Neppl RL, Wu CL, Walsh K. lncRNA Chronos is an aging-induced inhibitor of muscle hypertrophy[J]. J Cell Biol, 2017, 216(11):3497-3507.
[63] Sun L, Si M, Liu X, et al. Long-noncoding RNA Atrolnc-1 promotes muscle wasting in mice with chronic kidney disease[J]. J Cachexia Sarcopenia Muscle, 2018, 9(5):962-974.
[64] Cai B, Li Z, Ma M, et al. Long noncoding RNA SMUL suppresses SMURF2 production-mediated muscle atrophy via nonsense-mediated mRNA decay[J]. Mol Ther Nucleic Acids, 2021, 23:512-526.
[65] Alessio E, Buson L, Chemello F, et al. Single cell analysis reveals the involvement of the long non-coding RNA Pvt1 in the modulation of muscle atrophy and mitochondrial network[J]. Nucleic Acids Res, 2019, 47(4):1653-1670.
[66] Edlich F. BCL-2 proteins and apoptosis: recent insights and unknowns[J]. Biochem Biophys Res Commun, 2018, 500(1):26-34.
[67] Marzetti E, Calvani R, Cesari M, et al. Mitochondrial dysfunction and sarcopenia of aging: from signaling pathways to clinical trials[J]. Int J Biochem Cell Biol, 2013, 45(10):2288-2301.
[68] Zhang P, Du J, Guo X, et al. LncMyoD promotes skeletal myogenesis and regulates skeletal muscle fiber-type composition by sponging miR-370-3p[J]. Genes (Basel), 2021, 12(4):589-604.
[69] Zhu M, Liu J, Xiao J, et al. Lnc-mg is a long non-coding RNA that promotes myogenesis[J]. Nat Commun, 2017, 8:14718.
[70] Li J, Yang T, Tang H, et al. Inhibition of lncRNA MAAT controls multiple types of muscle atrophy by- and-regulatory actions[J]. Mol Ther, 2021, 29(3):1102-1119.
[71] d'Ydewalle C, Ramos DM, Pyles NJ, et al. The antisense transcript SMN-AS1 regulates SMN expression and is a novel therapeutic target for spinal muscular atrophy [J]. Neuron, 2017, 93(1):66-79.
[72] Zhou L, Sun K, Zhao Y, et al. Linc-YY1 promotes myogenic differentiation and muscle regeneration through an interaction with the transcription factor YY1[J]. Nat Commun, 2015, 6:10026.
[73] Jin JJ, Lv W, Xia P, et al. Long noncoding RNA SYISL regulates myogenesis by interacting with polycomb repressive complex 2[J]. Proc Natl Acad Sci U S A, 2018, 115(42):e9802-e9811.
[74] Zhan S, Qin C, Li D, et al. A novel long noncoding RNA, lncR-125b, promotes the differentiation of goat skeletal muscle satellite cells by sponging miR-125b[J]. Front Genet, 2019, 10:1171-1184.
[75] Cesana M, Cacchiarelli D, Legnini I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147(2):358-369.
[76] Lu L, Sun K, Chen X, et al. Genome-wide survey by ChIP-seq reveals YY1 regulation of lincRNAs in skeletal myogenesis[J]. EMBO J, 2013, 32(19):2575-2588.
[77] Wang J, Gong C, Maquat LE. Control of myogenesis by rodent SINE-containing lncRNAs[J]. Genes Dev, 2013, 27(7):793-804.
[78] 韩泽平, 何金花, 黎毓光. 长链非编码RNA与糖尿病关系的研究进展[J]. 中国病理生理杂志, 2014, 30(4):751-756, 762.
Han ZP, He JH, Li YG. Long non-coding RNA and diabetes mellitus[J]. Chin J Pathophysiol, 2014, 30(4):751-756, 762.
[79] 覃伟峰, 仉红刚. lncRNA在心血管疾病中作用的研究进展[J]. 中国病理生理杂志, 2016, 32(8):1471-1477.
Tan WF, Zhang HG. Progress in role of lnc RNA in cardiovascular diseases[J]. Chin J Pathophysiol, 2016, 32(8):1471-1477.
Progress in mechanism of lncRNA-mediated ceRNA network in sarcopenia
FU Ze-ting, LI Lun-yu, GUAN Xiao-tian, LIU Qing-song, LIU Ying, DING Hai-li△
(,610041,)
Long non-coding RNAs (lncRNAs) are non-protein-coding transcripts longer than 200 nucleotides.Studies illustrate that lncRNAs are associated with skeletal muscle dysfunction and muscle diseases such as sarcopenia. lncRNAs regulate microRNA (miRNA) levels by acting as sponges, suggesting the regulatory mechanism of competing endogenous RNA (ceRNA) network linked by lncRNA-miRNA-mRNA. Therefore, exploring the role of lncRNA-mediated ceRNA network in sarcopenia is expected to lead to the discovery of potential novel diagnostic markers and therapeutic targets.
Sarcopenia; Long non-coding RNA; Competing endogenous RNA; Aging
1000-4718(2022)09-1694-08
2022-05-09
2022-06-22
028-85051087; E-mail: dinghaili@cdsu.edu.cn
R685; R363.2
A
10.3969/j.issn.1000-4718.2022.09.020
[基金项目]国家重点研发计划(No. 2018YFF0300604);国家自然科学基金资助项目(No. 81904318);运动医学四川省重点实验室暨国家体育总局运动医学重点实验室资助项目(No. 2021-A021)
(责任编辑:余小慧,李淑媛)
