白色放线动孢菌DSM 45114T r pf基因的克隆表达及结构预测
2022-08-31张宇帆刘菁菁曹敏刘彤彤陈雪张秀敏
张宇帆刘菁菁曹敏刘彤彤陈雪张秀敏
(河北大学 生命科学学院,生命科学与绿色发展研究院,河北省微生物育种与保育工程实验室, 河北省微生物多样性研究与应用重点实验室, 河北 保定 071002)
活的非可培养(viable but non culturable,VBNC)状态是某些细菌在恶劣环境下形成的一种休眠状态,自然界中90%以上的微生物是不可培养的[1],在某些特定情况下可以使这些细菌恢复为可培养状态,即复苏[2].1998年,Mukamolova[3]等发现藤黄微球菌(Micrococcusluteus)中的Rpf蛋白可以复苏其休眠细胞.随后,科学家们发现,rpf基因广泛存在于高G+C含量的革兰氏阳性菌中,并且都具有70个氨基酸左右的保守区域,即Rpf域,该结构域是起复苏促进作用的重要结构[4-5].不同物种具有不同拷贝数和不同结构的rpf基因.目前,Rpf蛋白是第1 个在细菌中发现的复苏促进因子,其复苏促进作用现常被用在疫苗研制[6-7]、诊断检测[8-9]和污水处理[10-13]等方面.也有研究表明,Rpf蛋白可以有效促进放线菌在土壤中的分离和培养,是环境放线菌资源挖掘的有效手段[14-15].
为探索不同物种Rpf蛋白的功能特性,本研究设计了rpf基因特异性引物,扩增得到了白色放线动孢菌(Actinokineosporaalba)DSM 45114T的3 个rpf基 因(rpf-2、rpf-E-1、rpf-E-2),构 建 了 载 体p ET-28a(+)-rpf-2,p ET-28a(+)-rpf-E-1,p ET-28a(+)-rpf-E-2,在EscherichiacoliBL21(DE3)中进行了诱导表达,并对其编码蛋白氨基酸序列、结构域、功能等进行了生物信息学分析和预测,为后续探讨白色放线动孢菌(A.alba)Rpf蛋白的功能奠定了基础.
1 材料与方法
1.1 菌株来源
白色放线动孢菌(A.alba)DSM 45114T,E.coilDH5α、BL21(DE3),表达质粒p ET-28a(+)来源于河北省微生物育种与保育工程实验室,克隆质粒p MD18-T 购自大连宝生物工程有限公司.
1.2 Rpf蛋白结构及性质预测
通过NCBI、Protparam[16]、Protscale[16]、Signal P[17]、SOPMA[18]、CDD[19]、SWISS-MODEL[20]、DNAMAN 等软件对rpf-E-1、rpf-E-2、rpf-2基因进行了一系列生物信息学分析,具体预测项目与网址见表1.
表1生物信息学预测项目及网址Tab.1 Bioinformatics testing programs and websites
1.3 白色放线动孢菌的复苏及培养
从甘油管中活化白色放线动孢菌(A.alba)DSM 45114T,待菌株纯化后挑取单菌落接种于10 m L NA液体培养基中,28℃振荡培养48 h.待菌体长好后,12 000 r/min离心1 min,收集菌体,-20℃保藏备用.
1.4 白色放线动孢菌基因组DNA的提取
采用细菌基因组DNA 提取试剂盒(北京康为世纪生物科技有限公司)提取.
1.5 rpf 基因的扩增及克隆

1.6 rpf 基因表达载体的构建及诱导表达
提取验证正确的质粒和p ET-28a(+)质粒,使用限制性内切酶EcoRⅠ和NotⅠ对其进行酶切.用T4 DNA 连接酶连接,转化E.coliBL21(DE3)感受态细胞,然后涂布于LB平板(含质量浓度为100 mg/L的卡那霉素),37℃培养14~16 h,随机筛选2个菌落,分别接种于5 m L含卡那霉素的LB培养液中,37℃振荡培养过夜,提取质粒DNA,酶切进行验证.经验证,rpf基因插入正确的阳性克隆,命名为p ET-28a(+)-rpf-2,p ET-28a(+)-rpf-E-1,p ET-28a(+)-rpf-E-2.将验证正确的转化子转接于含卡那霉素的LB液体培养基中,37℃摇床振荡培养过夜.正交实验条件见表2,取500μL转接于50 m L的LB液体培养基中,每个条件接种2 瓶.当OD600值达到0.8 时,按照3 因素3 水平正交实验表(表2)中温度、诱导剂IPTG 终浓度、时间在摇床上以160 r/min的速度诱导.

表2 蛋白诱导条件的3因素3水平正交实验Tab.2 Three factors and three levels orthogonal test table of protein induction conditions
将培养好的菌液倒入50 m L的离心管中,4℃下10 000 r/min离心10 min,收集菌体,用无菌去离子水重新悬浮菌体后再次离心.最后将菌体重悬于10倍体积的IMAC裂解缓冲液(含DNase 100 U/m L和尿素8 mol/L)中,使用超声波细胞破碎仪对菌体进行破碎.在冰上超声处理15 min,工作5 s,间隔5 s.然后于高速冷冻离心机中4℃下10 000 r/min离心20 min,收集上清液和沉淀,置于-20℃冰箱保存.取10μL上清液和等体积的2×SDS-PAGE Loading Buffer,用涡旋混合器混匀,做好标记,煮沸5 min.用SDS-PAGE 进行检测.电泳结束后,用质量分数为0.1%的考马斯亮蓝R-250染液染色30 min,之后用去离子水洗去浮色,加入脱色液,摇床脱色至条带清晰,拍照记录.
1.7 Rpf蛋白的复性与纯化
将收集的沉淀用体积分数为1%的Triton X-100 蛋 白 洗 涤 液 洗 涤3 次,4℃下10 000 r/min 离心20 min,重新收集沉淀.然后用含尿素的缓冲液重新悬浮沉淀,置于4℃条件下溶解过夜,为后续纯化做准备.使用伯乐蛋白纯化试剂盒(6200239)和IMAC柱(6200241)对蛋白进行纯化.
1.8 VBNC状态的诱导
将保存于甘油管的藤黄微球菌(M.luteus)和滨海红球菌(Rhodococcusmarinonascens)接种于NA 培养基中重新纯化复壮,然后分别挑取单菌落接种于NA 液体培养基中,28℃下200 r/min摇床培养过夜.收集菌液4℃下6 000 r/min离心10 min后收集菌体,然后用生理盐水重悬菌体,重复3次,最后将洗过的细胞重新接种于生理盐水中于4℃条件下静置培养至少90 d,之后取菌液涂布于平板,当不能检测到细菌时,则认为其已进入VBNC状态.
1.9 Rpf对VBNC状态菌复苏的验证
为探讨获取的纯化重组Rpf蛋白对藤黄微球菌(M.luteus)和滨海红球菌(R.marinonascens)VBNC菌体的复苏促生长能力,使用0.22μm 的滤膜对之前纯化的重组蛋白进行过滤,然后添加体积分数为10%、1%、0.1%的重组Rpf蛋白至含有1%藤黄微球菌(M.luteus)或滨海红球菌(R.marinonascens)的NA 液体培养基中,并以添加体积分数为10%121℃高压灭活15 min的重组Rpf蛋白做空白对照,不添加重组Rpf蛋白的为阴性对照组,置于Bioscreen多功能微生物生长自动培养分析仪中.每组重复3次,分别在培养0、4、8、12、24、36、48、60、72 h时测定OD600吸光值.
2 结果
2.1 白色放线动孢菌(A.alba)DSM 45114T r pf 基因编码蛋白的理化性质及亲/疏水性分析
使用Expasy在线软件的Protparam功能,对目的蛋白理化性质进行了预测分析(表3).由表3可知,3个蛋白都具有亲水性.不稳定系数(Ⅱ)临界值为40,3个蛋白的此值均小于40,所以3个蛋白都是稳定性蛋白.用ProtScale功能对3个蛋白进行疏水性和亲水性分析(图1).综合整条肽链来看,3个蛋白分值绝大部分为负值,所以整条肽链表现为亲水性,没有明显的疏水区.因此3个蛋白均表现出一定的亲水性,与理化分析的结果一致.

图1 白色放线动孢菌DSM 45114T Rpf蛋白的疏水性和亲水性Fig.1 Hydrophobic and hydrophilic properties of Actinokineospora alba DSM 45114T Rpf protein

表3 氨基酸序列的理化性质分析Tab.3 Analysis of physicochemical properties of amino acid sequences
2.2 白色放线动孢菌(A.alba)DSM 45114T Rpf蛋白信号肽及磷酸化位点
通过在线软件Signal P-5.0的预测分析了Rpf-E-1、Rpf-E-2和Rpf-2的信号肽,结果如图2所示,Rpf-E-1蛋白在位点21和22(AAA-DP)间存在信号肽;Rpf-E-2蛋白在位点40和41(ASA-SS)间存在信号肽;Rpf-2蛋白不存在信号肽.使用NetPhosBac 1.0程序对Rpf蛋白的氨基酸序列进行磷酸化修饰位点的预测分析,结果如表4所示,3个Rpf蛋白氨基酸序列均只含有丝氨酸和苏氨酸磷酸化位点.Rpf-E-1蛋白有1个丝氨酸和1个苏氨酸共2个磷酸化修饰位点.Rpf-E-2蛋白中含有2个丝氨酸和1个苏氨酸,共3个磷酸化修饰位点.Rpf-2蛋白中含有4个丝氨酸和2个苏氨酸共6个磷酸化修饰位点.蛋白质磷酸化是蛋白质翻译后修饰的重要可逆机制[21],是在蛋白质生物合成后的一种有关活性调节的化学修饰.

图2 白色放线动孢菌DSM 45114 T Rpf蛋白的信号肽分析Fig.2 Signal peptide analysis of Actinokineospora alba DSM 45114 T Rpf protein

表4 蛋白磷酸化位点分析Tab.4 Analysis of protein phosphorylation sites
2.3 白色放线动孢菌(A.alba)DSM 45114T Rpf蛋白的二级结构
用SOPMA 对白色放线动孢菌(A.alba)DSM 45114T的3个Rpf 蛋白进行预测,结果表明,Rpf-E-1二级结构由26.09%的α-螺旋、7.73%的β-转角、16.91%延伸链和49.28%无规则卷曲构成;Rpf-E-2二级结构由26.39%的α-螺旋、10.65%的β-转角、12.04%的延伸链和50.93%的无规则卷曲构成;Rpf-2二级结构由24.23%的α-螺旋、9.03%的β-转角、18.28%的延伸链和48.46%的无规则卷曲构成.
2.4 白色放线动孢菌(A.alba)DSM 45114T Rpf蛋白保守结构域组成


图3 白色放线动孢菌、藤黄微球菌、结核分枝杆菌Rpf结构域分析Fig.3 Function domain analysis of Rpf proteins among Actinokineospora alba,Micrococcus luteus and M ycobacterium tuberculosis
2.5 Rpf蛋白三级结构预测
利用SWISS-MODEL在线软件对白色放线动孢菌(A.alba)DSM 45114T的3个Rpf蛋白的三维结构进行了同源建模(图4),使用X 线对模板序列进行结构预测:Rpf-E-1 的可信度为34.18%,分辨率为0.19 nm,覆盖率为33%左右,与Rpf C 的三级结构相似度为0.38;Rpf-E-2的可信度为62.96%,分辨率为0.19 nm,覆盖率为38%左右,与RpfC 的三级结构相似度为0.5;Rpf-2 的可信度为37.14%,分辨率为0.26 nm,覆盖率为50%左右,与Rpf B的三级结构相似度为0.4.因Rpf类蛋白大家族中已明确三级结构的较少,而不同物种Rpf蛋白在系统发育过程中又有所进化,导致3个蛋白的可信度和覆盖率并不高.
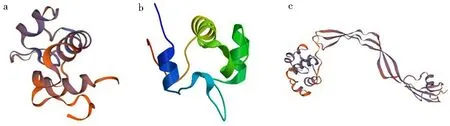
图4 Rpf三级结构预测模型Fig.4 Tertiary structure model of Rpf proteins
2.6 Rpf蛋白物种同源性分析
利用DNAMAN 软件,对白色放线动孢菌(A.alba)DSM 45114T的3个Rpf氨基酸序列与藤黄微球菌(M.luteus)Rpf、结核分枝杆菌(M.tuberculosis)Rpf A-E、红平红球菌(Rhodococcuserythropolis)Rpf氨基酸序列进行比对分析,发现它们均有1个由约70个氨基酸构成的Rpf特有保守区域(图5).Rpf-E-1与红平红球菌(R.erythropolis)Rpf同源性最高为22.84%,Rpf-E-2与藤黄微球菌(M.luteus)Rpf同源性最高为38.12%,Rpf-2与结合分枝杆菌(M.tuberculosis)Rpf B同源性最高为24.44%,这一结果与结构域的分析结果一致.

图5 白色放线动孢菌、藤黄微球菌、结核分枝杆菌、红平红球菌Rpf氨基酸序列对比Fig.5 Alignment of Rpf amino acid sequence among Actinokineospora alba,Micrococcus luteus,Mycobacterium tuberculosis and Rhodococcus erythropolis
2.7 rpf 基因的扩增及测序
用上述合成的3对引物,以白色放线动孢菌(A.alba)DSM 45114T的基因组为模板,扩增其rpf基因序列,大小均正确.使用纯化试剂盒纯化目的条带,经质量浓度为10 g/L的琼脂糖凝胶电泳检测,检测结果如图6.由图6可知,条带大小正确,并将纯化条带送至生工生物工程(上海)股份有限公司进行测序.

图6 rpf 基因扩增的电泳Fig.6 Electrophoretic map of rpf gene amplification
2.8 rpf 基因的克隆、表达载体的构建及鉴定
将PCR扩增产物克隆于p MD18-T 载体中,转化E.coliDH5α感受态细胞,挑选经酶切鉴定正确的克隆p MD18-T-rpf(图7),送至生工生物工程(上海)股份有限公司进行测序.提取测序的质粒p MD18-T-rpf双酶切后回收,与用相同酶切的pET-28a(+)质粒连接、转化,随机挑选2个克隆,提取质粒进行测序验证,测序结果均正确.

图7 p MD18-T-rpf 质粒酶切鉴定图谱Fig.7 Enzyme digestion map of p MD18-T-rpf plasmid
本研究克隆得到的序列已提交至GenBank数据库,登录号分别为Rpf-2(ON166660),G+C 的摩尔分数为67.62%;Rpf-E-1(ON166662),G+C的摩尔分数为72.97%;Rpf-E-2(ON166661),G+C 的摩尔分数为70.27%.
2.9 rpf 基因在大肠杆菌中的诱导表达及纯化
对rpf-2基因按照表2中的条件进行诱导表达(rpf-E-1、rpf-E-2未成功表达,分析其原因是因为2个基因在编码区均存在多个大肠杆菌的稀有密码子),结果见图8.泳道1~9分别是在正交实验表中的1~9号诱导条件下进行的蛋白诱导表达,泳道K 为不含质粒的E.coliBL21(DE3)菌体.在第8个诱导条件下,出现与阴性对照有明显区别的条带,条带大小52 ku左右,与预测条带大小相近,而在其他几个诱导条件下由于表达量低无法进行正交分析,所以采用第8个条件(25℃,IPTG浓度为0.5 mmol/L,诱导时间12 h)进行蛋白的纯化.经过蛋白的复性和纯化,得到了大小为52 ku的单一条带,纯化结果如图9.

图8 Rpf-2蛋白诱导表达的SDS-PAGEFig.8 SDS-PAGE of Rpf-2 protein induced expression

图9 纯化Rpf蛋白的SDS-PAGEFig.9 SDS-PAGE of purified Rpf protein
2.10 Rpf对VBNC状态菌复苏的验证
培养基中不同含量的重组Rpf蛋白对VBNC状态的藤黄微球菌(M.luteus)的复苏作用见图10,随着时间的增长,添加重组Rpf蛋白的实验组在600 nm 下的OD 值不断增长,并且明显高于未添加Rpf蛋白和添加10%(体积分数,下同)灭活Rpf蛋白的对照组.当向培养基中添加10%的Rpf蛋白时,复苏效果较明显.

图10 培养基中不同体积分数的重组Rpf蛋白对VBNC状态的藤黄微球菌的复苏促进作用Fig.10 Growth-promoting activity of different volume fraction Rpf protein on VBNC state M icrococcus luteus
不同体积分数重组Rpf蛋白对VBNC状态的滨海红球菌(R.marinonascens)的复苏作用见图11,向培养基中添加0.1%、1%和10%的重组Rpf蛋白均能使进入VBNC状态的滨海红球菌复苏生长,未添加Rpf蛋白和添加10%灭活Rpf蛋白的对照组生长情况基本一致.当向培养基中添加10%的Rpf蛋白时,复苏效果较明显.

图11 培养基中不同体积分数的重组Rpf蛋白对VBNC状态的滨海红球菌的复苏促进作用Fig.11 Growth-promoting activity of different volume fraction Rpf protein on VBNC state Rhodococcus marinonascens
3 讨论
自发现复苏促进因子以来,藤黄微球菌(M.luteus)[23-25]、结核分枝杆菌(M.tuberculosis)[26-28]、天蓝色链霉菌(Streptomycescoelicolor)[29]与红平红球菌(R.erythropolis)[30]等被研究得较多[31-32],且不同细菌Rpf种类和数量有一定差异.
为探究不同放线菌Rpf蛋白的种类、数量及功能,以白色放线动孢菌(A.alba)DSM 45114T为实验菌株,进行了生物信息学分析,发现白色放线动孢菌(A.alba)DSM 45114T中存在3个Rpf蛋白,基因所编码的蛋白均具有Rpf样结构域.这3个Rpf蛋白在保有自己独立特性的同时分别与结核分枝杆菌(M.tuberculosis)、红平红球菌(R.erythropolis)、藤黄微球菌(M.luteus)Rpf蛋白有较高同源性.成功克隆了rpf-2、rpf-E-1、rpf-E-2基因,并在大肠杆菌中成功表达了Rpf-2蛋白.将重组Rpf-2蛋白加入到VBNC状态的藤黄微球菌(M.luteus)和滨海红球菌(R.marinonascens)中,发现重组Rpf-2蛋白的添加量在体积分数10%时效果最好.此研究对白色放线动孢菌Rpf蛋白的后续功能探究奠定了理论和实验基础.
