基于分子对接技术双靶向PTP1B和GSK3β潜在活性小分子的发现
2022-05-30吴明凯张永豪姜德文龙治峰
吴明凯 张永豪 姜德文 龙治峰


摘要:目的 运用分子对接技术筛选出特异性作用于糖尿病双靶向PTP1B/GSK3β靶点的潜在活性小分子,为降糖药物的研究与开发提供依据和思路。方法 以PTP1B和GSK3β配体结合区域的晶体机构作为靶点,运用分子对接技术从小分子Specs和Chemdiv数据库中筛选双靶向PTP1B/GSK3β的小分子复合物的蛋白结构,删除晶体水分子,运用AutoDockTools以晶体小分子为质心定义对接的活性位点。结果 经过叠合与PTP1B/GSK3β对接打分,并进一步对潜在活性分子与PTP1B/GSK3β相互作用模式分析,筛选出22个小分子与PTP1B/GSK3β均具有较强的结合亲和力,其中小分子8019-7654、E589-4730和E589-4847三个化合物亲和性最优。结论 来源于Specs和Chemdiv数据库中的22种小分子化合物具有潜在双靶向于PTP1B/GSK3β的活性,其中小分子化合物8019-7654、E589-4730和E589-4847可优先选择开展降糖药物的研究,是潜在的双靶向于PTP1B/GSK3β活性小分子。
关键词:PTP1B;GSK3β;分子对接,糖尿病
糖尿病(DM)是一种由于胰岛素缺乏或胰岛素功能缺陷所致,以血糖浓度升高为特征的慢性代谢性疾病,其中以2型糖尿病(T2DM)最常见,即人体无法有效利用胰岛素。全球约4.22亿人患有糖尿病,每年约150万人死于糖尿病,且糖尿病病例数和发病率呈逐年增长趋势。另据国际糖尿病联盟(IDF)全球糖尿病概览2021年数据显示,在20歲到79岁的人群中,共有约5.37亿糖尿病患者,糖尿病总人数预计到2030年将增至6.43亿(11.3%),到2045年将增至7.83亿(12.2%)。其中,中国糖尿病人数以及未诊断糖尿病人数均排名第一,总数约为2.137亿人,糖尿病健康支出排名全球第二,糖尿病防控形势严峻,迫切需要有效的干预策略和政策来阻止糖尿病人数的增加。目前,常用的降糖药有胰岛素及其类似物、双胍类口服降糖药等,但疗效有限,无法防止胰岛β细胞的坏死和胰岛素的抵抗;并且,由于糖尿病患者需要长期用药,且发病机制复杂,易导致药物耐药性增加。因此,抗糖尿病治疗药物的研究一直是国际医学领域研究的热点,发现新的药物有助于缓解当前困局。
PTP1B(蛋白质酪氨酸磷酸酶-1B)是一种非跨膜蛋白酪氨酸磷酸水解酶,位于胞浆内质网,与蛋白络氨酸激酶(PTK)共同维持着络氨酸蛋白磷酸化的平衡,参与细胞信号转导。PTP1B是胰岛素信号转导通路中的关键的负性调控因子,能够阻断胰岛素信号传导,导致葡萄糖摄取减少和胰岛素抵抗,是糖尿病的有效治疗靶点。
GSK3β(糖原合成酶激酶3β)参与的AMPK/GLUT4/GSK3β信号通路与糖尿病息息相关。GSK-3β在骨骼肌组织和β-胰岛细胞中发挥着重要作用,GSK-3β的过表达可以阻断糖原合成酶的活化并增加血糖,通过抑制GSK3β解除其对糖原合成酶的阻断,并促进糖原合成,有效降低血糖浓度[1~2]。
反向虚拟筛选是一种对给定药物或活性小分子通过计算方法找到其潜在药物靶标的技术。本研究通过设计双靶向于PTP1B/GSK3β的小分子抑制剂,从两个调控位点控制糖尿病的发生和发展,针对糖尿病的耐药具有较好的缓解作用。从PTP1B/GSK3β结构出发,采用分子对接方法AutoDock Vina和构建PTP1B/GSK3β双靶标,采用分布式架构,构建了反向虚拟筛选平台,对潜在双靶向PTP1B/GSK3β小分子化合物进行有效性筛选,对于活性小分子还可以进行反向预测其潜在作用靶标,为活性化合物的后期改造和机制研究提供研究方向。该方法尤其适用于药理活性明确,而靶标未知的中药天然活性成分的研究,为开发新型抗糖尿病药物提供一定的参考和思路。
1方法
1.1 PTP1B和GSK3β受体准备
查阅文献,获得GSK3β(PDB ID:3I4B)和PTP1B(PDB ID:2VEY)与小分子复合物的蛋白结构,删除晶体水分子,运用AutoDockTools以晶体小分子为质心定义对接的活性位点。
1.2 配体准备
我们选择用小分子数据库是Specs公司和Chemdiv公司的数据库。其中,Specs数据库是由荷兰Specs公司开发的数据库,Chemdiv数据库是由美国Chemdiv公司开发的数据库,将筛选的小分子进行优化加氢,并用OpenBabel分子格式转换工具转成pdbqt格式文件。
1.3 分子对接
运用Autodock Vina软件进行分子对接,每个配体保留9个构象,提取打分最好的构象进行分析。小分子化合物与PTP1B/GSK3β双靶标相互作用模式用Pymol软件可视化处理。
2结果
2.1 小分子与PTP1B和GSK3β结合情况
叠合与PTP1B和GSK3β对接打分靠前的2000个小分子发现有22个小分子是交集(图1),即这些小分子与PTP1B和GSK3β均有较强的亲和力。它们为0407-0052、3241-0275、8003-5449、8019-7654、E589-4730、E589-4749、和E589-4761等。
2.2 潜在活性分子与PTP1B和GSK3β相互作用模式
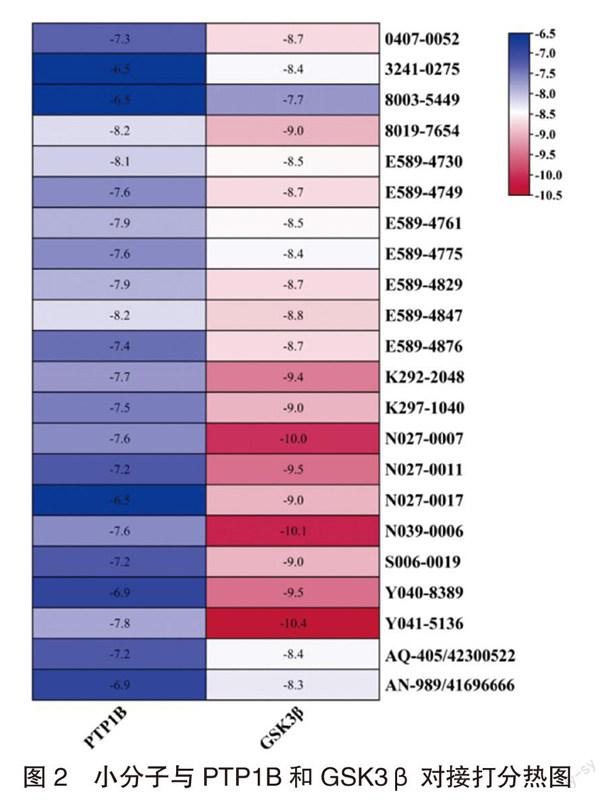
考察22个小分子与PTP1B和GSK3β的对接打分(图2)发现,这些小分子与GSK3β的对接打分在-7.7 kcal/mol ~ -10.4 kcal/mol之间,与PTP1B的对接打分在-6.5 kcal/mol ~ -8.2 kcal/mol之间。筛选对接打分同时优于-8.0 kcal/mol的小分子为:8019-7654、E589-4730和E589-4847。
为了考察小分子化合物8019-7654、E589-4730和E589-4847与PTP1B和GSK3β的相互作用,我们进一步分析了它们之间的作用模式(图3)。小分子化合物8019-7654、E589-4730和E589-4847与PTP1B的对接打分分别为-8.2 kcal/mol、-8.1 kcal/mol和-8.2 kcal/mol。在PTP1B体系中,8019-7654与PTP1B的Ile62、Arg141、Val135和Try134形成6个氢键;E589-4830与PTP1B的Ile62、Val135、Asn64和Lys85形成5个氢键;E589-4847与PTP1B的Ile62、Gln185和Lys85形成4个氢键。小分子化合物8019-7654、E589-4730和E589-4847与GSK3β的对接打分分别为-9.0 kcal/mol、-8.5 kcal/mol和-8.8 kcal/mol。在GSK3β体系中,8019-7654与GSK3β的Tyr46、Asp181、Phe182、Gln266和Arg221形成7个氢键;E589-4830与GSK3β的Asp48、Asp181、Ser216、Gln262和Arg221形成7个氢键;E589-4847与GSK3β的Asp48、Gly259、Asp181、Gln262和Arg221形成5个氢键。
3讨论
对实验结果进行综合分析,发现小分子化合物8019-7654、E589-4730和E589-4847具有较好的潜在双靶向于PTP1B和GSK3β受体。其中化合物8019-7654亲和力最强,双靶向于PTP1B/GSK3β受体对接打分为:-8.2 kcal/mol和-9.0kcal/mol。与PTP1B/GSK3β双靶标相互作用模式分析发现,与PTP1B的Ile62、Arg141、Val135和Try134形成6个氢键,与GSK3β的Tyr46、Asp181、Phe182、Gln266和Arg221形成7个氢键,均体现出较强的氢键亲和力。化合物E589-4730亲和力次之,双靶向于PTP1B/GSK3β受体对接打分为:-8.1 kcal/mol和-8.5kcal/mol。与PTP1B/GSK3β双靶标相互作用模式分析发现,与PTP1B的Ile62、Val135、Asn64和Lys85形成5个氢键,与GSK3β的Asp48、Asp181、Ser216、Gln262和Arg221形成7个氢键,均体现出较强的氢键亲和力。化合物E589-4847亲和力较前两者相当,双靶向于PTP1B/GSK3β受体对接打分为:-8.2 kcal/mol和-8.8kcal/mol。与PTP1B/GSK3β双靶标相互作用模式分析发现,与PTP1B的Ile62、Gln185和Lys85形成4个氢键,与GSK3β的Asp48、Gly259、Asp181、Gln262和Arg221形成5个氢键,氢键亲和力较前两者弱一些。可见以上三个小分子化合物具有进一步研究的价值。
分子对接模拟配体小分子物质与受体生物大分子物质相互作用的技术方法,实质就是配体与受体相互作用,包括静电作用、氢键作用、疏水作用、范德华作用分子识别的过程。小分子配体与大分子蛋白受体之间通过能量匹配和几何匹配,达到相互识别,用来模拟小分子和蛋白之间的相互作用,使我们可以通过模拟小分子在靶蛋白结合位点的行为来阐述基础生理过程,并通过计算预测两者间的结合模式和亲和力。
4结论
综合分析,运用分子对接Autodock软件从Specs和Chemdiv数据库中筛选到小分子化合物8019-7654、E589-4730和E589-4847与PTP1B/GSK3β具有较好的结合亲和力,体现出一定的有效抑制性,提示这三個小分子是潜在的双靶向PTP1B和GSK3β抑制剂,可优先选择开展降糖药物的研究,是潜在的双靶向于PTP1B/GSK3β活性小分子化合物。
参考文献
[1] 李超.海洋天然产物BDDPM靶向调控PTP1B的降糖作用及机制研究[D].北京:中国科学院大学(中国科学院海洋研究所),2019.
