三种四膜虫全基因组N6-甲基腺嘌呤比较研究
2022-05-27柴小翠杨文涛袁冬霞
王 粟 柴小翠, 张 晶 杨文涛, 袁冬霞 熊 杰
(1. 大连海洋大学, 大连 116023; 2. 中国科学院水生生物研究所, 武汉 430072; 3. 中国科学院大学, 北京 100049)
DNA甲基化(DNA methylation)是一类表观遗传修饰, 发生的常见位点为腺嘌呤外环氨基(N6)、胞嘧啶环5-碳(C5)和胞嘧啶外环氨基(N4)。DNA甲基化作用主要是DNA甲基转移酶以S-腺苷甲硫氨酸(SAM)为甲基供体, 将甲基共价结合到碱基特定位点上的过程[1], 而根据共价结合位点的不同被区分为N6-甲基腺嘌呤(6mA)、C5-甲基胞嘧啶(5mC)和N4-甲基胞嘧啶(4mC)[2—4]。基于二代测序技术建立的重亚硫酸盐测序技术极大促进了DNA甲基化的研究, 但仅局限于检测分析5mC[5]。近年来, 随着三代测序技术的发展, 另一种DNA甲基化修饰6mA的研究也取得了一系列重要的进展[6]。
对6mA的认识最早来源于原核生物的限制修饰(R-M)系统[7]。原核生物依靠自身基因组上的6mA修饰, 使自身基因组DNA具有抗限制性内切酶消化的能力, 同时消化未被6mA修饰的外源DNA,从而保护宿主基因组[8]。在真核生物中, 6mA广泛分布。对于真核生物6mA的认识近年来取得了一系列发现。研究认为, 6mA在转录调控、控制转座子表达、控制核小体定位和作为表观遗传标记方面均有作用。例如, 在秀丽隐杆线虫(Caenorhabditis elegans)中, H3K4me2去甲基化酶的缺失会导致H3K4me2累积从而丧失受精能力[9], 而6mA DNA去甲基化酶缺失会加速H3K4me2的积累, 加强线虫的生育缺陷, 而潜在的6mA DNA甲基转移酶缺失会降低线虫的6mA水平, 并抑制线虫的生育缺陷[10]。在莱茵衣藻(Chlamydomonas reinhardtii)中发现6mA在转录起始位点附近富集于核小体连接DNA上[11]。在黑腹果蝇(Drosophila melanogaster)中6mA被证明在胚胎时期是高度动态的, 并且可以被特异性转录因子识别, 并通过调节叉头框(forkhead box FOX)基因来影响胚胎发育的作用[12]。在阴道毛滴虫(Trichomonas vaginalis)中作为全基因组主要甲基化修饰标记, 富集于某些转座子元件超家族, 并在基因上起沉默作用[13]。
然而6mA相关的研究主要针对单个真核模式生物, 考察其分布特征、动态变化及相关甲基化酶的功能。通过对分属于不同进化类群的物种进行6mA研究, 已揭示一些远缘物种中6mA的分布特征和功能异同。尽管如此, 已研究的物种分化时间久远, 进化关系远, 难以在进化尺度上精细比较物种间的6mA分布和功能变化, 提示6mA这种分布和功能的变化发生在更小的进化尺度上。因此, 开展近缘物种间6mA的比较对理解其在进化上的动态变化具有重要意义。到目前为止, 仅Mondo等[14]在16种真菌的6mA单碱基分辨率比较研究中, 发现了早期分化真菌中高达2.8%的腺嘌呤被甲基化, 并且显著富集于转录起始位点附近且与5mC分布呈负相关。在其他类群近缘种中比较6mA甲基化组学研究尚未开展。
本研究利用PacBio单分子实时测序技术, 绘制了2种四膜虫Tetrahymena malaccensis和Tetrahymena pyriformis的全基因组单碱基分辨率6mA图谱, 结合Wang等[15]发表的Tetrahymena thermophila全基因组单碱基分辨率6mA数据, 在此基础上开展了四膜虫比较甲基化组分析, 揭示了6mA在四膜虫基因组中的分布特征、进化保守性和差异,帮助理解四膜虫中6mA修饰在进化上的变化及其功能。
1 材料与方法
1.1 实验材料
T. thermophila、T. malaccensis和T. pyriformis的基因组之前已经被测序, 可以在Tetrahymenacomparative genome database(http://ciliate.ihb.ac.cn/tcgd/)上获得。三伪尖毛虫(Oxytricha trifallax)的基因组数据来自OxyDB(https://oxy.ciliate.org/)。实验中用到的T. malaccensis和T. pryformis细胞株系与之前的研究相同[16]。
在无菌条件下, 将实验细胞株接种至新鲜Super Proteose Peptone(SPP)培养基[17]中, 置于温度为28℃, 转速为135 r/min的摇床内培养。待细胞生长至对数期, 细胞密度约为(3.2—3.8)×108cells/mL(采用Beckman Coulter Counter Z1进行细胞计数)用于分离大核。
1.2 四膜虫大核分离提纯
将准备好的实验材料用差速离心法分离四膜虫的大核: 试剂配方参考差速离心法[18]。离心收集(3.2—3.8)×108cells/L的对数期细胞, 在冰上用200 μL MediumA悬浮细胞, 倒入搅拌器(搅拌器采用8011s/1 L Waring匀浆仪), 加入2 mL PMSF(0.1 mol/L)和1 mL正辛醇, 18000 r/min破碎10s, 加入9 mL EDTA(0.5 mol/L), 22000 r/min破碎30s, 在冰上冷却1min, 再22000 r/min破碎20s。镜检确定细胞大小核分开后, 使用平转离心机(Allegra 25R Centrifuge Beckman离心机)600×g, 4℃, 10min, 样品沉淀为大核。若大核样品仍有较多小核污染, 则加入50 μL正辛醇, 充分颠倒混匀后1500×g, 4℃, 2min, 去上清。镜检确定样品合格, 然后将大核样品1500×g离心, 4℃, 3min, 弃上清, 再加入1 mL Nuclear Wash Buffer吹打混匀, 再次1500×g离心, 4℃, 3min, 弃上清, 重复2次, 最后一次将大核沉淀摇匀后, 使用QIAGEN®Genomic试剂盒进行DNA提取。
1.3 DNA建库和测序
根据建库的片段大小用g-TUBEs(Covaris,USA)对基因组DNA进行目的打断; 利用AMPure PB磁珠富集、纯化目的片段DNA并利用0.7%琼脂糖凝胶电泳对片段化DNA进行质检; 使用SMRT-bellTMExpress Template Prep Kit 2.0试剂盒对合格DNA样本进行文库构建。采用外切酶去除DNA单链末端, 随后对片段化的DNA进行损伤修复、末端修复并添加Poly-A尾, 进而进行茎环状测序接头连接。使用SMRTbell Enzyme Cleanup Kit, 对SMRT-bell文库进行核酸酶消化处理并纯化回收后, 再利用切胶仪BluePippin(Sage Science, USA)筛选目的片段, 纯化后得到文库。使用Agilent 2100 Bioanalyzer(Agilent technologies, USA)检测文库片段大小。
建库完成后利用Sequel Ⅱ Binding Kit 2.0上机试剂盒对DNA文库、测序引物Sequel Sequencing Primer v2及聚合酶进行一定比例混合, 并将文库转移到Sequel Ⅱ系列测序仪纳米孔内, 进行实时单分子测序。
1.4 数据分析
本文用到的T. thermophila三代测序数据从网上直接下载, 来自于Wang等[15]2017年公开发表的三代测序数据,O. trifallax的6mA位点注释数据来自于Beh等[19]2019年公开发表的数据。而T. malaccensis和T. pryformis为本研究新测三代数据。这3种四膜虫的基因组数据均来自于Tetrahymenacomparative genome database(http://ciliate.ihb.ac.cn/tcgd/)。O. trifallax的基因组数据来自OxyDB(https://oxy.ciliate.org/)。使用SMRTLINK v6 对PacBio三代数据进行6mA位点的鉴定, 并对每个位点的甲基化水平进行计算。同时, 利用SMRTLINK v6对6mA位点区域的保守基序(motif)进行分析。并通过Gbrowse对6mA位点注释到基因组的结果进行可视化。通过自写的Perl脚本对单碱基分辨率甲基化组上的所有6mA位点上下游序列的其他6mA位点进行分析, 统计出6mA的周期性分布规律。
同时, 利用自写的Perl脚本对AT基序中A和T(反义链的A)的甲基化水平进行比较。为了研究6mA位点在小核染色体水平上是否有分布差异, 参考我们之前的研究, 将嗜热四膜虫每条小核染色体平均分为长度相等的25个片段[20], 用自写的Perl脚本统计每个片段上的6mA甲基化水平, 计算出6mA位点在小核染色体上的6mA甲基化水平差异。3种四膜虫种间直系同源基因和种内并系同源基因基于OrthoMCL软件进行鉴定[20]。使用MUSCLE v3.8.31对直系同源基因和并系同源基因进行序列比对。根据单碱基分辨率6mA甲基化图谱, 对上述两类基因的甲基化数目进行统计, 并用R进行散点图绘制, 线性回归分析及相关性分析。对O. trifallax大核基因组的比较分析采用相同的分析方法。
2 结果
2.1 三种四膜虫全基因组6mA图谱
基于PacBio三代测序数据, 绘制了3种四膜虫全基因组单碱基分辨率6mA图谱(表 1)。T. thermophila原始数据约为8.8G, 测序深度85X;T. malaccensis原始数据约为11.2G, 测序深度105X;T. pyriformis原始数据14.8G, 测序深度127X。T. malaccensis中含有约61 万个6mA位点, 占大核基因组位点数的0.56%;T. pyriformis中含有约64万个6mA位点, 占大核基因组位点数的0.56%。T. thermophila大核基因组103M, 有约46万个6mA 位点, 占大核基因组位点数的0.44%, 相对比T. malaccensis和T. pyriformis中6mA少, 可能是由于测序深度相对较低导致一些低丰度的6mA位点没有被鉴定出来。整体上看, 6mA位点在全基因组中所占比例在3种四膜虫中类似。
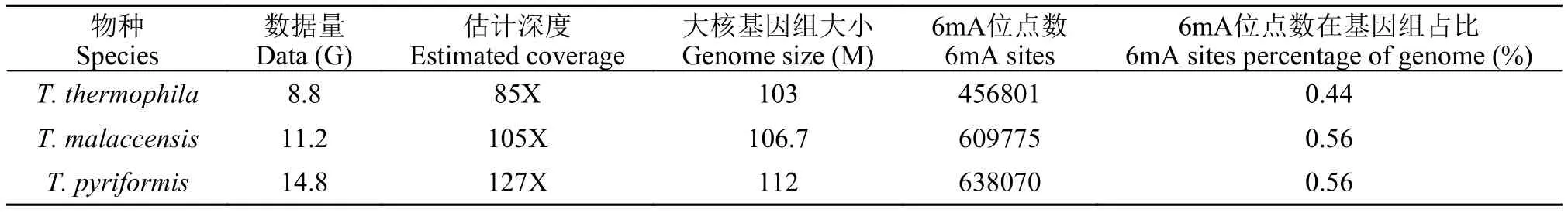
表1 三种四膜虫大核全基因组6mA图谱Tab. 1 The genome wide 6mA map of the macronuclei in three Tetrahymena species
2.2 三种四膜虫具有保守的6mA分布特征
以鉴定到的每一个单碱基6mA位点为原点, 在上、下游各1000 bp长度范围内统计6mA位点的出现频次, 发现6mA在四膜虫大核基因组上分布具有200 bp周期性(图 1A)。这种200 bp的周期性分布在3种不同的四膜虫中类似。而核小体的单元长度加上linker DNA的长度约为200 bp。这种周期性可以从6mA对组蛋白的排斥作用来理解, 核小体的内部并不能支持稳定的6mA存在[21]。
对鉴定到的6mA位点进行基序特征分析, 发现3种四膜虫的6mA位点显著富集于AT基序(图 1B),AT基序占到了整个基因组中6mA总数的86.42%—97.58%, 说明AT序列甲基化是四膜虫中的保守进化特征。对AT序列A和T(反义链A)的甲基化水平进行比较发现, A和T的甲基化水平的比值的分布峰为1(图 1C), 说明在大部分位点中, 同一个AT序列中A和T(反义链A)的甲基化水平相当。这一结果提示DNA在半保留复制时或者复制后, AT基序中反义链6mA的建立可能是以正义链6mA位点为“模板”, 最终形成对称的甲基化ApT二核苷酸。

图1 三种四膜虫中6mA的保守分布特征Fig. 1 Conserved 6mA distribution patterns in three Tetrahymena species
为了考察6mA的分布与基因保守性之间的关系, 对3种四膜虫中都存在的基因(保守基因)和仅在1种四膜虫中存在的基因(种特异基因)中6mA位点的数目进行了分析和比较, 发现保守基因中6mA位点数目显著高于种特异基因(图 2A), 且在3种四膜虫中均具有类似的结果。另外, 四膜虫中有一类特殊的基因—富亮氨酸重复基因(LRR基因), 我们之前的研究表明, 四膜虫中LRR基因从保守性上可以根据是否含有90 bp外显子, 按照是否含有输出一致性序列(Consensus repeat sequence, CRS)可分为3类。其中第I类不含有90 bp外显子的LRR最为保守; 第Ⅱ类含有至少一个90 bp外显子但不含有CRS, 保守性次之; 第Ⅲ类含有至少一个90 bp外显子和至少一个CRS, 为种特异基因[20]。对这三类基因中6mA位点数目的分析发现, 第Ⅰ类保守LRR基因中6mA位点数远远高于第Ⅱ类, 且均显著高于第Ⅲ类(图 2B)。更进一步, 我们将大核中6mA位点比对回小核染色体, 发现大核基因组的6mA在小核染色体上呈差异性分布, 即6mA在小核显著富集于染色体臂, 而在近着丝粒和亚端粒附近区域则分布较少(图 2C)。我们前期研究显示, 四膜虫中保守基因显著富集于小核染色体臂, 而种特异基因显著富集于近着丝粒和亚端粒区域, 这一分布与6mA的分布很类似。因此, 以上结果说明, 6mA更偏向于分布于保守性高的基因中。
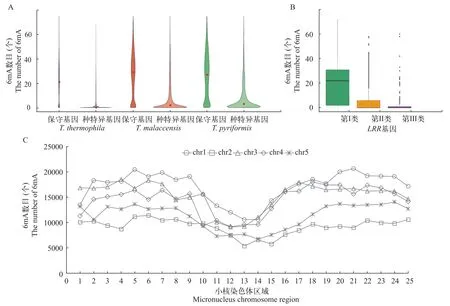
图2 三种四膜虫中6mA分布偏好Fig. 2 6mA distribution preferences in three species Tetrahymena
2.3 四膜虫6mA在同源基因中分布的区域近似性和单碱基水平的可变性
对四膜虫中的6mA位点分布特征分析结果表明, 四膜虫保守基因中具有更高的6mA位点数目。那么, 值得注意的是6mA的分布在不同物种间或者同一物种内的同源基因中具有什么特征? 为了回答这一问题, 我们首先对3种四膜虫种间一对一直系同源基因中6mA位点进行了分析和比较, 整体上看,物种间保守的直系同源基因中6mA位点数目具有显著正相关性(图 3A), 说明直系同源基因6mA的分布模式可能类似。因此, 我们进一步对6mA在基因上的分布进行了比较, 发现6mA主要分布于基因的5′区域, 且成簇分布(图 3B)。在不同物种间的直系同源基因中, 6mA的成簇分布模式类似, 主要表现为成簇的数目相同或类似, 如图 3B中基因对有明显的3个甲基化簇且分布类似。为了进一步考察6mA在直系同源基因分布特征, 在单碱基水平比较了一对一直系同源基因中6mA分布的异同, 发现6mA分布的在单碱基水平并不完全保守(图 3B)。以上结果表明6mA分布的在直系同源基因中具有区域的近似性, 但在单碱基水平存在可变性。
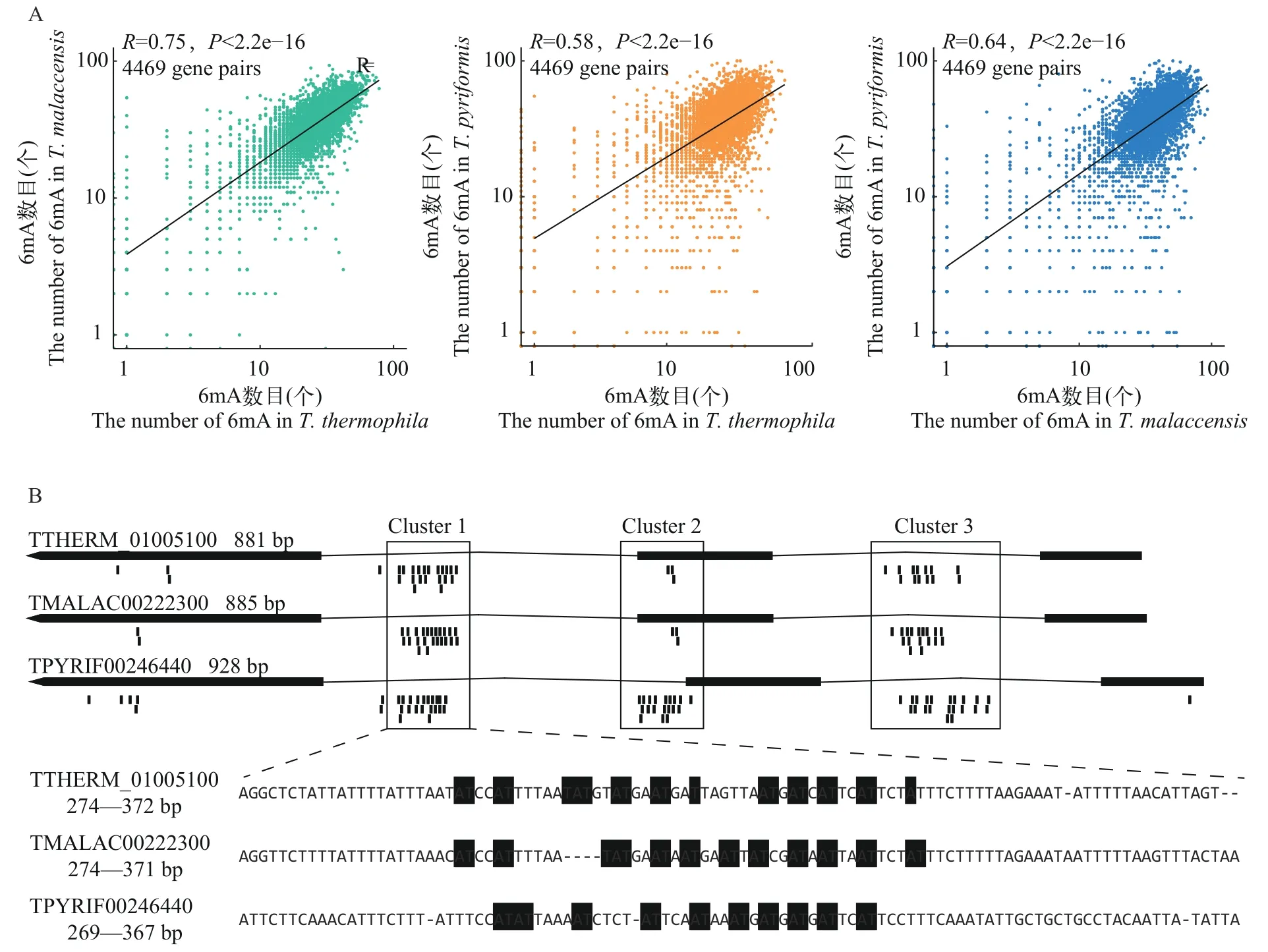
图3 三种四膜虫直系同源基因中6mA分布模式比较Fig. 3 Comparison of 6mA distribution patterns among orthologs in three Tetrahymena species
为了探究这种分布模式保守性在较远亲缘关系的纤毛虫中是否存在, 我们对已报道的O. trifallax大核基因组中的6mA位点进行分析[19], 发现O.trifallax具有与3种四膜虫类似的200 bp周期性分布(图 4A)。但在与T. thermophila的直系同源基因6mA甲基化水平比较中, 相关系数和显著性水平均低于3种四膜虫内部比较结果(图 4B)。在单碱基水平比较通过OrthoMCL鉴定出的直系同源基因对,发现两者间也存在区域近似性, 但相对于分歧时间较近3种四膜虫之间则较弱, 而单碱基水平的保守性则更弱(图 4C)。
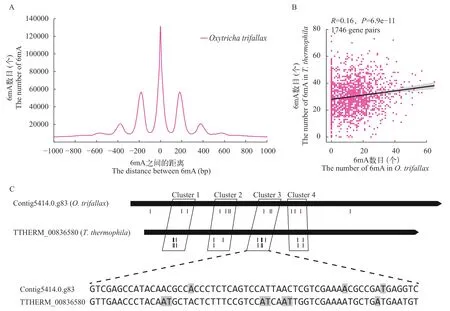
图4 在O. trifallax大核基因组中6mA的分布模式Fig. 4 6mA distribution patterns on macronuclear genome in O. trifallax
除了直系同源基因外, 我们也对3种四膜虫中,发生近期复制的(氨基酸序列一致性90%以上)的并系同源基因中的6mA分布进行了分析。我们发现两个近期复制的基因, 其所具有的6mA位点数目呈显著正相关(图 5A), 进一步对6mA位点在并系同源基因间的分布进行了比较, 发现其分布模式与直系同源基因类似, 也具有区域近似性, 如图 5B中基因对有明显的3个甲基化簇且分布类似。并且在较高同源性的并系同源基因中, 6mA位点在单碱基水平也具有较高的保守性(图 5B)。
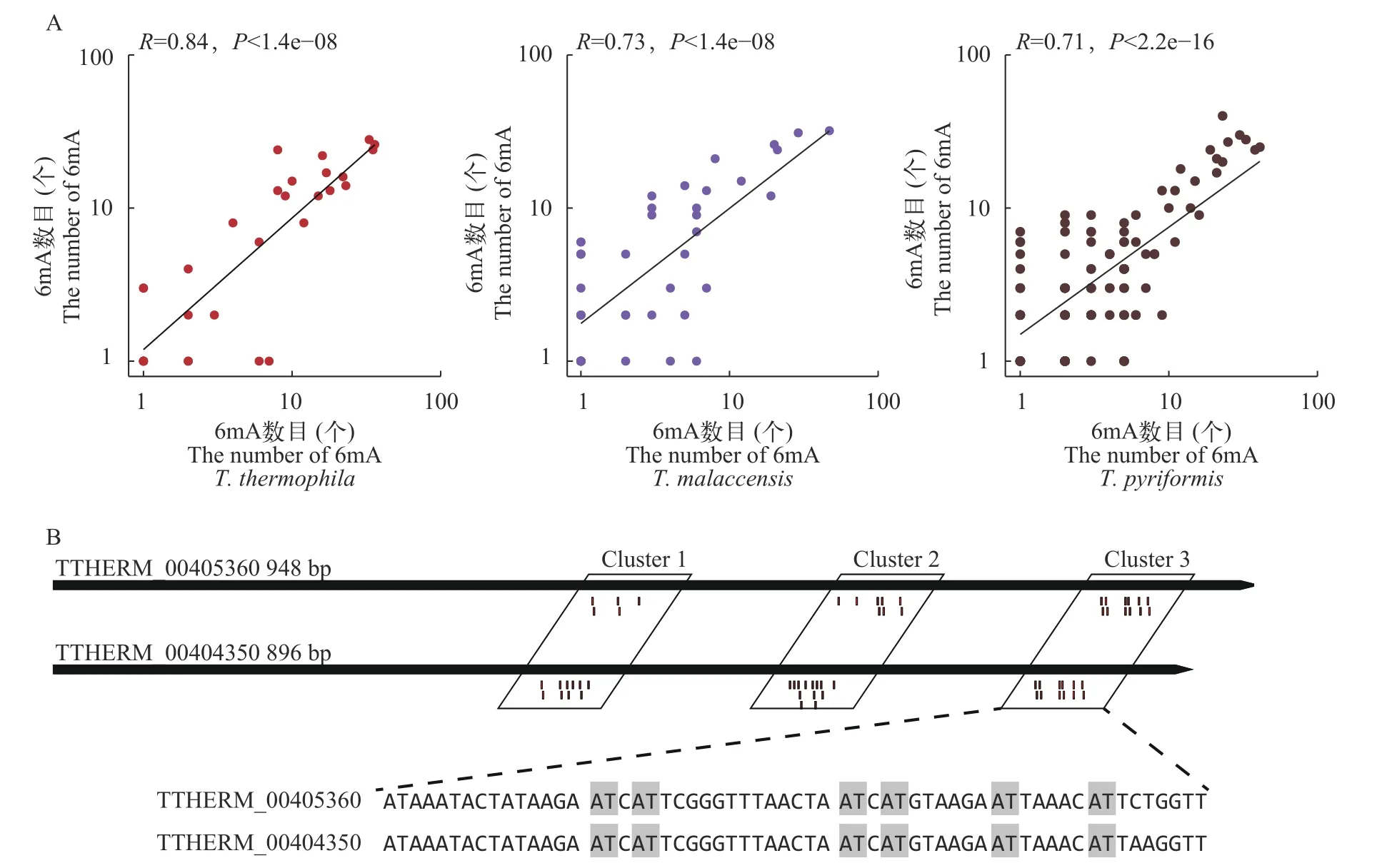
图5 三种四膜虫并系同源基因中6mA分布模式比较Fig. 5 Comparison of 6mA distributions in inparalogs genes of three Tetrahymena species
由于在物种分化和进化过程中, 基因组会不断累积DNA突变, A突变成其他任意3种碱基(T、G和C)都将会导致6mA修饰的消失, 甚至AT基序中任意一个突变都可能导致另一个位点上6mA修饰的消失。而上述我们发现的6mA分布的在直系同源基因中具有区域的近似性, 但在单碱基水平存在可变性这一结果提示进化上基因组中一个位点6mA消失后, 细胞可能在其附近区域重新建立6mA位点, 以维持相应的功能。
为了理解四膜虫分化过程中6mA的产生, 我们对四膜虫直系同源基因中的6mA进行了比较(图 6A),使用的同源基因序列总长度均为10 Mb左右。3种四膜虫的系统发育关系如图 6B所示,T. thermophila和T. malaccensis亲缘关系较近, 聚在一起, 分化时间约为22个百万年, 而T. pyriformis的分化时间更为久远, 约116个百万年。比较发现三种四膜虫共有的6mA位点有28769个;T. thermophila和T.malaccensis共有的44276个6mA位点被认为是和T.pyriformis分化后到T. thermophila和T. malaccensis分化前产生的新位点;T. pyriformis特异的6mA位点81083个,T. thermophila特异的6mA位点31121个,T. malaccensis特异的6mA位点49732个。
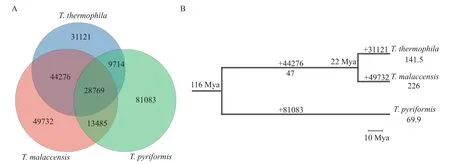
图6 6mA位点数目在3种四膜虫物种分化过程中的动态变化Fig. 6 Dynamic changes of 6mA sites during the evolution of three Tetrahymena species
因此, 计算出T. pyriformis的特异6mA位点产生速率为69.9个位点每Mb每百万年; 而T. thermophila和T. malaccensis在和T. pyriformis分化后且互相分化之前的94个百万年中6mA位点产生速率为47个位点每Mb每百万年;T. thermophila和T. malaccensis相互分化后,T. thermophila的特异6mA产生速率为141.5个位点每Mb每百万年,T. malaccensis的特异6mA产生速率为226个位点每Mb每百万年。从以上结果中可以估算出四膜虫进化过程中新6mA位点产生的速率大约为69.9—226个位点每Mb每百万年。
3 讨论
3.1 四膜虫大核基因组上6mA分布特征比较
随着三代测序技术普及, 报道全基因组6mA甲基化的物种数目越来越多, 包括黑腹果蝇、秀丽隐杆线虫、莱茵衣藻、嗜热四膜虫和三伪尖毛虫等[10,15,19,22,23]。在纤毛虫的不同类群中, DNA甲基化类型为5mC和6mA, 有的只存在一种, 有的两种都存在[24]。在O. fallax、T. thermophila、T. pyriformis和Stylonychia mytilus中只检测到6mA[25—27],而在Blepharisma japonicum的营养细胞大核中,5mC和6mA都能被观察到[28,29]。在T. thermophila的营养细胞中, 大约0.8%的大核基因组腺嘌呤被甲基化, 而在小核基因组中检测不到6mA[25,30]。而在接合生殖14—16h时可以在新大核中检测到新生6mA[31]。Paramecium大核和小核DNA中6mA的含量约为2.5摩尔百分比[32]。Kwok等[33]提出,6mA可能参与了Paramecium大核中一些序列的转录失活。
近些年, 通过单分子实时测序技术, 直接从O.trifallax和T. thermophila大核中获得了全基因组碱基对分辨率的6mA位点图谱[15,19]。Wang等[15]在T.thermophila中发现6mA修饰富集于一致序列5′-AT-3′中, 也定位于转录起始位点下游的DNA连接区(Polymerase Ⅱ转录基因的转录起始位点), 并直接影响核小体的定位。四膜虫的MT-A70同源物ATM1 (6mA DNA甲基转移酶)是结合生殖后细胞正常生长发育所必需的[34]。Beh等[19]认为O. trifallax中负责6mA修饰的酶MTA1c (DNA甲基转移酶)不利于核小体占据。6mA使AT碱基对失稳, 降低了DNA的熔化温度导致核小体占用率降低。
将已发表的黑腹果蝇、莱茵衣藻、秀丽隐杆线虫和3种四膜虫的6mA各方面数据进行比较, 从基因组中6mA位点含量上看, 果蝇6mA含量约占基因组中所有A的0.001%—0.07%, 线虫为0.01%—0.4%, 衣藻约为0.4%—0.8%, 四膜虫约为0.56%—0.77%。物种间6mA含量差异很大, 以单细胞衣藻的含量最高, 果蝇和线虫的含量较低, 四膜虫的含量更接近衣藻。
从6mA分布规律上看, 果蝇中6mA富集于转座子区域, 线虫中6mA无明显分布特征, 仅在传代过程中不同阶段表现出与H3K4me2组蛋白甲基化修饰的增加具有一定相关性[9,35], 衣藻中6mA则倾向于分布在核小体之间的linker DNA区域, 具有一定周期性, 并显著富集于转录起始位点附近[23]。四膜虫中6mA的分布规律和衣藻相似, 6mA分布倾向于核小体间linker DNA区域, 在编码基因上显著富集于5′端转录起始位点附近[15], 并且我们发现6mA在基因组上的分布具有200 bp周期性, 与四膜虫核小体长度与linker DNA长度之和相等, 也侧面支持了Wang等[15]发现嗜热四膜虫6mA位点富集于核小体Linker DNA的结果。并且有研究表明, 四膜虫中6mA对核小体的定位具有指导作用[36]。本研究发现在3种亲缘关系相近的四膜虫中均有此规律, 说明在四膜虫属中这种现象可能比较常见。
其中, 6mA对linker DNA的偏好性可以解释为对组蛋白的排斥作用, 这得到了体外重构实验和遗传学实验的支持, 当6mA水平改变时, 核小体的位置受到了可能产生排斥的空间位阻干扰, 因为包裹DNA的核小体周期性地位于组蛋白表面的主要沟槽一侧[36], 而有模型显示大多数6mA二核苷酸在两条链上的甲基之间存在空间位阻冲突[21]。
从6mA区域的基序特征上看, 物种间差异明显,线虫的6mA基序主要为AGAA/GAGG, 衣藻为NATN, 而果蝇中未见报道保守基序。在3种四膜虫中得到的保守基序均为AT, 占到四膜虫基因组中总6mA位点数的86.42%— 97.58%。并且3种四膜虫的基因组6mA甲基化水平也相当, 结合四膜虫属内普遍基因组高AT含量, 推测在四膜虫属内6mA甲基化基序也可能是保守的。并且, 对同一个AT序列A和T(反义链A)的甲基化水平进行比较发现, 3种四膜虫中大部分AT基序均形成对称的ApT甲基化二核苷酸, 意味着DNA半保留复制时或者复制后, 新生链上的6mA修饰可能通过其所处的AT基序中模板链的6mA得以重新建立。
3.2 6mA数目与基因保守性具有相关性
在比较3种四膜虫的直系同源基因的6mA甲基化水平、位点簇分布和位点详细分布后发现, 直系同源基因间甲基化水平类似, 位点簇分布保守而位点却不那么保守。因此我们推测四膜虫6mA甲基化位点序列发生突变时, 具体的6mA甲基化位点发生了某种代偿机制, 在突变位点附近建立了新的6mA甲基化位点, 使得基因上的6mA甲基化水平得以保持, 从而维持其相关功能。通过分析6mA在小核染色体上的分布, 发现基因上6mA甲基化水平与基因在小核染色体上的定位有关。为寻找这种甲基化水平差异的原因, 我们之前的研究[20]对于四膜虫不同保守程度的LRR基因和10个四膜虫种间的保守基因和种特异基因划分, 通过6mA位点图谱比较, 发现其分布偏好于保守基因和年龄更大的LRR基因。
在和嗜热四膜虫较远亲缘关系的纤毛虫O. trifallax中, 比较其6mA的分布模式, 发现O. trifallax大核基因组中6mA分布也具有200 bp周期性, 根据6mA与核小体的互斥关系, 推测这可能是具有6mA的真核细胞的共有特征。在直系同源基因的甲基化水平上,O. trifallax与T. thermophila的相关性要弱于3个四膜虫种内部的相关性, 说明6mA的分布在纤毛虫中存在一定保守性的同时, 随着同源基因序列的分歧度增加, 保守性减弱。
基因复制可以产生新的基因, 主要是通过产生与原基因拷贝的功能分化从而增加生物复杂性[37,38]。而在3种四膜虫的并系同源基因中, 在近期发生基因复制事件的基因, 表现出6mA位点具有高保守性,意味着在这类事件中, 6mA位点可一起复制, 保持其基因调控或者表观遗传标记作用。
本研究的结果表明6mA位点的数目与基因的保守性呈正相关。尽管如此, 由于基因的保守性和基因的表达也呈正相关, 在多个物种中报道了6mA位点富集于转录起始位点附近[14,15,23]。因此,难以确定的是, 6mA位点数目与基因保守性是直接相关还是间接相关。
3.3 DNA 6mA甲基化位点在物种分化中的动态变化
在已报道6mA位点的3种具有代表性的真核生物中, 线虫属于线虫动物门, 果蝇属于节肢动物门,衣藻属于绿藻门, 其中线虫和果蝇进化关系较近,均属于后生动物, 但分化时间也较为久远(约为744百万年)。而基于T. thermophila、T. malaccensis和T. pyriformis这3个四膜虫种, 能在更小的进化尺度上探究更精确的6mA位点动态变化。其中, 这3种四膜虫同属“Borealis”类群,T. thermophila和T.malaccensis分化时间约为22个百万年,T. thermophila和T. pyriformis分化时间约为161个百万年[20]。通过比较3者的基因组6mA图谱, 得出四膜虫中6mA甲基化修饰位点建立的速度约69.9—226个位点每Mb每百万年。在直系同源基因和旁系同源基因都呈现甲基化水平保守的基础上, 意味着基因拷贝数变化和基因功能变化会导致6mA甲基化修饰位点数目变化。当然, 这种数目变化也受到基因上的甲基化水平变化, 即具体的甲基化位点的消失和产生的影响。物种分化过程中6mA甲基化修饰变化应该是一个动态平衡的过程, 具体的机制还有待研究。
