茶树CsHIPP26.1互作蛋白的筛选与验证
2022-05-16范延艮王域刘富浩赵秀秀向勤锃张丽霞
范延艮,王域,刘富浩,赵秀秀,向勤锃,张丽霞
茶树CsHIPP26.1互作蛋白的筛选与验证
范延艮,王域,刘富浩,赵秀秀,向勤锃,张丽霞*
山东农业大学园艺科学与工程学院/作物生物学国家重点实验室,山东泰安 271018
【背景】‘黄金芽’属于光照敏感型黄化茶树()品种,叶片色泽呈现强光黄化、弱光复绿的特点,但叶色响应光照的黄化机制并不明确。前期通过对黄化叶片、遮阴复绿叶片以及常绿品种叶片的蛋白组研究发现,重金属相关异戊二烯化植物蛋白CsHIPP26.1(TEA000549)的表达响应光照强度,表明CsHIPP26.1可能参与调控‘黄金芽’叶色黄化的光响应过程。【目的】通过筛选与CsHIPP26.1互作的光信号响应相关的蛋白,为叶片色泽响应光照信号变化提供科学依据。【方法】以‘黄金芽’茶树1芽2叶为材料进行和互作基因的克隆,经酵母双杂交筛库,然后将筛选得到的目的蛋白进行酵母双杂交点对点验证、体内双分子荧光互补(BiFC)和体外pull-down等技术进行蛋白互作的进一步验证。【结果】通过酵母双杂交对茶树cDNA文库进行筛选,共筛选到26个候选互作蛋白,主要集中在细胞组分、结合以及催化活性方面发挥作用,其中生物素合成代谢过程富集程度较高,与光信号通路及叶绿素合成相关的蛋白只有编号为TEA026466.1的bHLH30转录因子。克隆bHLH30转录因子后发现,该转录因子与茶树光信号传导途径蛋白PIF4处于同一进化树分支,且含有与茶树PIF4蛋白相同的HLH和ACT结构域,将该bHLH30转录因子命名为CsPIF4.2,GenBank登记号为MW116834。进一步通过体外Pull-down蛋白互作和体内双分子荧光互补(BiFC)验证发现,CsHIPP26.1和CsPIF4.2蛋白能够发生互作,并且发生互作的部位在细胞核内。【结论】初步筛选出26个与CsHIPP26.1互作的蛋白,并验证发现CsHIPP26.1能够在细胞核内与其中一个光敏色素互作因子CsPIF4.2发生蛋白相互作用。
HIPP;PIF4;蛋白互作;黄化品种;光响应
0 引言
【研究意义】‘黄金芽’属于光照敏感型黄化茶树()品种,叶片色泽受光单一调控[1-2],呈现“强光黄化,弱光复绿”的特点[3],但其叶片色泽响应光照强度黄化的机制并不明确。‘黄金芽’新梢黄化性状稳定[4-5],目前以‘黄金芽’茶树品种为母本进行杂交选育的后代绝大部分为光照敏感型黄化茶树品种[6],因此,研究‘黄金芽’叶色黄化的分子机制,可以探究光照作为信号调控叶片黄化的分子机制,并且能够以叶片色泽作为标记性状,后续开发分子标记,加快优质品种的育种进程,提高育种效率。【前人研究进展】前期对光照敏感型黄化茶树品种‘黄金芽’不同叶色叶片的筛选以及后续的蛋白组分析[7-8],发现一个响应光照强度表达的重金属相关异戊二烯化植物蛋白(heavy metal-associated isoprenylated plant protein,HIPP),该蛋白N端包含金属结合域(heavy metal-associated domain,HMA,pfam00403.6),C端存在一个被异戊二烯化的保守位点[9]。目前对HIPP的研究较少,仅在模式植物拟南芥中鉴定到45个HIPP基因[10],根据已有的研究结果,拟南芥中HIPP的功能分为两种:(a)在重金属稳态和脱毒机制(特别是镉耐受性)中发挥作用[11-14];(b)作为响应低温、干旱和盐胁迫的调控元件[15-16]。除此之外,HIPP能够通过感知胁迫参与调控拟南芥开花时间[17]和拟南芥中植物-病原体相互作用的信号等[18],在植物生长发育和逆境适应性中发挥重要作用,但并没有HIPP缓解强光胁迫的相关研究。在前期研究中,将筛选到的克隆后发现,该基因cDNA全长为465 bp,编码154个氨基酸,蛋白分子量为17 kD;理论等电点为9.13,无跨膜结构域;共有16个磷酸化位点,位于Ser、Thr和Tyr三个氨基酸残基上;根据基因序列号,将该基因命名为(TEA000549),GenBank登录号:MK654903;亚细胞定位发现定位于细胞核,并且在‘黄金芽’中,的表达能够响应光照强度,同时能够显著提高转基因苹果()愈伤组织的强光耐受性[9],因此,可能参与了‘黄金芽’叶色黄化的光响应调控过程,但如何参与叶色的光响应调控途径并不清楚。【本研究切入点】叶色黄化主要是由于叶绿素含量降低造成的叶色失绿,因此,通过筛选与CsHIPP26.1互作且在调控叶绿素合成过程中发挥作用的蛋白,能够阐述CsHIPP26.1可能参与的叶色响应光照强度的调控途径。【拟解决的关键问题】以‘黄金芽’茶树品种为试验材料,利用前期克隆获得的序列,构建酵母双杂交诱饵蛋白载体,通过酵母双杂交筛库筛选与其互作的光信号响应相关蛋白,并通过体内、体外等方式验证蛋白相互作用,为进一步探究HIPP与光照强度调控‘黄金芽’叶色之间的关系提供科学依据。
1 材料与方法
试验材料取自山东省泰安市茶溪谷有限公司茶园,室内酵母双杂交、pull-down等于2019—2020年在山东农业大学茶学实验室进行。
1.1 试验材料
茶树材料:采摘种植于山东省泰安市茶园的五年生‘黄金芽’茶树()一芽二叶,迅速置于液氮中,-80℃保存。
载体:酵母双杂交载体:pGBKT7(BD载体)和pGADT7(AD载体),购自大连宝生物公司;双分子荧光互补(BiFC)载体:pSPYNE-35s和pSPYCE-35s,购自Biovector;Pull-down载体:pET-32a(带His标签)和PGEX-4T-1(带GST标签),购自上海信裕生物科技有限公司;克隆载体:pEASY BLUNT-Zero,购自北京全式金生物公司。
菌株:大肠杆菌DH5α、大肠杆菌BL21(DE3)、酵母Y2H、根癌农杆菌GV3101均购自上海维地生物技术有限公司。
试剂:Phusion高保真酶(F530S)、dNTP、I、R I、H I、I内切酶、T4连接酶购自美国赛默飞世尔科技公司,多糖多酚RNA提取试剂盒(DP441)和DNA回收试剂盒(DP219)均购自北京天根生化科技有限公司,反转录试剂盒RNA to cDNA EcoDry™ Premix购自宝生物工程(大连)有限公司。所有引物由上海生工生物工程股份有限公司合成,编号及序列见表1。

表1 CsHIPP26.1和CsPIF4.2引物
下划线为酶切位点。The underline is the digestion site.CATATG:I; GGATCC:H I; GAATTC:R I; GTCGAC:I
1.2 试验方法
1.2.1 基因克隆与载体构建 以‘黄金芽’茶树品种一芽二叶为材料,根据多糖多酚RNA提取试剂盒(DP441)和反转录试剂盒(RNA to cDNA EcoDryTMPremix)的说明书进行茶树RNA的提取和反转录的操作。以茶树小叶种基因组数据库(http://tpia.teaplant.org/)中的基因序列为模板设计(TEA000549)和(TEA026466)的特异性引物(表1)。PCR扩增体系为20 μL,包括Phusion(F530S)高保真酶0.2 μL、5×Phusion HF Buffer 4 μL、10 mmol∙L-1dNTPs 0.4 μL、正向和反向引物各1 μL、模板DNA 1 μL、ddH2O 12.4 μL。反应条件为98℃ 30 s;98℃ 10 s,60℃ 20 s,72℃ 30 s,35个循环;72℃延伸10 min。采用DNA回收试剂盒(DP219)回收PCR产物后连接至pEASY BLUNT-Zero载体上,然后转化到大肠杆菌感受态细胞,经筛选抽提质粒后送上海生工生物工程股份有限公式进行测序。
将测序正确的克隆载体质粒与相应的空表达载体分别进行双酶切,酶切体系为40 μL,包括质粒25 μL、10×FastDigest Buffer 4 μL、各自对应的两种内切酶各2 μL、ddH2O 7 μL。反应条件为37℃酶切30 min,然后用DNA回收试剂盒分别回收酶切好的目的基因和空表达载体。将回收好的目的基因和对应的载体用T4连接酶进行连接,连接反应体系为10 μL,包括T4 DNA Ligase 0.5 μL、10×T4 Buffer 1 μL、目的片段7.5 μL、表达载体1 μL。22℃反应30 min,转化大肠杆菌感受态细胞后,PCR筛选阳性重组克隆。
1.2.2 酵母双杂交 自主激活鉴定:根据赵强[19]的方法,将构建好的BD-载体转入Y2H酵母中,将转化好的酵母细胞分别平涂到酵母培养板SD/-Trp、SD/-Trp+X-α-Gal、SD/-Trp+X-α-Gal+100 mmol∙L-1金担子素(Aureobasidin A,AbA)上,30℃倒置培养2—3 d,如果SD/-Trp+X-α-Gal上菌落呈白色或红色,则无自主激活活性;如果SD/-Trp+X-α- Gal上菌落变蓝,则说明有自主激活活性;如果SD/-Trp+X-α-Gal上菌落变蓝,而SD/-Trp+X-α-Gal+ 100 mmol∙L-1AbA上不生长,则说明自主激活活性可以被AbA抑制,后续双杂交验证时需添加一定量的AbA。
酵母双杂交相互作用的验证:将转化好的酵母细胞(双杂交筛库以及验证)平涂到酵母培养板SD/-Trp/-Leu(二缺)上;培养2—3 d后,再次活化到酵母培养板SD/-Trp/-Leu/-His/-Ade(四缺)上;如果四缺板上不长,则说明蛋白与蛋白可能不互作;如果在四缺板上正常生长,并且在加入X-α-gal和AbA的四缺平板上菌落变蓝,则表示蛋白与蛋白发生互作。
1.2.3 蛋白互作验证 BiFC和Pull-down蛋白互作验证根据Su等[20]的方法进行。
BiFC体内蛋白互作验证:分别构建pSPYNE- 35s-和pSPYCE-35s-载体,将pSPYNE-35s、pSPYCE-35s空载体和构建好的pSPYNE- 35s-、pSPYCE-35s-载体分别转入根癌农杆菌GV3101中,并侵染洋葱()表皮,暗培养2—3 d后,在激光共聚焦显微镜下观察结果。
Pull-down体外蛋白互作验证:分别构建pET-32a- CsHIPP26.1(CsHIPP26.1-His)和PGEX-4T-1-CsPIF4.2(CsPIF4.2-GST)载体,将pET-32a-CsHIPP26.1、PGEX-4T-1-CsPIF4.2、pET-32a和PGEX-4T-1分别转入大肠杆菌BL21中,用IPTG法诱导蛋白,破碎大肠杆菌并用磁珠纯化蛋白后,分别上柱共孵化,洗脱后用Western blotting进行蛋白检测。
1.2.4 生物信息学预测 筛选到的互作蛋白使用OmicShare Tools网站(https://www.omicshare.com/ tools/home/soft/getsoft/p/7.html)进行GO功能分析。通过茶树基因组数据库(http://tpia.teaplant.org/)检索茶树中光敏色素互作因子(phytochrome interacting factor,PIF)的同源家族基因,根据酵母双杂交筛选到的bHLH30(TEA026466.1)蛋白,用MEGA7软件通过极大似然法(Maximum likelihood)构建PIF家族进化树,通过Clustalx软件进行蛋白序列比对,通过MEME网站(http://meme-suite.org/tools/meme)预测同源家族基因蛋白序列的保守位点。
2 结果
2.1CsHIPP26.1茶树中互作蛋白筛选
通过自激活验证发现,转入BD-载体的Y2H酵母能够在SD/-Trp酵母培养基上正常生长,而且能够在SD/-Trp+x-α-gal的酵母培养基上生长且变蓝。x-α-gal是酵母半乳糖苷酶(MEL1)的显色底物,GAL4酵母双杂交系统的报告基因激活后,能够发生互作的阳性克隆直接在含有x-α-gal的固体培养基上呈蓝色[21]。因此,单转BD-的Y2H变蓝表明构建于BD载体的具有自激活反应;AbA是酵母双杂交阳性克隆子筛选用的药物选择性标记,能够有效抑制酵母双杂交中的自激活反应[22],在添加了100 mmol∙L-1AbA的SD/-Trp+x-α- gal+100 mmol∙L-1AbA酵母培养基上,酵母的生长被抑制,表明100 mmol∙L-1浓度的AbA能够抑制BD-的自激活反应(图1)。
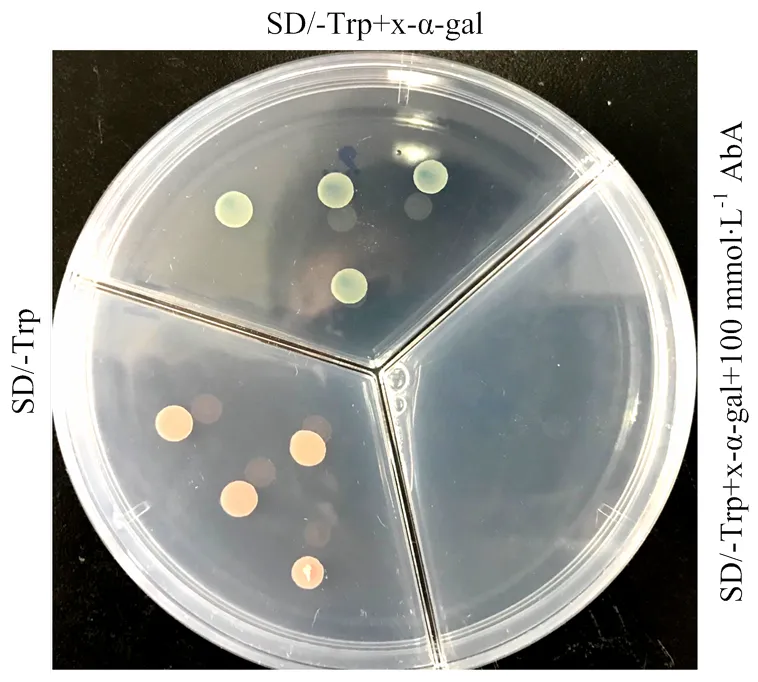
图1 BD诱饵蛋白自激活检测
以AbA浓度为100 mmol∙L-1的酵母筛库培养基(二缺与四缺)进行茶树基因文库筛选,经酵母菌落PCR检测并送样测序后,初步获得候选互作蛋白26个(表2)。分析发现,筛选到的26个互作蛋白中,共有16个蛋白能够注释到具体KEGG通路中,其中有3个信号通路相关蛋白,3个类黄酮、花青素相关通路蛋白,2个核糖体相关蛋白,2个转运相关蛋白,2个叶酸合成相关蛋白和1个生物素合成相关蛋白;对26个筛选蛋白进行GO分析发现(图2),26个候选蛋白主要集中在细胞组分、结合以及催化活性方面,其中生物素合成代谢过程富集程度较高。但与光信号通路及叶绿素合成相关的蛋白只有编号为TEA026466.1的bHLH30转录因子,因此选择该蛋白作进一步分析。
2.2 CsPIF4.2的克隆与鉴定
为进一步验证筛选到的与CsHIPP26.1互作的bHLH30(PIF4-like)转录因子蛋白,首先将该转录因子(TEA026466)进行基因克隆,克隆后发现其全长1 098 bp,编码365个氨基酸;然后将茶树小叶种基因组数据库(http://tpia.teaplant.org/)找到的PIF蛋白与该转录因子(TEA026466)进行进化树与结构域分析,发现筛库得到的bHLH30蛋白(TEA026466)与茶树PIF4蛋白(TEA016807)处于同一进化树分支,具有相似的保守结构位点(Motif),并含有与PIF4相似度极高的bHLH和ACT结构域(图3)。根据茶树小叶种基因组数据库的预测名称以及基因序列号大小排序,将筛库得到的该蛋白命名为CsPIF4.2(TEA026466),并将克隆到的基因序列提交到NCBI,GenBank登记号:MW116834。
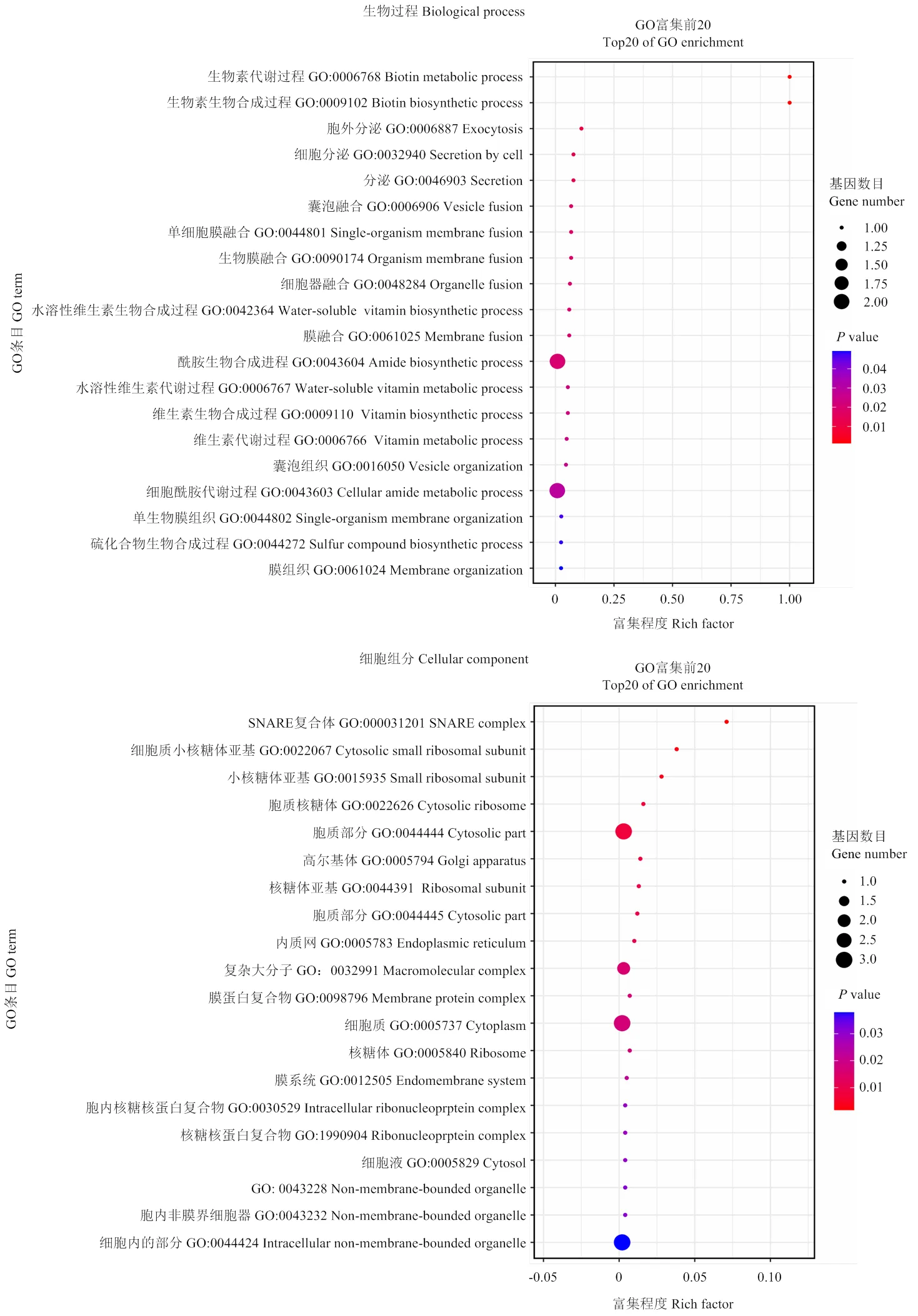
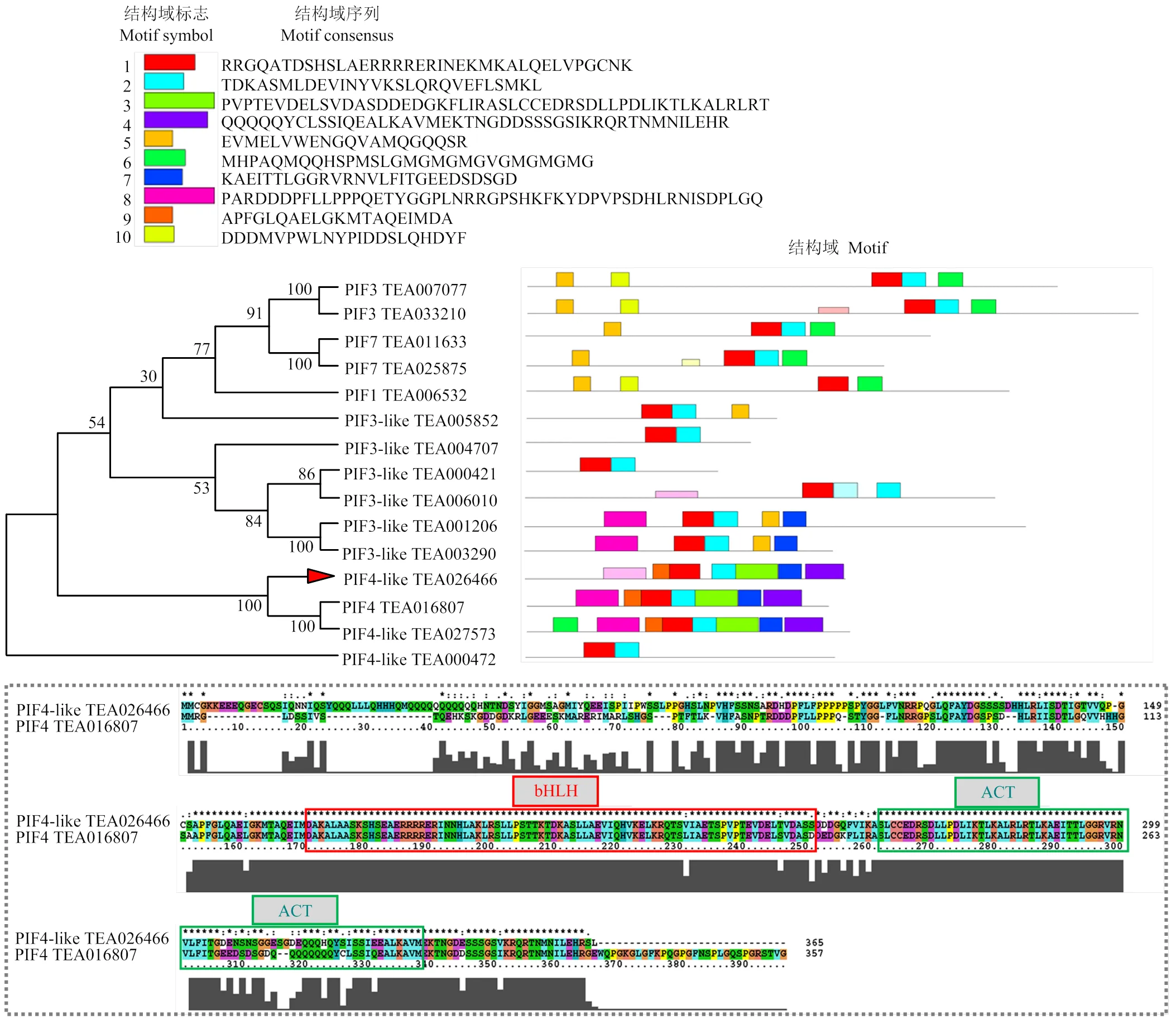
图3 茶树PIFs进化树分析与蛋白序列比对
2.3 CsHIPP26.1和CsPIF4.2蛋白互作验证
将克隆到的构建到pGADT7(AD)质粒载体中,然后将pGBKT7::(- BD)和pGADT7::(-AD)质粒共同转化进酵母感受态。结果显示,在100 mmol∙L-1AbA的选择下,只有共转了-BD和- AD载体的酵母才能正常生长变蓝,而共转- BD和空AD载体的酵母以及共转空BD和- AD载体的酵母在选择培养基上均不能正常生长(图4-A),以上结果说明CsHIPP26.1和CsPIF4.2蛋白能够在酵母体内结合并启动下游报告基因的表达,可以在酵母内发生互作。
为进一步验证蛋白互作的真实性,采用体外蛋白互作的方法(Pull-down)和体内蛋白互作的方法(双分子荧光互补技术,BiFC)进行互作的进一步验证。从图4-B可以看出,在Input中,第一列CsHIPP26.1蛋白和CsPIF4.2蛋白都加入,但并未经过洗脱,因此上清液中存在这两种蛋白;第二列CsHIPP26.1蛋白与GST蛋白在没有洗脱的情况下也能共存。在Output中,CsPIF4- GST能够将CsHIPP26.1蛋白拉下来,而单独的GST蛋白不能将CsHIPP26.1蛋白拉下来,说明CsHIPP26.1蛋白能够在体外与CsPIF4.2蛋白结合。通过BiFC验证,激光共聚焦显微镜下观测发现(图4-C),在pSPYNE- 35s::与pSPYCE-35s以及pSPYNE-35s与pSPYCE-35s::共同侵染的洋葱表皮细胞中,在激发光下并无荧光发生,说明单独的N端或者C端并不能引起黄色荧光的激发,从而排除BiFC体系本身失效的影响。而在pSPYNE-35s::与pSPYCE-35s::共同侵染的洋葱表皮细胞中,在激发光下能够在细胞核中看到黄色荧光,说明CsHIPP26.1蛋白和CsPIF4.2蛋白可以在洋葱表皮细胞内发生互作,且发生互作的部位位于细胞核内。
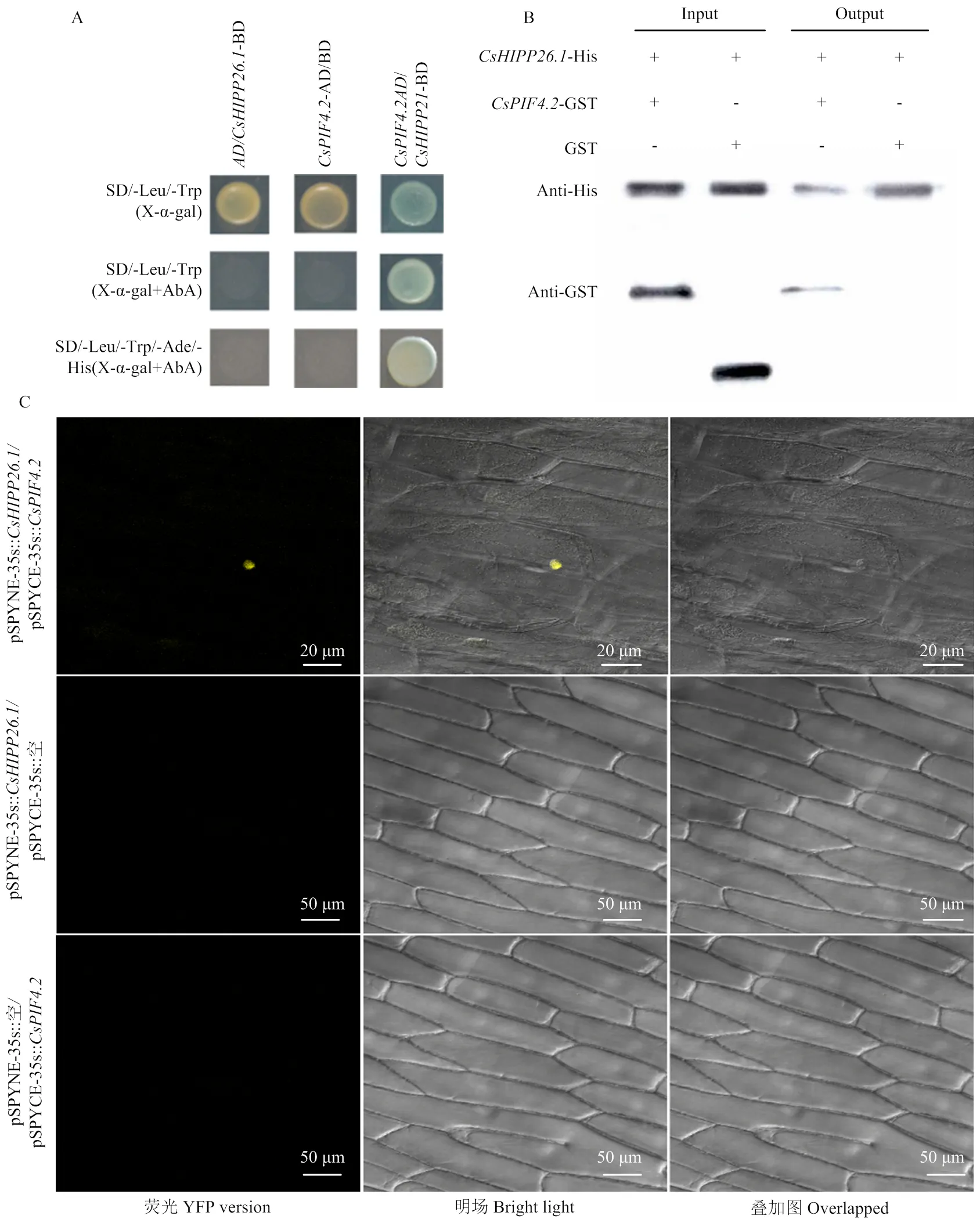
“+”表示加入,“-”代表不加入,只有CsHIPP26.1-His和CsPIF4.2-GST共同加入后才可以在最后的洗脱液中检测到;同时只有在pSPYNE-35s::CsHIPP26.1与pSPYCE-35s::CsPIF4.2共同侵染的洋葱表皮细胞中才能观察到黄色荧光,说明CsHIPP26.1和CsPIF4.2能够互作
3 讨论
‘黄金芽’属于光照敏感型黄化茶树品种,强光下叶片易出现生理胁迫现象[3,23],而重金属相关异戊烯化蛋白(HIPP)则能够缓解低温、盐和干旱等逆境胁迫对植物的伤害[10],还能够通过感知胁迫参与多种植物生理调控过程[18]。笔者前期通过蛋白组分析得到一个定位于细胞核且在‘黄金芽’黄化叶片中高表达的重金属相关异戊烯化蛋白CsHIPP26.1(TEA000549),其基因表达量随着光照强度的增强而增加[9]。通过酵母双杂交对CsHIPP26.1蛋白进行了茶树cDNA文库筛选(表2),得到一个与其互作的光敏色素互作因子(CsPIF4.2,TEA026466)蛋白。前人研究发现,PIF4对光敏色素B(PhyB)介导的光信号对细胞核中叶绿素合成以及光合系统基因的表达具有负调控作用[24-27],例如,在光诱导拟南芥的去黄化过程中,PIF4能够抑制叶绿素合成前期、的表达,从而负调控叶绿素的合成[28-29]。PIF4作为PhyB直接结合的下游信号因子,在细胞核中与处于生物激活状态的PhyB-Pfr结合后,PIF4蛋白发生磷酸化,随后26S蛋白酶体降解磷酸化的PIF4蛋白,被PIF4蛋白抑制的光形态建成相关基因开始表达[30-31],由于CsHIPP26.1与CsPIF4.2在细胞核中发生蛋白的相互作用,推测CsPIF4.2通过与CsHIPP26.1互作以稳定CsPIF4.2的蛋白结构,从而降低光下26S介导的CsPIF4.2蛋白的降解,最终抑制了‘黄金芽’叶绿素合成以及叶绿体发育和光合系统的组装。此外,PIF4在植物抵御逆境胁迫过程中也发挥了重要作用,番茄植株中PIF4通过感知光信号正调控植物的耐低温能力[32];高温下,PIF4主要通过影响IAA水平进而调控拟南芥下胚轴伸长等形态特征;PIF4还能抑制气孔发育正向调节因子SPCH(Speechless)的表达,进而抑制气孔张开,减少高温对植物的伤害[33];植株耐高温特性还受到植株体内PIF4积累量以及PIF4对靶标基因的激活能力的制约[34-35]。综上,CsHIPP26.1通过与CsPIF4.2发生蛋白的相互作用,一方面可能通过稳定CsPIF4.2的蛋白结构,从而负调控叶绿素合成及光和系统基因表达等光形态建成过程;另一方面,还有可能通过稳定蛋白结构,从而增强‘黄金芽’黄化叶片的抗逆性。本研究发现,CsHIPP26.1蛋白能够在细胞核中与CsPIF4.2蛋白发生互作,推测CsHIPP26.1蛋白通过在细胞核中结合CsPIF4.2蛋白并稳定其结构。这一结果为CsHIPP26.1响应光强从而调控下游相关生理过程变化提供了科学依据,但CsHIPP26.1是否能够促进光下CsPIF4.2蛋白的稳定性还需做进一步探究。
4 结论
通过酵母双杂交,从茶叶cDNA文库中筛选到与CsHIPP26.1互作的候选互作蛋白26个。经验证,CsHIPP26.1能够与光敏色素互作因子CsPIF4.2蛋白互作,并且两者的蛋白相互作用发生在细胞核内。
[1] TIAN Y Y, WANG H Y, SUN P, FAN Y G, QIAO M M, ZHANG L X, ZHANG Z Q.Response of leaf color and the expression of photoreceptor genes ofcv.Huangjinya to different light quality conditions.Scientia Horticulturae, 2019, 251: 225-232.
[2] TIAN Y Y, WANG H Y, ZHANG Z Q, ZHAO X X, WANG Y, ZHANG L X.An RNA-seq analysis reveals differential transcriptional responses to different light qualities in leaf color ofcv.Huangjinya.Journal of plant growth regulation.Journal of Plant Growth Regulation, 2001, 19(4): 463-470.doi: 10.1007/s003440010017.
[3] 王开荣, 李明, 梁月荣, 张龙杰, 沈立铭, 王盛彬.茶树新品种黄金芽选育研究.中国茶叶, 2008, 30(4): 21-23.doi: 10.3969/j.issn.1000-3150.2008.04.007.
WANG K R, LI M, LIANG Y R, ZHANG L J, SHEN L M, WANG S B.Research on the breeding of new tea variety Huangjinya.China Tea, 2008, 30(4): 21-23.doi: 10.3969/j.issn.1000-3150.2008.04.007.(in Chinese)
[4] 刘丁丁, 梅菊芬, 王君雅, 汤榕津, 陈亮, 马春雷.茶树白化突变研究进展.中国茶叶, 2020, 42(4): 24-35.doi: 10.3969/j.issn.1000- 3150.2020.04.006.
LIU D D, MEI J F, WANG J Y, TANG R J, CHEN L, MA C L.Research progress on albino trait of tea plant.China Tea, 2020, 42(4): 24-35.doi: 10.3969/j.issn.1000-3150.2020.04.006.(in Chinese)
[5] 王开荣, 李明, 张龙杰, 梁月荣.白化茶种质资源分类研究.茶叶, 2015, 41(3): 126-129.doi: 10.3969/j.issn.0577-8921.2015.03.002.
WANG K R, LI M, ZHANG L J, LIANG Y R.Studies on classification of albino tea resources.Journal of Tea, 2015, 41(3): 126-129.doi: 10.3969/j.issn.0577-8921.2015.03.002.(in Chinese)
[6] 王开荣, 李明, 梁月荣.光照敏感型白化茶.杭州: 浙江大学出版社, 2014.
WANG K R, LI M, LIANG Y R.Light-sensitive albino tea.Hangzhou: Zhejiang University Press, 2014.(in Chinese)
[7] 范延艮, 赵秀秀, 王翰悦, 田月月, 向勤锃, 张丽霞.黄金芽不同色泽叶片生理特性研究.茶叶科学, 2019, 39(5): 530-536.doi: 10.13305/j.cnki.jts.2019.05.004.
FAN Y G, ZHAO X X, WANG H Y, TIAN Y Y, XIANG Q Z, ZHANG L X.Study on physiological characteristics of leaves with different colors of ‘huangjinya’.Journal of Tea Science, 2019, 39(5): 530-536.doi: 10.13305/j.cnki.jts.2019.05.004.(in Chinese)
[8] FAN Y G, ZHAO X X, WANG H Y, TIAN Y Y, XIANG Q Z, ZHANG L X.Effects of light intensity on metabolism of light-harvesting pigment and photosynthetic system inL.cultivar ‘Huangjinya’.Environmental and Experimental Botany, 2019, 166: 103796).
[9] 范延艮, 刘富浩, 赵秀秀, 王域, 张丽霞.茶树‘黄金芽’基因克隆与光响应表达分析.植物生理学报, 2021, 57(5): 1087-1097.doi: 10.13592/j.cnki.ppj.2021.0018.
FAN Y G, LIU F H, ZHAO X X, WANG Y, ZHANG L X.Cloning of‘Huangjinya’ and analysis of its expression level in response to light intensity.Plant Physiology Journal, 2021, 57(5): 1087-1097.doi: 10.13592/j.cnki.ppj.2021.0018.(in Chinese)
[10] DE ABREU-NETO J B, TURCHETTO-ZOLET A C, DE OLIVEIRA L F V, ZANETTINI M H B, MARGIS-PINHEIRO M.Heavy metal-associated isoprenylated plant protein (HIPP): Characterization of a family of proteins exclusive to plants.The FEBS Journal, 2013, 280(7): 1604-1616.doi: 10.1111/febs.12159.
[11] TEHSEEN M, CAIRNS N, SHERSON S, COBBETT C S.Metallochaperone-like genes inthaliana.Metallomics, 2010, 2(8): 556-564.doi: 10.1039/c003484c.
[12] GAO W, XIAO S, LI H Y, TSAO S W, CHYE M L.acyl-CoA-binding protein ACBP2interacts with heavy-metal-binding farnesylated protein AtFP6.The New Phytologist, 2009, 181(1): 89-102.doi: 10.1111/j.1469-8137.2008.02631.x.
[13] CHANDRAN D, SHAROPOVA N, IVASHUTA S, GANTT J S, VANDENBOSCH K A, SAMAC D A.Transcriptome profiling identified novel genes associated with aluminum toxicity, resistance and tolerance in.Planta, 2008, 228(1): 151-166.doi: 10.1007/s00425-008-0726-0.
[14] SUZUKI N, YAMAGUCHI Y, KOIZUMI N, SANO H.Functional characterization of a heavy metal binding protein CdI19 from.The Plant Journal, 2002, 32(2): 165-173.doi: 10.1046/ j.1365-313x.2002.01412.x.
[15] BARTH O, VOGT S, UHLEMANN R, ZSCHIESCHE W, HUMBECK K.Stress induced and nuclear localized HIPP26 frominteracts via its heavy metal associated domain with the drought stress related zinc finger transcription factor ATHB29.Plant Molecular Biology, 2009, 69(1/2): 213-226.doi: 10.1007/ s11103-008-9419-0.
[16] BARTH O, ZSCHIESCHE W, SIERSLEBEN S, HUMBECK K.Isolation of a novel barley cDNA encoding a nuclear protein involved in stress response and leaf senescence.Physiologia Plantarum, 2004, 121(2): 282-293.doi: 10.1111/j.0031-9317.2004.00325.x.
[17] WIEBKE Z, OLAF B, KATHARINA D, SANDRA B, JULIANE R, KLAUS H.The zinc-binding nuclear protein HIPP3acts as an upstream regulator of the salicylate-dependent plant immunity pathway and of flowering time in.The New Phytologist, 2015, 207(4): 1084-96.doi: 10.1111/nph.13419.
[18] RADAKOVIC Z S, ANJAM M S, ESCOBAR E, CHOPRA D, CABRERA J, SILVA A C, ESCOBAR C, SOBCZAK M, GRUNDLER F M W, SIDDIQUE S.HIPP27 is a host susceptibility gene for the beet cyst nematode.Molecular Plant Pathology, 2018: 1917-1928.
[19] 赵强.苹果BTB-TAZ蛋白MdBTs调控MdbHLH104影响PMH+-ATP酶的活性和铁的动态平衡[D].泰安: 山东农业大学, 2015.
ZHAO Q.BTB-TAZ protein MdBTs target MdbHLH104 to regulate the activity of PMH+-ATPase and the homeostasis of iron in apple [D].Taian: Shandong Agricultural University, 2015.(in Chinese)
[20] SU M Y, WANG N, JIANG S H, FANG H C, XU H F, WANG Y C, ZHANG Z, ZHANG J, XU L, ZHANG Z Y, CHEN X S.Molecular characterization and expression analysis of the critical floral gene MdAGL24-like in red-fleshed apple.Plant Science, 2018, 276: 189-198.doi:10.1016/j.plantsci.2018.08.021.
[21] 崔红军, 魏玉清.酵母双杂交系统及其应用研究进展.安徽农业科学, 2015, 43(13): 45-47.doi: 10.13989/j.cnki.0517-6611.2015.13.152.
CUI H J, WEI Y Q.Review on application status of yeast two hybrid system.Journal of Anhui Agricultural Sciences, 2015, 43(13): 45-47.doi: 10.13989/j.cnki.0517-6611.2015.13.152.(in Chinese)
[22] HASHIDA-OKADO T, OGAWA A, ENDO M, YASUMOTO R, TAKESAKO K, KATO I.AUR1, a novel gene conferring aureobasidin resistance on Saccharomyces cerevisiae: a study of defective morphologies in Aur1p-depleted cells.Molecular and General Genetics, 1996, 251(2): 236-244.doi: 10.1007/BF02172923.
[23] 田月月, 张丽霞, 张正群, 李智, 侯剑, 乔明明.主要气象因子对‘黄金芽’茶树叶片日灼伤害的影响.山东农业科学, 2017, 49(7): 42-48.doi: 10.14083/j.issn.1001-4942.2017.07.009.
TIAN Y Y, ZHANG L X, ZHANG Z Q, LI Z, HOU J, QIAO M M.Influences of main meteorological factors on leaf sunburn of tea cultivar ‘huangjinya'.Shandong Agricultural Sciences, 2017, 49(7): 42-48.doi: 10.14083/j.issn.1001-4942.2017.07.009.(in Chinese)
[24] XU D Q.Multifaceted roles of PIF4 in plants.Trends in Plant Science, 2018, 23(9): 749-751.doi: 10.1016/j.tplants.2018.07.003.
[25] HUQ E, QUAIL P H.PIF4, a phytochrome-interacting bHLH factor, functions as a negative regulator of phytochrome B signaling in.The EMBO Journal, 2002, 21(10): 2441-2450.doi: 10.1093/emboj/21.10.2441.
[26] INAGAKI N, KINOSHITA K, KAGAWA T, TANAKA A, UENO O, SHIMADA H, TAKANO M.Phytochrome B mediates the regulation of chlorophyll biosynthesis through transcriptional regulation of ChlH and GUN4 in rice seedlings .PLoS ONE, 2015, 10(8): e0135408.
[27] 赵杰, 周晋军, 顾建伟, 钱凤芹, 谢先芝.光敏色素B正调控水稻叶绿素合成和叶绿体的发育.中国水稻科学, 2012, 26(6): 637-642.doi: 10.3969/j.issn.1001-7216.2012.06.001.
ZHAO J, ZHOU J J, GU J W, QIAN F Q, XIE X Z.Phytochrome B positively regulates chlorophyll biosynthesis and chloroplast development in rice.Chinese Journal of Rice Science, 2012, 26(6): 637-642.doi: 10.3969/j.issn.1001-7216.2012.06.001.(in Chinese)
[28] Toledo-ortiz G, JOHANSSON H, LEE K P, BOU-TORRENT J, STEWART K, STEEL G, RODRIGUEZ-CONCEPEION M, HALLIDAY K J.The HY5-PIF regulatory module coordinates light and temperature control of photosynthetic gene transcription .PLoS Genetics, 2014, 10(6): e1004416.
[29] TANG W J, WANG W Q, CHEN D Q, JI Q, JING Y J, WANG H Y, LIN R C.Transposase-derived proteins FHY3/FAR1 interact with PHYTOCHROME-INTERACTING FACTOR1 to regulate chlorophyll biosynthesis by modulating HEMB1 during deetiolation in.The Plant Cell, 2012, 24(5): 1984-2000.doi: 10.1105/tpc.112.097022.
[30] LORRAIN S, ALLEN T, DUEK P D, WHITELAM G C, FANKHAUSER C.Phytochrome-mediated inhibition of shade avoidance involves degradation of growth-promoting bHLH transcription factors.The Plant Journal, 2008, 53(2): 312-323.doi: 10.1111/j.1365-313X.2007.03341.x.
[31] MIGUEL D L, JEAN-MICHEL D, MARIANA R F, MARIELA P, MANUEL I P J, SÉVERINE L, CHRISTIAN F, ANGEL B M, ELENA T, SALOMÉ P.A molecular framework for light and gibberellin control of cell elongation.Nature, 2008, 451(7177): 480-484.doi: 10.1038/nature06520.
[32] 陈笑笑.PIF4介导光质调控番茄低温抗性的作用机制[D].杭州: 浙江大学, 2019.
CHEN X X.The mechanisms of PIF4-mediated light quality- regulated cold tolerance in tomato [D].Hangzhou: Zhejiang University, 2019.(in Chinese)
[33] LAU O S, SONG Z J, ZHOU Z M, DAVIES K A, CHANG J, YANG X, WANG S Q, LUCYSHYN D, TAY I H Z, WIGGE P A, BERGMANN D C.Direct control of SPEECHLESS by PIF4 in the high-temperature response of stomatal development.Current Biology, 2018, 28(8): 1273-1280.doi: 10.1016/j.cub.2018.02.054.
[34] BO Z, MATTIAS H, SEVERINE L, MIKAEL N, LÁSZLÓ B, CHRISTIAN F, OVE N.Blade-on-petiole proteins act in an e3 ubiquitin ligase complex to regulate phytochrome interacting factor 4 abundance.eLife, 2017, 6: e26759.doi: 10.7554/eLife.26759.
[35] NIETO C, LÓPEZ-SALMERÓN V, DAVIÈRE J M, PRAT S.ELF3-PIF4 interaction regulates plant growth independently of the evening complex.Current Biology, 2015, 25(2): 187-193.doi: 10.1016/j.cub.2014.10.070.
Screening and Verification of CsHIPP26.1 Interaction Protein in Tea Plant
FAN YanGen, WANG Yu, LIU FuHao, ZHAO XiuXiu, XIANG QinZeng, ZHANG LiXia*
College of Horticulture Science and Engineering, Shandong Agricultural University/State Key Laboratory of Crop Biology, Tai’an 271018, Shandong
【Background】Huangjinya is a light sensitive chlorotic tea () variety, the leaf color of which presents yellow under strong light and presents green under weak light, but the chlorotic mechanism of leaf color in response to light is not clear.Previous proteomic studies on etiolated leaves, shaded green leaves and evergreen leaves found that the expression of heavy metal-associated isoprenylated plant protein CsHIPP26.1 (TEA000549) responded to light intensity, indicating that CsHIPP26.1 may be involved in regulating the light response process of leaf color etiolation of Huangjinya.【Objective】The proteins related to the light signal response interacting with CsHIPP26.1 were screened to provide a scientific basis for leaf color response to light signal changes.【Method】The gene were cloned from one bud and two leaves of Huangjinya.And the screened target protein was further verified by yeast two hybrid point-to-point verification,bimolecular fluorescence complementarity (BiFC), andpull-down techniques.【Result】The tea cDNA library was screened by yeast two hybrid, and a total of 26 candidate interaction proteins were screened, which mainly played a role in cell components, binding and catalytic activity.Among them, the enrichment degree of biotin anabolism process was high, and the proteins related to light signal pathway and chlorophyll synthesis were only the bHLH30 transcription factor, and its gene ID was TEA026466.1.After cloning the gene of bHLH30 transcription factor, it was found that the transcription factor was in the same evolutionary tree branch as tea light signal transduction pathway protein PIF4, and contained the same HLH and ACT domains as tea PIF4 protein.Therefore, the bHLH30 transcription factor was named CsPIF4.2, GenBank registration number: MW16834.And through the pull-down protein interactionand bimolecular fluorescence complementarity (BiFC), it was found that CsHIPP26.1 and CsPIF4.2 proteins could indeed interact, and the site of interaction was in the nucleus.【Conclusion】26 proteins interacting with CsHIPP26.1 were preliminarily screened, and it was found that CsHIPP26.1 could interact with one of the phytochrome interacting factors CsPIF4.2 in the nucleus.
HIPP; PIF4; protein interaction; yellowing varieties; light response
2021-08-20;
2021-12-10
山东省现代农业产业技术体系创新团队项目(SDAIT-19-05)、山东省“双一流”奖补资金项目(SYL2017YY03)、鲁渝科技协作计划项目(2020LYXZ005)
范延艮,E-mail:876562801@qq.com。王域,E-mail:1113405407@qq.com。范延艮和王域为同等贡献作者。通信作者张丽霞,E-mail:lxzhang@sdau.edu.cn
(责任编辑 赵伶俐)
