细胞重编程过程中核小体定位改变研究进展
2022-03-26崔浩亮史佩华高锦春张新博赵顺然陶晨雨
崔浩亮,史佩华,高锦春,张新博,赵顺然,陶晨雨
综 述
细胞重编程过程中核小体定位改变研究进展
崔浩亮,史佩华,高锦春,张新博,赵顺然,陶晨雨
河北农业大学动物科技学院,保定 07100
细胞重编程是指在精卵结合或核移植过程中,核遗传物质的表观遗传标记发生删除和重塑,从而使已分化的细胞成为具有全能性的过程。发生细胞重编程的方法主要有细胞融合、体细胞核移植以及诱导多能干细胞等。核小体是染色质的基本结构及功能单位,是染色质的一级结构,核小体定位对基因的表达及细胞的状态有着重要的调控作用。细胞重编程过程中核小体的含量和位置也会发生剧烈的变化,同时在相关基因启动子位置的核小体含量也会降低从而促进多能性基因的表达。本文综述了核小体定位在基因激活与抑制、染色质重塑以及转录因子识别中的作用,旨在为深入解析细胞重编程机制提供重要依据。
细胞重编程;核小体定位;染色质重塑;转录起始位点
细胞重编程(cellular reprogramming)是指已经分化的细胞在一些特殊条件下重新恢复到分化前的全能性状态的过程,此过程包括多潜能基因的活跃表达、表观遗传标记改变、核小体定位变化以及染色质重塑等。实现细胞重编程的方法包括体细胞核移植、细胞融合以及诱导多能干细胞等,细胞重编程在医学领域以及干细胞研究领域具有巨大应用前景。但目前人们对重编程作用机制尚不清楚,同时细胞重编程效率低下极大限制了重编程的研究,因此探明重编程机制对当前动物遗传育种与繁殖研究具有重大意义。
核小体的动态变化是基因组调节生物活动基础特征之一,核小体定位在调节核小体结构和功能方面发挥重要作用。有研究表明,在真核生物中大约75%~90%的基因组被包装成核小体[1],细胞重编程过程中核小体定位发生明显变化,且核小体分布呈现不均匀性,许多转录因子结合位点及转录起始位点存在明显的核小体缺失区域[2]。核小体定位的改变是基因表达调控的重要方式之一,在细胞重编程过程中的核小体定位模式是目前研究热点之一。
1 细胞重编程过程中核小体定位
1.1 细胞重编程过程中核小体覆盖率变化
已分化细胞可以通过核移植、细胞融合以及诱导多能干细胞(induced pluripotent stem cell, iPSC)技术重编程回到多能性细胞状态[3](图1)。向小鼠胚胎成纤维细胞(mouse embryonic fibroblasts, MEF)添加4种转录因子(OCT4、SOX2、KLF4和C-MYC),可以将MEF重编程为诱导多能干细胞及其前体细胞(pre-iPSC)。iPSC是一种研究细胞重编程的重要材料[4],核小体是染色质的基本结构单位,是基因表达的重要调节因子。iPSC的核小体定位模式与胚胎干细胞(embryonic stem cell, ESC)极其相似,而与MEF存在很大差别。DNA序列核小体覆盖率一般通过与DNA序列结合的核小体数量表示。Huang等[5]分别比较了MEF、pre-iPSC和iPSC的全基因组核小体覆盖率情况,从MEF到pre-iPSC,只有17.32%的基因组显示较高的核小体覆盖率,52.71%的基因组显示较低核小体覆盖率,这表明细胞重编程过程染色质结构倾向开放状态。然而pre-iPSC进一步诱导成iPSC,近50%的基因组区域倾向于获得核小体,同时基因的开放程度降低,为解释这一现象,该研究通过利用核小体二元体周围标签位置的标准偏差计算核小体模糊度,发现pre-iPSC的核小体平均模糊度(26 bp)远低于MEF(31 bp)和iPSC(32 bp)的核小体模糊度,表明在pre-iPSC表现出更紧密的核小体定位,在完成重编程的iPSC中恢复为稀松的核小体定位模式。Tao等[6]通过体细胞核移植(somatic cell nuclear transplantation, SCNT)构建猪()早期胚胎细胞,并对1000个受精卵(fertilized zygote, FZ)进行了微球菌核酸酶测序(MNase-seq),相比发现,由SCNT构建的胚胎细胞整体核小体覆盖率下降,编码区的核小体覆盖率由猪胚胎成纤维细胞(pig embryonic fibroblasts, PEF)的29.58%上升到31.97% (SCNT)、30.31% (FZ),而启动子区的核小体覆盖率从PEF的1.42%减少到SCNT的1.37%和FZ的1.3%,这种核小体定位阶段性变化可能是为了胚胎阶段的大规模转录做准备。在细胞重编程过程中,转录因子结合位点以及转录起始位点核小体含量变低,这可能导致某些基因的表达量上升从而促进细胞重编程的进行。Jose等[7]将OCT4和KLF4转导到脑源性神经干细胞(neural stem cell, NSC)中,发现NSC比MEF更容易被诱导成iPSC,而且这一过程不需要外源SOX2或C-MYC[8]。这可能说明相比于核小体覆盖率高的体细胞,具有较低核小体覆盖率的干细胞更有利于细胞重编程发生[9]。总之,核小体定位变化是细胞重编程的重要标志之一。

图1 细胞重编程方法
A:在体细胞培养过程中,通过添加4种转录因子,诱导重编程发生,使其转变成诱导多能性细胞;B:通过将体细胞核移植到去核卵母细胞中,诱导细胞重编程,使其形成新的胚胎个体;C:通过细胞融合使体细胞来源的细胞核发生重编程。根据参考文献[3]总结绘制。
1.2 细胞重编程中转录起始位点核小体排布变化
在探究核小体定位是如何影响细胞重编程过程中,发现在转录起始位点(turn start site, TSS)存在特定的核小体规范排列(–1, NDR (nucleosome deletion region), +1, +2……),这对基因表达起到至关重要的作用[10]。Tao等[11]通过利用3种体细胞重编程诱导成iPSC,发现iPSC的核小体定位模式与ESC几乎相同,同时在活性基因TSS周围有典型的核小体(–1、NDR、+1、+2、+3……)排布。而在小鼠() ESC和MEF中,高表达的基因比低表达的基因TSS周围有更明显的NDR[12]。类似的,在iPSC中多能性基因具有较高的表达水平,基因表达水平高低与NDR数量呈正相关[5,12]。Tao等[6]比较了若干个多能性基因、成纤维细胞特异性基因以及管家基因TSS周围核小体定位模式,发现所选多能性基因TSS周围的核小体定位模式在PEF是典型的沉默基因,但在SCNT和FZ中是典型的活跃基因,而成纤维细胞特异性基因TSS周围的核小体定位模式则正好相反。这可能说明多能性基因可能在重编程的早期阶段表达或准备高表达。总之,比较PEF、SCNT和FZ,沉默基因依旧沉默,多潜能基因TSS周围趋向更加开放的染色质结构(NDR),使其转录活性升高。CpG岛区域通常被认为是哺乳动物的基因启动子标志,启动子可以根据CpG岛数量分为High-CG启动子和Low-CG启动子[13]。Huang等[5]通过观察重编程过程中High-CG启动子和Low-CG启动子的核小体定位模式与基因表达之间的关系,发现Low-CG启动子处存在明显的核小体占据,类似于沉默基因的核小体占据模式。相比之下,High-CG启动子在TSS处存在明显NDR和侧翼区域的核小体高度富集,类似于活性基因的模式。如图2A所示,在重编程过程中,多能性基因TSS周围的核小体占据模式类似活性基因,表现典型的核小体定位模式(–1、NDR、+1、+2……)。成纤维细胞特异性基因TSS周围的核小体占据模式类似沉默基因,表现典型的核小体占据模式。虽然许多因素都可以参与基因表达的调控,但本课题组观察到核小体定位与基因表达之间存在很强的相关性,表明基因表达调控的灵活性在一定程度上受到核小体调控的影响。
2 转录因子特异性识别核小体并促进细胞重编程
转录因子结合位点具有独特的核小体结构,它们在转录过程中受核小体定位模式的调控[11]。干细胞多能性、细胞分化和细胞重编程过程需要转录因子(transcription factors, TFs)参与进行,在细胞重新编程过程中,TFs通过与核小体覆盖的DNA结合,引起染色质封闭区域结构改变,从而启动新的表达模式[14,15]。在细胞重编程中Foxa1、OCT4、Ascl1/ E12a、PU1和Cebpa显示很高的核小体亲和力[16]。Foxa1可以取代组蛋白连接体,促进局部染色质开放,从而有利于其他转录因子与基因结合。Ebf1可以与致密染色质结合,并诱导染色质重塑[17,18]。SOX2是胚胎干细胞多能性的重要先导因子,SOX2可以在超螺旋位置结合并局部扭曲DNA,这有助于核小体组织与核小体末端DNA分离,从而增加DNA可及性[19]。与之相似,Michael等[20]发现OCT4与SOX2会不同程度地扭曲核小体结合的DNA,促进DNA从组蛋白H2A和H3上脱离。OCT4、SOX2和KLF4能够靶向优先结合复合核小体的沉默位点,从而激活基因[19,21],同时在另一个转录因子C-MYC的共同参与下,OCT4、SOX2和KLF4在重新编程的最初48小时与封闭染色质紧密结合[22]。Run等[23]发现先锋转录因子Leafy与核小体结合并与包括AP1在内的大多数靶基因结合,随后取代连接组蛋白H1并募集Swi/Snf,在局部“解锁”染色质。相似的,Gata3与染色质结合,促使局部染色质发生重塑以及提高增强子结合能力[24]。此外,在细胞重编程过程中,孤核受体Essrb可以募集转录因子OCT4、SOX2和NANOG进入封闭染色质区域,与包含稳定核小体组织及高甲基化的DNA的增强子结合。如图2B所示,这些结果说明TFs可以特异性识别重编程重要基因,虽然这些基因在已分化细胞中属于核小体高覆盖的沉默基因,但TFs可以识别核小体并促使核小体组织与DNA分离,以及驱动局部染色质重构促进核小体重塑以及随后的转录。
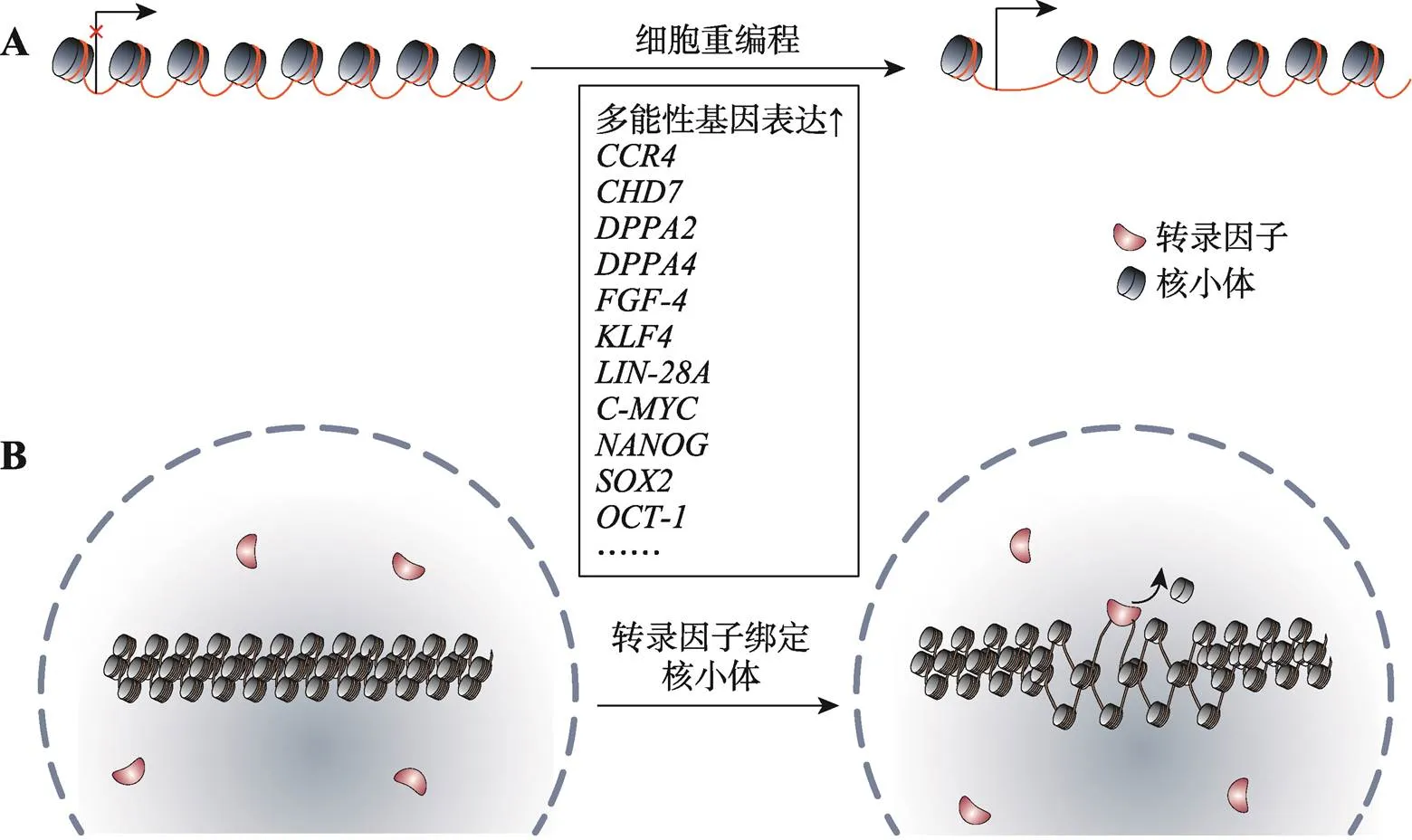
图2 细胞重编程中多能性基因及转录因子变化
A:细胞重编程过程中多能性基因转录起始位点发生核小体缺失;B:转录因子结合靶核小体并改变染色质结构,从而刺激转录。
3 核小体定位变化促进染色质重塑
染色质重塑是细胞重编程的重要标志之一[25],染色质/核小体重塑因子是利用ATP水解产生的能量来移动和调整核小体,从而调控DNA功能(转录、复制和DNA修复)[26](图3)。染色质重塑因子可以分为4个家族:Swi/Snf (switch/sucrose non fermentable) 家族、Iswi (imitation switch)家族、Ino80 (inositol requiring 80)家族以及Chd (chromo helicase domain)家族。最初的重塑复合物是在酵母()中发现的Swi/Snf复合物[27]。这些重塑因子可以协同驱逐启动子处核小体,从而创造NDR以实现基因表达[28]。Nap111通过偶联染色质重塑复合物Swi/Snf和Ino80降低核小体覆盖率,促进ES细胞分化[29]。Asf1是一种组蛋白伴侣,在和突变的细胞内染色质重组程度显著降低[30]。总之,Swi/Snf家族通过核小体滑动和移除改变染色质状态,以此调节靶基因的激活和抑制[31]。染色质重塑因子Ino80可维持胚胎干细胞的多能性以及调节体细胞重编程为多能细胞,Cao等[32]发现Ino80可以调节滋养外胚层上皮通透性以促进猪胚泡发育。此外,Ino80还被证明通过影响组蛋白乙酰化来维持代谢平衡,将其破坏有概率导致疾病发生[33]。染色质可及性复合体(chromatin accessible complex, CHRAC)是一种典型的核小体滑动因子,由ATP酶ISWI、ACF1亚基和一对组蛋白样蛋白CHRAC-14/16组成[34],能调节核小体定位,改变染色质的规则性和完整性。这可能有助于抑制性染色质的形成。标志性亚单位ACF1在胚胎发育过程中的表达受到限制,但在原始生殖细胞中的表达仍然很高[35]。核小体重塑和去乙酰化(nucleosome remodeling and deacetylation, NuRD)复合物[36]在基因表达调控和干细胞自我更新中起着重要作用。在小鼠胚胎干细胞(ESC)和神经祖细胞(neural progenitor cell, NPCs)分化过程中中,NuRD的基因组靶点是动态变化的,大多数结合发生在细胞特异基因的启动子和增强子上[37]。Maud等[38]发现在ES中,染色质重塑因子Chd1、Chd2、Chd4、Chd6、Chd8、Chd9、Brg1以及Ep400靶向结合NDR边缘核小体,从而调节ES转录程序。核小体定位是建立染色质结构的基础。在体细胞中,iPS技术可以诱导沉默的X染色体重新活化。在小鼠细胞重编程过程中,全能性的获得与X染色体重新激活之间有很强关联性[39]。Tao等[6]在对猪卵母细胞进行SCNT也发现相似结果,同时在SCNT前期核小体占有率降低也可能是为X染色质活化做准备。这些结果表明X染色体的活化可能对细胞重编程的发生起到积极作用。综上所述,染色质重塑对细胞重编程起着至关重要的作用,染色质重塑因子通过介导核小体定位动态变化(滑动、去除、解离、重新组装)来实现染色质重塑,从而影响细胞重编程的发生。
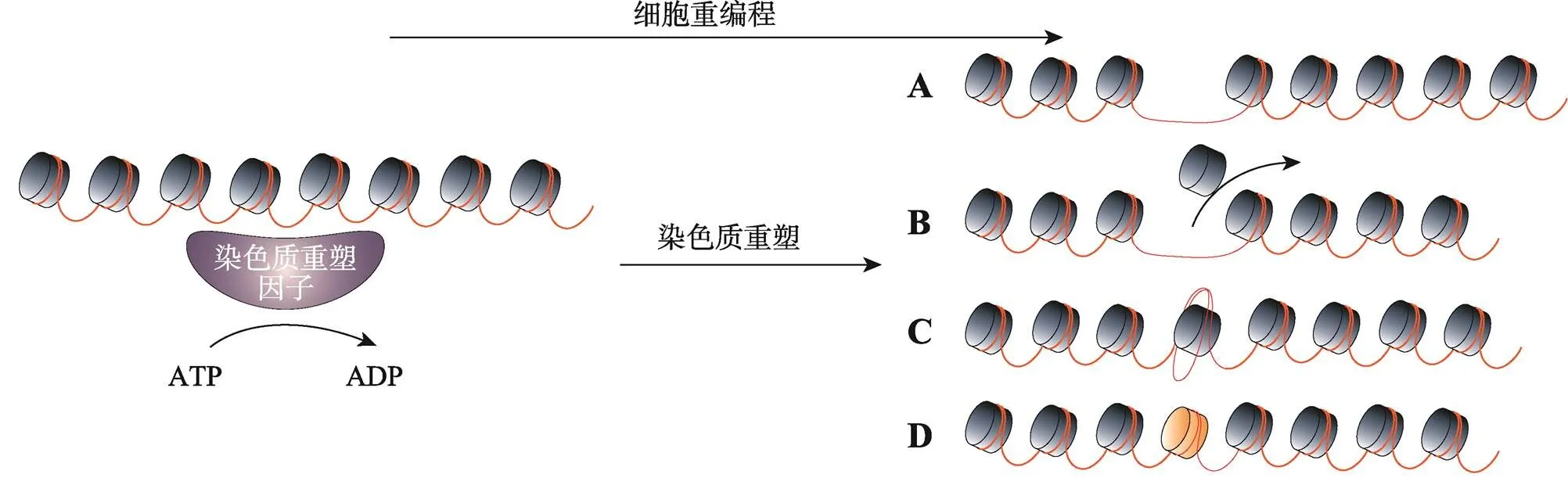
图3 染色质重塑过程中的核小体定位变化
A:细胞重编程过程中染色质重塑因子结合核小体,并促进核小体滑动,暴露出DNA片段;B:靶标核小体去除;C:DNA与核小体发生解离;D:核小体变体替换原有核小体,改变染色质结构。
4 组蛋白变体在细胞重编程中的作用
核小体是由长度约147 bp的DNA片段以及4种组蛋白(H2A、H2B、H3和H4)构成的八聚体共同组成。组蛋白变体是一类特殊的组蛋白,其与常规组蛋白相差一个到几百个氨基酸,组蛋白变体在细胞重编程过程中具有重要作用。在SCNT过程前期连接组蛋白H1会和异染色质蛋白1 (HP1)发生置换;随后组蛋白变体H3.3 (一种与基因激活相关的组蛋白变体)会参与重编程过程。在ESC中,组蛋白变体H3.3促使基因启动子处建立H3K27三甲基而在低表达发育基因中建立双价染色质,从而使特定基因在分化过程中表达[40]。连接组蛋白H1促进高阶染色质折叠,对哺乳动物的发育至关重要,在基因的高GC区域和活性启动子中富含H3K4三甲基,缺乏H1d和H1c,这与特征序列特征相关[41]。成熟精子和卵子染色质包装存在很大差异,但这些差异在脊椎动物早期胚胎中通过染色质重新编程得到协调[42]。在斑马鱼受精卵中,处于转录静止的雌原核DNA甲基化模式被重编程以匹配雄原核。在精子发生过程中,通过体细胞组蛋白与睾丸特异性组蛋白变体的交换完成染色质重构[43]。和功能相同且在卵母细胞和受精卵中高度表达[44,45],研究表明,它们的表达水平会随着分化程度升高而降低。变异体诱导开放的染色质结构,并在染色体上均匀分布;同时这些组蛋白变体与4种转录因子OCT4、SOX2、KLF4和C-MYC (OSKM)共表达时会增强iPSC的诱导成功率[46]。组蛋白变体H2A.Z通常定位在TSS的两侧,Bagchi等[47]发现H2A.Z在NDR两侧是其双向转录的特异性标志。在iPSC重编程过程中,H2A.Z倾向于整合到高转录活性基因的TSS中[48]。综上所述,组蛋白变体作为一种特殊的组蛋白,它们通过与常规组蛋白发生调换从而实现核小体定位改变以及组蛋白翻译后修饰影响染色质重塑,此外在胚胎形成过程中通过使配子的表观遗传修饰趋向一致来调控早期胚胎重编程过程。
5 结语与展望
随着现代生物技术的快速发展,人们通过体细胞核移植技术、细胞融合技术以及诱导多能干细胞技术实现了人为细胞重编程,通过对细胞重编程过程及其调控机制的研究,发现核小体定位动态变化在细胞重编程过程具有十分关键的作用。细胞重编程过程中会激活多能性基因以及沉默另外一些特异性基因,其中就需要基因启动子可以实现“自动开关”,这一过程是通过启动子TSS位置的核小体动态变化来实现的。除此之外,在细胞重编程过程中,一些特殊转录因子以及染色质重塑因子可以特异性结合核小体以及改变核小体定位,从而达到激活某些特定基因的目的,促进细胞重编程。同时,一些染色质重塑因子会引起组蛋白变体置换,改变核小体结构,从而促进细胞重编程。现如今全基因组测序技术和分析方法的飞速发展使得人们对核小体定位的调控机制有了更深的了解,有利于发现更多的基因功能和表观遗传修饰。此外,关于细胞重编程过程的研究主要集中于小鼠与大鼠()这些模型动物上,相比之下在其他大型哺乳动物如猪、牛()、马()的研究很少,在未来畜牧领域的研究具有巨大的应用前景。
[1] Baldi S. Nucleosome positioning and spacing: from genome-wide maps to single arrays., 2019, 63(1): 5–14.
[2] Klemm SL, Shipony Z, Greenleaf WJ. Chromatin accessibility and the regulatory epigenome., 2019, 20(4): 207–220.
[3] Jullien J, Pasque V, Halley-Stott RP, Miyamoto K, Gurdon JB. Mechanisms of nuclear reprogramming by eggs and oocytes: a deterministic process?, 2011, 12(7): 453–459.
[4] Kwon D, Kim JS, Cha BH, Park KS, Han I, Park KS, Bae H, Han MK, Kim KS, Lee SH. The effect of fetal bovine serum (FBS) on efficacy of cellular reprogramming for induced pluripotent stem cell (iPSC) generation., 2016, 25(6): 1025–1042.
[5] Huang KM, Zhang XB, Shi JJ, Yao MZ, Lin JN, Li J, Liu H, Li HH, Shi G, Wang ZB, Zhang BL, Chen JK, Pan GJ, Jiang CZ, Pei DQ, Yao HJ. Dynamically reorganized chromatin is the key for the reprogramming of somatic cells to pluripotent cells., 2015, 5:17691.
[6] Tao CY, Li J, Zhang X, Chen BB, Chi DM, Zeng YQ, Niu YJ, Wang CF, Cheng W, Wu WJ, Pan ZX, Lian JM, Liu HL, Miao YL. Dynamic reorganization of nucleosome positioning in somatic cells after transfer into porcine enucleated oocytes., 2017, 9(2): 642– 653.
[7] Silva J, Barrandon O, Nichols J, Kawaguchi J, Theunissen TW, Smith A. Promotion of reprogramming to ground state pluripotency by signal inhibition., 2008, 6(10): e253.
[8] Kim JB, Zaehres H, Wu GM, Gentile L, Ko K, Sebastiano V, Araúzo-Bravo MJ, Ruau D, Han DW, Zenke M, Schöler HR. Pluripotent stem cells induced from adult neural stem cells by reprogramming with two factors., 2008, 454(7204): 646–650.
[9] Luo M, Ling T, Xie WB, Sun H, Zhou YG, Zhu QY, Shen ML, Zong L, Lyu GL, Zhao Y, Ye T, Gu J, Tao W, Lu ZG, Grummt I. NuRD blocks reprogramming of mouse somatic cells into pluripotent stem cells., 2013, 31(7): 1278–1286.
[10] Taberlay PC, Statham AL, Kelly TK, Clark SJ, Jones PA. Reconfiguration of nucleosome-depleted regions at distal regulatory elements accompanies DNA methylation of enhancers and insulators in cancer., 2014, 24(9): 1421–1432.
[11] Tao Y, Zheng WS, Jiang YH, Ding GT, Hou XF, Tang YT, Li YY, Gao S, Chang GC, Zhang XB, Liu WQ, Kou XC, Wang H, Jiang CZ, Gao SR. Nucleosome organizations in induced pluripotent stem cells reprogrammed from somatic cells belonging to three different germ layers., 2014, 12: 109.
[12] Teif VB, Vainshtein Y, Caudron-Herger M, Mallm JP, Marth C, Höfer T, Rippe K. Genome-wide nucleosome positioning during embryonic stem cell development., 2012, 19(11): 1185–1192.
[13] Lenhard B, Sandelin A, Carninci P. Metazoan promoters: emerging characteristics and insights into transcriptional regulation., 2012, 13(4): 233–245.
[14] Iwafuchi-Doi M, Zaret KS. Cell fate control by pioneer transcription factors., 2016, 143(11): 1833–1837.
[15] Duan JL, Li BX, Bhakta M, Xie SQ, Zhou P, Munshi NV, Hon GC. Rational reprogramming of cellular states by combinatorial perturbation., 2019, 27(12): 3486–3499.e6.
[16] Fernandez Garcia M, Moore CD, Schulz KN, Alberto O, Donague G, Harrison MM, Zhu H, Zaret KS. Structural features of transcription factors associating with nucleosome binding.,2019, 75(5): 921–932.e6.
[17] Boller S, Ramamoorthy S, Akbas D, Nechanitzky R, Burger L, Murr R, Schübeler D, Grosschedl R. Pioneering activity of the C-terminal domain of EBF1 shapes the chromatin landscape for B cell programming., 2016, 44(3): 527–541.
[18] Li R, Cauchy P, Ramamoorthy S, Boller S, Chavez L, Grosschedl R. Dynamic EBF1 occupancy directs sequential epigenetic and transcriptional events in B-cell programming., 2018, 32(2): 96–111.
[19] Dodonova SO, Zhu FJ, Dienemann C, Taipale J, Cramer P. Nucleosome-bound SOX2 and SOX11 structures elucidate pioneer factor function.,2020, 580(7805): 669– 672.
[20] Michael AK, Grand RS, Isbel L, Cavadini S, Kozicka Z, Kempf G, Bunker RD, Schenk AD, Graff-Meyer A, Pathare GR, Weiss J, Matsumoto S, Burger L, Schübeler D, Thomä NH. Mechanisms of OCT4-SOX2 motif readout on nucleosomes., 2020, 368(6498): 1460–1465.
[21] Soufi A, Garcia MF, Jaroszewicz A, Osman N, Pellegrini M, Zaret KS. Pioneer transcription factors target partial DNA motifs on nucleosomes to initiate reprogramming., 2015, 161(3): 555–568.
[22] Soufi A, Donahue G, Zaret KS. Facilitators and impediments of the pluripotency reprogramming factors' initial engagement with the genome., 2012, 151(5): 994–1004.
[23] Jin R, Klasfeld S, Zhu Y, Fernandez Garcia M, Xiao J, Han SK, Konkol A, Wagner D. LEAFY is a pioneer transcription factor and licenses cell reprogramming to floral fate., 2021, 12(1): 626.
[24] Tanaka H, Takizawa Y, Takaku M, Kato D, Kumagawa Y, Grimm SA, Wade PA, Kurumizaka H. Interaction of the pioneer transcription factor GATA3 with nucleosomes., 2020, 11(1): 4136.
[25] Zaret KS. Pioneer transcription factors initiating gene network changes., 2020, 54: 367–385.
[26] Smolle MM. Chd1 bends over backward to remodel., 2018, 25(1): 2–3.
[27] Kingston RE, Tamkun JW. Transcriptional regulation by trithorax-group proteins., 2014, 6(10): a019349.
[28] Rawal Y, Chereji RV, Qiu HF, Ananthakrishnan S, Govind CK, Clark DJ, Hinnebusch AG. SWI/SNF and RSC cooperate to reposition and evict promoter nucleosomes at highly expressed genes in yeast., 2018, 32(9–10): 695–710.
[29] Li ZY, Gadue P, Chen KF, Jiao Y, Tuteja G, Schug J, Li W, Kaestner KH. Foxa2 and H2A.Z mediate nucleosome depletion during embryonic stem cell differentiation., 2012, 151(7): 1608–1616.
[30] Tolkunov D, Zawadzki KA, Singer C, Elfving N, Morozov AV, Broach JR. Chromatin remodelers clear nucleosomes from intrinsically unfavorable sites to establish nucleosome- depleted regions at promoters., 2011, 22(12): 2106–2118.
[31] Clapier CR, Iwasa J, Cairns BR, Peterson CL. Mechanisms of action and regulation of ATP-dependent chromatin-remodelling complexes., 2017, 18(7): 407–422.
[32] Cao ZB, Gao D, Yin HQ, Li H, Xu TT, Zhang MY, Wang X, Liu QC, Yan YL, Ma YY, Yu T, Li YS, Zhang YH. Chromatin remodeler INO80 mediates trophectoderm permeability barrier to modulate morula-to-blastocyst transition., 2021, 42(5): 562–573.
[33] Beckwith SL, Schwartz EK, García-Nieto PE, King DA, Gowans GJ, Wong KM, Eckley TL, Paraschuk AP, Peltan EL, Lee LR, Yao W, Morrison AJ. The INO80 chromatin remodeler sustains metabolic stability by promoting TOR signaling and regulating histone acetylation., 2018, 14(2): e1007216.
[34] Scacchetti A, Brueckner L, Jain D, Schauer T, Zhang X, Schnorrer F, van Steensel B, Straub T, Becker PB. CHRAC/ACF contribute to the repressive ground state of chromatin., 2018, 1(1): e201800024.
[35] Börner K, Jain D, Vazquez-Pianzola P, Vengadasalam S, Steffen N, Fyodorov DV, Tomancak P, Konev A, Suter B, Becker PB. A role for tuned levels of nucleosome remodeler subunit ACF1 duringoogenesis., 2016, 411(2): 217–230.
[36] Shao SM, Cao HW, Wang ZK, Zhou DM, Wu CS, Wang S, Xia D, Zhang DY. CHD4/NuRD complex regulates complement gene expression and correlates with CD8 T cell infiltration in human hepatocellular carcinoma., 2020, 12(1): 31.
[37] Kloet SL, Karemaker ID, van Voorthuijsen L, Lindeboom RGH, Baltissen MP, Edupuganti RR, Poramba-Liyanage DW, Jansen PWTC, Vermeulen M. NuRD-interacting protein ZFP296 regulates genome-wide NuRD localization and differentiation of mouse embryonic stem cells., 2018, 9(1): 4588.
[38] de Dieuleveult M, Yen K, Hmitou I, Depaux A, Boussouar F, Bou Dargham D, Jounier S, Humbertclaude H, Ribierre F, Baulard C, Farrell NP, Park B, Keime C, Carrière L, Berlivet S, Gut M, Gut I, Werner M, Deleuze JF, Olaso R, Aude JC, Chantalat S, Pugh BF, Gérard M. Genome-wide nucleosome specificity and function of chromatin remodellers in ES cells., 2016, 530(7588): 113–116.
[39] Ohhata T, Wutz A. Reactivation of the inactive X chromosome in development and reprogramming., 2013, 70(14): 2443–2461.
[40] Banaszynski LA, Wen DC, Dewell S, Whitcomb SJ, Lin MY, Diaz N, Elsässer SJ, Chapgier A, Goldberg AD, Canaani E, Rafii S, Zheng DY, Allis CD. Hira-dependent histone H3.3 deposition facilitates PRC2 recruitment at developmental loci in ES cells., 2013, 155(1): 107–120.
[41] Cao KX, Lailler N, Zhang YZ, Kumar A, Uppal K, Liu Z, Lee EK, Wu HW, Medrzycki M, Pan CY, Ho PY, Cooper GP Jr, Dong X, Bock C, Bouhassira EE, Fan YH. High-resolution mapping of h1 linker histone variants in embryonic stem cells., 2013, 9(4): e1003417.
[42] Dahl JA, Jung I, Aanes H, Greggains GD, Manaf A, Lerdrup M, Li GQ, Kuan S, Li B, Lee AY, Preissl S, Jermstad I, Haugen MH, Suganthan R, Bjørås M, Hansen K, Dalen KT, Fedorcsak P, Ren B, Klungland A. Broad histone H3K4me3 domains in mouse oocytes modulate maternal-to-zygotic transition., 2016, 537(7621): 548–552.
[43] Shaytan AK, Landsman D, Panchenko AR. Nucleosome adaptability conferred by sequence and structural variations in histone H2A-H2B dimers., 2015, 32: 48–57.
[44] Huh NE, Hwang IW, Lim K, You KH, Chae CB. Presence of a bi-directional S phase-specific transcription regulatory element in the promoter shared by testis-specific TH2A and TH2B histone genes.,1991, 19(1): 93–98.
[45] Padavattan S, Thiruselvam V, Shinagawa T, Hasegawa K, Kumasaka T, Ishii S, Kumarevel T. Structural analyses of the nucleosome complexes with human testis-specific histone variants, hTh2a and hTh2b., 2017, 221: 41–48.
[46] Shinagawa T, Takagi T, Tsukamoto D, Tomaru C, Huynh LM, Sivaraman P, Kumarevel T, Inoue K, Nakato R, Katou Y, Sado T, Takahashi S, Ogura A, Shirahige K, Ishii S. Histone variants enriched in oocytes enhance reprogramming to induced pluripotent stem cells., 2014, 14(2): 217–227.
[47] Bagchi DN, Battenhouse AM, Park D, Iyer VR. The histone variant H2A.Z in yeast is almost exclusively incorporated into the +1 nucleosome in the direction of transcription., 2020, 48(1): 157–170.
[48] Dong FL, Song ZW, Yu JL, Zhang BL, Jiang BC, Shen Y, Lu YD, Song CL, Cong PQ, Liu HL. Dynamic changes in occupancy of histone variant H2A.Z during induced somatic cell reprogramming.,2016, 2016: 3162363.
Progress on the study of nucleosome reorganization during cellular reprogramming
Haoliang Cui, Peihua Shi, Jinchun Gao, Xinbo Zhang, Shunran Zhao, Chenyu Tao
Cellular reprogramming is the process during which epigenetic markers of nuclear genome are deleted and remodeled during sperm-egg binding or nuclear transplantation, thereby rendering differentiated cells totipotent. The main cellular reprogramming methods are cell fusion, somatic cell nuclear transplantation, and induced pluripotent stem cells. Nucleosomes are the basic structural and functional units of chromatin, and nucleosome localization has an important role in regulating gene expression and the state of the cell. The occupancy and location of nucleosomes also change dramatically during cellular reprogramming, while the occupancy of nucleosomes around the transcriptional start site also decreases to promote the expression of pluripotency genes. In this review, we summarize the role of nucleosome localization in gene activation and repression, chromatin remodeling, and transcription factor recognition, with the aim of providing an important basis for an in-depth analysis of cellular reprogramming mechanisms.
cellular reprogramming; nucleosome localization; chromatin remodeling; transcription initiation sites
2021-08-13;
2022-01-07;
2022-02-24
国家自然科学基金项目(编号:31802063),河北省自然科学基金项目(编号:C2020204058)和河北农业大学引进人才科研专项(编号:ZD201718)资助 [Supported by the National Natural Science Foundation of China (No. 31802063), Natural Science Foundation of Hebei Province (No. C2020204058) and Scientific Research Foundation for the Introduction of Talent in Hebei Agricultural University (No. ZD201718)]
崔浩亮,在读硕士研究生,专业方向:动物遗传育种与繁殖。E-mail: 1522942442@qq.com
陶晨雨,副教授,硕士生导师,研究方向:动物遗传育种与繁殖。E-mail: taochenyuty@163.com
10.16288/j.yczz.21-299
(责任编委: 赵建国)
