大熊猫遗传多样性评估的微卫星分型体系优化
2022-03-26寇洁李严王鹏刘红刘佳文王涓王也张亮沈富军
寇洁,李严,王鹏,刘红,刘佳文,王涓,王也,张亮,沈富军
技术与方法
大熊猫遗传多样性评估的微卫星分型体系优化
寇洁1,2,李严1,2,王鹏3,刘红1,2,刘佳文1,2,王涓1,2,王也1,2,张亮1,2,沈富军1,2
1. 成都大熊猫繁育研究基地, 成都 610081 2. 四川省濒危野生动物保护生物学重点实验室,成都 610081 3. 西南科技大学生命科学与工程学院,绵阳 621010
微卫星作为重要的分子标记之一,已被证明在大熊猫种群规模评估、亲子鉴定和遗传多样性分析方面是有效的。目前微卫星标记在大熊猫()染色体上物理定位方面的报道较少,而且缺乏微卫星基因分型系统的效能评估以及PCR扩增条件的优化。本研究基于大熊猫基因组参考序列(ASM200744v2),分析了34个大熊猫微卫星位点的染色体定位特征并评价了位点的应用价值。通过优化34个STR-PCR反应体系和扩增程序,结合微卫星的染色体定位数据确定了Ame-μ10标记的较低应用价值以及gpz-6重新筛选引物的必要性。本研究有助于提高基因分型结果的重复性和可靠性,对促进《大熊猫种群遗传档案建立技术规程》规范化应用和制定大熊猫保护策略具有重要意义。
大熊猫;遗传多样性;微卫星标记;STR-PCR
微卫星标记也被称作短串联重复序列(simple sequence repeats, SSRs)或简单重复序列(short tandem repeats, STRs)。因其具有多等位基因、杂合度高等特点,在种群遗传变异研究方面应用广泛,是目前大熊猫()个体识别、亲子鉴定、遗传多样性研究、种群遗传管理、种群数量调查等领域[1~5]最常用的遗传标记。一些广泛使用的微卫星位点及其配套引物[6~9]已被初步筛选并收录于国家林业和草业局最新颁布的《大熊猫种群遗传档案建立技术规程》(LY/T 2896—2017),用于推动不同科研机构检测结果的重复性和可比性,实现数据共享。
由于微卫星分型体系缺乏检测效能的评估以及STR-PCR扩增条件的优化,在使用这套技术规程时各科研机构之间检测结果的重复性和数据可比性仍然有限,推广应用的问题颇多。首先是收录微卫星标记进行组合应用时未做组合应用价值的细致评估。仅有少数微卫星位点用荧光原位杂交法明确了染色体定位[10],或用基因分型实验验证了彼此间的连锁情况[11]。但大多数微卫星的染色体定位、微卫星位点之间的连锁关系以及同源序列干扰情况仍不明确。用于个体识别和遗传多样性评估的多个微卫星可能位于同一染色体上处于连锁遗传状态,导致分析结论支撑力不足。其次是技术规程对实验背景条件的限制不足。技术规程推荐的反应体系中模板DNA加入量较大,而且反应体系和扩增程序的通用性不足,可能需要针对性地优化扩增条件。野生大熊猫保护研究领域严重依赖于非损伤性样品采集,而这类样品通常含有一定程度的降解DNA和PCR抑制物,模板加入量较多时易导致PCR扩增失败和基因分型错误[12]。为了提高分型结果的可靠性,需对每个位点进行多次独立的PCR扩增,直至获得分型结果的一致性才能确定基因型[12,13]。这就要求PCR模板加入量必须预先得到优化,使反应体系能够在不影响扩增的抑制物浓度和足以进行扩增的目标DNA浓度之间获得平衡。而扩增稳定性和特异性的实现还依赖于反应体系中各组分的配合[14]。提高引物设计质量、调整体系组分之间的加入量通常能有效改善PCR扩增效果。在完整度更高的基因组参考序列中评估引物扩增特异性,并在优化后的PCR扩增条件中验证引物应用价值,将有助于提高检测结果的重复性和可靠性。
为了更好的满足实际应用需求,本研究以2020年新组装到染色体层次的大熊猫基因组参考序列(ASM200744v2)[15]为基础进行微卫星分型体系的位点特征研究和应用价值评估,并对34个常用大熊猫微卫星的PCR反应体系和扩增程序进行优化。本研究可为大熊猫种群遗传保护工作提供研究基础,对促进《大熊猫种群遗传档案建立技术规程》规范化应用和制定大熊猫保护策略具有重要意义。
1 材料与方法
1.1 大熊猫参考基因组
本研究涉及的大熊猫基因组参考序列为最新的ASM200744v2版本[15]。该序列发布于2020年4月20日,已组装到染色体层次。下载路径为:https:// www.ncbi.nlm.nih.gov/assembly/GCF002007445.1。
1.2 拟评估的大熊猫微卫星分型体系
本研究拟评估的常用微卫星标记来源于中华人民共和国林业行业标准《大熊猫种群遗传档案建立技术规程》(LY/T 2896—2017) (https://www.forestry.gov. cn/uploadfile/lykj/2017-11/file/2017-11-2-b7b9c90cc9aa49f0a90c8d68d556d9b9.pdf)和国家发明专利《一种应用于大熊猫亲子鉴定的试剂盒》(ZL201010277132.0),具体信息见表1。这些微卫星标记已被证明在大熊猫群体中有较高的遗传多态性,并被广泛应用于大熊猫个体识别和遗传多样性评估。34对微卫星引物由上海生工生物技术服务有限公司合成,并依据相应参考文献在上游引物或下游引物的5′端添加荧光标记。

表1 大熊猫微卫星标记信息
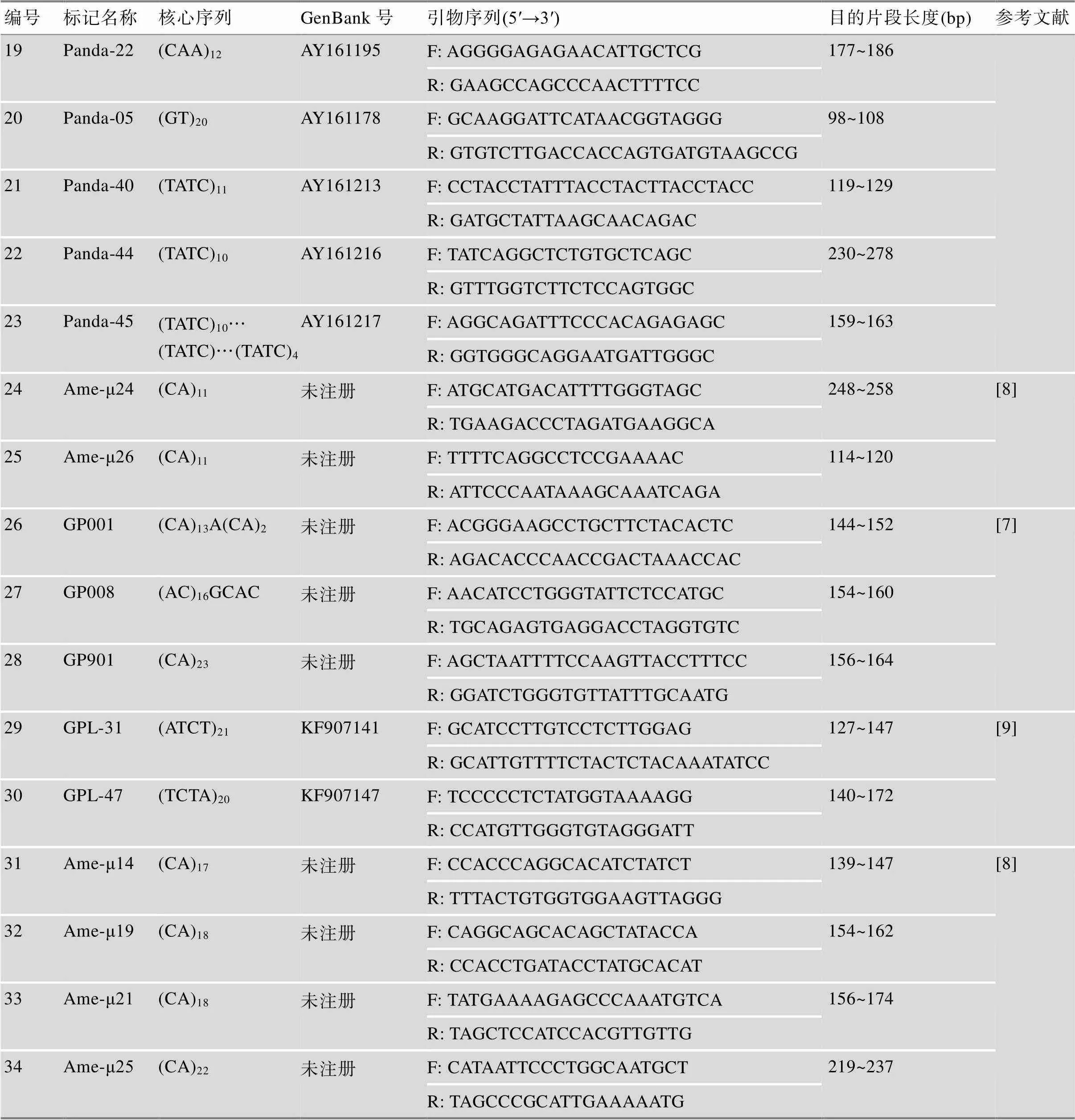
续表
编号1~30为《大熊猫种群遗传档案建立技术规程》(LY/T 2896—2017)收录的30对大熊猫微卫星引物,编号31~34为国家发明专利《一种应用于大熊猫亲子鉴定的试剂盒》(ZL201010277132.0)中参考Lu等[8]补充的4对大熊猫微卫星引物。
1.3 微卫星分型体系的预测和分析
在参考基因组(ASM200744v2版本)上,利用基于基因组信息预测微卫星标记扩增特异性的Python程序(SPE.V1.py)(《微卫星PCR引物特异性鉴定程序》,软件著作权登记号为2021SR0150624)对表1中的微卫星位点及其配套引物进行分析和预测。目标为:(1)确定微卫星位点在参考基因组上的染色体定位;(2)在基因组水平上查找引物同源序列并确定它们在染色体上的位置;(3)模拟上述引物可能引起的多位点扩增结果。按照上述目标,该程序具体分析过程如下:
(1)将基因组序列存贮在一个单独的字典中,其中字典的键是染色体的编号,字典的值是染色体的序列。
(2)根据3′端碱基完全匹配个数,提取出4种形式的引物3′端碱基序列:第一种形式是上游引物3′端序列在基因组中的所有结合位点;第二种形式是下游引物反向互补后3′端序列在基因组中的所有结合位点;第三种形式是下游引物3′端序列在基因组中的所有结合位点;第四种形式是上游引物反向互补后3′端序列在基因组中的所有结合位点。
(3)罗列出(2)中第一种形式和第二种形式所有的组合结果。该组合能扩增出PCR产物满足的条件为:上游引物和下游引物在同一条染色体上,且上游引物位于参考基因组序列的上游。
(4)进一步罗列出(2)中第三种形式和第四种形式所有的组合结果,该组合能扩增出PCR产物满足的条件为:下游引物和上游引物在同一条染色体上,且下游引物位于参考基因组序列的上游。
(5)根据设定好的参数(扩增出的最大PCR产物长度),预测并分析引物的扩增情况,判定正常、疑似多位点扩增、疑似连锁的引物。
(6)预测微卫星位点的染色体物理定位情况。
1.4 PCR扩增条件优化
以1个大熊猫血液纯化DNA样品(谱系号678,浓度20 ng/µL,纯度260/280=1.83)为模板,选择DNA Polymerase (recombinant) (EP0406) (美国Thermo Fisher Scientific公司)、dNTP Mix (10 mmol/L each) (美国Thermo Fisher Scientific公司)和BSA (美国Promega公司)试剂,利用ProFlex PCR system (美国Thermo Fisher Scientific公司)仪器,将表1中34个微卫星的PCR反应体系和扩增程序重新进行优化。扩增产物经2%琼脂糖凝胶电泳检测合格后,采用ABI3500xl遗传分析仪(美国Thermo Fisher Scientific公司)进行扩增产物的电泳分离与检测,并在仪器自带GeneMapper@ ID-X软件下分析等位基因型情况。
20 µL PCR反应体系优化:在《大熊猫种群遗传档案建立技术规程》对PCR产物质量要求的基础上,以扩增重复性好、目的片段亮度好、大小符合预期、目的片段附近无明显非特异性扩增带、操作方便为原则,在技术规程推荐PCR反应体系的基础上,控制34对引物的试剂环境,组合各变量因素条件,分别筛选34对引物各自的最适反应体系。设计单因素实验分别依次优化最适退火温度范围、反应Buffer类型、引物工作液用量、酶用量、dNTP用量、Mg2+用量和模板DNA用量。
PCR扩增程序优化:以分型结果中特异性峰峰型正常且信号强度好、无明显异常峰型、操作方便为原则,以《大熊猫种群遗传档案建立技术规程》推荐PCR扩增程序为对照,在优化后的最适反应体系条件下,针对34对引物分别优化延伸时间和扩增循环次数。
1.5 最优PCR扩增条件的电泳效果验证
以1个大熊猫血液纯化DNA样品(谱系号678,浓度20 ng/µL,纯度260/280=1.83)为模板,分别在两种PCR扩增条件下进行优化效果的验证。对照组按照《大熊猫种群遗传档案建立技术规程》中“10.2 PCR扩增的反应体系和反应程序”执行,待测组则按照本研究优化的PCR扩增条件执行。在各自要求的试剂品牌条件中配制反应体系,在 ProFlex PCR system (美国Thermo Fisher Scientific公司)仪器环境下设置退火温度为55℃,然后进行扩增。5 µL扩增产物在1.5%琼脂糖凝胶中电泳检测。
1.6 基因分型分析
根据1.3软件预测结果,随机选取软件预测结果为正常的两对引物作为对照组,疑似多位点扩增引物和疑似连锁引物分别作为两类待测组,以30个大熊猫血液纯化DNA样品(浓度20~60 ng/µL,纯度260/280=1.13~1.97,谱系关系见附图1)为模板,按照优化后的最适反应体系和扩增程序条件进行扩增。扩增产物经2%琼脂糖凝胶电泳检测合格后,采用ABI3500xl遗传分析仪(美国Thermo Fisher Scientific公司)进行扩增产物的电泳分离与检测,并在仪器自带GeneMapper@ ID-X软件下分析等位基因型情况。先根据微卫星目的片段长度范围和核心序列碱基重复数,确定理论上可能的目的片段长度。结合软件自动判定基因型结果和分型图谱,在考虑酶加A碱基效应的前提下,对最终分型结果进行人工校正。
2 结果与分析
2.1 微卫星分型体系的预测和分析结果
引物分析结果显示,30对引物被判定为高应用价值的正常引物;引物Ame-µ11与panda-44被判定为疑似连锁引物,应用价值待实验评估;引物Ame- µ10和gpz-6的3′端碱基结合参考基因组非目标位置的能力强,被判定为应用价值不高的疑似多位点扩增引物(附表1)。
位点的染色体分布预测结果(表2)显示,32个位点匹配上参考基因组特定区域的可能性高,被判定为单位点微卫星。但Ame-µ11与panda-44在参考基因组上存在连锁不平衡可能性,两个预测目标位置之间的距离为3,514,165 bp,位点应用价值待实验评估。另外2个位点Ame-µ10和gpz-6在染色体上有较大概率呈现多位点分布。
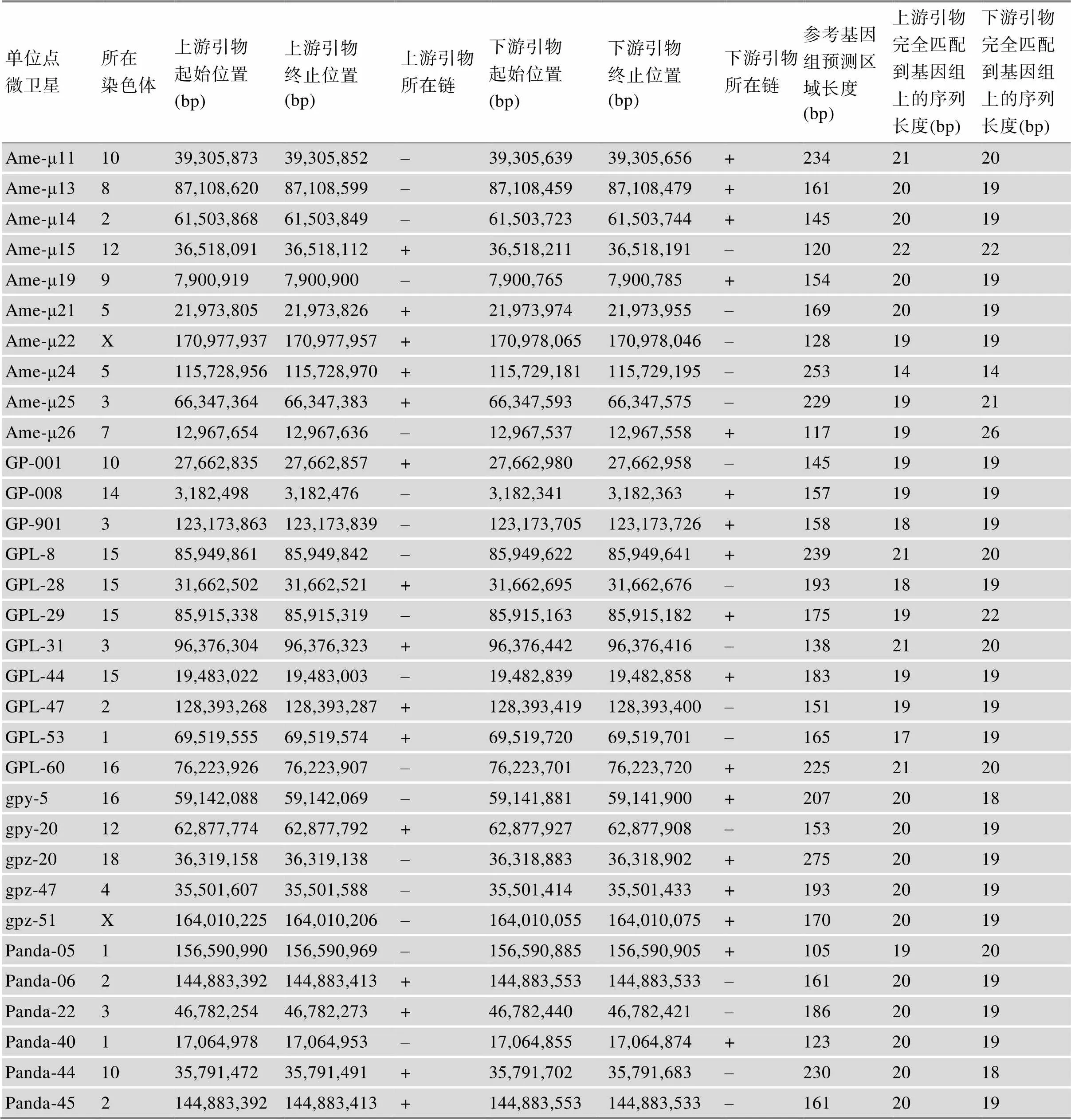
表2 大熊猫染色体中32个微卫星分布情况
“+”表示正义链,“–”表示反义链。
2.2 优化的PCR扩增条件及其验证结果
优化的PCR反应体系及扩增程序见表3和表4。用大熊猫血液DNA对优化获得的STR-PCR反应体系及扩增程序进行优化效果验证,琼脂糖凝胶电泳结果见图1。电泳结果显示,待测组和对照组电泳条带均符合《大熊猫种群遗传档案建立技术规程》对PCR产物的质量检测要求。但待测组模板用量为对照组的16%,扩增循环数相对更少,非特异性扩增程度更轻。
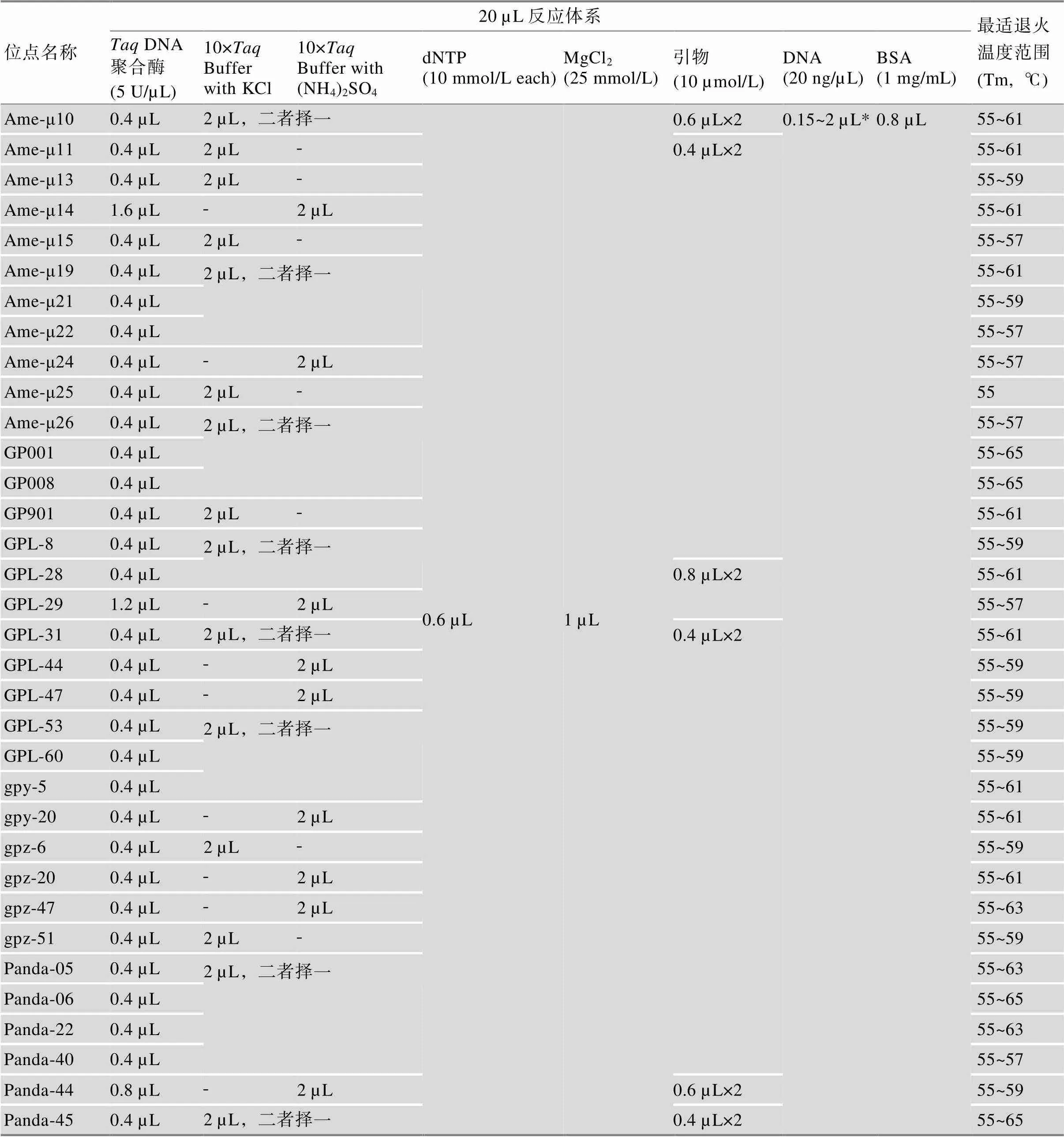
表3 优化的PCR反应体系
*建议Ame-μ11、Panda-44和Ame-μ14按照最大DNA参考加入量执行。
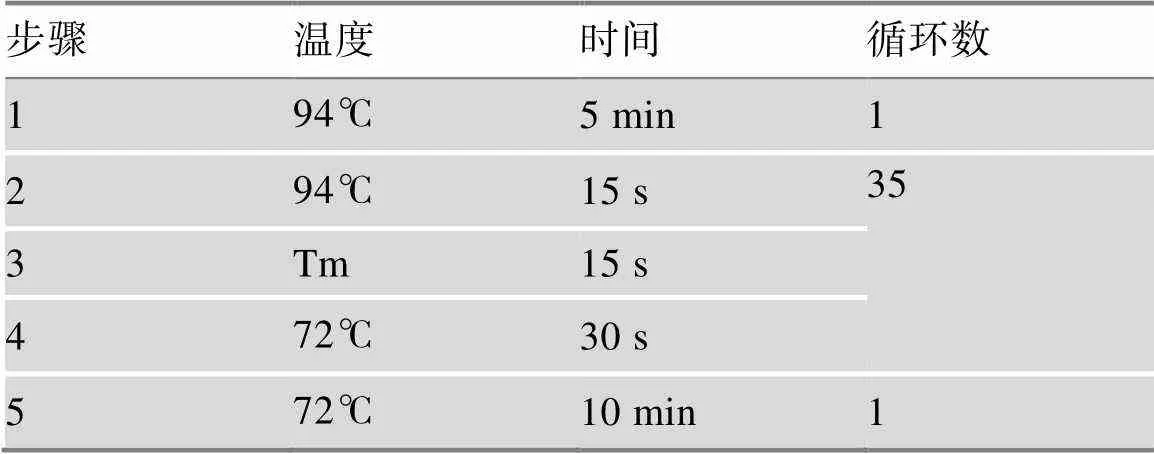
表4 优化的PCR扩增程序
2.3 基因分型结果
30个大熊猫血液纯化DNA样品的6个微卫星分型结果见表5,根据表5和附图1整理出的11组亲代及其子代的微卫星分型结果见表6,典型分型图谱见图2。分型结果显示(表5),所有位点均获得有效扩增。2个疑似连锁微卫星(Panda-44和Ame-μ11)的分型结果不符合连锁遗传规律,位点和引物的应用价值再评估结果良好。11组亲代及其子代的微卫星分型结果显示(表6),疑似多位点微卫星gpz-6出现了2个PCR复制滑脱现象;对照微卫星Ame-μ19出现了3个PCR复制滑脱现象;在11个分组内部,其余的分型结果均符合预期,亲子关系成立。分型图谱结果显示,扩增产物片段长度符合预期,各基因座之间扩增产物相对平衡。2个疑似连锁微卫星(Panda-44见图2A,Ame-μ11见图2B)、2个对照微卫星(GPL-29见图2C,Ame-μ19见图2D)均有信号明显的主峰,在主峰小于核心序列重复单位的位置无明显PCR复制滑脱所形成的“stutter峰”,也无明显非特异性峰,表明引物特异性好且PCR反应体系组分添加比例适宜;没有出现由3′端加A碱基不全所形成的比目的峰少一个碱基的“双尖峰”,表明扩增程序中延伸时间设置合适。2个疑似多位点微卫星(Ame-μ10见图2E、F、G和H,gpz-6见图2I、J、K和L)均有连续的多峰;主峰附近杂峰(图2箭头所示)的峰高或峰面积为主峰的25%~50%;在所有检测个体的分型结果中,杂峰在主峰位置少2~8 bp处固定出现,排除“stutter峰”或“双尖峰”可能性,判定为非特异性扩增峰;非特异性峰与目的峰有融合现象(图2L圆圈所示)。Ame-μ10和gpz-6的分型结果表明,优化PCR反应体系和扩增程序不能消除非特异性扩增;部分非特异性扩增峰信号可能干扰分型结果判读。提示Ame-μ10和gpz-6需要提升扩增特异性,引物直接应用价值不高。
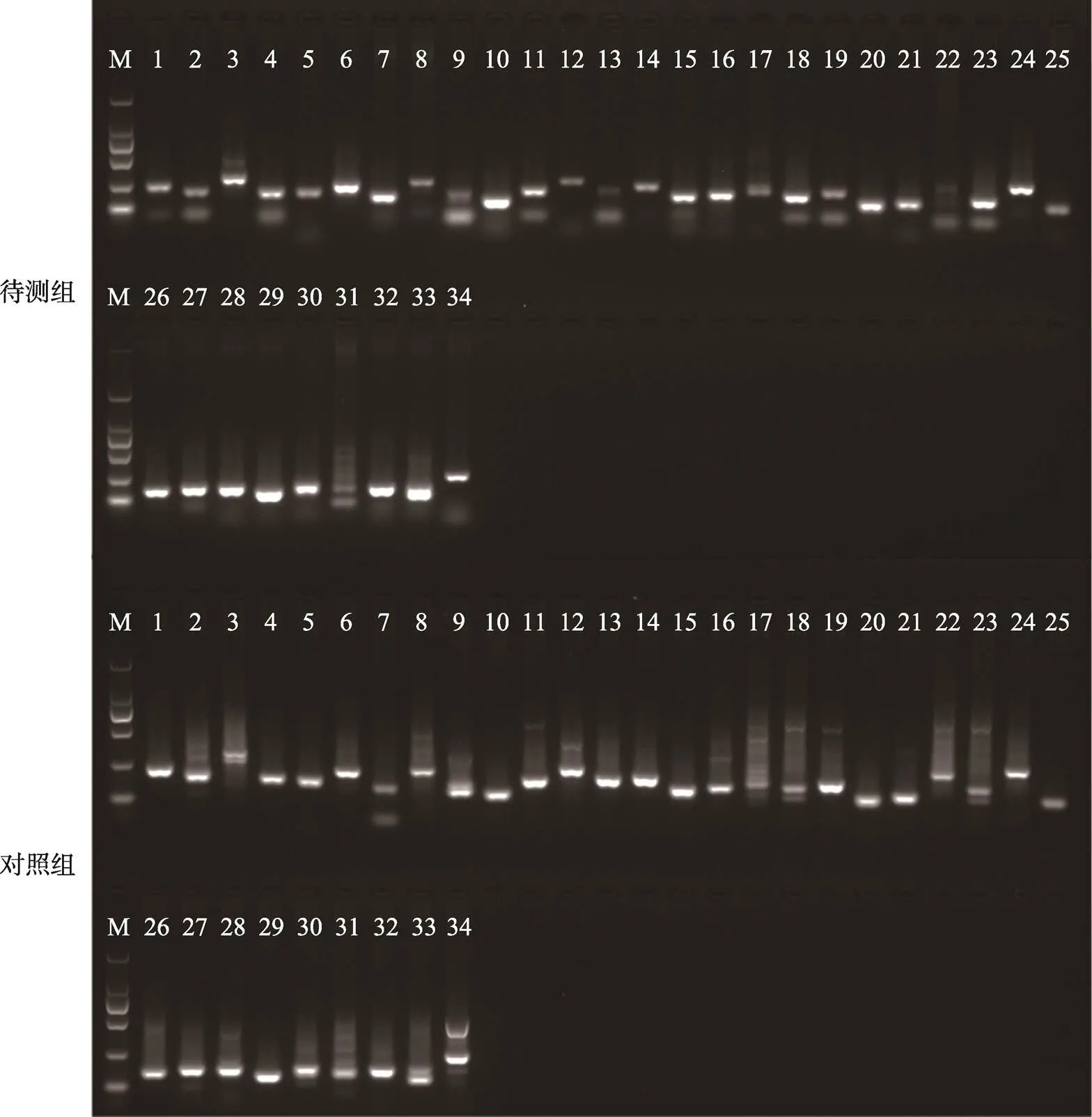
图1 1.5%琼脂糖凝胶电泳检测图
M:D2000分子质量标准;1~34:与表1微卫星编号相对应。
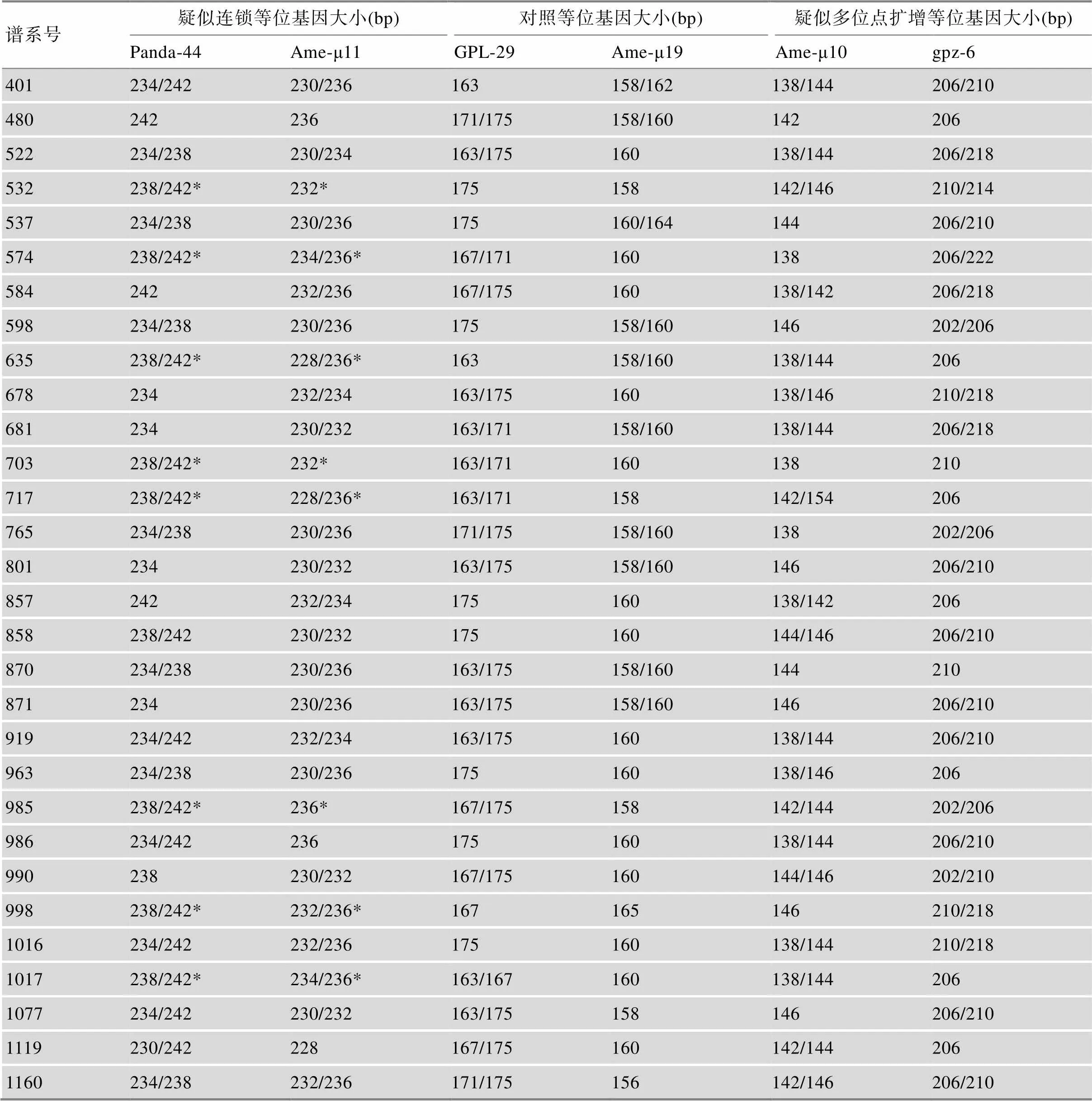
表5 6个微卫星标记的基因分型结果
*表示分型结果不符合连锁遗传规律。
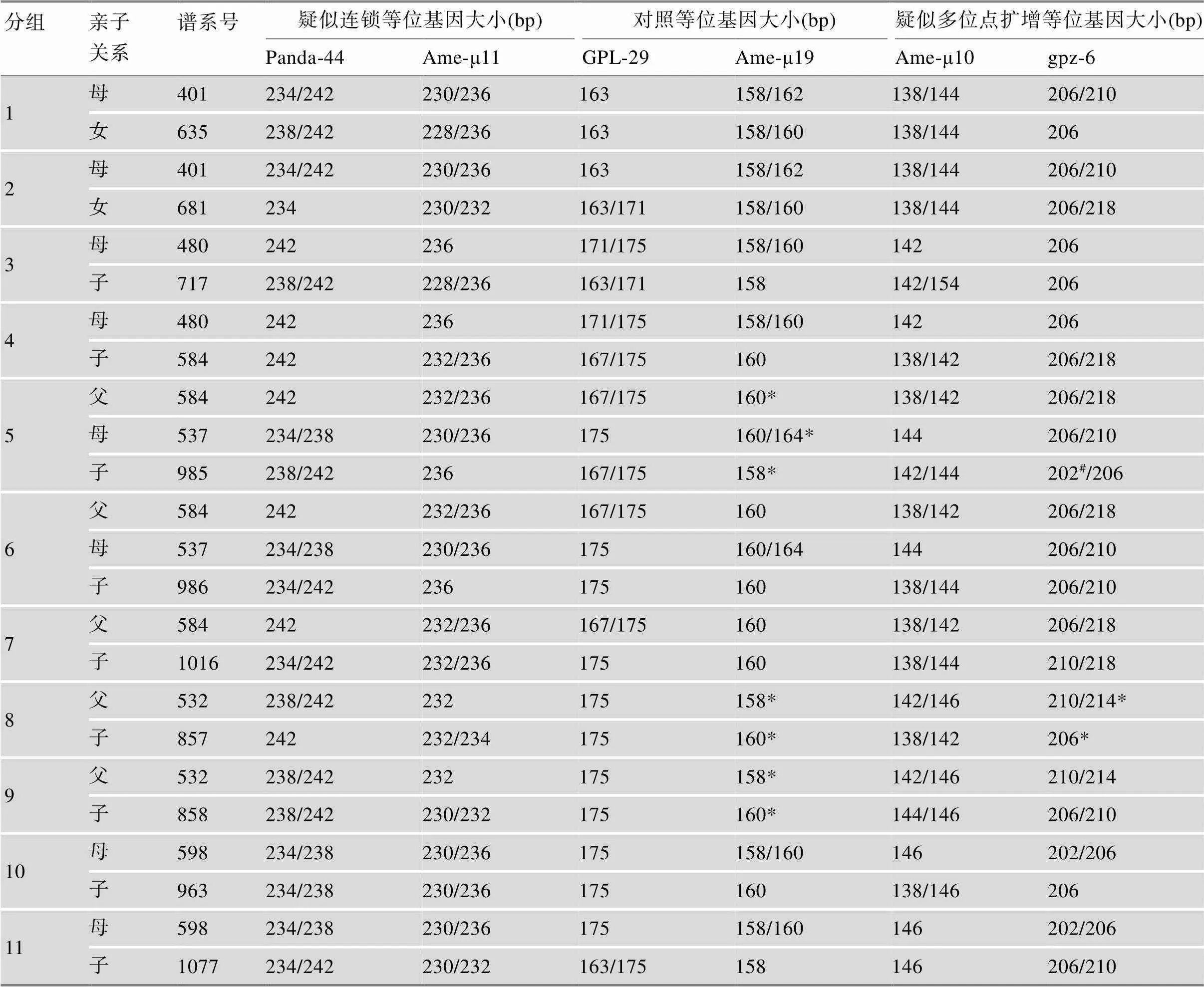
表6 11组亲代及其子代的基因分型结果
*表示该分组内引物的亲子鉴定结果异常,可能存在一个或数个重复单元的PCR复制滑脱现象;#表示该等位基因均未在双亲中检出,可能存在一个或数个重复单元的PCR复制滑脱现象。
3 讨论
借助测序技术和各种生物信息学工具,大量微卫星位点被识别并筛选用于大熊猫遗传多样性分析[9,16,17]。尽管相关实验验证了这些微卫星位点的多态性,但在使用成套微卫星标记分析大熊猫遗传多样性时,结论的客观可靠性仍是个问题。一部分原因是利用传统的微卫星分子标记开发方法挖掘了大量两碱基重复标记。而这类标记发生滑链效应的可能性较高[18]。另一部分原因是研究机构选取的标记组合不统一。由于缺乏微卫星在染色体上的物理定位研究,选取标记组合中的多个位点可能在同一染色体上连锁。因此需要在设计微卫星组合进行个体识别、遗传多样性评估之前,先分析各位点的特点,再筛选出独立表达的优质微卫星组合。但目前仅有少量微卫星位点通过实验明确了其在染色体上的定位或者相互之间的连锁关系[10,11],远远不能满足应用终端的鉴定需求。与其他模式动物大量的成熟微卫星相比,大熊猫优质微卫星标记的数量有限,不利于现有微卫星分型系统效能的进一步提升。大熊猫基因组在染色体层次上完整度更高的组装,弥补了“晶晶”基因组草图的缺陷,为解决微卫星物理定位需求、再评估位点和引物质量提供了便利。本研究通过软件分析,获得了32个微卫星位点在大熊猫参考基因组上的定位,并证实这些微卫星具有一定应用价值。该结果将为今后各机构研究大熊猫遗传数据奠定理论基础。
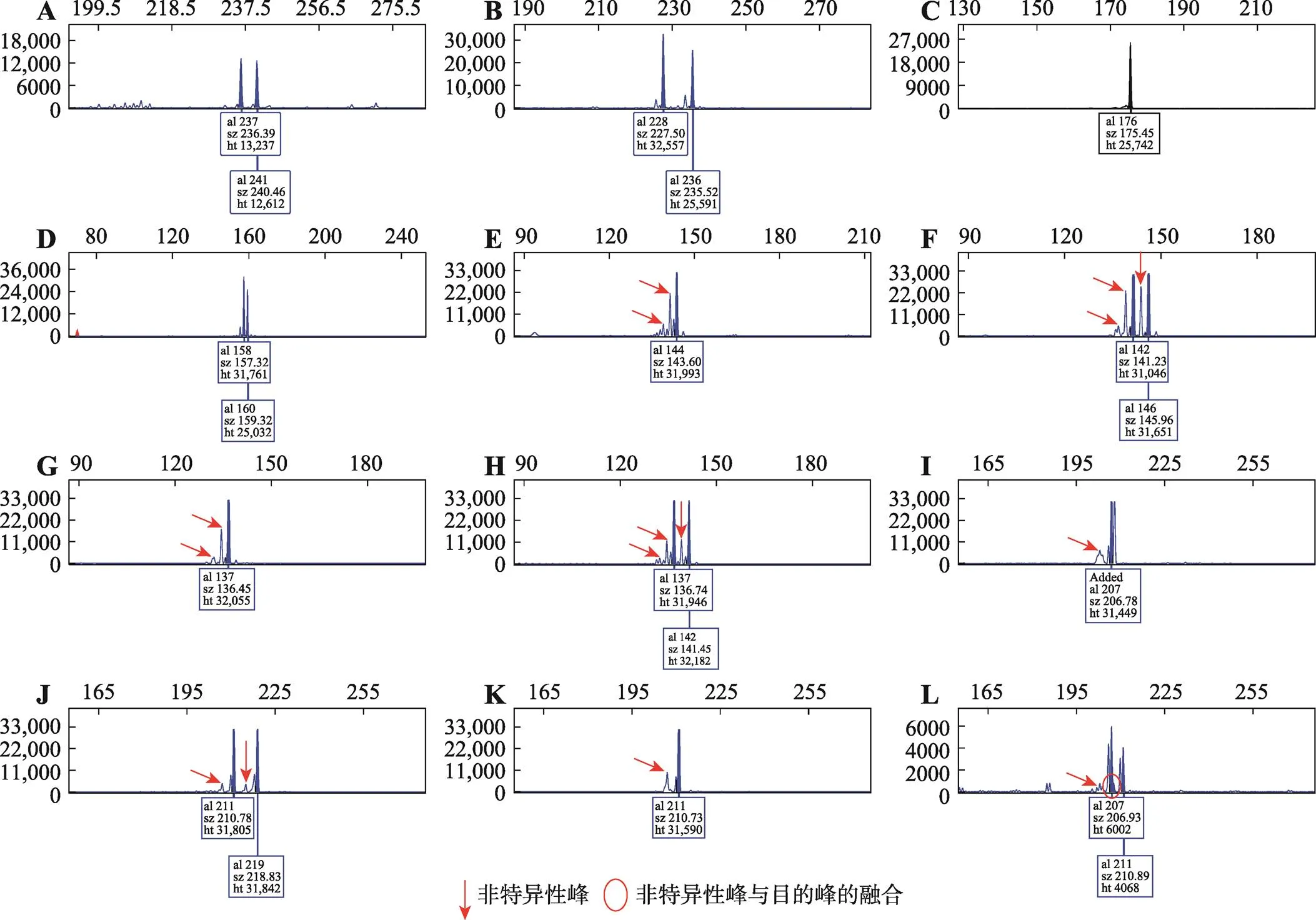
图2 6个微卫星标记典型分型图谱
A:Panda-44,谱系号717;B:Ame-μ11,谱系号717;C:GPL-29,谱系号537;D:Ame-μ19,谱系号598;E:Ame-μ10,谱系号537;F:Ame-μ10,谱系号532;G:Ame-μ10,谱系号765;H:Ame-μ10,谱系号857;I:gpz-6,谱系号1017;J:gpz-6,谱系号1016;K:gpz-6,谱系号703;L:gpz-6,谱系号801。红色箭头表示非特异性峰,红色圆圈表示非特异性峰与目的峰的融合现象;图横坐标为片段大小(bp),纵坐标为荧光信号强度。
作为全国第4次大熊猫调查的辅助调查手段,微卫星与非损伤性遗传取样法的联用对大熊猫保护研究做出了巨大贡献。在大熊猫遗传样本采集压力较大的现状下,采取措施提升现有微卫星分型体系的扩增效率是必要的。PCR扩增可能会因为各种原因而失败。即使是同一物种也会因为实验背景条件的不同而存在差异性的最适反应体系[19~21]。因此几乎每个实验室都必须根据实验室硬件、模板质量和试剂供应情况,预先优化PCR扩增条件。缺乏统一的实验背景导致各科研机构的分型数据不能被有效共享。对此,国家林业局制定并推动《大熊猫种群遗传档案建立技术规程》应用,以期提高数据可比性。但该技术规程对试剂品牌、操作规程等实验背景的限制不够明确,在强调灵活操作的同时也未细致筛选标记组合以及提出针对性的优化建议。在这种变量因素较多的条件下,模板质量和加入量可能会显著影响PCR扩增结果[14,22]。因此,本研究通过控制试剂、仪器等背景条件,采取单因素考察方法调整包括酶、模板、Mg2+、引物、dNTP等在内的多个变量因素,获得了34套微卫星分型体系的最适扩增条件供研究人员参考。相对于原技术规程,不仅增加了DNA利用效率,而且提升了PCR扩增质量,更有利于各种应用场景下鉴定结果的重现性和数据可比性。但需要指出的是,本研究中使用的模板为大熊猫血液纯化DNA,在反应体系中的模板加入量为参考加入量。对于非损伤性的粪便样本,大熊猫DNA在背景DNA中的相对含量取决于研究者富集动物细胞的效率,整体质量则取决于样本保存质量以及PCR抑制物去除效率[23]。建议根据前人的方法[24~26]先尽量富集和纯化大熊猫DNA并确定其在提取产物中的大致浓度,结合本研究中的参考加入量,再开展后续实验。另外,相对于其他引物,Ame-μ11、Panda-44和Ame-μ14虽有一定的扩增特异性,但目的条带亮度难以大幅度优化提升。这提示上述引物的检测灵敏度相对较低,建议涉及非损伤性DNA时慎重使用。
Ame-μ10为定位在大熊猫3号染色体近着丝粒长臂端的微卫星标记[10],是由Lu等[8]基于克隆和探针杂交技术从DNA文库中筛选所得,近年来在大熊猫遗传保护领域应用频率相对较高[1,4,27~29]。但本研究证实,引物Ame-μ10易导致多位点扩增,且利用最优扩增条件不能完全消除分型结果中非特异性扩增峰的影响。两碱基重复的微卫星标记更有可能发生PCR滑链效应,产生相对较多的“stutter”峰产物[18]。而过多的“stutter”峰产物将会增加峰型判断难度,导致分型错误率上升[30,31]。在电泳分析杂合子时,两碱基重复的微卫星标记也会因等位基因碱基差异较小而增加分型判断的难度。因此乔麦菊等[5]主张加大对稳定性更强的四碱基标记的使用。本研究分析了3对两碱基重复的微卫星引物,其中Ame-μ19出现了相对高频的PCR滑链效应,导致这部分分型结果不符合亲子关系的理论预期(表6)。而已经开展的野外大熊猫遗传多样性研究大多以两碱基微卫星为主,从提升技术规程系统效能的角度考虑,继续利用两碱基重复微卫星位点以及重新筛选引物的意义不大。四碱基重复的gpz-6是Huang等[9]根据大熊猫“晶晶”基因组草图筛选所得,因其在非损伤性样本来源DNA的PCR扩增中表现良好而备受关注[3,4,9,32]。本研究结果中,引物gpz-6理论上结合参考基因组非目标位置的能力较强;分型结果中非特异性扩增峰不易被消除且存在干扰目的峰信号的可能性(图2L);在11组亲子关系的鉴定结果中,有2组亲子(表6)的基因分型结果不符合理论预期。gpz-6位点应用价值虽高,但有重新提升引物设计质量的必要性。
综上所述,本研究通过序列比对,获取了32个微卫星标记的染色体定位信息;优化微卫星分型的PCR扩增条件,确定了Ame-μ10标记的较低应用价值以及引物gpz-6重筛选的必要性。但本研究涉及的样本量较少,对标记应用价值的评估方法较为单一。大熊猫参考基因组序列完整度还存在继续提升的可能性。在未来的研究中可以借助大量个体的基因组重测序数据,应从多个角度综合分析微卫星位点的应用价值,并采用其他测序数据解读工具辅助判断微卫星组合的检测系统效能。需要指出的是,本研究中部分引物的设计长度与匹配到参考基因组上的序列长度有出入(表2),优化的PCR扩增条件不能完全消除非特异性扩增情况(图1)。这提示过去一直使用的微卫星引物可能需要继续优化设计质量。为了进一步验证软件预测结果,本研究分析了6对微卫星引物在30个DNA中的基因分型图谱。受研究条件限制,未能继续分析其余28对微卫星引物。在未来的研究中,还需要进一步评估这些引物的应用价值,继续提升微卫星分型的系统效能。
附加材料详见文章电子版www.chinagene.cn。
[1] Ma TX, Hu YB, Russo IRM, Nie YG, Yang TY, Xiong LJ, Ma S, Meng T, Han H, Zhang XM, Bruford MW, Wei FW. Walking in a heterogeneous landscape: dispersal, gene flow and conservation implications for the giant panda in the Qinling Mountains., 2018, 11(10): 1859– 1872.
[2] Qiao MJ, Zhou YM, Connor T, Li RG, Tang D, Zhang HM, Ran JH. Diagnosing zygosity in giant panda twins using short tandem repeats., 2018, 21(6): 527–532.
[3] Qiao MJ, Connor T, Shi XG, Huang J, Huang Y, Zhang HM, Ran JH. Population genetics reveals high connectivity of giant panda populations across human disturbance features in key nature reserve., 2019, 9(4): 1809–1819.
[4] Dai QL, Li JW, Yang Y, Li M, Zhang K, He LY, Zhang J, Tang B, Liu HP, Li YX, Zhu LF, Yang ZS, Dai Q. Genetic diversity and prediction analysis of small isolated giant panda populations after release of individuals., 2020, 16: 1176934320939945.
[5] Qiao MJ, Ran JH, Zhang HM. The application of microsatellite markers in giant panda research., 2019, 39(1): 103–110.
乔麦菊, 冉江洪, 张和民. 微卫星标记在大熊猫研究中的应用进展. 兽类学报, 2019, 39(1): 103–110.
[6] Shen FJ, Watts P, Zhang ZH, Zhang AJ, Sanderson S, Kemp SJ, Yue BS. Enrichment of giant panda microsatellite markers using dynal magnet beads., 2005, 32(5): 457–62.
沈富军, Phill Watts, 张志和, 张安居, Stephanie Sanderson, Steve J. Kemp, 岳碧松. Dynal磁珠富集大熊猫微卫星标记. 遗传学报, 2005, 32(5): 457–462.
[7] Zhang YP, Wang W, Su B, Fan ZY, Zhang HM, He TM, Ryder OA. Screening and application of giant panda microsatellite DNA., 1995, 16(4): 301–306.
张亚平, 王文, 宿兵, 范志勇, 张和民, 何廷美, Oliver A. Ryder. 大熊猫微卫星DNA的筛选及其应用. 动物学研究, 1995, 16(4): 301–306.
[8] Lu Z, Johnson WE, Menotti-Raymond M, Yuhki N, Martenson JS, Mainka S, Huang SQ, Zheng ZH, Li GH, Pan WS, Mao XR, O’Brien SJ. Patterns of genetic diversity in remaining giant panda populations., 2001, 15(6): 1596–1607.
[9] Huang J, Li YZ, Du LM, Yang B, Shen FJ, Zhang HM, Zhang ZH, Zhang XY, Yue BS. Genome-wide survey and analysis of microsatellites in giant panda (), with a focus on the applications of a novel microsatellite marker system., 2015, 16(1): 61.
[10] 田亮. 应用微卫星进行大熊猫亲子鉴定及微卫星在染色体上的物理定位[学位论文]. 中国农业大学, 2006.
[11] Wu H, Zhan XJ, Zhang ZJ, Zhu LF, Yan L, Li M, Wei FW. Thirty-three microsatellite loci for noninvasive genetic studies of the giant panda ()., 2009, 10: 649–652.
[12] Taberlet P, Griffin S, Goossens B, Questiau S, Manceau V, Escaravage N, Waits LP, Bouvet J. Reliable genotyping of samples with very low DNA quantities using PCR., 1996, 24(16): 3189–3194.
[13] Navidi W, Arnheim N, Waterman MS. A multiple-tubes approach for accurate genotyping of very small DNA samples by using PCR: statistical considerations., 1992, 50(2): 347–359.
[14] Ghaffari S, Hasnaoui N. Microsatellite amplification in plants: optimization procedure of major PCR components., 2013, 1006: 139–146.
[15] Fan HZ, Wu Q, Wei FW, Yang FT, Ng BL, Hu YB. Chromosome-level genome assembly for giant panda provides novel insights into Carnivora chromosome evolution., 2019, 20(1): 267.
[16] Tu HM, Zhou C, Wang GN, Cheng ML, Yue BS, Meng Y. Development of polymorphic microsatellite markers forbased on RNA-sequencing., 2020, 39(1): 15–22.
涂洪梅, 周闯, 王冠楠, 成美玲, 岳碧松, 孟杨. 基于转录组测序的大熊猫多态性微卫星标记筛选. 四川动物, 2020, 39(1): 15–22.
[17] Song XH, Shen FJ, Huang J, Huang Y, Du LM, Wang CD, Fan ZX, Hou R, Yue BS, Zhang XY. Transcriptome-Derived tetranucleotide microsatellites and their associated genes from the giant panda ()., 2016, 107(5): 423–430.
[18] Morin PA, Chambers KE, Boesch C, Vigilant L. Quantitative polymerase chain reaction analysis of DNA from noninvasive samples for accurate microsatellite genotyping of wild chimpanzees ()., 2001, 10(7): 1835–1844.
[19] Wang ZY, Guo HL, Liu JX. Optimization of SSR-PCR system onspp. by orthongonal design., 2007, 5(z1): 201–206.
王志勇, 郭海林, 刘建秀. 正交设计优化狗牙根SSR-PCR反应体系. 分子植物育种, 2007, 5(z1): 201–206.
[20] Patiguli M, Zhang YH, LI PY, Abulaiti. Preliminary analysis on optimization of EST-SSR PCR reaction system and genetic diversity forL., 2014, 37(2): 112–118.
帕提古丽·麦麦提敏, 张延辉, 李培英, 阿不来提. 狗牙根EST-SSR反应体系优化及其遗传多样性初步分析. 新疆农业大学学报, 2014, 37(2): 112–118.
[21] Long T, Huang CQ, Liu GD. Optimization of SSR-PCR reaction system forby orthogonal design., 2016, 35(1): 198–205.
龙萄, 黄春琼, 刘国道. 正交设计优化狗牙根SSR-PCR反应体系. 基因组学与应用生物学, 2016, 35(1): 198– 205.
[22] Johnson PCD, Haydon DT. Maximum-likelihood estimation of allelic dropout and false allele error rates from microsatellite genotypes in the absence of reference data., 2007, 175(2): 827–842.
[23] Shan L, Hu YB, Wei FW. Opportunities and challenges of fecal DNA technology in molecular ecology researches., 2018, 38(3): 235–246.
单磊, 胡义波, 魏辅文. 粪便DNA分析技术在分子生态学研究中的机遇与挑战. 兽类学报, 2018, 38(3): 235–246.
[24] Zhu Y, Liu HY, Yang HQ, Li YD, Zhang HM. Factors affecting genotyping success in giant panda fecal samples., 2017, 5: e3358.
[25] Wan QH, Zhu L, Wu H, Fang SG. Major histocompatibility complex class II variation in the giant panda ()., 2006, 15(9): 2441–2450.
[26] Zhang BW, Wei FW, Li M, Lü XP. A simple protocol for DNA extraction from faeces of the giant panda and lesser panda., 2004, 50(3): 452–458.
张保卫, 魏辅文, 李明, 吕晓平. 大熊猫和小熊猫粪便DNA提取的简易方法. 动物学报, 2004, 50(3): 452–458.
[27] Zhu LF, Hu YB, Qi DW, Wu H, Zhan XJ, Zhang ZJ, Bruford MW, Wang JL, Yang XY, Gu XD, Zhang L, Zhang BW, Zhang SN, Wei FW. Genetic consequences of historical anthropogenic and ecological events on giant pandas., 2013, 94(10): 2346–2357.
[28] Shan L, Hu YB, Zhu LF, Yan L, Wang CD, Li DS, Jin XL, Zhang CL, Wei FW. Large-scale genetic survey provides insights into the captive management and reintroduction of giant pandas., 2014, 31(10): 2663–2671.
[29] Hu YB, Nie YG, Wei W, Ma TX, Van Horn R, Zheng XG, Swaisgood RR, Zhou ZX, Zhou WL, Yan L, Zhang ZJ, Wei FW. Inbreeding and inbreeding avoidance in wild giant pandas., 2017, 26(20): 5793–5806.
[30] Taberlet P, Waits LP, Luikart G. Noninvasive genetic sampling: look before you leap., 1999, 14(8): 323–327.
[31] Bowler DE, Benton TG. Causes and consequences of animal dispersal strategies: relating individual behaviour to spatial dynamics., 2005, 80(2): 205–225.
[32] Guo W, Mishra S, Wang CD, Zhang HM, Ning RH, Kong FL, Zeng B, Zhao JC, Li Y. Comparative study of gut microbiota in wild and captive giant pandas ()., 2019, 10(10): 827.
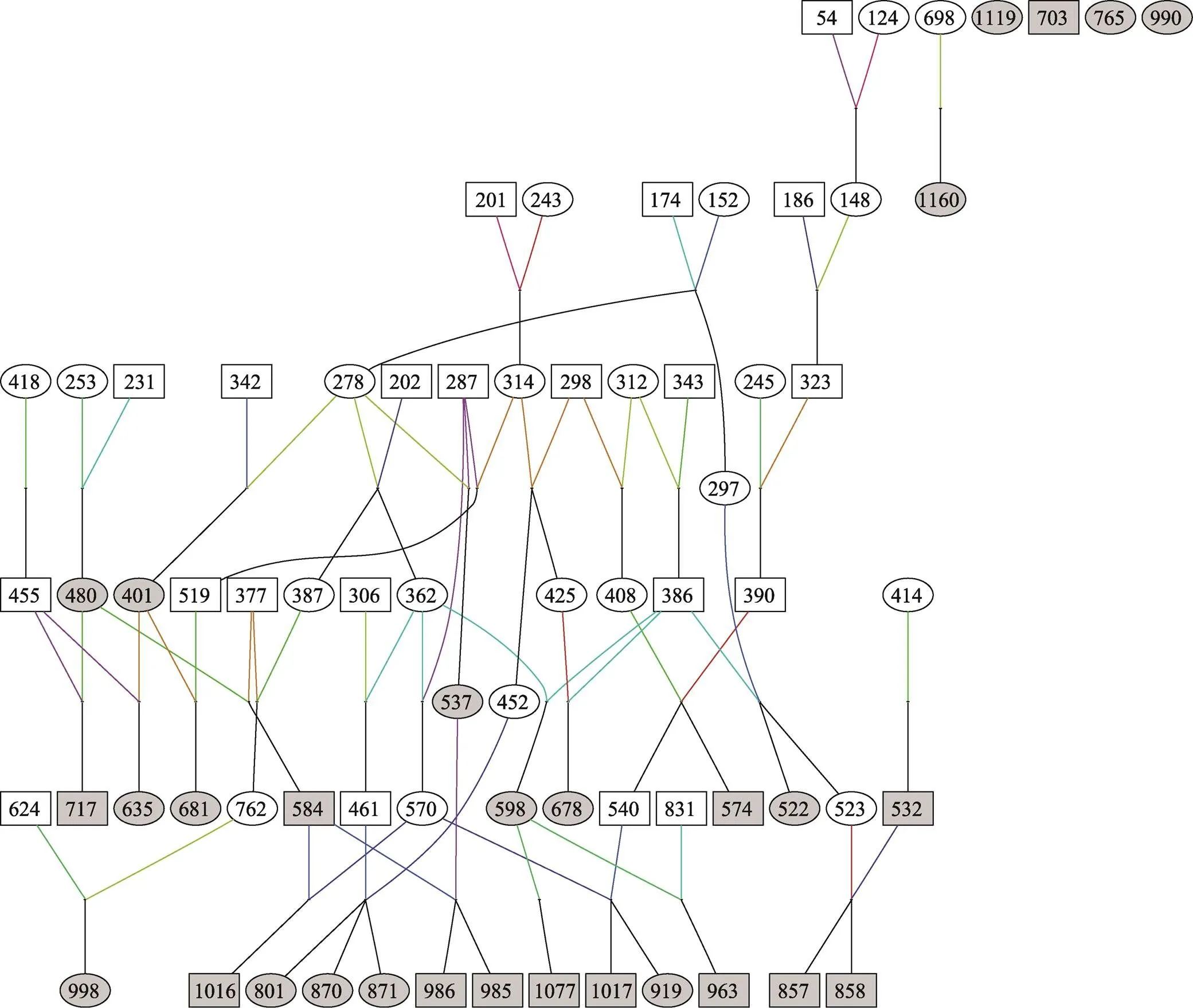
附图1 30个大熊猫的谱系关系图
Supplementary Fig. 1 The pedigree diagram of 30 giant pandas
在Pedigraph软件(https://animalgene.umn.edu/download-pedigraph)制作的谱系关系图中,每个个体都由一个节点来表示。节点内的标签是个体的谱系号。圆圈表示雌性,方形表示雄性。用彩色线将子代与其亲代联系起来。灰色节点代表本实验涉及的30只大熊猫。
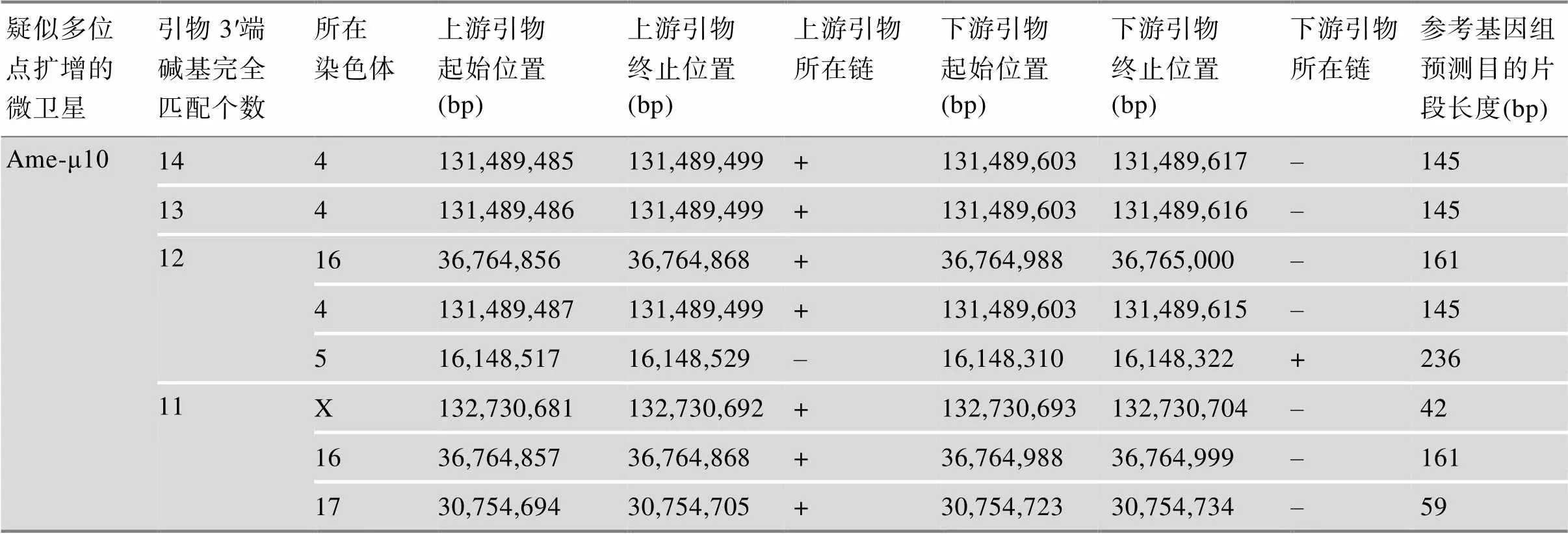
附表1 两对微卫星引物(Ame-µ10、gpz-6)的扩增情况
