关中奶山羊CSN1S1基因克隆、生物信息学及组织表达谱分析
2022-03-17侯金星王战航朱俊儒刘淑娟安小鹏
侯金星,王战航,朱俊儒,江 悦,刘淑娟,安小鹏
(1.杨凌职业技术学院,杨凌 712100;2.西北农林科技大学动物科技学院,杨凌 712100)
山羊奶营养成分丰富,营养价值接近人乳,被营养学界评价为唯一可与人乳相媲美的功能性食品[1]。与牛奶相比,羊奶还具有抗氧化、消炎作用,可改善婴儿的轻微消化紊乱,预防过敏性疾病[2]。羊奶中具有更开放的酸诱导凝固结构和更细的蛋白质链,婴幼儿食用后胃蛋白酶可以更好地扩散,与牛奶相比,羊奶在婴幼儿的胃中更容易被消化[3]。羊奶含有丰富的营养保健功能,其短链脂肪酸含量丰富,不易在体内沉积,且因其核苷酸含量丰富,还具有益脑健智的功能[4]。αs1-酪蛋白(alpha-s1 casein,CSN1S1)基因位于山羊6号染色体一段长250~300 kb区域中,与它共同组成基因座的还有β-酪蛋白(CSN2)、αS2酪蛋白(CSN1S2)和κ-酪蛋白(CSN3)。山羊CSN1S1基因延伸出约16 785 bp的片段,其中外显子区域长1 138 bp、内含子区域长15 647 bp,与牛CSN1S1基因序列相似性约为57%。编码酪蛋白的基因普遍具有多态性,研究表明,CSN1S1基因的多态性不仅会影响羊奶中酪蛋白的含量,而且会影响羊奶的结构和营养特性[5]。研究发现,CSN1S1基因中11 bp的突变与关中奶山羊羊奶中的酸度有显著关系[6];CSN1S1基因多态性对挪威奶山羊的游离脂肪酸浓度有显著影响[7]。本试验以关中奶山羊为研究对象,PCR扩增并克隆了关中奶山羊CSN1S1-1和CSN1S1-2 2种突变形态,利用多种生物信息学软件及在线工具对突变型和野生型序列进行分析比对,同时分析其在关中奶山羊各组织中的表达情况,为进一步研究CSN1S1基因在奶山羊乳成分合成中的调控作用提供理论依据。
1 材料与方法
1.1 材料
于西北农林科技大学关中奶山羊养殖场,选取泌乳期的健康关中奶山羊,屠宰后采集肝脏、脾脏、乳腺、肾脏、子宫、输卵管组织1~2 g,置于DEPC水中漂洗后迅速放入液氮罐中保存。采集乳腺组织用PBS冲洗2~3次,保存于含有双抗的PBS中,带回实验室后在超净工作台中处理乳腺组织,组织块培养法培养原代乳腺上皮细胞。
高纯总RNA快速提取试剂盒购自北京百泰克生物技术有限公司;反转录试剂盒、荧光定量试剂盒、pMD19-T载体、RNAiso Plus均购自TaKaRa公司;琼脂糖凝胶回收试剂盒、大肠杆菌DH5α感受态细胞均购自天根生化科技(北京)有限公司;氨苄青霉素溶液(AMP)购自Solarbio公司。
1.2 CSN1S1基因CDS区克隆
1.2.1 引物设计及合成 根据GenBank中CSN1S1基因变体1的CDS区序列(登录号:XM_018049127.1),利用Primer Premier 5.0软件设计CSN1S1基因CDS区扩增引物:F:5′-CCCAAGC-TTATGAAACTTCTCATCCTTACCTGTCTT-3′;R:5′-CCCTCGAGTCACCACAGTGGCATAGTA-GTCTTTC-3′,预计扩增产物大小为645 bp。引物由生工生物工程(上海)股份有限公司合成。
1.2.2 RNA提取 按照总RNA快速提取试剂盒说明书从乳腺上皮细胞及奶山羊肝脏、脾脏、乳腺、肾脏、子宫、输卵管中提取总RNA,用核酸分光光度仪检测RNA的纯度和浓度。
1.2.3 PCR扩增及克隆 根据反转录试剂盒说明书将RNA反转成cDNA,反转录体系20 μL:Reaction Solution from Step 1 10 μL,5×Prime Script Buffer 2(for Real-time) 4 μL,Prime Script RT Enzyme Mix 1 1 μL,RT Prime Mix 1 μL,DEPC水4 μL。反转录反应条件:37 ℃ 15 min,85 ℃ 5 s,4 ℃保存。
以反转录得到的cDNA作为模板进行PCR扩增。 PCR反应体系20 μL:上、下游引物(10 μmol/L)各1 μL,模板(50 μmol/L)2 μL,Primer Star MAX酶10 μL,DNase-free ddH2O 6 μL。 PCR反应条件:94 ℃预变性3 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,共35个循环;72 ℃延伸5 min;4 ℃保存。
采用1.0%琼脂糖凝胶电泳对PCR产物进行检测,切胶回收正确条带,与pMD19-T载体连接。连接体系10 μL:胶回收产物(DNA片段)3 μL,Solution Ⅰ 5 μL,pMD19-T载体1 μL,DEPC水1 μL。16 ℃过夜连接。连接产物转化大肠杆菌DH5α感受态细胞,取100 μL菌液涂布含1% Amp的固体LB培养基,37 ℃培养12 h后挑取单克隆菌落,摇床4~5 h后进行菌液PCR鉴定,取阳性单克隆菌液送生工生物工程(上海)股份有限公司测序。
1.3 生物信息学分析
通过NCBI中ORF Finder在线软件(https:∥www.ncbi.nlm.nih.gov/orffinder/)鉴定开放阅读框;用ProtParam在线软件(https:∥www.expasy.org/resources/protparam)分析CSN1S1基因野生型和突变型编码蛋白的基本理化性质和结构特征;采用NetPhos在线软件(http:∥www.dabi.temple.edu/disphos/)进行蛋白磷酸化位点预测;通过TMHMM Server v.2.0在线软件(http:∥www.cbs.dtu.dk/services/TMHMM/)进行蛋白跨膜结构预测;利用SingalP 4.1 Server在线软件(http:∥www.cbs.dtu.dk/services/SignalP/)进行蛋白信号肽预测;利用YLoc在线软件(https:∥kohlbacherlab.org/Software/)进行蛋白亚细胞定位分析;利用NPS(https:∥npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)和Phyre2(http:∥bioinf.cs.ucl.ac.uk./psipred/)在线软件分别预测蛋白的二级结构和三级结构。
1.4 CSN1S1基因组织表达谱分析
将关中奶山羊肝脏、脾脏、乳腺、肾脏、子宫、输卵管6个组织的总RNA反转录为cDNA,通过实时荧光定量PCR检测其相对表达量,引物信息见表1,引物均由生工生物工程(上海)股份有限公司合成。 PCR反应体系25 μL:SYBR Premix ExTaqⅡ (2×) 12.5 μL,上、下游引物(10 μmol/L)各1 μL,cDNA模板2 μL,ddH2O 8.5 μL。PCR反应程序:95 ℃ 10 min;95 ℃ 15 s,60 ℃ 30 s,共40个循环。每个样品做3个重复,各表达量采用2-ΔΔCt法计算。

表1 引物信息

续表
1.5 统计分析
利用SPSS 25.0软件分析试验数据,通过独立样本t检验分析结果的差异性,P<0.05表示差异显著。
2 结 果
2.1 CSN1S1基因2种新序列
PCR产物采用1.0%琼脂糖凝胶电泳检测,结果显示,目的条带清晰,条带大小分别为645和639 bp(图1),分别命名为CSN1S1-1和CSN1S1-2。测序结果显示,关中奶山羊乳腺上皮细胞中存在CSN1S1-1和CSN1S1-2两种突变形态,其中CSN1S1-1在92 bp处T突变成C,108、274 bp处C突变成G;CSN1S1-2除在上述位点发生突变外,还在242 bp处缺失C碱基,249-253 bp处缺失TGAGG序列(表2)。
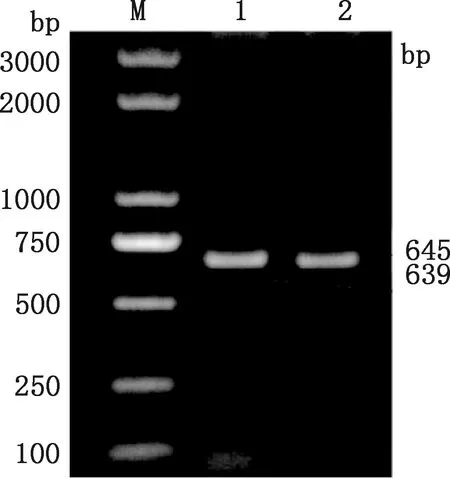
M,Direct-load StarMarker D2000 Plus;1,CSN1S1-2基因PCR扩增产物;2,CSN1S1-1基因PCR扩增产物M,Direct-load StarMarker D2000 Plus;1,PCR amplification product of CSN1S1-2 gene;2,PCR amplification product of CSN1S1-1 gene图1 CSN1S1-1/-2基因PCR扩增结果Fig.1 PCR amplification results of CSN1S1-1/-2 genes
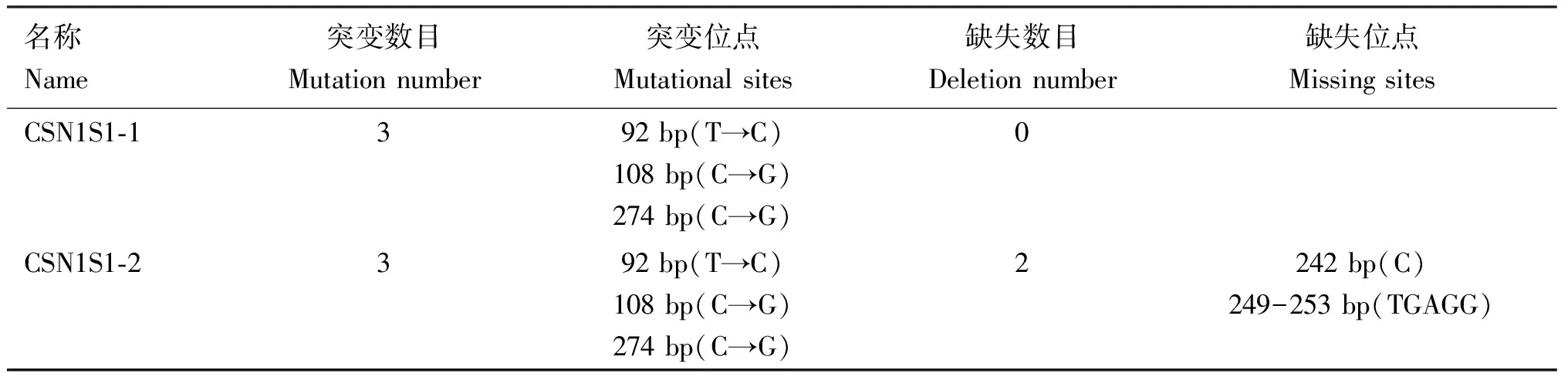
表2 CSN1S1-1/-2基因CDS区序列突变及缺失
2.2 生物信息学分析
2.2.1 蛋白理化性质分析 蛋白理化性质分析结果显示,CSN1S1和CSN1S1-1均含有214个氨基酸,相对分子质量分别为24 276.59和24 261.53,CSN1S1-2含有212个氨基酸,相对分子质量为24 102.45;CSN1S1和扩增的2种突变形态均为酸性蛋白,且不稳定系数均>40,为不稳定蛋白,具体见表3。BLAST比对结果显示,CSN1S1野生型与CSN1S1-1蛋白相似性为99.07%,与CSN1S1-2蛋白相似性为97.20%。与CSN1S1相比,CSN1S1-1中3个碱基的突变导致第31位亮氨酸变成脯氨酸,第92位谷氨酰胺变成谷氨酸;CSN1S1-2中除上述氨基酸改变之外,还出现了第81、82位2个丝氨酸缺失,第83位丝氨酸变成半胱氨酸,第84位谷氨酸变成谷氨酰胺。3种蛋白中氨基酸含量占比相对较多的均为亮氨酸和谷氨酸,CSN1S1中占比分别为10.7%和8.9%,CSN1S1-1中占比为10.3%和9.3%,CSN1S1-2中占比分别为10.4%和9.0%;氨基酸含量占比较少的均为色氨酸和半胱氨酸,其中色氨酸占比均为0.9%(表4)。

表3 CSN1S1及CSN1S1-1/-2蛋白理化性质对比
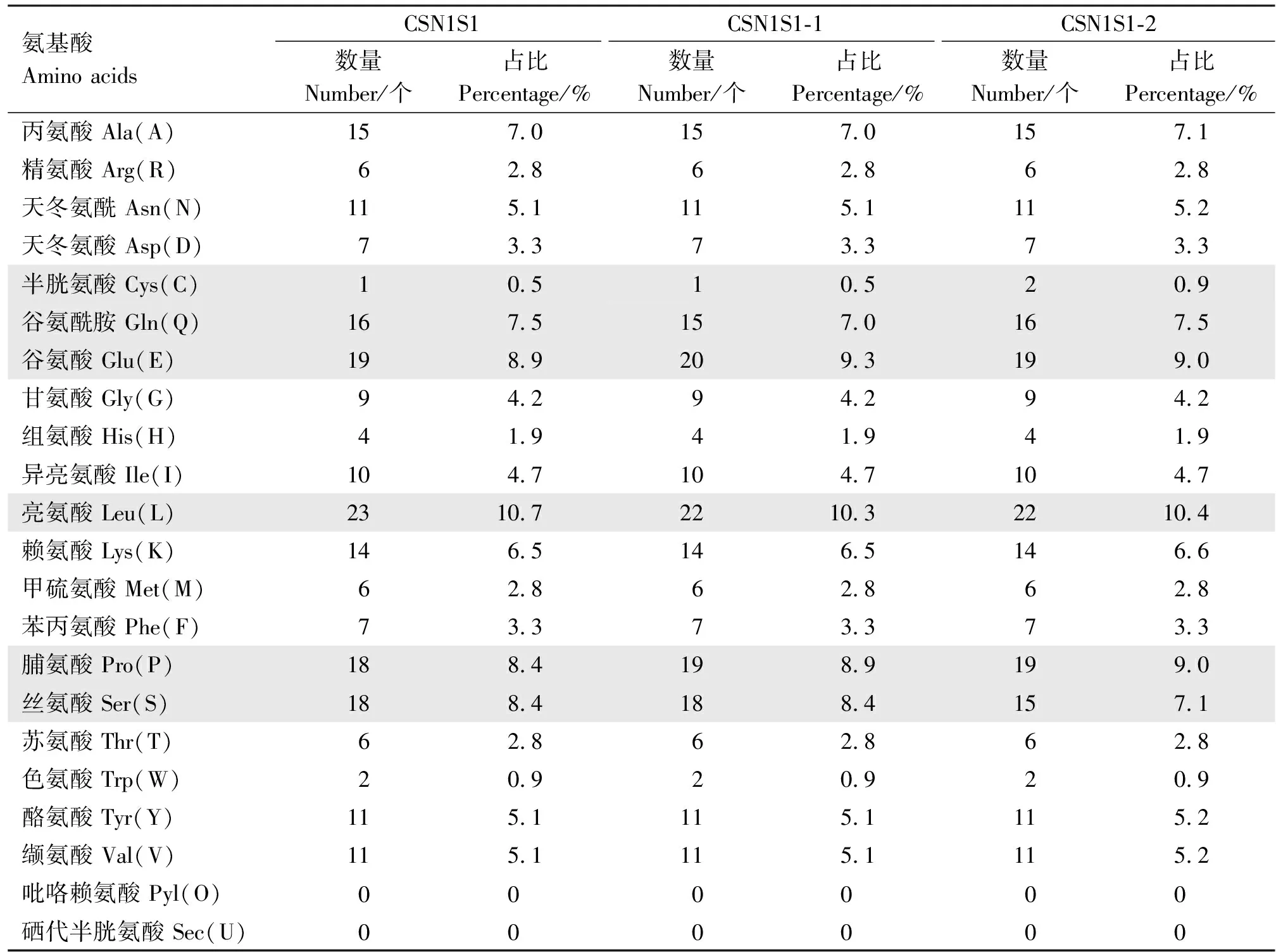
表4 CSN1S1及CSN1S1-1/-2蛋白的氨基酸组成
2.2.2 磷酸化位点预测 通过DISPHOS 1.3软件分析3种蛋白的磷酸化位点分布情况,结果见图2。由图2可知,CSN1S1和CSN1S1-1蛋白共有15个丝氨酸磷酸化位点、3个苏氨酸磷酸化位点和4个酪氨酸磷酸化位点;CSN1S1-1蛋白共有16个丝氨酸磷酸化位点、3个苏氨酸磷酸化位点和4个酪氨酸磷酸化位点;CSN1S1-2由于碱基的缺失与CSN1S1相比少了3个丝氨酸磷酸化位点。3种蛋白中苏氨酸和酪氨酸的数量虽然一致,但由于突变其分布位点却有所差异。
2.2.3 亚细胞定位分析 根据YLoc在线软件预测发现,CSN1S1及CSN1S1-1/-2蛋白均在细胞外分布(表5)。
2.2.4 二级结构和三级结构预测 经NPS和Phyre2分析发现,CSN1S1蛋白二级结构中α-螺旋、延伸链和无规则卷曲占比分别为42.99%、8.88%和48.13%;CSN1S1-1中占比分别为40.19%、7.48%和52.34%;CSN1S1-2中占比分别为40.09%、8.49%和51.42%(图3)。三级结构预测结果与二级结构一致(图4)。
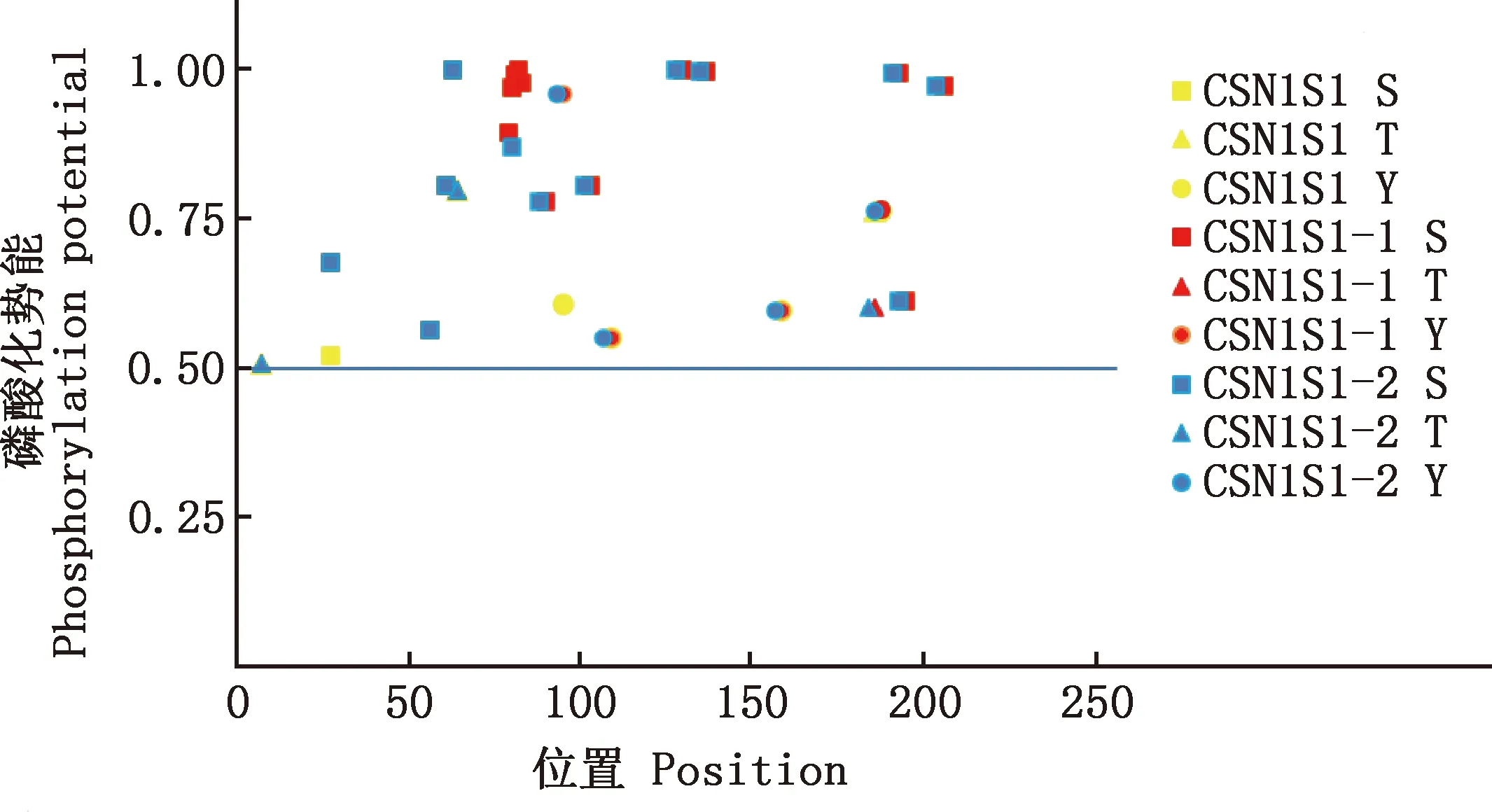
正方形,丝氨酸磷酸化位点;三角形,苏氨酸磷酸化位点;圆形,酪氨酸磷酸化位点Square,Ser phosphorylation site;Triangle,Thr phosphorylation site;Round,Tyr phosphorylation site图2 CSN1S1及CSN1S1-1/-2蛋白的磷酸化位点预测Fig.2 Phosphorylation site prediction of CSN1S1 and CSN1S1-1/-2 proteins

表5 CSN1S1及CSN1S1-1/-2蛋白亚细胞定位分布
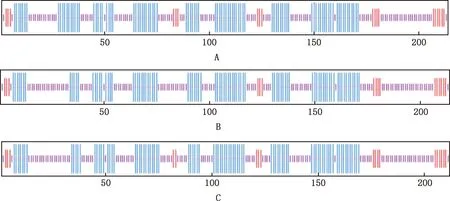
长竖线,α-螺旋;中竖线,延伸链;短竖线,无规则卷曲Long vertical line,Alpha helix;Middle vertical line,Extended chain;Short vertical line,Random coil图3 CSN1S1(A)、CSN1S1-1(B)和CSN1S1-2(C)蛋白二级结构预测Fig.3 Secondary structure prediction of CSN1S1 (A),CSN1S1-1 (B) and CSN1S1-2 (C) proteins
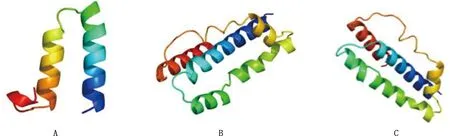
图4 CSN1S1(A)、CSN1S1-1(B)和CSN1S1-2(C)蛋白三级结构预测Fig.4 Tertiary structure prediction of CSN1S1 (A),CSN1S1-1 (B) and CSN1S1-2 (C) proteins
2.3 组织表达谱分析
由图5可知,CSN1S1-1/-2基因在关中奶山羊各组织中的相对表达趋势基本相同,子宫中CSN1S1-2基因表达量显著高于CSN1S1-1基因(P<0.05),2种突变形态均在乳腺中的表达量最高,其次是输卵管和子宫,而肝脏、肾脏和脾脏中表达量均较低。

*,差异显著(P<0.05)*,Significant difference (P<0.05)图5 CSN1S1-1/-2基因在关中奶山羊各组织中的相对表达量Fig.5 Relative expression of CSN1S1-1/-2 genes in various tissues of Guanzhong dairy goats
3 讨 论
3.1 CSN1S1基因及CSN1S1-1/-2 2种突变形态
CSN1S1编码αs1-酪蛋白基因,与CSN2、CSN1S2和CSN3构成一个基因座,聚集在6号染色体300 kb内[8],以上4种酪蛋白基因的遗传变异可以影响羊乳乳蛋白产量、乳成分、奶酪加工特性、人体营养消化率和耐受性等[5]。这4个基因编码的酪蛋白在山羊中有很高的遗传变异性[9],目前在山羊、奶牛、绵羊、骆驼和马身上已经发现了几十个酪蛋白等位基因,这些序列变异与基因表达和牛奶蛋白含量的改变有关[10]。Luigi-Sierra等[11]分析了来自非洲、欧洲、南亚、东亚和西亚的伊朗绵羊和家羊的全基因组序列,鉴定出CSN1S1的248个SNPs、CSN1S2的268个SNPs、CSN2的146个SNPs和CSN3的112个SNPs,并发现4个家羊群体共享44.2%~57.4%的蛋白多态性位点。Zhang等[12]研究表明,CSN1S1基因是调节哺乳动物泌乳性能的主要基因之一,也与机体发育有关,同时提出CSN1S1基因11 bp的插入或缺失对山羊泌乳性能和体测性状存在潜在影响。本研究通过PCR扩增并克隆关中奶山羊中普遍存在的CSN1S1-1和CSN1S1-2 2个CSN1S1基因的突变形态,比对发现,2个突变形态均发生了3个碱基的转换和颠换,CSN1S1-2还存在2处共6个碱基的缺失。推测CSN1S1-1和CSN1S1-2中由于突变导致的氨基酸种类和数量的变化可能影响奶山羊乳中蛋白成分。
3.2 CSN1S1-1/-2生物信息学分析
研究表明,细胞中蛋白质的结合、生物活性及蛋白质和其他生物分子的相互作用等功能都可以由蛋白质的磷酸化-去磷酸化来调控[13],且每个组织内部都有专门的、相互关联的磷酸化网络,许多蛋白质由磷酸化直接调控[14]。酪蛋白的磷酸化通常发生在丝氨酸、苏氨酸和酪氨酸的侧链,由磷酸激酶催化[15]。本研究发现,CSN1S1-1在第90位氨基酸残基处多了1个丝氨酸磷酸化位点,而CSN1S1-2由于碱基的缺失反而缺少了3个丝氨酸磷酸化位点,且存在大范围的酸磷酸化位点前移,CSN1S1-1和CSN1S1-2丝氨酸磷酸化位点数量的变化预示着其具有调控CSN2表达的潜能,为CSN2合成调控的研究提供了新思路。
基因邻域(gene neighborhood)指基因组中共同表达的空间集群[16]。CSN1S1、CSN2、CSN3、CSN1S2等基因都表达酪蛋白,这些基因功能相关,但不具有同源性,构成一个基因邻域[17]。对蛋白质相互作用分析发现,本试验未直接找到关中奶山羊CSN1S1蛋白互作图,而绵羊和牛CSN1S1与CSN2、CSN3、CSN1S2、LALBA、PAEP、CYM、PRR12、SCAF1存在互作关系,推测关中奶山羊中也存在CSN1S1及其突变形态与以上蛋白的互作。Gilmanov等[18]研究发现,CSN1S1、CSN2、CSN3基因复杂型对奶牛产奶量具有显著影响。Song等[19]研究证实,奶山羊CSN1S1基因可以通过调节JAK2/STAT5a信号通路抑制CSN2启动子活性和CSN2合成。 此外,miR-204-5p通过CSN1S1/STAT5a信号轴增强了CSN2表达,并通过激活位于CSN2启动子中的STAT5响应元件促进了CSN2的转录,进一步确定了奶山羊中CSN1S1与CSN2的互作关系[20]。在幼龄反刍动物中,凝乳酶(CYM)可以通过水解CSN2产生1个抑制金黄色葡萄球菌、肺炎双球菌的小肽[21-22],由此推测,CYM与CSN1S1基因可能存在潜在互作关系。目前对于奶山羊中CSN1S1基因与其他基因互作的研究还较为缺乏,CSN1S1-1和CSN1S1-2的功能也有待进一步挖掘。
3.3 CSN1S1基因组织表达谱分析
酪蛋白是主要的乳蛋白,可以改善肠道钙吸收,在营养方面具有进化上的保守作用。酪蛋白基因的序列变化直接影响动物的乳成分,酪蛋白基因的调控元件可用于在转基因动物乳汁中直接表达所需蛋白成分[23]。研究发现,CSN1S1可使单核细胞分化向巨噬细胞样表型倾斜[24],还可通过TLR4和炎症小体通路以磷酸化依赖的方式发挥促炎作用[25],且在原代人单核细胞中可检测到CSN1S1基因mRNA表达[26]。表明CSN1S1基因不仅能表达乳蛋白,还可能在其他组织中发挥免疫调节作用。本研究构建的关中奶山羊组织表达谱中CSN1S1-1和CSN1S1-2在乳腺中表达量最高,子宫和输卵管中也有表达,且子宫中CSN1S1-2的表达量显著高于CSN1S1-1的表达,提示CSN1S1-2可能在子宫中发挥其他功能,其具体功能还有待进一步探索。
4 结 论
本研究扩增出2种(CSN1S1-1、CSN1S1-2)与CSN1S1蛋白相似性均达到97%以上的突变形态,其中CSN1S1-1存在3个碱基突变,在蛋白结构上与CSN1S1已有明显区别,而CSN1S1-2中6个碱基的缺失是导致氨基酸改变、磷酸化位点缺失与移位的直接原因。CSN1S1-1和CSN1S1-2均在关山奶山羊乳腺、子宫和输卵管中高表达,CSN1S1-2在关中奶山羊子宫中表达量显著高于CSN1S1-1。
