肿瘤免疫治疗中新生抗原预测与筛选技术研究进展
2022-01-07顾芳玲姚文兵田浤
顾芳玲,姚文兵,田浤
(中国药科大学生命科学与技术学院,江苏 南京 211198)
肿瘤新生抗原是由体细胞突变产生的能被特异性T细胞识别的多肽,是一种肿瘤特异性抗原。由于肿瘤新生抗原不在胸腺中表达,不受中枢耐受性的影响,与非突变的肿瘤相关抗原相比具有更强的免疫原性[1];同时,正常组织细胞不表达肿瘤新生抗原,故靶向肿瘤新生抗原的免疫治疗不会引起对非肿瘤组织的脱靶损伤,具有更好的安全性[2]。因此,从免疫学角度而言,肿瘤新生抗原是肿瘤免疫治疗的理想靶点。靶向肿瘤新生抗原的疫苗、T细胞受体嵌合T细胞疗法和肿瘤浸润淋巴细胞疗法均已进入了临床研究阶段。准确快速地鉴定肿瘤新生抗原对于成功的免疫治疗至关重要,也是目前个性化免疫治疗中的一个难点。本文对肿瘤新生抗原的各种来源、基于高通量测序数据进行肿瘤新生抗原预测的算法以及肿瘤新生抗原筛选和鉴定方法进行综述,以期为靶向肿瘤新生抗原的免疫治疗提供参考。
1 肿瘤新生抗原的来源
1988年,de Plaen等[3]研究团队发现,来自小鼠P815肿瘤细胞经诱变处理产生的突变肽可被T细胞识别。随后一系列研究证明,肿瘤体细胞突变也能产生可被T细胞识别的突变肽,证实了肿瘤新生抗原的存在[4-6]。自2012年始,高通量测序技术被广泛应用,进一步证明了肿瘤突变可以从各种来源获得,且来源于这些突变的新生抗原已被证明能够引起 CD8+T或CD4+T淋巴细胞的有效应答[7-9]。
根据主要组织相容性复合体(major histocompatibility complex,MHC)的限制性,可以将肿瘤新生抗原分为MHCⅠ类和MHCⅡ类2种。经MHCⅠ类分子提呈的表位被称为CD8+T细胞表位。抗原提呈细胞表面的MHC-肽复合物与T细胞表面受体结合从而激活了T细胞。被激活的细胞毒性T细胞可以在体内直接杀伤肿瘤细胞,是最受研究者关注的肿瘤新生抗原类型。然而,深入研究发现,单一的MHCⅠ类表位难以产生持续的抗肿瘤作用,越来越多的证据表明CD4+T细胞表位(即MHCⅡ类表位)在抗肿瘤方面也起到积极的作用[10-12]。有研究人员在3种不同的小鼠肿瘤模型中发现,大多数具备免疫原性的肿瘤新生抗原是由CD4+T细胞识别的[13]。
肿瘤新生抗原的来源可以分为5类:1)基因组变异,包括单核苷酸变异(single-nucleotide variations,SNVs)和插入/缺失突变(insertions and deletions,Indels);2)基因融合;3)转录组选择性剪接;4)RNA 编辑;5)转录组非编码区突变。
1.1 单核苷酸变异和插入/缺失突变
SNVs是肿瘤中最常见的基因组水平突变类型,非同义突变编码蛋白产生的变异肽可能通过 MHC分子呈递到肿瘤细胞表面,形成肿瘤新生抗原。近来研究表明,基于SNVs的新生抗原负荷与经免疫检查点抑制剂治疗患者的临床获益具有相关性。Charoentong等[14]基于肿瘤基因组图谱数据库(The Cancer Genome Atlas,TCGA)对20种实体肿瘤的免疫微环境景观和肿瘤新生抗原进行了研究,并创建了肿瘤免疫组图谱。
处于外显子区域的Indels突变会引起移码,从而形成一个新的开放阅读框,并可能产生大量与自身高度不同的新生抗原。尽管Indels的发生频率比SNVs低,但对来自TCGA的19种癌症类型的5 777个实体瘤的大规模分析显示,Indels产生的新生抗原比SNVs衍生的新生抗原更具免疫原性[8]。同样,研究发现,基于Indels的新生抗原负荷也与黑素瘤患者对免疫检查点抑制剂治疗应答存在显著相关性[8]。
1.2 基因融合
结构变异是大多数肿瘤基因组的标志,包括倒位、缺失、重复和易位。其中易位导致的基因融合会改变阅读框架,从而形成肿瘤新生抗原。Yang等[9]研究证明了基因融合是免疫原性肿瘤新生抗原的重要来源,并报道了DEK-AFF2和NFIB-MYB的基因融合,可以被MHCⅠ类复合物呈递,从而引起有效的CD8+T淋巴细胞免疫反应。类似的,最近研究人员证实,CBFB-MYH11融合基因产生的新生抗原能够被递呈于肿瘤细胞表面,并激活T细胞识别和杀死肿瘤细胞,可见靶向性新生抗原的治疗在低突变频率的癌症中也具有临床意义[15]。
1.3 转录组选择性剪切
选择性剪接是细胞中维持所产生蛋白多样性的一种常见机制。通过选择性剪接,基因的特定外显子可能被排除在成熟的mRNA之外或保留特定内含子,导致基因组编码的蛋白质多样性增加。剪接因子(如U2AF1和SF3B1)发生突变可能导致肿瘤中剪接事件的广泛改变。Kahles等[16]对TCGA数据库中8 705例患者的泛癌分析表明,选择性剪接也是肿瘤新生抗原的重要来源。Smart等[17]研究证实,保留内含子也是肿瘤新生抗原的来源之一。
1.4 RNA编辑
RNA编辑是一种常见的转录后修饰,可以改变RNA序列中的特定核苷酸,也可以导致非同义替换,从而产生新的蛋白质。A ~Ⅰ的RNA 编辑是人类最常见的RNA编辑类型,它会提高包括癌症在内的多种疾病的蛋白质组的多样性[18]。由A ~Ⅰ的RNA编辑产生的多肽由MHCⅠ类分子自然呈递并引发CD8+T细胞反应,表明RNA编辑扩展了人类白细胞抗原(human leukocyte antigen,HLA)呈递的自身抗原类别,并且这些抗原可被免疫系统识别[19]。
1.5 转录组非编码区突变
99%的癌症突变发生在非编码区域,而外显子组仅占人类基因组的2%;然而,高达75%的基因组可以被转录并潜在地翻译成蛋白质。最近的研究表明,许多所谓的非编码区实际上也具有编码蛋白质的能力,可以产生许多由MHCⅠ类分子递呈的肽。Laumont等[20]用液相色谱串联质谱和RNA测序对白血病和肺癌患者的非编码序列进行了研究,在非编码区发现了大量的突变和未突变的抗原,其中一些已被确定为肿瘤浸润淋巴细胞的靶标。值得注意的是,来源于非编码区的MHCⅡ类分子限制性新生抗原尚未有文献报道。
2 肿瘤新生抗原的预测
随着高通量测序技术的发展,研究人员可以分析肿瘤的基因组与转录组数据,并借助计算机预测潜在的肿瘤新生抗原,这是个性化免疫治疗中极为关键的环节。肿瘤新生抗原预测的流程如图1所示,这一流程中的每一步骤均涉及到生物信息学工具的使用和需要权衡的因素。
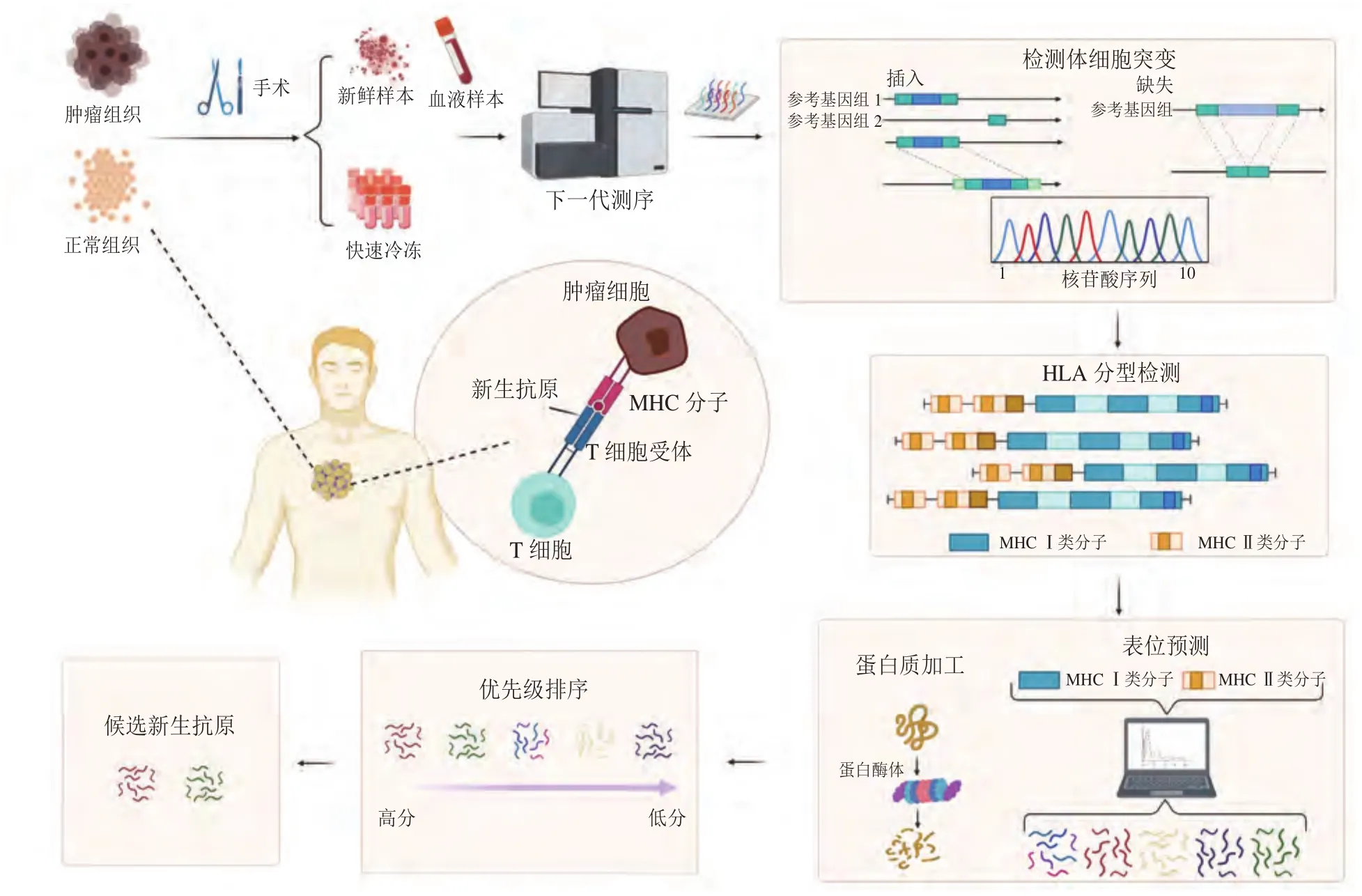
图1 肿瘤新生抗原预测流程示意图Figure 1 Schematic diagram of tumor neoantigen prediction
2.1 突变序列的识别与筛选
新生抗原的预测需要获得肿瘤细胞与非肿瘤组织的基因组和转录组信息。可用于测序的样本类型包括新鲜冷冻的组织、cDNA、血液等。高异质性的瘤内组织需要收集多个活检部位进行测序[21]。全外显子测序因其高效性、高覆盖的优势,是用于预测肿瘤新生抗原的标准测序方法,但也可以使用RNA测序方法。RNA测序包括了整个转录组RNA测序,因此可以用于分析RNA编辑、转录后修饰等产生的突变肽[22]。
已有研究证明, SNVs和Indels衍生的突变蛋白可以同时被 MHCⅠ类和MHCⅡ类分子呈递,并引起足够的 CD8+T和 CD4+T细胞免疫应答。这2种类型的基因组变体是基因组水平上肿瘤新生抗原的主要来源,目前针对这2种变异已开发了大量的分析算法。
进行SNVs分析需要解决3个问题:低频突变的检测、区分种系变异和消除测序伪迹。MuTect2和Strelka在检测低等位基因分数的SNV时具有很高的灵敏度,从而可进行精确的亚克隆变异检测。VarScan2和SomaticSniper需要更高的等位基因分数来识别变异体,但在区分种系变异和肿瘤变异中可以提高性能。同时运行多种算法可以提高检测精度,例如,Callari等[23]通过将来自多个比对管线的结果取交集,然后合并来自MuTect2和Strelka的相交结果,在不增加假阳性率的情况下使检测灵敏度提高了17.1%。
2.2 HLA分型的计算与分析
由于HLA基因广泛的个体化,准确的HLA单倍分型对于准确预测新生抗原至关重要。该过程的金标准是利用序列特异性聚合酶链式反应(PCR)扩增进行临床HLA分型。然而,临床分型可能费时、费力且价格昂贵,所以一个常见的替代方法是使用病人的测序数据集计算 HLA 分型。
与临床分型结果相比,Ⅰ类HLA分型算法的预测准确率可达99%[24-25]。其中,OptiType、Polysolver和PHLAT工具目前报告的准确率最高。与Ⅰ类HLA 分型算法相比,Ⅱ类HLA分型算法仍需进一步开发以提高其预测精度。最近,Orenbuch等[26]发布了arcasHLA工具,其对Ⅰ类基因的双字段分辨准确率为100%,对Ⅱ类基因的准确率超过99.7%。
除了对HLA分型进行计算之外,HLA变异也是肿瘤新生抗原预测中值得关注的问题。HLA基因拷贝数变异导致的杂合性缺失会致使肿瘤免疫逃逸[27]。针对肿瘤中HLA突变的分析显示,HLA突变可能会导致功能丧失,从而影响抗原的递呈[28]。抗原递呈相关的其他蛋白组分出现突变也会影响抗原递呈。在肺癌和结肠癌中,B2M的突变或双等位基因丢失会导致缺乏Ⅰ类HLA[29-30]。因此,在肿瘤新生抗原的预测和筛选中,需要优先考虑能结合未突变HLA基因的新生抗原。
2.3 突变肽与MHC分子亲和力的预测
通常MHC分子与肽的结合被认为是抗原呈递过程中最具选择性的步骤。目前MHCⅠ类分子与肽的亲和力预测已能达到较高的精度,大多数等位基因的典型曲线下面积分数大于 0.90。这些预测算法已成为传染病、变态反应、自身免疫、肿瘤免疫和疫苗开发领域的标准工具。
2.3.1 突变肽与MHCⅠ类分子的亲和力预测 NetMHC 4.0是基于人工神经网络的算法所开发,NetMHCpan 4.0在其训练集中包括了质谱鉴定的MHC配体[31]。NetMHCpan 4.0模型提供2个输出,一个是预测的肽/MHC结合的亲和力,另一个是在质谱-HLA配体洗脱实验中鉴定的可能性,其4.1版本在独立软件包的基础上提供了简易的网页模式。MHCflurry是采用类似的机器学习算法建立的开源预测工具[32]。Boehm等[33]用质谱鉴定的肽配体作为训练数据,建立了基于随机森林算法MHCⅠ类配基预测算法。尽管这些工具并未显示出明显优于NetMHCpan 4.0的准确性,但它们的优点是开源,更容易集成到生物信息处理的管道中。
一些研究已表明,肽/MHC 复合物的半衰期比肽与MHC 亲和力更能预测其免疫原性[34]。虽然有netMHCstab 等工具可以用于预测pMHC稳定性,但因为亲和力预测工具具有更大的训练集的优势,大多数研究者还是采用了亲和力算法。然而,最近报道的肽/MHC稳定性的高通量分析方法可能为通过肽/MHC 复合物稳定性预测肿瘤新生抗原创造了机会[35]。
2.3.2 突变肽与MHCⅡ类分子的亲和力预测 尽管已有研究表明,Ⅱ类限制性新生抗原反应对某些抗肿瘤反应是至关重要的,但目前大多数预测新生抗原的管道并不根据MHCⅡ类分子的亲和力预测结果来筛选肿瘤新生抗原。一方面是由于缺乏训练数据,IEDB数据库中MHCⅡ类配体的数据远远低于MHCⅠ类配体;另一方面,由于MHCⅡ的肽结合槽在两端是开放的,允许长度可变的长肽结合,导致对MHCⅡ类配体建模更加困难。
最常用的MHCⅡ配体预测器是NetMHC套件中的NetMHCIIpan[36]。NetMHCIIpan使用与 NetMHCpan相似的神经网络结构,但在训练过程中使用迭代方法预测训练期间每个肽的结合核心。使用NetMHCIIpan 的分析与使用 NetMHCpan的分析非常相似,结果报告为每个肽和等位基因的亲和力值。NetMHCIIpan 4.0的训练数据集已经覆盖了116 个不同的 MHCⅡ类分子。MixMHC2pred工具使用多等位基因质谱数据集训练Ⅱ类预测因子,通过基序聚类分析质谱数据,然后根据具有共同等位基因的个体之间的共享聚类模式将聚类与等位基因相关联[37]。NetMHC 套件的开发人员也采用了这种方法,将单等位基因和多等位基因的质谱训练数据混合在一起,并使用神经网络进行基序识别[38]。
2.3.3 突变肽的加工预测 目前,肿瘤新生抗原的研究重点大部分集中在预测病人的MHC分子和特定突变肽的亲和力上。然而,即使一个肽具有很强的MHC 结合能力,如果上游处理阻止了该肽的实际负载,那么这个预测可能是毫无意义的。肽的加工,特别是免疫蛋白酶体加工和肽裂解,是新生抗原预测中必须考虑的因素。
对于MHCⅠ类和 MHCⅡ类抗原递呈途径来说,在肽和MHC分子相互作用之前的一个重要步骤是蛋白质通过免疫蛋白酶体降解成肽。现在可以利用NetChop20S、NetChopCterm、ProteaSMM等工具来获得蛋白酶体的特异性,并预测不同蛋白酶靶向的蛋白裂解位点[39]。在该领域发展起来的算法通常是根据体外蛋白酶体消化数据或体内MHCⅠ和MHCⅡ配体洗脱数据进行训练。基于神经网络的预测方法 NetChop-3.1 Cterm 已被证明在预测体内蛋白质水解方面有较好的表现[40]。
2.4 候选抗原肽的优先级排序
对于最终候选抗原肽的选择,目前并没有非常公认的选择标准。候选抗原肽的选择需要综合考虑以下几点因素:首先,突变序列能否在肿瘤细胞内转录、被翻译成蛋白质且成功被转运加工进而呈递;其次,应该考虑突变肽与相应MHC分子的结合能力和稳定性,MHC分子自身的表达与突变情况,还需要考虑不同突变肽对MHC分子结合的竞争;最后,T细胞受体库是否足够多样、肽/MHC复合体和T细胞受体之间的结合能力也应该被纳入考量[41]。
2.5 集成的预测管道
由于在肿瘤新生抗原的生成、加工、结合和识别过程中涉及到许多因素,研究人员开发了许多生物信息学管道以汇集现有的工具,为不同的临床目的简化新生抗原识别过程。例如:预测对免疫检查点阻断疗法的反应、设计基于肽或载体的疫苗等。表1列出了一些常用的预测管道,这些管道将为肿瘤新生抗原预测提供一个全面的关键信息汇总。

表1 肿瘤新生抗原预测常用管道Table 1 Tumor neoantigen prediction and prioritization pipelines
pVACtools 是一套集成的计算框架,可以为肿瘤新生抗原的预测提供端到端的解决方案[42]。pVACtools支持识别不同来源的突变肽,包括:点突变、移码插入和缺失以及基因融合。预测肽和MHC分子的结合是通过支持 MHCⅠ类和 MHCⅠ类结合算法集合在一个框架内完成的。通过整合不同的数据,包括突变等位基因的表达,肽结合的亲和力,以及确定一个突变是克隆还是亚克隆,可以对预测的多肽进行优先排序。这个工具还包括了一些支持新生抗原疫苗设计的模块。
MuPeXI 工作流程与pVACtools中的pVACseq模块相似[43]。MuPeXI 需要用户输入包含体细胞突变调用的文件、 HLA 类型列表和可选的基因表达谱。MuPeXI 输出结果是一个表格,其中列出了所有来自SNVs和Indels的肿瘤特异性突变肽以及全面的注释,包括HLA结合和与正常肽的相似性,这些肽将按照一个优先分值进行分类,以便粗略地预测免疫原性。MuPeXI工具的一个优势是其使用 NetMHCpan 4.0进行MHCⅠ类结合预测,使用户能够从结合亲和力和洗脱配体数据训练的预测器中受益。MuPeXI可以作为独立软件或网络服务器使用。
TIminer集成了一组生物信息学工具来分析单一样本的 RNA-seq 数据和体细胞 DNA 突变,包括:1)从高通量测序数据中分析 HLA 分型;2)利用突变数据和 HLA 类型预测肿瘤新生抗原;3)从转率组测序数据中分析鉴定肿瘤浸润免疫细胞;4)从表达数据中定量分析肿瘤免疫概况。TIminer的一个优势是能够处理原始 RNA测序数据并提取与新生抗原预测的信息[44]。
OpenVax新生抗原预测管道是从早期的Epidisco工作流程演变而来,后者更关注计算机集群并行性。OpenVax是流水线式的自动化读取处理和体细胞变体调用,支持SNVs和Indels突变体。OpenVax也是一个端到端的工作流程,从原始DNA和RNA FASTQ数据开始,生成包含突变的肽,将其列为疫苗包含的最终输出。目前,使用OpenVax必须向软件提供 HLA 类型。OpenVax管道是可配置的,允许用户指定使用哪个 MHCⅠ类结合预测器,以及 MHC结合亲和力和变异基因特异性表达的阈值。OpenVax管道的最终结果是一组按用户指定长度排列的合成长肽(synthetic long peptides,SLPs),优化后用于SLP疫苗。值得关注的是,目前有3个用于肿瘤新生抗原疫苗的Ⅰ期临床实验(NCT02721043、T03223103和 NCT03359239)使用的长肽疫苗是基于该OpenVax管道设计的[44]。
NeoFuse是一种从肿瘤RNA-seq数据中预测融合新生抗原的计算管道,可识别可能作为免疫治疗合适靶点的融合新生抗原[49]。NeoFuse主要包括五大模块,分别为HLA分型检测、预测融合肽、肽与MHC结合亲和力、基因表达水平量化和对候选融合肽排序。NeoFuse管道可以在隔离环境中运行,防止与托管环境中的其他程序发生冲突。
3 肿瘤新生抗原的筛选与验证
各种针对肿瘤新生抗原的免疫治疗策略的最终目的是引发和(或)扩大肿瘤特异性T细胞应答。由于疫苗的制造周期和患者病情发展的限制,很多针对肿瘤新生抗原的临床研究很难具备充分的时间对预测得到的候选肿瘤新生抗原进行免疫学性质的验证。然而,在一项关于患者对预测的肿瘤新表位疫苗接种反应的报告中,3名黑色素瘤患者分别接种了与MHC亲和力预测结果为IC50小于500 nmol · L-1的肽;在测试的21种肽中,只有9种诱导了T细胞应答[51]。此外,新近研究报道中,研究人员从B16F10黑色素瘤细胞株中鉴定出一个“抑制性”肿瘤新生抗原表位,该表位可促进荷瘤小鼠体内的肿瘤生长[52]。由此可见,肿瘤新生抗原表位的验证需要进行更多的研究。
3.1 常规免疫分析方法
基于已被激活的T细胞接受相同抗原再刺激时可以释放大量的干扰素-γ(IFN-γ)的作用机制,IFN-γ ELISPOT是目前最为常用的验证各种抗原肽免疫原性的分析方法。该方法普遍用于验证患者经过免疫治疗,对各种预测的肿瘤新生抗原的应答情况[53-54]。MHC多聚体技术灵敏度较高,是检测抗原特异性T细胞的常规方法,能检测到所含比例低于0.1%的新生抗原特异性T细胞。该技术被广泛用于检测特定新生抗原特异性T细胞,以评价免疫治疗效果。该技术也可用于筛选具有免疫原性的新生抗原[55-56]。
3.2 基于TCR谱的分析方法
肿瘤特异性T细胞应答也可通过表征患者在免疫治疗前后T细胞受体(T cell receptor,TCR)库的变化来进行分析。目前关于TCR谱的研究主要集中在代表独特T细胞克隆的CDR3序列的鉴定和表征上,这一过程称为TCR克隆分型,已被用于鉴定个体化癌症疫苗接种后或检查点封锁治疗后对新生抗原的克隆性T细胞应答。研究发现,患者TCR库的大小和多样性与患者对免疫疗法的反应之间存在相关性[57]。从外周血或肿瘤浸润淋巴细胞中观察到的 TCR 谱系的克隆性和多样性的变化表明正在发生抗肿瘤T细胞应答。Lu 等[58]最近开发了一种单细胞转录组测序方法,通过用串联小基因转染或肽脉冲自体抗原递呈细胞培养肿瘤浸润性淋巴细胞来鉴定新生抗原特异性TCRs,然后利用单个新生抗原的实验验证数据来训练和改进当前的新生抗原优先策略。
3.3 基于患者记忆性T细胞的分析方法
有研究人员借助T细胞对肿瘤新生抗原具有特异性记忆的方法,建立了离体筛选肿瘤新生抗原的方法(见图2)。 该方法的第一步也是利用计算机预测肿瘤新生抗原,然后将全部候选新生抗原的基因片段重组到大肠埃希菌中表达;同时收集患者的CD4+T和CD8+T细胞作为后续产生记忆应答的细胞,将大肠埃希菌和体外诱导的树突状细胞共孵育之后,与T细胞亚群隔夜孵育;以非免疫原性蛋白产生的背景分泌作为对照,通过比较候选新生抗原刺激下细胞因子的分泌来检测新生抗原特异性记忆应答,从而筛选出真正的新生抗原。
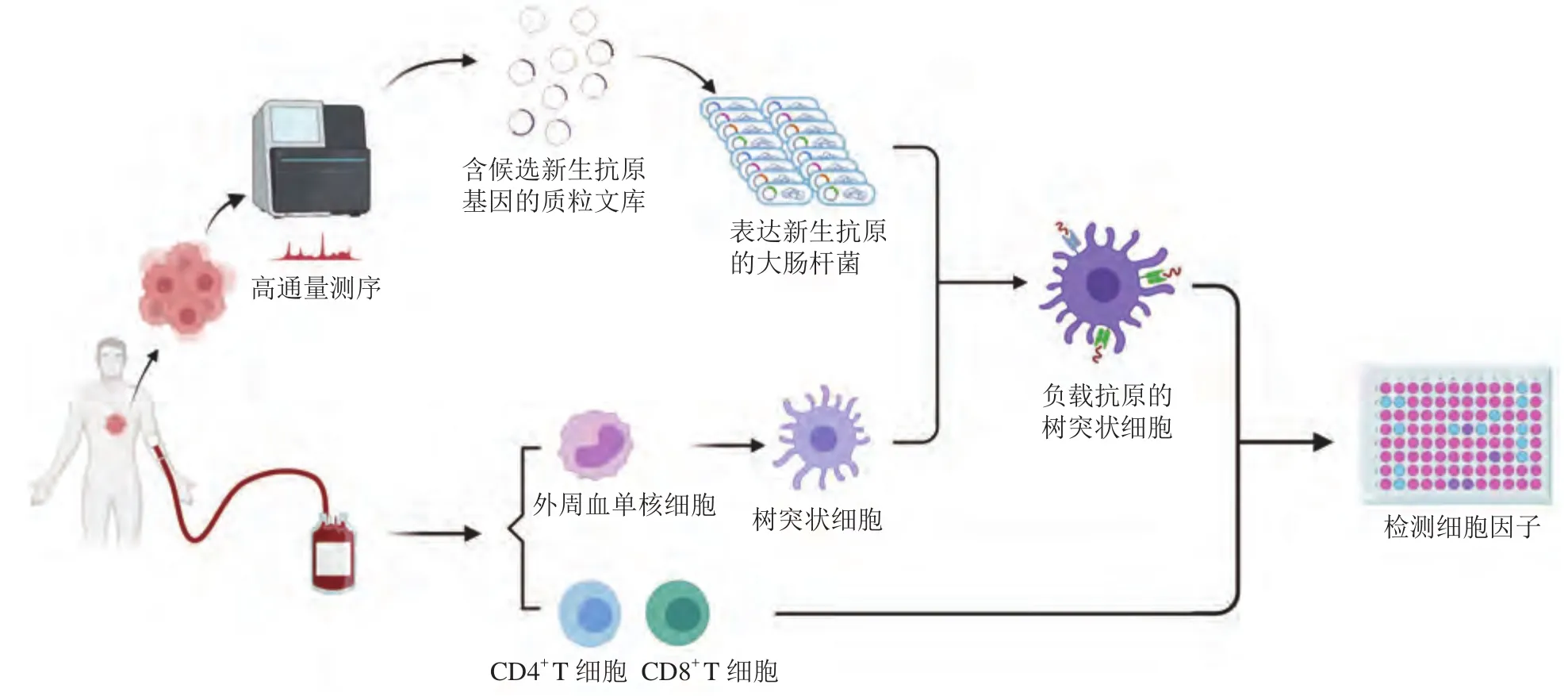
图2 基于患者记忆性T细胞的分析方法原理示意图Figure 2 Schematic diagram of analytical method based on patient memory T cells
这种基于患者记忆性T细胞的分析方法的优势是可以筛选到真正能够在患者体内激发免疫反应的肿瘤新生抗原。一项多中心Ⅰ/Ⅱa期在研临床试验(NCT03633110)的中期结果显示,患者接受疫苗免疫后对99%的抗原肽具有免疫反应,而依靠计算机预测获得的候选抗原,免疫患者仅有60%的抗原肽具有免疫反应[52]。
4 存在的问题与展望
高通量测序平台的出现,加上多种生物信息学算法层出不穷,为检测体细胞的遗传改变和肿瘤浸润淋巴细胞的表型提供了独特的机会,并利用这些研究结果进行新一代的癌症免疫治疗。尽管如此,在肿瘤新生抗原预测与筛选方面但仍有很大的改进空间。目前不断有各种肿瘤新生抗原的预测工具被开发出来,但一些有益的工具尚未被纳入分析工作流程。同时也存在由于缺乏有效预测工具,致使重要的因素没有被考虑的情况,例如SNVs 和 Indels以外的一些变异类型已被证实为新生抗原来源,但在目前的预测管道中对它们几乎没有支持。
Ⅱ类HLA分型算法的低精度也阻碍了Ⅱ类新生抗原的广泛预测。当Ⅱ类HLA分型临床数据可用时,应该使用它们代替管道中的计算HLA预测,以提高预测的可靠性。由于MHCⅡ类新生抗原的结合亲和性训练数据较少,且肽与MHC 结合复杂性增加,所以 MHCⅡ类新生抗原的算法也较少,限制了MHCⅡ类新生抗原表位的预测精度。通过目前的算法预测的肽库仍需实验的验证才可以确定哪些新生抗原会引发免疫反应,但实验的时间和经费成本较大,仍旧需要继续寻找确定新生抗原的指标。在肿瘤的免疫逃逸之下,新生抗原疫苗也面临着诱导特异性T细胞发挥作用有限的问题。
随着对肿瘤和免疫学的进一步探索,将会有更多的预测和筛选工具被开发出来,并纳入到临床前和临床研究中,对肿瘤新生抗原的预测与筛选将会更加精准,为肿瘤的个性化免疫治疗提供有效的支持。
