碳青霉烯耐药大肠埃希菌的基因组学特点分析*
2022-01-05曹小利张葵周辉郑洁周万青张之烽沈瀚南京大学附属鼓楼医院检验科南京210008
曹小利,张葵,周辉,郑洁,周万青,张之烽,沈瀚(南京大学附属鼓楼医院检验科,南京210008)
近年来,由于产超广谱β-内酰胺酶(extended-spectrum β-lactamases, ESBLs)和/或AmpC型β-内酰胺酶的产生,碳青霉烯类抗菌药物成为临床治疗多药耐药菌感染有效的抗菌药物[1]。碳青霉烯耐药大肠埃希菌(carbapenem-resistantEscherichiacoli, CREC)在世界范围内频繁出现,已成为威胁公众健康的主要病原菌。
目前已知,碳青霉烯酶的产生是肠杆科细菌对碳青霉烯类抗菌药物耐药的主要机制[2],而新德里金属β-内酰胺酶(NewDelhi metallo-β-lactamase, NDM)是大肠埃希菌中的主要碳青霉烯酶[2]。值得注意的是单菌可产多个β-内酰胺酶,尤其是碳青霉烯酶的共同产生,如NDM-1和肺炎克雷伯菌碳青霉烯酶2(KlebsiellaPneumoniaecarbapenemase-2, KPC-2)的共产生[3]。迄今为止,KPC-2和OXA-48在多种临床肠杆科细菌如肺炎克雷伯菌、阴沟肠杆菌和弗氏柠檬酸杆菌中的共存已被频繁报道[4]。这种多个碳青霉烯酶编码基因在单菌中的频繁共存,不仅会导致碳青霉烯耐药性迅速上升,也会导致发病率和死亡率增加[5],亟需关注。
中国碳青霉烯耐药肠杆科细菌监测网的报告和有关研究显示,各地区CREC感染率存在显著差异[6-7]。此外,多项研究表明,blaNDM通常位于IncX3质粒上[8],但关于这类菌株的毒力基因、血清分型和菌毛(Fim)分型的信息有限。本研究主要对CREC菌株的基因组流行病学进行研究,包括耐药基因、毒力因子、血清分型、Fim分型、质粒复制子和进化关系。此外,基于blaNDM和blaKPC的频繁共存,筛查该类细菌在南京鼓楼医院的分布,并分析了其播散特点。
1 材料和方法
1.1菌株来源 本研究的分离株包括两部分。(1)南京市6家医院2015年6—12月期间收集的11株非重复CREC分离株,进行基因组特征分析。其中,4株分离自尿液,3株分离自痰液,1株来自血液,1株来自胆汁,1株来自子宫颈分泌物,还有1株来源不明;1株分离自南京明基医院,2株分离自南京儿童医院,1株分离自南京溧水医院,4株分离自南京鼓楼医院,1株分离自南京金域医院,另外2株分别来自南京市妇幼保健院和南京市第一医院。(2)南京鼓楼医院2013—2017年间收集的43株连续非重复CREC菌,以确定共携带blaKPC-2和blaNDM的菌株。其中,分离时间为2013年的有4株,2014年10株,2015年2株,2016年11株,2017年16株; 18株来自中段尿,9株来自血,6株来自痰,3株来自分泌物,3株来自胆汁,2株来自腹水,2株来自脓液。
1.2仪器与试剂 Vitek 2.0细菌鉴定仪(法国生物梅里埃公司)、引物合成及测序(擎科生物公司)、PCR扩增仪(美国ABI公司)、脉冲场凝胶电泳仪及配套试剂(Bio-Rad公司)、叠氮钠和美罗培南(Sigma公司)、细菌基因组提取试剂盒DP705(北京天根公司)、NanoDrop 2000分光光度计(德国Thermo公司)、全基因组测序(广东美格基因公司)、CLC genomics workbench软件(苏州协云基因公司)、限制性内切酶XbaⅠ(德国ABI公司)、 BioNumerics软件(上海一贝公司)、肠杆科细菌药敏平板(温州康泰生物公司)、分光光度计(美国赛默飞世尔科技公司)。
1.3抗菌药物敏感性试验 采用微量肉汤稀释法测定11株CREC菌株对抗菌药物的敏感性。肠杆科细菌药敏平板中的抗菌药物包括厄他培南、亚胺培南、美罗培南、头孢吡肟、头孢他啶、头孢噻肟、头孢呋辛、头孢唑林、哌拉西林/他唑巴坦、阿米卡星、庆大霉素、呋喃妥因、甲氧苄啶和磺胺甲噁唑、氨曲南、哌拉西林、环丙沙星、左氧氟沙星、氨曲南/阿维巴坦、头孢他啶/阿维巴坦和替加环素,大肠埃希菌ATCC 25922为质量控制菌株。药敏结果根据美国临床和实验室标准协会(Clinical and Laboratory Standards Institute, CLSI)2020指南进行解释[9]。替加环素和黏菌素的药敏判读参考欧洲委员会的抗菌药物敏感性试验折点 (http://www.eucast.org/clinical_breakpoints)。CREC菌株定义为对亚胺培南耐药的菌株。
1.4基因组的DNA提取、测序、注释、递交 采用磁珠法通用型基因组DNA提取试剂盒(DP705)提取细菌基因组DNA后,使用分光光度计测定DNA浓度和纯度。用于全基因组测序DNA样品必须满足下列条件:浓度≥20 ng/μL,总量≥2 μg,吸光度值在1.8~2.0之间。提取合格的DNA 样本送广东美格基因科技有限公司测序。测序过程如下:经电泳检测合格的DNA样品用Covaris超声波破碎仪随机打断成长度约为350 bp的片段,再经末端修复、加A尾、加测序接头、纯化、PCR扩增等步骤完成文库制备;先使用Qubit 2.0进行初步定量,稀释文库至2 ng/μL,随后使用Agilent 2100对文库的插入片段进行检测,片段符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量,以保证文库质量。测序平台采用Illumina Hiseq PE150,建库类型为350 bp小片段文库,细菌重测序策略为350 bp文库≥10。原始下机数据过滤测序质量值低的reads(Qs≥20),保留高质量reads后,由CLC Genomics Workbenchv7.0.4进行配对末端读取的从头组装。然后将基因组提交至NCBI进行注释。
1.5基因组流行病学分析 用Resfinder v2.1(http://cge.cbs.dtu.dk/services/ResFinder-2.1/)和Virulence Finder 2.0 (https://cge.cbs.dtu.dk/services/VirulenceFinder/)鉴定抗菌药物的耐药基因和毒力因子;用多位点序列分型法(MLST) 2.0 (https://cge.cbs.dtu.dk/services/MLST/)进行菌株的序列分型(sequence type, ST);用Plasmid Finder 2.1(https://cge.cbs.dtu.dk/services/PlasmidFinder/)进行质粒复制子分型,用Serotype Finder 2.0(https://cge.cbs.dtu.dk/services/SerotypeFinder/)进行血清学分型,用 FimTyper 1.0(https://cge.cbs.dtu.dk/services/FimTyper/)进行Fim分型。
1.6系统发育树构建 利用细菌的核心基因(同一性>95%;覆盖率=100%)中检测到的单核苷酸多态性位点,构建11株CREC的核心基因组系统发育树[10]。使用raxml 8.2.8软件版本,采用一般时间可逆模型和100次引导计算最大似然树[11]。使用Interactive Tree Of Life (https://itol.embl.de) 产生进化树[12]。
1.7共携带blaNDM和blaKPC-2大肠埃希菌的筛选 用PCR方法检测blaKPC(KPC-Fm:5′-CGTCTAGTTCTGCTGTCTTG-3′;KPC-Rm:5′-CTTGTCATCCTTGTTAGGCG-3′)和blaNDM(NDM-F:5′-GGTTTGGCGATCTGGTTTTC-3′;NDM-R:5′-CGGAATGGCTCATCACGATC-3′)基因[13]。PCR阳性产物送至擎科生物公司(中国南京)进行纯化和测序。使用chromas pro应用程序和BLAST(www.ncbi.nlm.nih.gov/BLAST)进一步分析序列。
1.8脉冲场凝胶电泳(pulsed field gel electrophoresis, PFGE) 6株共携带blaNDM和blaKPC-2的大肠埃希菌分离株(包括11个CREC中的3株和南京鼓楼医院43株CREC中的3株)通过PFGE进一步分析遗传相关性[14]。将新鲜菌落与蛋白酶K混合制成模块,用限制性内切酶XbaⅠ消化模块,在PFGE CHEF-DRⅢ系统中,在0.5×Tris-borate-EDTA缓冲液中进行电泳,电泳条件为:切换时间2.2~54.2 s,电场强度6 V/cm,电场夹角120°,电泳时间18.5 h。使用BioNumerics 8.0软件分析条带模式。
1.9接合试验 对于共携带blaKPC和blaNDM的6株细菌,进行肉汤法接合以分析这些基因的可转移性[7],抗叠氮钠大肠埃希菌J53作为受体菌。将新鲜菌落接种至5 mL LB肉汤中,在37 ℃、200 r/min下培养。5 h后,将500 μL受体细胞和100 μL供体混匀于5 mL LB肉汤中,37 ℃过夜培养,然后取100 μL涂布于含有30 mg/L头孢西丁和100 mg/L叠氮钠的LB平板上。为筛选接合子,对于筛选平板上长出来的菌落,每株细菌挑5个菌落转种至含30 mg/L头孢西丁和100 mg/L叠氮钠的LB平板上分纯扩增后,水煮法提取基因组DNA,使用PCR法检测菌落中blaKPC和blaNDM基因;使用Eric-PCR(引物序列为EricF:5′-ATGTAAGCTCCTGGGGATTCAC-3′和EricR:5′-AAGTAAGTGACTGGGGTGAGCG-3′)检测接合子与供体菌和受体菌之间的遗传相关性。如果检测菌落与受体菌有遗传相关性,并且携带有blaKPC和/或blaNDM基因,即本实验所获得的接合子。
2 结果
2.111株CREC的药物敏感性 11株CREC菌株对β-内酰胺类(包括碳青霉烯类)、β-内酰胺酶抑制剂(包括头孢他啶/阿维巴坦)和氟喹诺酮类药物均耐药。对甲氧苄啶磺胺甲噁唑、庆大霉素、阿米卡星和呋喃妥因的耐药率分别为72.7%、36.4%、18.1%和9.1%。11株细菌对替加环素、多黏菌素和氨曲南/阿维巴坦均敏感(表1)。与携带blaNDM菌株的最低抑菌浓度(minimum inhibitory concentration, MIC)相比,共携带blaNDM和blaKPC的菌株对所有抗菌药物的MIC并没有明显升高。

表1 11株碳青霉烯耐药大肠埃希菌对抗菌药物的最低抑菌浓度(mg/L)
2.211株CREC的基因组流行病学特点 见表2。基因组分析显示,11株CREC菌株中,9株细菌携带blaNDM-5基因,2株携带blaNDM-1,3株共携带blaNDM-5和blaKPC-2;10株细菌携带blaCTX-M,其中,4株携带blaCTX-M-55,2株携带blaCTX-M-65,2株携带blaCTX-M-14,2株携带blaCTX-M-15;3株细菌产AmpC酶,其中2株产blaCMY-2,1株产blaCMY-43;11株细菌中检出5种质粒介导的喹诺酮类耐药(PMQRs),包括2株携带oqxAB,1株携带qnrA1,4株携带qnrS1,4株携带aac(6′)-Ib-cr,1株携带qepA;4株细菌携带质粒介导的谷胱甘肽S转移酶(PMGST)基因fosA3(图1)。毒力基因分析发现12种毒力基因,其中gad分布于9株细菌中,lpfA和iss分布于7株细菌中,其他毒力基因包括astA(分布于3株细菌)、cma(分布于3株细菌)、capU(分布于3株细菌)、iroN(分布于2株细菌)、sat(分布于1株细菌)、senB(分布于1株细菌)、sepA(分布于1株细菌)、iha(分布于1株细菌)和cnf1(分布于1株细菌)(表3)。
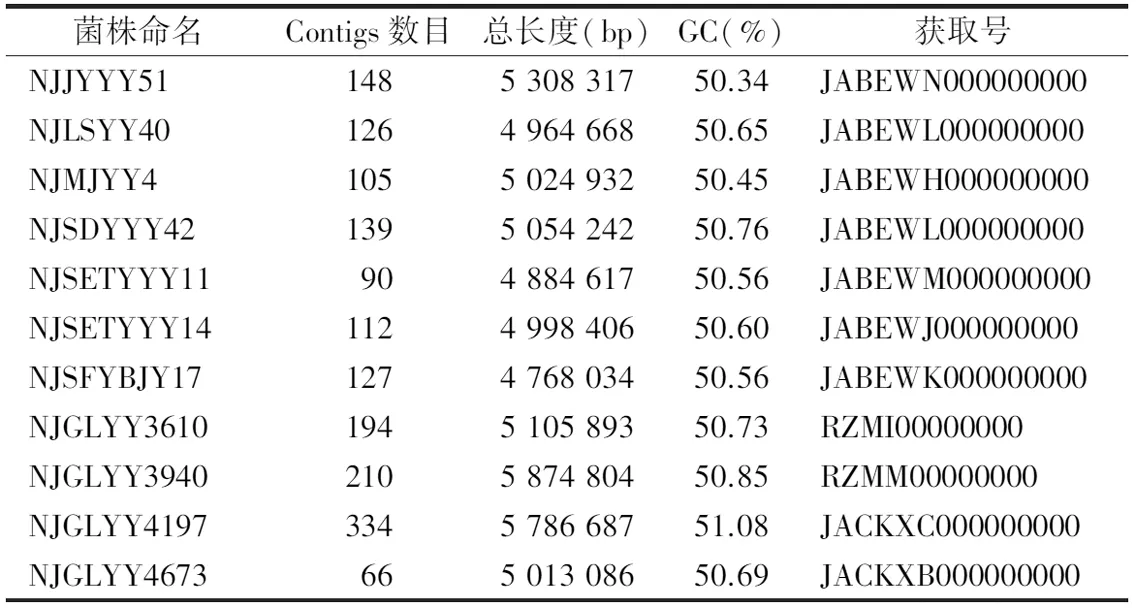
表2 11株碳青霉烯耐药大肠埃希菌的基因组学特征
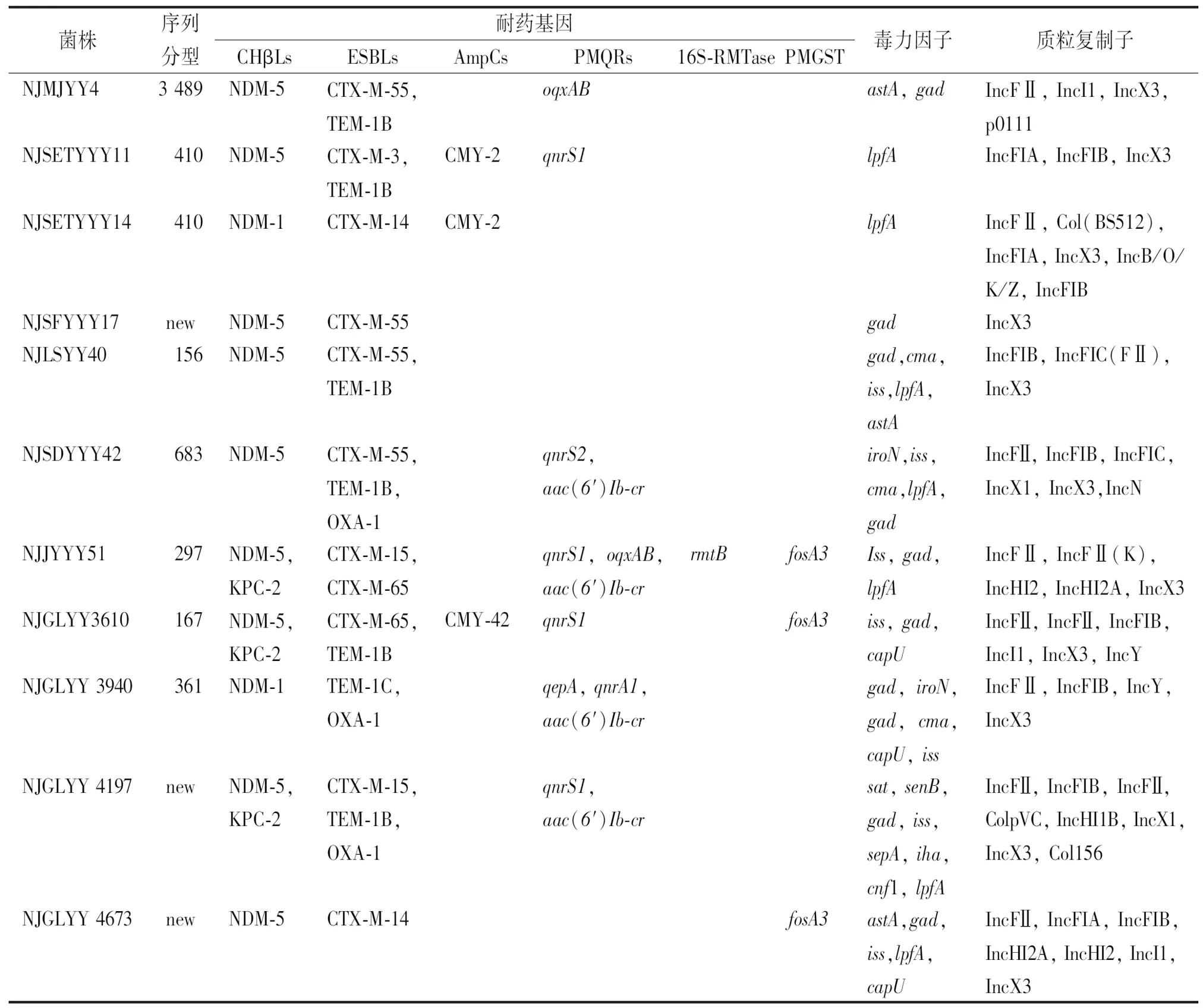
表3 11株碳青霉烯大肠埃希菌中耐药基因、毒力基因和质粒复制子的分布
MLST分析发现7个不同的ST,2株细菌为ST410,6株细菌的ST分别为ST156、ST167、ST297、ST361、ST683和ST3489,另外3株细菌的ST为新型,尚未命名(图1)。11株细菌的血清型分为 5个O群(O8、O9、O25、O30、O89)和8个H群(H4、H9、H10、H21、H26、H30、H40和H45)。FimH分型鉴定了8 种Fim型, 包括2个Fim23,2个 Fim24,1个 Fim31,1个Fim34,1个Fim38,1个 Fim54,1个Fim121和1个Fim276。

注:CHβLs,碳青霉烯水解β-内酰胺酶; ESBLs,超广谱β-内酰胺酶; pAmpCs,质粒介导AmpC酶; 16S-RMTase,外源性获得的16S rRNA甲基转移酶; PMGST,质粒介导的谷胱甘肽S转移酶。图1 11株碳青霉烯耐药大肠埃希菌的热图
共检测到7种质粒复制子。其中,11株细菌均携带IncX3复制子。7株细菌携带IncFⅡ复制子,6株细菌携带IncFIB复制子,3株携带IncI1复制子,3株携带IncFIA复制子,2株携带IncY复制子,2株携带IncFIC复制子。
2.311株CREC菌株的系统发育特征 系统发育树虽然显示11株CREC菌株有很大的遗传多样性,但从整体上看,11株CREC进化成2个主要分支(图1),其中南京儿童医院分离到的2株细菌具有密切的进化关系。
2.4共携带blaNDM-5和blaKPC-2菌株的流行情况 在南京鼓楼医院2013—2017年收集的43株CREC中,6株细菌携带blaKPC-2,23株细菌携带blaNDM基因,2株共携带blaNDM-5和blaKPC-2,1株共携带blaNDM-1和blaKPC-2。共携带blaNDM和blaKPC的3株细菌中,1株于2014年从ICU住院患者尿液中分离,另2株于2017年从不同患者的腹水和痰中分离。
2.5blaKPC和blaNDM共携带菌株的遗传相关性 PFGE显示共携带blaKPC和blaNDM的6株大肠埃希菌的遗传呈高度的多样性(图2),表明这些菌株不是来自同一克隆。

注:树形图是基于Dice相似系数由UPGMA算法生成,依据PFGE图谱85%的相似性定义为6个PFGE组。图2 基于脉冲场凝聚凝聚电泳图谱产生的6株共携带blaKPC-2和 blaNDM大肠埃希菌的树状图
2.6blaKPC和blaNDM的可转移性 接合试验表明,6株菌株的blaNDM均能转移到大肠埃希菌J53中。然而,接合子中并没有检测到blaKPC基因,表明blaKPC可能和blaNDM不在同一质粒上。
3 讨论
本研究中来自6家医院的11株CREC细菌对临床治疗中常用的抗菌药物表现出的高耐药性与以往的报道一致[15],表明对此类菌株引起的感染所能选择的抗菌药物相当有限。替加环素、多黏菌素和氨曲南/阿维巴坦的敏感性很好。当比较只携带blaNDM的菌株与共携带blaKPC和blaNDM的菌株对β-内酰胺类抗菌药物的MIC时,单个分离株中blaKPC和blaNDM的同时存在似乎并不能导致对β-内酰胺类抗菌药的更高耐药性,但本研究中此类细菌数目有限,结果有待验证。此外,据报道,厄他培南对产生NDM-5菌株的MIC比对产NDM-1菌株的MIC高4倍或8倍[16],但本研究没有观察到这样的现象,这可能与其他耐药机制如ESBLs和AmpC酶的产生、外排泵的过表达、外膜通透性降低等的存在有关。
在本研究中,11株CREC中blaNDM的高流行率与以往的报道一致[8],表明NDM是介导大肠埃希菌对碳青霉烯类耐药的主要碳青霉烯酶,这可能与大肠埃希菌和blaNDM质粒的适应性有关。此外,本研究发现NDM-5是最常见的NDM,这与目前的流行病学数据一致[8],表明NDM-5是大肠埃希菌中的主要NDM酶。在医院内,NDM-5主要存在于高危流行克隆ST167、ST410和ST101中[17],这可能与大肠埃希菌克隆群的成功播散和NDM-5表达质粒的水平基因转移有关。虽然ST131作为一种多药耐药克隆型已在全世界广泛传播,而在本研究中没有检测到ST131。本研究发现了多个不同的STs,表明这些CREC的克隆多样性,也与本研究系统发育树结果一致。虽然NDM-5已在大肠埃希菌ST410、ST156和ST167中被报道[7],但产NDM-5的大肠埃希菌ST297、ST683和ST3489以及产NDM-1的大肠埃希菌ST361以前未见报道。
尽管在CREC中发现了多个质粒复制子,但携带blaNDM的质粒主要有IncX3、IncFⅡ或IncC。考虑到在所有携带blaNDM的大肠埃希菌中均有IncX3质粒复制子,推测IncX3是blaNDM的主要宿主[18]。本研究中接合实验表明,blaNDM的传播并没有伴随blaKPC-2的转移,表明blaNDM和blaKPC-2可能并没有在同一质粒上。
毒力基因分析显示CREC中有几个主要的毒力基因,其中gad编码谷氨酸脱羧酶,是耐酸系统的主要结构成分,保护大肠埃希菌免受强酸(pH<3.0)压力,通常在哺乳动物胃肠道中[19];lpfA是一个假定的黏附基因,编码肠出血性大肠埃希菌O157:H7菌毛,与宿主肠道定植相关,在细菌感染过程中发挥重要作用[19];iss是最常见的禽致病性大肠埃希菌编码基因,已被鉴定为与大肠埃希菌毒力相关的一种毒力特征,可引起家禽大肠埃希菌病[20]。这些毒力基因在CREC中的高流行率可能表明CREC主要定植于宿主肠道。1株从尿液中分离的CREC菌株不仅携带gad、iss和lpfA,而且还携带sat(分泌型自体转运毒素)、senB(质粒编码肠毒素)、sepA(志贺氏菌胞外蛋白a)、iha(黏附蛋白)和cnf1(细胞毒性坏死因子)。研究表明,sat可促进一些未分化上皮细胞的细胞毒性作用,并在某些引起泌尿和肠道感染的大肠埃希菌中高度流行[21];cnf1经常在临床尿致病性大肠埃希菌分离株中表达,产Cnf1和β-溶血的大肠埃希菌常显著引起人类的尿路和脑膜感染[22]。总之,这些毒力基因在CREC中的存在可能表明该菌株具有较高的致病性。
此外,发现O8:H9是最常见的血清型,这与以往的报道一致[23],表明O8:H9是一种与多药耐药相关的临床相关血清型。
综上所述,基因组分析发现NDM是11株CREC菌株的主要碳青霉烯酶,KPC共存的发生率较高。这些CREC菌株的MLST、Fim分型、血清分型和系统发育关系表现出遗传多样性,IncX3可能是NDM的主要质粒,gad、iss和lpfA是主要的毒力基因;尽管检测到blaNDM和blaKPC-2共携带菌株,但未发现克隆传播;基于这些菌株对公众健康的潜在威胁,应进一步加强感染控制措施。
