基于转录组学的绿豆改善肥胖小鼠肝脂肪变性的潜在机制
2021-12-31侯殿志郑博妍贾秋菊周素梅
侯殿志, 唐 健, 郑博妍, 贾秋菊, 周素梅, 沈 群
(1.北京工商大学 食品与健康学院, 北京 100048;2.中国农业大学 食品科学与营养工程学院, 北京 100083)
非酒精性脂肪性肝(NAFLD)是指排除继发性原因或酒精滥用所致的以肝脏脂肪过度积累为主要特征的临床病理综合征[1]。目前,NAFLD已成为世界范围内的一种常见的肝脏疾病,其流行率在22.10%~28.65%。NAFLD对人类健康造成了严重的影响,同时也对社会造成了巨大的经济负担[2]。研究发现,NAFLD的发生也增加了其他一些慢性疾病的风险,如糖尿病和心血管疾病等[3]。然而,NAFLD发生和发展的病理机制较为复杂,目前尚无明确的针对NAFLD治疗的药物。生活方式改变和饮食调整是目前缓解NAFLD的主要策略[4]。近年来,作为饮食干预策略的功能性食品或其成分因在治疗和管理NAFLD方面表现出的有益效果而受到广泛的关注[5]。大量的流行病学调查显示,一定量杂豆类食物的摄入可以降低高脂膳食引发的NAFLD的发病率[6-7]。
绿豆是我国大部分地区普遍种植且经常食用的豆类作物之一。绿豆含有丰富的营养物质,包括蛋白质(14.6%~32.6%)、膳食纤维(14%~25%)、矿物质(3.6%~4.2%)和大量的植物化学物[8],可满足人体正常的营养需求。除了营养方面的性质外,古代医学典籍和现代实验研究均证实绿豆作为一种功能性食品,具有多种生理功效,如降糖、降脂及抗炎等[9-10]。在日常生活中,人们对绿豆主要是以全绿豆和去皮绿豆两种形式进行食用。我们前期的研究发现,全绿豆和去皮绿豆均可显著改善肥胖小鼠和糖尿病前期小鼠肝脂肪变性和肠道菌群紊乱[11-12],但是,全绿豆和去皮绿豆对于肝脂肪变性改善作用的具体调控作用途径以及它们之间的差异化机制还待阐明。
转录组学作为一个在mRNA表达水平上研究生物在某一生理进程中所有基因转录及其转录调控规律的测序手段,可提供大量代谢信息,用于帮助预测响应外部刺激而激活的单个基因作用[13]。目前,转录组学已被广泛用于寻找疾病的治疗靶标[14]。本研究以高脂饲养的肥胖小鼠为模型,利用转录学鉴定和筛选全绿豆和去皮绿豆改善肥胖小鼠肝脂肪变性的差异表达基因和富集的信号调控途径,以期揭示绿豆缓解肝脂肪变性的潜在作用机制。希望本研究结果可为绿豆基功能性食品的开发提供数据支撑。
1 材料与方法
1.1 材料与试剂
明绿豆,购自山西东方物华农业科技有限责任公司;4周龄雄性C57/BL6J小鼠[(16±2) g],动物伦理审查编号为AW08089102-1- 4,北京维通利华实验动物技术有限公司;高脂饲料(D12492)和低脂饲料(D12450J),美国Research Diets公司;TRIzol试剂,赛默飞世尔科技有限公司;DNase Ⅰ(脱氧核糖核酸酶Ⅰ),日本TaKara公司;TruSeqTM RNA sample preparation kit试剂盒,美国Illumina公司;SuperScript double-stranded cDNA synthesis kit试剂盒,赛默飞世尔科技有限公司;BeyoFastTMSYBR Green qPCR Mix,碧云天生物技术有限公司;肿瘤坏死因子(TNF-α)和白细胞介素6(IL-6)ELISA检测试剂盒,南京建成生物工程研究所。
1.2 仪器与设备
NanoDrop 2000型超微量分光光度计,赛默飞世尔科技公司;Agilent 2100 型生物分析仪,安捷伦科技有限公司;CFX96型 Real-Time PCR 检测系统,伯乐生命医学产品有限公司;NovaSeq 6000型测序系统,美国Illumina公司。
1.3 实验方法
1.3.1去皮绿豆的制备及特种饲料加工
将绿豆常温下于蒸馏水中浸泡8 h,手动揉搓去除种皮,然后冷冻干燥。将干燥的全绿豆和去皮绿豆研磨成粉,过80目筛,于-20 ℃条件下保存备用。
根据高脂饲料配方(D12492),将质量分数为30%的全绿豆(可利用碳水化合物64.78%,灰分3.36%,脂肪2.58%,蛋白质25.81%,粗纤维3.48%,以干基计)和去皮绿豆(可利用碳水化合物66.28%,灰分2.99%,脂肪2.56%,蛋白质27.32%,粗纤维0.85%,以干基计)以等宏量营养素和等热量的形式配比到高脂饲料中,具体的配比参考文献[15-16]。特种饲料委托常州鼠一鼠二生物科技有限公司加工完成。
1.3.2动物实验设计与分组
整个动物实验方案由中国农业大学实验动物福利和动物实验伦理委员会批准,并严格按照相关的规定执行。将32只小鼠进行适应性喂养1周后,根据小鼠的体质量按照区组随机分组的方法分为4组,每组8只,分别为对照组(NCD)、模型组(HFD)、全绿豆干预组(HFD- WMB)、去皮绿豆干预组(HFD- DMB)。4只小鼠1笼,自由取食和取水,干预周期为12周。饲养环境为SPF级,温度为(22±2) ℃,相对湿度为55%±10%,12 h光照交替。
1.3.3肝组织收集
小鼠麻醉处死后,进行解剖处理,取小鼠肝大叶于冻存管中,立即放入液氮,随后转移到-80 ℃冰箱储存。每组随机取3只小鼠的肝脏样本用于转录组测序分析。
1.3.4血清中炎症因子的测定
使用市售的ELISA检测试剂盒测定血清肿瘤坏死因子(TNF-α)和白细胞介素6(IL-6)的浓度。测定方法根据制造商的使用说明书进行。
1.3.5RNA的提取和转录组测序
1)RNA提取。根据制造商的说明,使用TRIzol®试剂从组织中提取总RNA。具体方法:将100 mg肝组织样品在液氮中研磨后,加入1.0 mL Trizol(静置10 min)。随后,加入200 μL氯仿(混匀30 s,静置5 min),1 000 r/min离心15 min(4 ℃)。上层水相(约0.4 mL)转移至灭菌EP管,以1∶1的体积比加入异丙醇(静置20 min)。1 000 r/min再次离心15 min(4 ℃),弃上清液,自然风干(约10 min),得到的RNA呈半透明状,并且在超净工作台中完成。加入20~50 μL DEPC水溶解RNA沉淀,使用ND- 2000(NanoDrop Technologies)对其进行定量,1%凝胶电泳测定所提取RNA质量。仅使用高质量的RNA样品[OD260/280=1.8~2.2,OD260/230≥2.0,RIN(表示RNA样品的完整度)≥6.5,28S:18S≥1.0(衡量提取RNA完整性的指标),样品总量>1 μg]用于后续实验。
2)文库制备和测序。按照Illumina(San Diego, CA)的TruSeqTMRNA样品制备试剂盒使用1 μg总RNA制备RNA-seq转录组文库。简单来说,根据polyA选择方法,通过oligo(dT)磁珠分离信使RNA,用片段缓冲液将其片段化。使用SuperScript双链cDNA合成试剂盒(Invitrogen,CA)和随机六聚体引物(Illumina)合成双链cDNA。根据Illumina的文库构建方案对合成的cDNA进行末端修复,磷酸化和“A”碱基添加。选择质量分数为2%的低范围超琼脂糖上300 bp cDNA靶片段大小的文库,然后使用Phusion DNA聚合酶(NEB)进行15个PCR周期的PCR扩增。通过TBS380定量后,使用Illumina HiSeq xten/NovaSeq 6000测序仪(2×150 bp读取长度)对配对末端的RNA-seq测序文库进行测序。测序过程由上海美吉生物医药科技有限公司完成。
1.3.6转录组测序分析
原始测序数据以fastq格式保存,使用fastx_toolkit_0.0.14软件对原始数据进行测序质量评估,包括:碱基质量分布统计;碱基错误率分布统计;A/T/G/C碱基含量分布统计。由SeqPrep(https:∥github.com/jstjohn/SeqPrep)和Sickle(https:∥github.com/najoshi/sickle)使用默认参数进行质量控制。使用HISAT2(http:∥ccb.jhu.edu/software/hisat2/index.shtml)软件以定向模式将Clean Reads与指定的参考基因组进行序列比对,比对率从92.82%到96.63%不等[17]。获得用于后续转录本组装、表达量计算等的Reads,在已有参考基因组基础上,将Reads进行组装拼接获得参考基因,使用表达量计算软件对参考基因表达水平进行定量差异分析。
为了鉴定两个不同样品之间的DEG(差异表达基因),根据TPM的方法计算每个转录本的表达水平。RSEM(http:∥deweylab.biostat.wisc.edu/rsem/)用于定量基因丰度。在计算每个基因的表达水平后,利用edgeR进行差异表达分析。利用错误发现率(FDR),确定多次测试中P值的阈值,并进行分析,使用FDR阈值≤0.01和绝对值log2FC(上下调倍数)≥2来判断重要性基因表达之间的差异。利用差异表达基因(DEGs)建立基因集,并与京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)数据库比对,对基因集中的基因进行KEGG功能富集分析。Q值≤0.05的GO和KEGG途径均被认为在DEGs中显著富集。
1.3.7qPCR检测差异基因的表达
qPCR检测差异基因表达参考Hou等[18]的方法进行。TNF-α的引物序列为5′-TGGCCCAGACC CTCACACTC-3′和5′-CTCCTGGTATGAAATGGCAA ATC-3′。
1.4 数据分析
利用单因素方差分析(one-way ANOVA,Tukey test)计算组间差异。P<0.05代表存在显著性差异,具有统计学意义。采用Graphpad 8.0软件进行图像的绘制。
2 结果与分析
2.1 转录组测序数据及比对分析
我们前期的实验结果显示,绿豆可显著缓解肥胖小鼠的体质量、体质量增量和肝脂肪变性情况[11],但相比于去皮绿豆,全绿豆的作用效果更为显著。为了进一步探究绿豆改善肝脂肪变性的潜在机制,我们以高脂饲养的肥胖小鼠为模型,采用转录组技术对对照组(NCD)、模型组(HFD)、全绿豆干预组(HFD- WMB)和去皮绿豆干预组(HFD- DMB)小鼠的肝脏组织进行RNA测序分析。
不同组别中肝样品转录组测序质量和序列比对结果见表1。表1显示,经转录组测序后,NCD组,HFD组,HFD- WMB组和HFD- DMB组分别平均获得50 937 731、48 910 641、51 540 116和52 250 552条原始目数,经质控去除低质量的测序目数后,分别为50 555 963、48 502 161、51 156 807和51 853 601条测序目数。Q20、Q30分别指测序质量在99.0%和99.9%以上的碱基占总碱基的百分比。在本研究中采用Q20和Q30对原始测序数据进行质量评估,一般Q20在85%以上,Q30在80%以上可满足测序的质量要求。表1中的结果显示,Q20值均大于98%,Q30值均大于94%。测序所得的总条目数和质量评估结果表明,本研究测序的数量和质量达到了后续分析的要求。另外,测序结果与参考基因组的比对结果也显示,所有样品的唯一比对率均达到83%以上,证实本次测序结果的有效性。
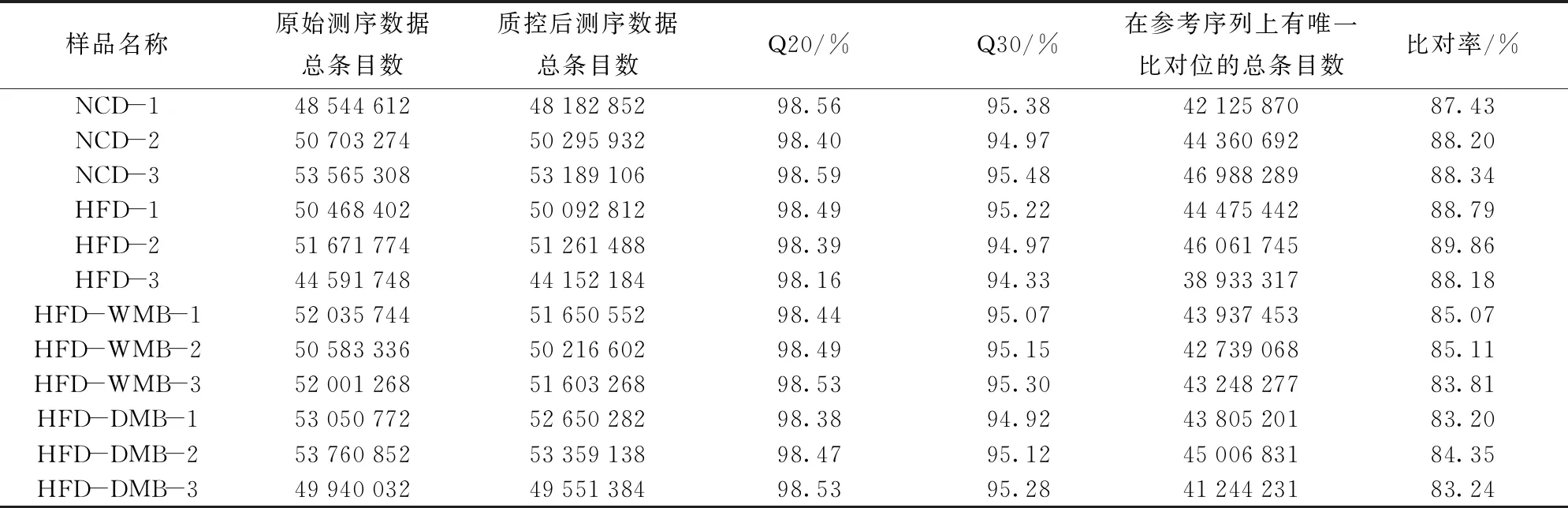
表1 不同组别中肝样品转录组测序质量和序列比对
2.2 主成分分析
根据基因在不同样本间的表达情况,对4个组别的基因组样本进行主成分分析(PCA),分析结果见图1。图1显示,4组基因组样本之间各自明显聚集且表现出显著分离。具体而言,NCD组与HFD组之间明显分离,表明高脂饲养显著改变了小鼠肝脏的基因表达谱。经绿豆干预后,HFD- WMB和HFD- DMB两组与HFD组呈现出的显著分离暗示绿豆干预可显著逆转部分高脂饲养导致的基因表达改变。尽管HFD- WMB组和HFD- DMB组之间也显示各自聚集和分离的现象,但是距离较为接近。

图1 不同组别基因表达主成分分析
2.3 差异表达基因分析
NCD和HFD组、HFD和HFD- WMB组,HFD和HFD- DMB组之间的表达量差异基因(P<0.05,|log 2FC|≥2.0)分析结果见图2。图2显示,NCD组和HFD组之间共有819个表达量差异基因。与NCD组相比,HFD组有606个表达量差异基因显示出显著上调,213个表达量差异基因显示出显著下调[图2(a)和图2(d)]。经绿豆干预后,与HFD组相比,HFD- WMB组有306个表达量差异基因显示出显著上调,596个表达量差异基因显示出显著下调[图2(b)和图2(e)],而HFD- DMB组有461个表达量差异基因显示出显著下调,1 132个表达量差异基因显示出显著上调[图2(c)和图2(f)]。这些显著上调和下调的表达量差异基因表明绿豆在一定程度上可通过调控相关基因的表达改善小鼠的肝脂肪变性。

图2 不同组别的差异表达基因分析
2.4 KEGG功能注释分析
KEGG是系统分析基因功能、联系基因组信息和功能信息的知识库。利用KEGG数据库,可将基因集中的基因按照参与的信号通路或行使的功能进行分类。为了进一步明确组间表达量差异基因的功能,对HFD和HFD- WMB组、HFD和HFD- DMB组之间的表达量差异基因进行KEGG功能注释分析。KEGG通路的功能注释分析结果见图3。图3显示,根据KEGG分类,HFD和HFD- WMB组、HFD和HFD- DMB组之间的表达量差异基因分别注释到了代谢、遗传信息处理、环境信息处理、细胞过程、生物体系统和人类疾病中。在代谢相关的功能中,全绿豆和去皮绿豆分别有30和52个表达量差异基因显著注释到了脂质代谢相关的通路上,其次有23和29个表达量差异基因注释到了碳水化合物代谢相关的通路上。脂质代谢和碳水化合物代谢被认为与肝脂肪变性之间存在密切联系,因此,基于KEGG功能注释分析的结果,进一步证实了绿豆改善肥胖小鼠肝脂肪变性与调控一些表达量差异基因存在显著的相关性。
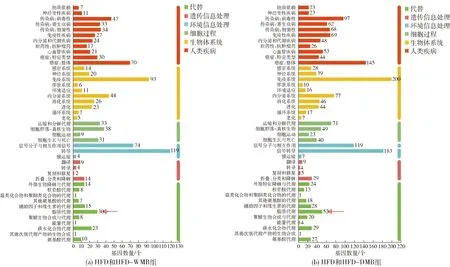
图3 KEGG通路的功能注释分析
2.5 KEGG途径富集分析
基于KEGG数据库,对表达量差异基因可能参与的信号通路进行富集分析,结果见图4。图4显示了HFD和HFD- WMB组、HFD和HFD- DMB组之间表达量差异基因富集的相关信号通路。以HFD与HFD- WMB之间表达量差异基因富集的前20条信号通路为基准,表达量差异基因参与了多种不同疾病的调控,其中涉及的与肝脂肪变性调控相关的信号通路,包括NOD样受体信号通路和Toll样受体信号通路。不同组别中与肝脂肪变性调控相关的KEGG信号通路见表2。从表2中可以看出,3组之间的表达量差异基因显著富集到了2种信号通路中。Liu等[18]采用比较转录组分析发现,虾青素对小鼠酒精性肝的保护作用主要是通过调控NOD样受体信号通路和Toll样受体信号通路完成的。NOD样受体信号通路和Toll样受体信号通路与调节免疫和炎症反应密切相关[19]。最近的研究表明,炎症小体激活引发的炎症应答与非酒精性脂肪性肝病的发生发展密切相关[20]。Kwon等[21]的研究结果显示,木犀草素可通过靶向Toll样受体信号通路预防饮食诱导肥胖小鼠肝和脂肪细胞纤维化及胰岛素抵抗。基于图4和表2的分析可以看出,尽管绿豆可通过调控多信号通路的协同作用改善小鼠肝脂肪变性,但是,绿豆对肥胖小鼠肝脂肪变性的改善作用可能主要侧重于炎症和免疫相关的NOD样受体信号通路和Toll样受体信号通路上。
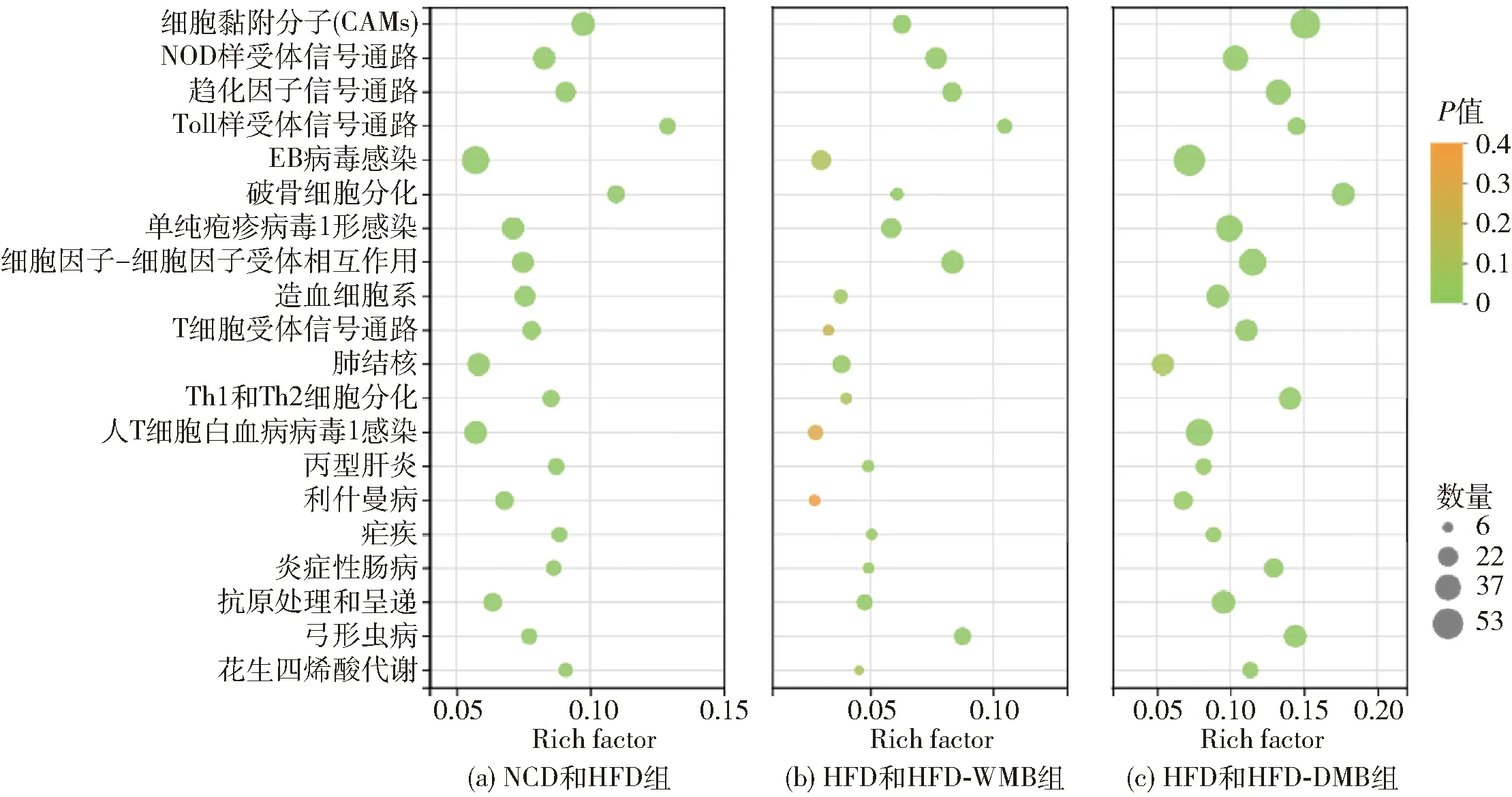
Rich factor表示该通路中富集到的基因/转录本数目与注释基因/转录本数目的比值。

表2 不同组别中与肝脂肪变性调控相关的KEGG信号通路
另外,需要注意到的是,NOD样受体信号通路中所含有的表达量差异基因的数量几乎是Toll样受体信号通路的两倍。结合我们的研究目的以及考虑到NOD样受体信号通路在HFD模型组和绿豆干预组中均可显著富集且在肝脂肪变性中发挥的重要作用,在接下来的研究中选择NOD样受体信号通路进一步分析绿豆改善肥胖小鼠肝脂肪变性的潜在机制。
2.6 NOD样受体信号通路中调控肝脂肪变性的表达量差异基因分析
绿豆干预后富集在NOD样受体信号通路上的表达量差异基因见表3。由表3可知,对NOD样受体信号通路中的表达量差异基因进行分析发现,NCD和HFD组,HFD和HFD- WMB组,HFD和HFD- DMB组之间有12个共有表达量差异基因,即干扰素激活基因206,干扰素激活基因211,鸟苷酸结合蛋白7,鸟苷酸结合蛋白5,鸟苷酸结合蛋白3,2′~5′寡腺苷酸合成酶3,鸟苷酸结合蛋白2,干扰素激活基因208,2′~5′寡腺苷酸合成酶1G,干扰素调节因子7,肿瘤坏死因子,信号转导和转录激活因子1。与NCD组相比,这12个表达量差异基因在HFD组显著上调,但经过绿豆干预后,它们均得到显著下调。这表明绿豆改善高脂饲养肥胖小鼠肝脂肪变性的效果很大可能与调控这些基因的表达有关。
2.7 小鼠血清中炎症因子的测定及qPCR验证差异基因的表达
从2.5和2.6结果的分析可以看出,绿豆有可能主要是通过调控炎症相关的信号通路和基因完成肝脂肪变性的改善作用。我们前期的实验结果已经证实,绿豆可显著改善肥胖小鼠肝脏组织中炎性细胞浸润[11]。为了更好地支撑本研究的实验结果,我们对小鼠血清中的炎症因子TNF-α和IL-6进行了测定,炎症因子水平见图5。图5显示,与NCD组相比,尽管HFD组中的炎症因子TNF-α和IL-6的水平显著升高,但是经过全绿豆(HFD-WMB)的干预后,两种炎症因子的水平显著降低[图5(a),(b)]。去皮绿豆对于炎症因子TNF-α和IL-6的干预仅仅只是显示出改善的趋势,但不显著。这一结果基本与转录组测序的结果一致。另外结合血清炎症因子TNF-α的变化以及NOD信号通路中差异表达基因TNF的表达情况(表3),我们重点对NOD样受体信号通路中TNF-α转录因子进行了验证。结果显示,相比NCD组,HFD组小鼠肝脏中TNF-α mRNA的表达水平显著提升。全绿豆显著降低了高脂饲养肥胖小鼠肝组织中的TNF-α mRNA的表达情况(如图1),而去皮绿豆对TNF-α mRNA的表达量只表现出下降的趋势,但并不显著。在之前的研究中,牡荆素被发现可通过降低肝脏中TNF-α等炎症因子mRNA的表达水平显著改善小鼠的肝损伤[22],而牡荆素也被认为是绿豆发挥功效作用的主要黄酮类物质[9]。
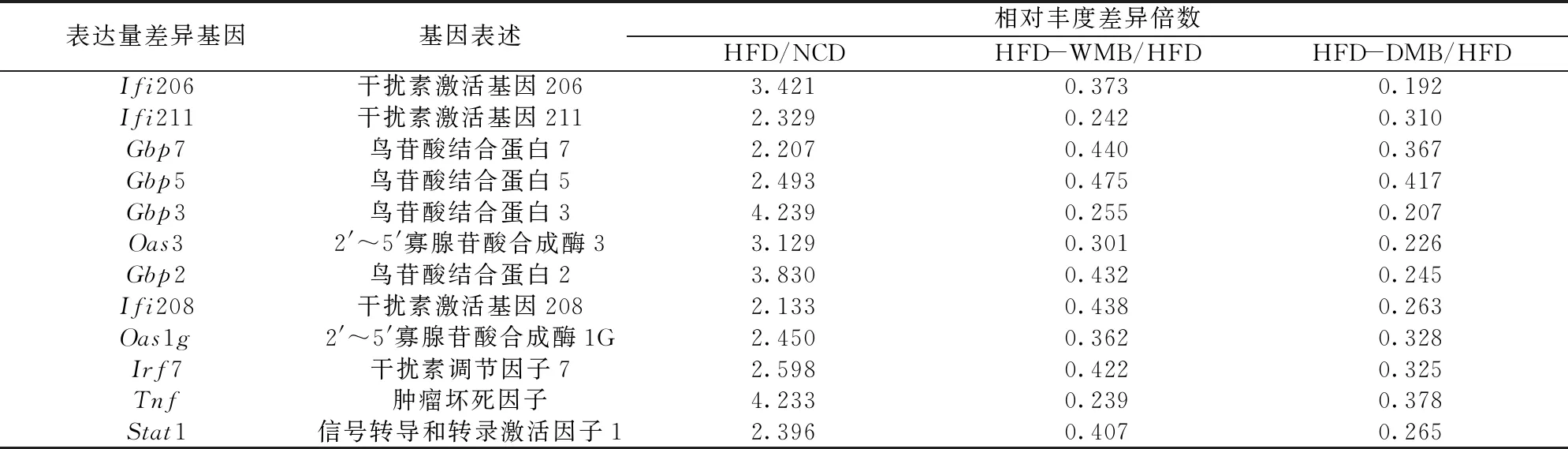
表3 绿豆干预后富集在NOD样受体信号通路上的表达量显著差异基因及其差异倍数

不同小写字母表示组间差异显著。
3 结 论
绿豆对肥胖小鼠肝脂肪变性的改善作用与其调控肝脏中相关的基因表达有关。绿豆可显著改变肥胖小鼠肝脏的基因表达谱,且与模型组小鼠相比,全绿豆和去皮绿豆干预组分别有902个(306个显著上调和596个显著上调)和1 593个(461个显著上调和1 132个显著下调)表达量差异基因。30和52个表达量差异基因显著注释到了脂质代谢相关的通路上。KEGG通路富集分析结果表明,绿豆干预后的表达量差异基因发生了以NOD样受体信号通路和Toll样受体信号通路激活为特征的功能转变。绿豆引起了NOD样受体信号通路中12个表达量差异基因的显著下调。肥胖小鼠血清中炎症因子TNF-α和IL-6水平的显著降低以及肝脏TNF-α mRNA表达水平的降低对转录组测序的结果起到了很好的支撑作用。未来仍需要进一步的实验研究验证转录组学的推测,以及更多的证据来阐释绿豆通过NOD样受体信号通路缓解肝脂肪变性的分子机制。
