西藏林芝3种市售牦牛奶渣的细菌多样性研究
2021-11-22杨林龚小曼池福敏辜雪冬张昊张哲
杨林,龚小曼,池福敏,辜雪冬,张昊,张哲
(1.西藏农牧学院 食品科学学院,西藏林芝 860000;2.陕西师范大学食品工程与营养科学学院,西安 710119)
0 引言
牦牛奶是一种营养丰富、品质优良的特色奶源,被称为“天然浓缩乳”,是藏族同胞聚居地区的人们重要的食品和乳制品加工原料,奶渣、酸奶、酥油、干酪等便是其常见的加工制品,其中,奶渣是一种发酵乳制品,含有丰富的微生物资源[1-4]。常见对牦牛奶渣及其它发酵乳制品中微生物资源的研究报道,但偏向对其中优良乳酸菌、酵母菌的筛选分离及其生理功能特性的研究。如万金敏等人以西藏牦牛奶渣对象,对奶渣中优势乳酸菌的产胞外多糖及其耐受性进行了探讨[5];张晓旭则以西部牧区酸马奶和奶渣为对象,对其中的优势酵母菌进行分离筛选,探讨了其应用特性[6];杨俊俊也以牦牛奶渣为对象,对其中的乳酸菌、酵母菌进行了研究、鉴定、筛选[7];肖秋颖等则以川西高原发酵牦牛乳为对象,分离筛选降解亚硝酸盐能力强的乳酸菌,并得到了降解能力极强的菌株[8]。然而,对市场销售的各种有着地区差异的牦牛奶渣的细菌菌群多样性还鲜有报道。
本研究以西藏林芝市场销售的3 种不同形式的牦牛奶渣为研究对象,采用Illumina HiSeq 高通量测序检测技术,提取牦牛奶渣中微生物宏基因组DNA,对16S rRNA 基因的V3~V4 区域[9]进行特异性扩增、高通量测序,研究牦牛奶渣中细菌的组成及丰度,分析牦牛奶渣中细菌群落的多样性,以便全面认识牦牛奶渣的价值,也为进一步开发、利用西藏特色发酵乳制品中的微生物提供参考。
1 材料与方法
1.1 牦牛奶渣样品的采集
本研究所采用的样品均购自西藏林芝市巴宜区,3 组供试奶渣样品分别来自3 个不同的市售摊点,每组样品采用无菌包装取样3 份后在无菌操作环境下混匀,小心转至无菌离心管中,按照要求分别编号为M01、M02、M03后置于冰箱(-20 ℃)中保存[10]。

图1 3种市售奶渣外观图
1.2 主要实验试剂
E.Z.N.ATM Mag-Bind Soil DNA Kit,OMEGA公司;Qubit3.0 DNA 检测试剂盒,Life 公司;2×Taq Master Mix,Vazyme 公司;MagicPure Size Selection DNA Beads,Transgen公司。
1.3 实验设备
Pico-21 型台式离心机,Thermo Fisher 公司;TND03-H-H 混匀型干式恒温器,深圳拓能达科技有限公司;DYY-6C 型电泳仪电源,北京市六一仪器厂;DYCZ-21 电泳槽,北京市六一仪器厂;凝胶成像系统,美国UVP 公司;Q32866Qubit®3.0 荧光计,Invitro⁃gen公司;T100TM Thermal Cyeler PCR仪,BIO-RAD公司;Research plus 0.5~10μL移液器,Eppendorf公司。
1.4 实验方法
所有奶渣样品进行DNA 提取和高通量测序。
1.4.1 DNA 提取及纯度鉴定
参照OMEG 公司的E.Z.N.ATMMag-Bind 土壤基因组提取试剂盒提取奶渣样品中DNA,使用1%琼脂糖凝胶电泳对抽提后的基因组DNA 进行检测。
1.4.2 16S rDNA 基因的V3~V4 区域特异性扩增及高通量测序
使用Qubit 2.0 DNA 检测试剂盒精确定量供试奶渣样品DNA,以纯化的DNA 为模板,采用通用引物341F(CCTACGGGNGGCWGCAG)和805R(GAC⁃TACHVGGGTATCTAATCC)扩增目的片段16S rD⁃NA V3~V4区。
PCR 扩增反应体系:5 μL10× PCR buffer,0.1 mmol/LdNTP,10 ngGenomic DNA,0.5 μmol/L341F,0.5 μmol/L805R,0.05U Plantium Taq,ddH2O 补充至50 μL。
PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s,45 ℃退火20 s,65 ℃延伸30 s,共计5 个循环;然后再94 ℃变性20 s,45 ℃退火20 s,65 ℃延伸30 s,共计20个循环;最后72 ℃延伸5 min。PCR 产物进行琼脂糖电泳后,使用SanPrep 柱式DNA 胶回收试剂盒回收DNA。再使用Qubit2.0 DNA 检测试剂盒精确定量回收的DNA,1∶1 的等量混合后,利用Illumina HiSeq 平台进行测序。
重大突破出现了:他的团队在一个极小的模糊热团块中探测到了热斑(或者说“耀斑”)存在的证据,这直接标明了那个疑似黑洞天体的位置。一个重达400万个太阳质量的黑洞应该有一张“嘴”(或者说“视界”),其直径大约是1 600万英里 ——实在是太小了,小到地球上的“引力”干涉仪都无法将其分辨出来。
1.4.3 生物信息学分析
使用QIIME(version1.8.0)软件中的UCLUST 对Tags 在相似度为97%水平下聚类、获得OTU[11]。根据OTU 的序列组成得到种属地位,并进行分类学分析,同时利用Alpha 多样性指数分析、Beta 多样性指数分析等评价微生物群落的丰度和多样性,挖掘样品间的差异。
2 结果及分析
2.1 OTU 聚类分析
3 个样品测序共产生238 280 对Reeds,对其进行双端Reeds拼接、过滤后,每个样品至少产生42 912 条Clean tags,平均产生74 702 条Clean tags,进一步将得到的优化序列聚类,划分OTU 数。样本间OTU 数组成的相似性及其重叠情况见图2。由图可知,样品M01 中OTU 总数目为190,其中独有的OTU 数为6;样品M02 中OTU 总数为81,其中独有的OTU 数为3;样品M03 中OTU 总数为226,其中独有的OTU 数为37,M01 和M02、M01 和M03、M02 和M03 分别共有的OTU 数为73、184、78,3 组样品中共有的OTU 数为73。经过分析,样品M03 中细菌的种类比另外两种更丰富,且样品M03与样品M01中共有微生物数目较多。聚类的OTU 数表现出一定的差异性,在数量上表现为样品M03>样品M01>样品M02。
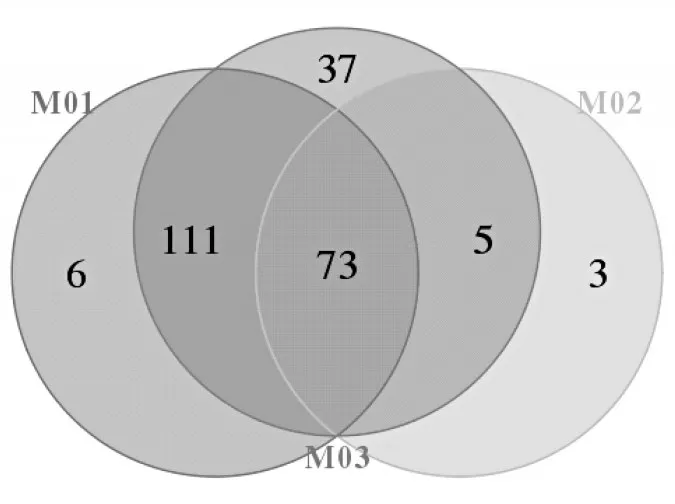
图2 3种市售奶渣OTU Venn图
2.2 Alpha多样性指数分析
3种市售奶渣的物种多样性指数见表1和图3。
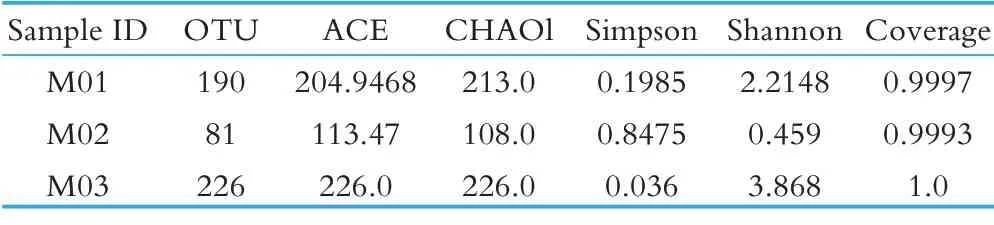
表1 Alpha多样性指数统计
样本在不同测序数量时的生物物种信息见图3a。由图3a 可知,其斜率在0~10 000 的测序数量范围内逐渐上升,后趋势平缓,进入平台期,说明测序量再增加,而OTU 种类不会再随着增长,表明样品中绝大多数的微生物信息在使用研究中的测序数据量情况下已经能够被充分反映。
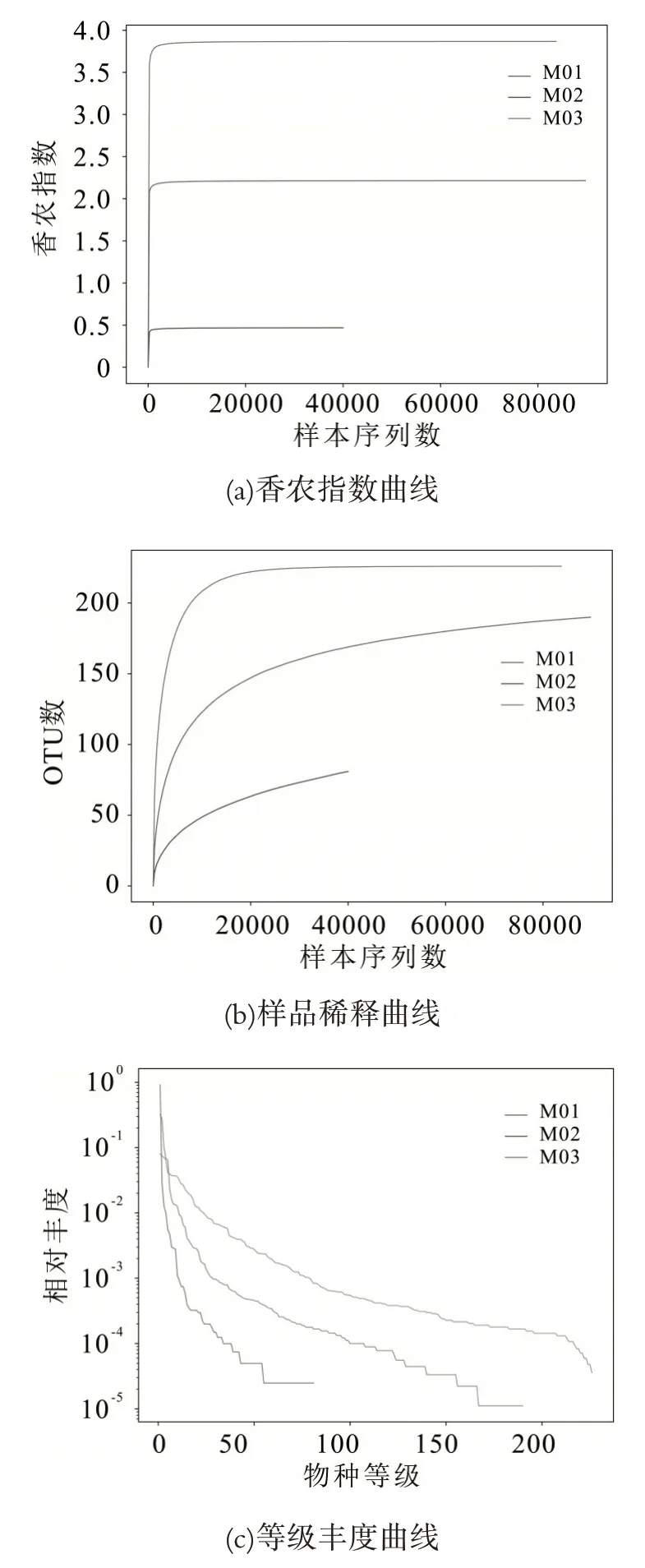
图3 3种市售奶渣物种信息图
验证测序数据量反映样品物种多样性结果见图3b。从图中可发现,当测序范围在0~20 000,曲线随着测序条数增加出现了急剧上升的趋势,这表明有大量物种在群落中被发现。从各样品该曲线的斜率来看,M03>M01>M02,说明各物种的多样性为M03>M01>M02。随着测序数量的进一步增加,稀释曲线斜率逐渐降低,但未进入平台期,说明再增加测序数量也只会产生少量新的OTUs。结合表1 种牦牛奶渣样品M01、M02、M03 在Alpha 多样性分析中的Simp⁃son 指数结果0.1985、0.8475、0.036,Chao1 指数213、108、226,Coverage 指数0.9997、0.9993、1.0,表明测序数据量合理,可以反映样品中绝大多数的微生物信息。
不同物种的分布规律见图3c。由图3c 可知,M01 样品的曲线比其他样品的曲线更平坦且在横坐标上长度越宽,说明M03 中的微生物组成更丰富、分布更均一,其次是M01、M02。
2.3 物种注释及分类学分析
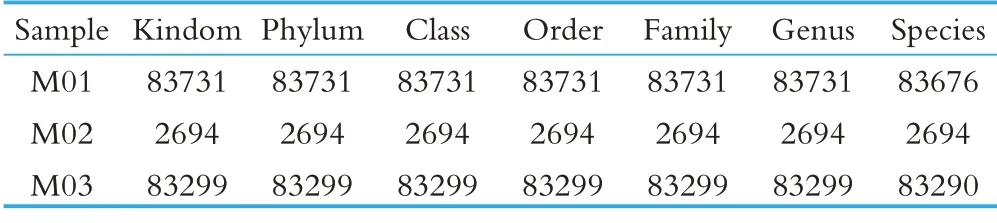
表2 样品各等级Tags统计表

表3 样品各等级物种统计表
由表2 可知,在门、科、属水平上,M01、M02、M03物种tags 数均分别为83731、2694、83299。由表3 可知,在门水平上,物种组成分析共获得15 个门,在科水平上,物种组成分析共获得89 个科,在属水平上,物种组成分析共获得156 个属。其中样品M01、样品M03物种类型丰富且相似,样品M02物种较少。
3 种不同牦牛奶渣样品中不同门水平下物种分布见图4。由图4a 可知,样品M01 中厚壁菌门(Fir⁃micutes)丰度最大,占细菌群落52.47%;其次是变形杆菌门(Proteobacteria),为38.38%,未识别的菌群占比6.87%,其他检测出来的菌种含量极低。样品M02 中变形杆菌门(Proteobacteria)为可检测出来菌群中丰度最大的菌种,占比5.38%;厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、蓝藻菌门(Cyanobacteria)、放线菌门(Actinobacteria)含量极低,检测出未识别的菌群占比93.3%。样品M03 中,变形菌门Proteobacteria 为其优势菌种,细菌含量占比55.91%,其次是厚壁菌门(Firmicutes),占比26.74%。M03 样品可检测出的菌种相较另外两种而言,含量与种类均更丰富多样。
牦牛奶渣样品中细菌群落在科分类水平的分布情况见图4b。由图4b 可知,样品M01 中乳酸杆菌科(Lactobacillaceae)丰度最大,在细菌群落中占比49.18%,为其优势菌种,其次是醋菌科(Acetobactera⁃ceae),占比27.12%,肠杆菌科(Enterobacteriaceae)、假单胞菌科(Pseudomonadaceae)占比分别为2.11%、2.33%,检测出未识别的菌群占比6.87%,Others 菌群占比5.7%。样品M02 中肠杆菌科(Enterobacteriaceae)有一定丰度,占比3.12%,为其优势菌种,Others 菌群占比0.88%,但其检出却未识别的菌群占比最大,为93.30%。样品M03 中,细菌群落丰富且均有一定丰度,其中,乳酸杆菌科(Lactobacillaceae)丰度最大,在细菌群落中占比13.42%,其次为假单胞菌科(Pseudo⁃monadaceae),占比10.00%,所检出的其他微生物均有一定丰度,为2.76%~8.47%,除此之外,Others 菌群占比26.24%,远远超出M01、M02 样品,检测出未识别的菌群仅有0.57%,少于M01、M02样品。
牦牛奶渣样品中细菌群落在属分类水平的分布情况见图4c。由图4c 可知,样品M01 中乳酸菌属(Lactobacillus)丰度最高,细菌群落占比49.18%,其次是醋酸杆菌属(Acetobacter)占比27.12%,Others 菌群占比9.85%,检测出未识别的菌群占比6.87%。样品M02 中所检测出来的微生物丰度极小,占比0.03%~0.99%;M02 中Others 菌群占比3.70%,检测出未识别的菌群占比极大,为93.30%。样品M03 中,乳酸菌属(Lactoba⁃cillus)丰度最大,为13.42%,其次为假单胞菌属(Pseudo⁃monas),占比10.00%,所检测出来的其他微生物有一定丰度,为2.70%~8.02%,Others 菌群占比极大,为34.27%,检测出未识别的菌群占比较低,为0.57%。其变化规律与科分类水平相似。
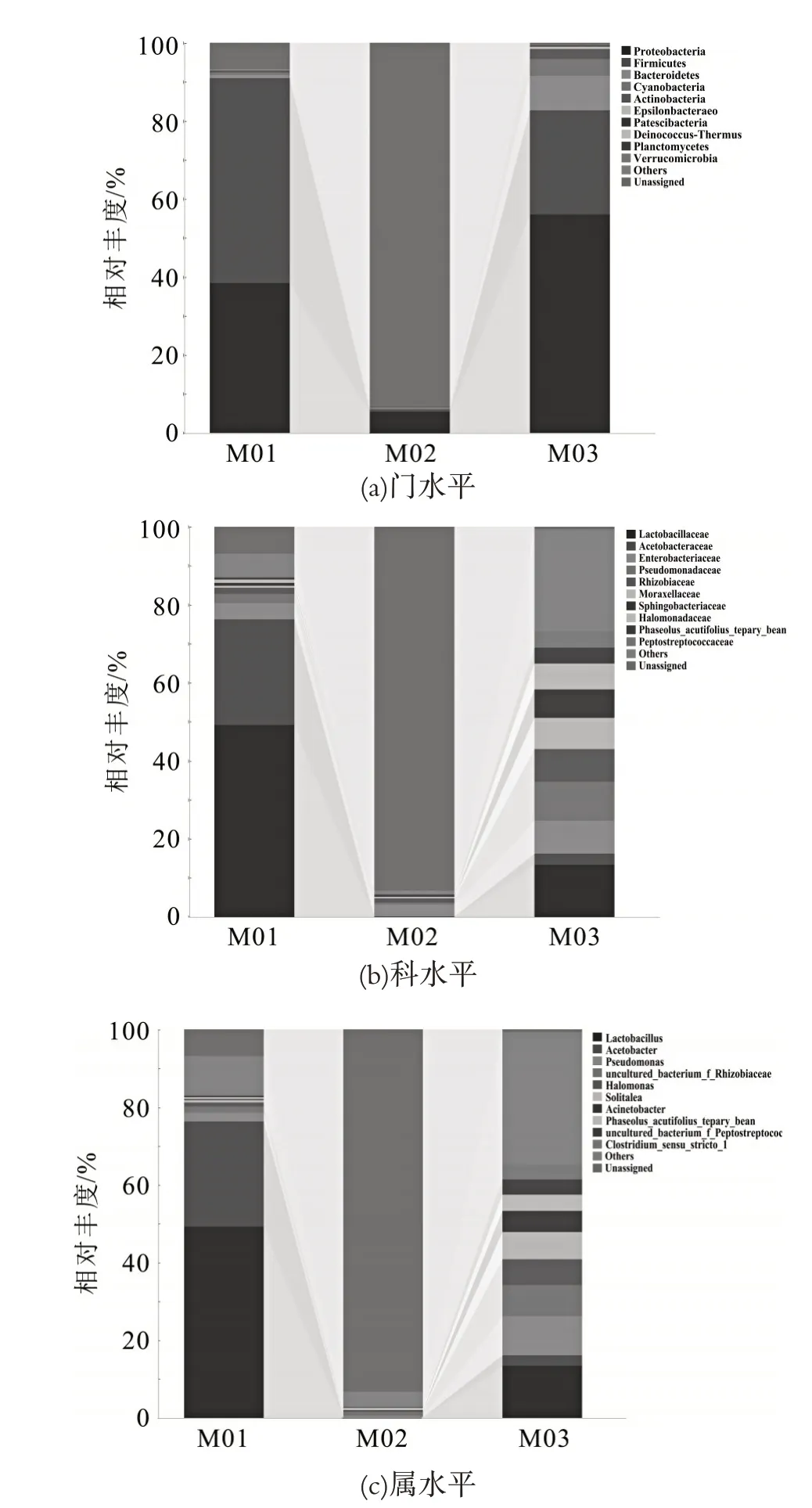
图4 不同水平下物种分布柱状图
综上所述,在门水平上,3 种牦牛奶渣中样品M01中厚壁菌门(Firmicutes)丰度最大,其次是变形杆菌门(Proteobacteria),样品M02、M03 中变形杆菌门(Pro⁃teobacteria)为优势菌种;在科水平上,样品M01、M03中乳酸杆菌科(Lactobacillaceae)丰度最大,样品M02中肠杆菌科(Enterobacteriaceae)为其优势菌种。在属水平上,样品M01、M03 中优势菌群都是乳酸菌属(Lactobacillus),与万金敏[12]、南志强[13]、刘洁洁[14]等众多研究发酵乳制品中优势菌群的学者的研究结果一致,但样品M02 中无显著的优势菌群。三水平下的细菌群落分布均说明M03 样品可检测出的菌种相较另外两种而言,含量与种类均更丰富多样,而样品M02 中极大部分微生物是检出未识别状态。分析由于其自然环境、地理位置的不同,各个地区制备传统发酵乳制品所采用的牦牛奶、加工工艺等方面也存在差异[15]。因此,造成不同地区牦牛奶渣制品中微生物结构、物种组成与含量存在一定差异。
3 结论
本研究采用高通量测序法,对林芝市售3 种牦牛奶渣中的细菌多样性进行分析,共获得有效序列224 106 条,OTU 数为235;对不同市售牦牛奶渣产品中细菌群落组成的分析发现,牦牛奶渣样品中细菌门有15 种,厚壁菌门(Firmicutes)和变形杆菌门(Proteo⁃bacteria)丰度最高;细菌科有89 种,乳酸杆菌科(Lac⁃tobacillaceae)的丰度最高;细菌属有156 种,乳酸菌属(Lactobacillus)和醋酸杆菌属(Acetobacter)为优势菌属。另外,细菌群落丰富度和多样性高低、分布均匀性上来说,M03样品>M01样品>M02样品。
