基于多工具的山羊circRNA-miRNA靶向关系分析
2021-11-18许铭洙刘泽林陈明新
陶 虎,杨 娟,许铭洙,刘泽林,张 年,刘 洋,陈明新*
(1.湖北省农业科学院畜牧兽医研究所/动物胚胎工程及分子育种湖北省重点实验室,湖北武汉 430064;2.西南科技大学生命科学与工程学院,四川绵阳 621010)
CircRNA 是一类以共价键首尾连接成环的非编码RNA 分子,在结构上不包含5'端帽子和3' poly A 尾。自1976 首次发现以来,circRNA 一直被认为是转录的副产物,并无生物学功能[1]。随着高通量测序技术的兴起,大量circRNA 在各类生物体内被发现,研究人员也逐渐揭示了circRNA 在各种生命过程中发挥的重要调控作用。CircRNA 的环状结构具有很高的稳定性,可以作为各类癌症的标志物[2];Piwecka 等人[3]研究发现,circRNA Cdr1as 能调控哺乳动物大脑中的miRNA 水平,缺失会导致小鼠出现神经元异常和行为障碍;山羊中新发现的circ_ZCCHC24 可以促进山羊卵泡颗粒细胞的增殖[4]。CircRNA 主要有4 种作用机制:1)竞争性吸附miRNA,调控其靶基因的表达[5];2)调节RNA 结合蛋白,可作为蛋白分子的海绵体[6];3)与RNA 聚合酶II 相互作用,调控基因转录[7];4)编码蛋白质发挥功能[8]。本研究前期以高低繁殖力的麻城黑山羊为研究对象,分析高低产羔数山羊排卵前卵泡中circRNA 的表达情况,筛选出差异表达的circ_0008219 作为调控山羊卵泡发育的候选因子,拟下一步开展深入研究[9]。
MiRNA 靶标预测工具众多,在动物中常用的工具包括miRWalk[10]、PicTar[11]、miRanda[12]、Targetscan[13]、RNAhybrid[14]、PITA[15]、miRmap[16]、DIANA-microT[17]和RNA22V2[18]等,但这些工具主要针对人、小鼠等热点物种开发,其中一些预测工具参数设置有限或无法输入序列,只能通过已有数据库中选取相应ID 进行分析,这对于山羊一类家畜则无法进行靶向关系的准确预测。
本研究选择5 款支持输入序列分析的本地化miRNA靶标预测工具(miRanda、targetscan、RNAhybrid、PITA 和RNA22V2)来进行数据分析,利用山羊circ_0008219 全长序列和山羊所有miRNA 种子序列信息进行二者靶向关系的联合预测。通过对上述结果取交集,获得了10 个潜在的候选miRNA,选取其中5 个8mer site 类型进行后续研究。利用双荧光素酶报告实验发现miR-130b-5p、miR-423-3p、miR-326-3p 和miR-671-5p 可与载体靶序列结合。本研究表明利用支持序列分析的多工具对miRNA 靶标进行联合预测,具有较高的阳性率,可为circRNA 与miRNA 靶向关系预测提供新的思路和方法。
1 材料与方法
1.1 实验动物 在湖北省农业科学院种羊场选择12 月龄的成年母羊,采集血液样品置于抗凝采血管中迅速带回实验室。样品利用基因组DNA 提取试剂盒(天根公司)进行提取,-20℃保存备用。
1.2 主要试剂 聚合酶PrimeSTAR®Max DNA Polymerase购自中国的宝生物工程(大连)有限公司。Ecos感受态细胞和pmirGLO 双荧光表达载体由湖北省农业科学院畜牧兽医研究所保存;pMD18-T Vector 购自宝生物工程(大连)有限公司;限制性内切酶PmeI 和NheI、T4 DNA 连接酶购自NEB 公司;凝胶回收试剂盒和质粒提取试剂盒购自Omega 公司;Dual-Luciferase®Reporter Assay System 购 自Promega 公 司;miRNA mimics 和NC 由广州锐博生物技术有限公司合成;山羊皮肤成纤维细胞由本实验室分离培养;引物序列由北京奥科生物科技公司合成。
1.3 实验方法
1.3.1 软件的下载与安装 miRanda v3.3a、targetscan 7.2、RNAhybrid 2.1.2、PITA 1.6 和RNA22 V2 5 款miRNA靶标预测工具均安装于linux 系统中。
1)miRanda
miRanda 的下载地址:http://cbio.mskcc.org/micror na_data/miRanda-aug2010.tar.gz
miRanda 的安装命令如下:
tar -xzvf miRanda-aug2010.tar.gz
cd/path/to/miRanda-3.3a4
./configure --prefix=/path/to/miRandada-aug2010
sudo make
sudo make install
2)Targetscan
Targetscan 的下载地址:http://www.targetscan.org/vert_72/vert_72_data_download/targetscan_70.zip
Targetscan 的安装命令如下:
unzip targetscan_70.zip
cd/path/to/targetscan_70
perl targetscan_70.pl
其中,targetscan_70.pl 为perl 脚本文件,可在perl 语言环境下运行。
3)RNAhybrid
RNAhybrid 的下载地址:https://bibiserv.cebitec.uni-bielefeld.de/applications/rnahybrid/resources/downloads/RNAhybrid-2.1.2.tar.gz
RNAhybrid 的安装命令如下:
tar -xzvf RNAhybrid-2.1.2.tar.gz
cd/path/to/RNAhybrid-2.1.2
./configure
sudo make
sudo make install
4)PITA
PITA 的下载地址:http://genie.weizmann.ac.il/pubs/mir07/64bit_exe_pita_prediction.tar.gz
PITA 的安装命令如下:
tar -zxvf 64bit_exe_pita_prediction.tar.gz
cd/path/to/ 64bit_exe_pita_prediction
./configure
sudo make
sudo make install
5)RNA22V2
RNA22V2 的下载地址:https://cm.jefferson.edu/rna22/Interactive/remoteRNA22v2.zip
RNA22V2 的安装命令如下:
unzip remoteRNA22v2.zip
cd/path/to/remoteRNA22v2
java RNA22v2
将miRNA 种子序列和circ_0008219 全长序列文件导入Parameters.properties 和RNA22v2.class 所在文件夹中,打开Parameters.properties 文件,根据注释信息修改后运行程序。
1.3.2 原始数据整理 数据均储存于txt 文档内,miRanda、RNAhybrid、PITA 和RNA22V2 4 款工具所需数据为FASTA 格式,包括2 个文件:山羊所有miRNA 种子序列文件和circ_0008219 全长序列文件。其中,山羊miRNA 的种子序列文件可从miRbase 数据库中下载,下载网址为ftp://mirbase.org/pub/mirbase/CURRENT/mature.fa.gz。Targetscan 工具的数据格式有别于其他软件,miRNA 序列格式为“miRNAID 种子序列物种ID”,circ_0008219 的全长序列格式为“基因ID 物种ID 序列。”
1.3.3 数据分析 在linux 系统中,运行安装好的5 款工具,对miRNA 的种子序列文件和circ_0008219 的全长序列文件进行处理。程序运行方法如下:
1)miRanda 程序运行命令如下:
miranda -sc 140 -en -7 miRNA.txt circ_0008219.txt-out miRanda_result.txt
其中,-sc 指分数阈值,-en 指能量阈值。
2)Targetscan 程序运行命令如下:
Perl targetscan_50.pl miRNA.txt circ_0008219.txt targetscan_result.txt
3)RNAhybrid 程序运行命令如下:
RNAhybrid -s 3utr_human -t circ_0008219.txt -q miRNA.txt
4)PITA 程序运行命令如下:
perl pita_prediction.pl -utr circ_0008219.txt -mir miRNA.txt -prefix PITA_result
5)RNA22V2 程序运行命令如下:
java RNA22v2
上述5 款工具所得结果在Excel 2019 中进行数据汇总,然后利用Calculate and draw custom Venn diagrams工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)绘制韦恩图、取交集。
1.3.4 载体构建 根据circ_0008219 的全长序列设计合成包含PmeI 和NheI 酶切位点的引物对Full-PF、Full-PR(表1)。以山羊DNA 为模板,利用上述引物进行PCR 扩增,产物长度为2 045 bp。PCR 扩增反应程序:98℃预变性2 min;98℃变性10 s,58℃变性10 s,72℃变性20 s,共35 个循环;72℃延伸7 min;16℃保存。琼脂糖凝胶电泳检测PCR 产物,利用DNA快速纯化回收试剂盒回收目的片段。pmirGLO 质粒经双酶切线性化后切胶回收。上述2 种胶回收产物利用T4 连接酶16℃连接2 h。连接体系加入50 μLEcos感受态细胞,均匀涂布于LB 固体培养基(氨苄)上,于37℃培养箱中过夜。筛选阳性克隆送北京奥科生物科技公司测序验证,提取重组载体质粒备用。

表1 引物与探针信息
1.3.5 细胞培养与转染 山羊皮肤成纤维细胞由湖北省农业科学院畜牧兽医研究所分离保存,细胞培养基为DMEM-F12 中加入10% 的胎牛血清,培养箱设置为5%CO2、37℃。待细胞培养至80%的融合度,使用胰酶消化后均匀接种于24 孔细胞培养板中。每孔加入2 μL 的lipofectmin 3000、0.2 μL mimics 和1/20 μgpmir-8219 双荧光载体质粒共转染于细胞,24 h 后提取蛋白,利用Dual-Luciferase®Reporter Assay System 在多功能酶标仪中检测相对荧光活性。
2 结果
2.1 与circRNA 靶向结合的miRNA 预测 利用miRanda、targetscan、RNAhybrid、PITA 和RNA22V2 等5 款miRNA 靶标预测工具来进行山羊circ_0008219 序列中miRNA 靶位点的预测,结果发现miRanda 有68 个miRNA 结合,targetscan 有308 个,RNAhybrid 有47 个,PITA 有174 个,RNA22V2 有333 个,上述结果取交集后,共得到10 个miRNA 与circ_0008219 结合(图1A),13 个结合位点信息(图1B)。

图1 circ_0008219 与miRNA 互作结果
如表2 所示,预测结果中包含8mer site 和7mer-m8 site 2 种匹配类型,属于8mer site 类型的有:miR-423-3p、miR-330-5p、miR-326-3p、miR-671-5p 和miR-130b-5p。属于7mer-m8 site 类型的有:miR-485-5p、miR-483、miR-330-5p、miR-326-3p、miR-130b-5p、let-7g-3p、miR-1343 和miR-29b-5p。此外,miR-330-5p、miR-326-3p 和miR-130b-5p 同时具有8mer site 和7mer-m8 site 2 种匹配类型。8mer site 的特异性高,假阳性率低于7mer-m8。

表2 miRNA 匹配类型
2.2 山羊circ_0008219 双荧光素酶载体构建 为了验证预测的circRNA-miRNA 的靶向关系,利用酶切重组法构建了circ_0008219 的pmirGLO 双荧光素酶重组载体。利用PCR 扩增circ_0008219 的全长序列,长度为2 045 bp。凝胶电泳结果显示,片段长度与预期结果相符,无杂带(图2)。

图2 山羊circ_0008219 基因全长序列的扩增
山羊circ_0008219 全长序列的PCR 回收产物和双荧光素酶载体pmirGLO 质粒(图3)经PmeI 和NheI双酶切后,琼脂糖凝胶电泳后切胶回收。利用T4 连接酶连接线性化pmirGLO 质粒和circ_0008219 序列的双酶切回收产物,随后克隆转化入Ecos感受态细胞,培养过夜后挑选阳性克隆进行鉴定。将阳性菌液进行测序,序列比对结果表明,目的片段与模板序列完全一致,该载体质粒命名为pmir-8219。
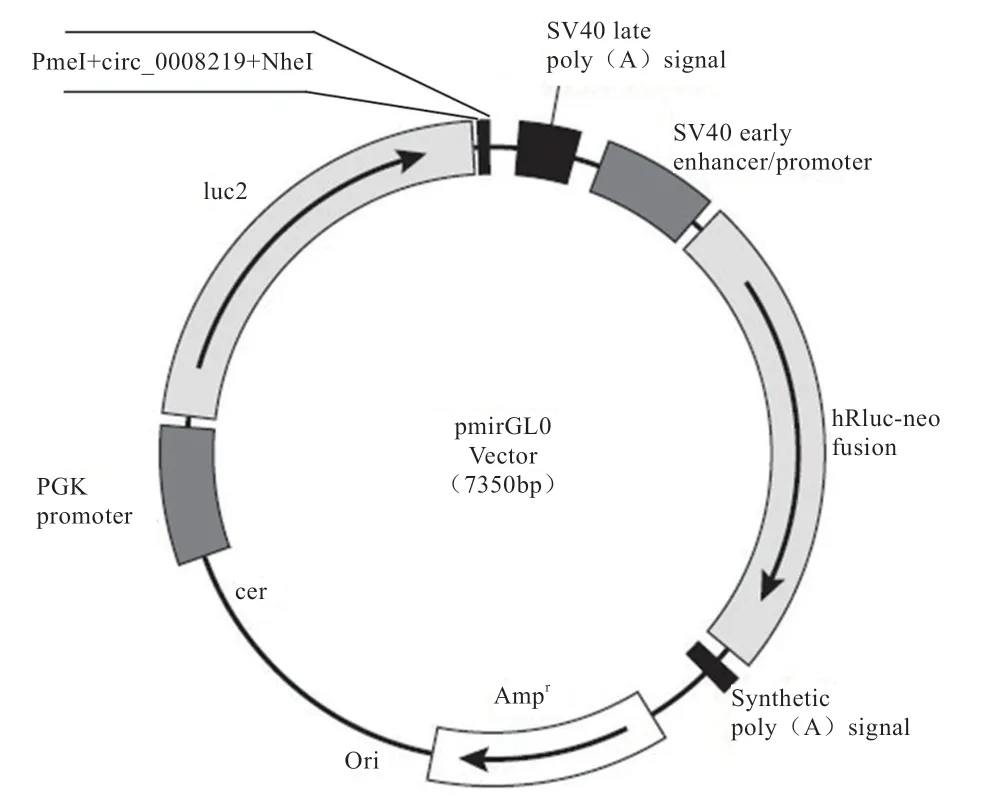
图3 双荧光素酶载体pmirGLO 的模式图
2.3 候选miRNA 的验证 选取8mer site 匹配类型的miR-423-3p、miR-330-5p、miR-326-3p、miR-671-5p和miR-130b-5p 等5 个miRNA,合成mimics 和NC,与pmir-8219 载体质粒共转染于山羊皮肤成纤维细胞,检测pmir-8219 质粒的荧光活性变化。结果发现,miR-130b-5p 可以抑制pmir-8219 质粒的荧光活性(P<0.01),miR-423-3p、miR-326-3p 和miR-671-5p 则发挥抑制作用(P<0.05)(图4)。结果表明,circ_0008219 可以通过“分子海绵”作用吸附miR-130b-5p、miR-326-3p、miR-671-5p 和miR-423-3p,从而发挥调控作用。此外,也证明了利用5 款miRNA 靶标预测工具进行联合预测有较高的阳性率。

图4 候选miRNA 对pmir-8219 双荧光素酶载体荧光活性的调控
3 讨 论
近年来,非编码RNA 已成为各类生命活动中基因表达调控的明星因子,在疾病、生长发育等领域已有大量重要研究成果相继被报道[19]。CircRNA 作为ncRNA的重要成员之一,在山羊研究领域中仅在乳腺上皮细胞增殖[20]、子宫内膜容受[21]、骨骼肌发育[22]等方面有相关报道。因此,本研究前期以高繁殖力的麻城黑山羊为研究对象,分析高低产羔数山羊排卵前卵泡中circRNA的表达情况,筛选出差异表达的circ_0008219 作为候选因子拟开展深入研究[9]。
CircRNA 通过吸附miRNA 发挥调控作用是其最重要的作用形式之一。利用miRNA 靶标预测工具进行生物信息学分析是研究的关键步骤。现有的miRNA 靶标预测工具有数十种之多,分为离线型和在线型[23]。预测工具主要通过4 个方面信息来进行miRNA 的靶基因预测:1)miRNA 与靶基因之间的互补配对关系;2)靶基因的热力学稳定性;3)靶基因的物种保守性特征;4)机器学习方法[24]。但这些预测工具大多针对人、小鼠等物种开发,仍存在无法输入已知序列、参数设置较为有限、只能输入miRNA 和靶基因ID 等问题,这都导致了在进行家畜miRNA 研究过程中,只能以人或小鼠的基因来进行预测,如果同源性不高,将导致预测结果出现假阳性。
CircRNA 最常见的调控机制——“miRNA 分子海绵”需要进行互作位点的预测。CircRNA 与miRNA 互作关系预测仍然遵循miRNA 种子序列中5'端2-7nt 与circRNA 序列形成Watson-Crick 配对这一重要原则。根据上述配对的匹配情况可分为以下形式:1)8mer site:miRNA 第2-8 nt 与circRNA 序列完全配对,circRNA 序列对应miRNA 第1 nt 为A;2)7mer-m8 site:miRNA第2~8 nt 与circRNA 序列完全配对;3)7mer-1A site:miRNA 第2~7 nt 与circRNA 序列完全配对,circRNA序列对应miRNA 第1 nt 为A;4)6mer site:miRNA 第2~7 nt 与circRNA 序列完全配对[25]。匹配类型的特异性为:8mer site>7mer-m8 site>7mer-1A site>6mer site。在分析预测结果时,尽量选择8mer site 类型,以提高实验成功率。
CircRNA 与miRNA 互补配对的紧密程度直接影响其作用机制。Cdr1as 是在哺乳动物大脑中高表达的一种circRNA,可吸附miR-7 发挥重要作用[26]。随后,Piwecka 等人[3]发现Cdr1as 可以结合另一种miR-671,且miR-7 下调表达,miR-671 上调表达。Cdr1as与miR-7 不是完全互补配对,导致Cdr1as 与miR-7 结合后不会被Ago2 蛋白剪切降解,起到稳定miR-7 的作用;Cdr1as 与miR-671 完全互补配对,导致Cdr1as与miR-671 结合后被Ago2 蛋白剪切降解,无法发挥吸附作用。该研究揭示了circRNA 作为ceRNA 作用的新机制,提示研究者在选择与circRNA 靶向作用的miRNA 时,需要考虑结合的紧密程度,根据circRNA和miRNA 的表达情况,明确circRNA 的作用机制。
4 结 论
本研究选择了miRanda、targetscan、RNAhybrid、PITA 和RNA22V2 等5 款支持输入序列分析的本地化miRNA 靶标预测工具来进行山羊circ_0008219 序列中miRNA 靶向关系的联合预测,共得到10 个候选miRNA,13 个结合位点信息。验证实验选择了结合类型为8mer 的5 个miRNA,利用双荧光素酶报告系统检测发现miR-130b-5p、miR-423-3p、miR-326-3p 和miR-671-5p 可与载体靶序列结合。本研究表明利用支持序列分析的多工具进行联合预测miRNA 靶位点具有较好的阳性率,为circRNA 与miRNA 靶向关系预测提供了新的思路和方法。
