细菌分型方法的研究进展
2021-09-30吴雨辰胡燕燕
吴雨辰, 胡燕燕, 张 嵘
细菌分型是区分菌株并研究其相关性的手段,是疫情调查、监测和系统发育研究的重要工具。早期的分型技术基于被测细菌的表型,如抗生素敏感性(耐药谱分型)、对噬菌体的敏感性(噬菌体分型)和表面抗原(血清分型)等进行区分。随着分子生物学的进步,基于DNA的分型方法得到了普及。相比于检测表型,DNA的稳定性使得测定结果具有高度的可重复性。后期质谱与振动光谱的发展使得检测更为快速和简便。本文将对常用细菌分型技术原理、应用以及优缺点进行简要阐述。
1 分子生物学技术
1.1 脉冲场凝胶电泳(PFGE)
PFGE技术主要原理是用适当的核酸内切酶对细菌的基因组序列进行消化[1],产生若干限制性片段,在脉冲电场中将大小片段分离,并进行应用分析。因其重复性好、分辨率高、结果稳定且易于标准化,被认为是细菌分型的“金标准”[2]。在耐万古霉素肠球菌暴发的流行病学调查中,PFGE相比于单核苷酸多态性(single nucleotide polymorphism, SNP)分析和基质辅助激光解吸电离飞行时间质谱法(matrix-assisted laser desorption/ionization time of flight mass spectrometry,MALDI-TOF MS)分析有着更高的分辨率[3]。在测定布鲁菌分离株的遗传相似性方面也比PCR-限制性片段长度多态性(restriction fragment length polymorphism,RFLP)法更优[4]。目前PFGE已广泛应用于流行病学、微生物学和进化生物学的研究,同时促进了细菌分型的PFGE协议标准化。CDC已发布统一的PFGE协议(https://www.cdc.gov/hai/pdfs/labsettings/unified_pfge_protocol.pdf),用于对革兰阳性细菌进行分型。国际食源性疾病监测网络PulseNet International在其网站(http://www.pulsenetinternational.org/protocols/)上发布了重要食源性病原体的PFGE协议,并在CDC PulseNet网站上发布了更新版本(https://www.cdc.gov/pulsenet/pathogens/pfge.html)[5]。 不 过 PFGE实验周期长,操作要求高,并且缺乏对几乎相同大小条带的分辨能力[6]。
1.2 限制性片段长度多态性(RFLP)
利用限制性内切酶(restriction endonuclease)切割目的基因片段,再利用标记探针对其中部分片段进行Southern印迹分析,就可以通过简化的数据进行菌株的区分并分析遗传相关性。插入序列(insertion sequences,IS)-RFLP模式主要是通过限制性内切酶和核酸探针的特定组合进行鉴定[7]。其中,使用IS6110探针进行RFLP分析,已是结核分枝杆菌复合体分型的“金标准”方法[8-9]。但RFLP对样品纯度要求较高,用量较大,RFLP的多态信息含量低且依赖于限制性内切酶的种类与数量。与IS-RFLP原理类似的核糖体分型根据核糖体操纵子基因的保守序列设计探针,作为艰难梭菌分型的重要手段[10]。
1.3 随机扩增多态性DNA(random amplified polymorphic DNA, RAPD)
该技术通过设计一些非特异性小片段随机引物,其中部分可与DNA序列结合并通过PCR扩增,对产物进行多态性分析,从而得出细菌的分型鉴定结果。在对28株泛耐药肺炎克雷伯菌的分子分型中,RAPD-PCR揭示了18种相似度≥80%的不同分离株,且分型结果与耐药模式显著相关[11]。在铜绿假单胞菌的分型中,反应条件标准化的RAPDPCR技术,比基于宏基因组的PFGE技术分辨率更高[12]。RAPD对艰难梭菌的分型分辨率也高于其他分子分型方法[13]。
1.4 重复序列PCR(repetitive element PCR, rep-PCR)
一些短重复序列广泛分布于细菌基因组中,尽管功能未知,但可以作为细菌的指纹图谱。通过扩增这些短重复序列,可以揭示基因组间的差异。常见的重复序列家族有基因外重复回文序列(REP)、肠杆菌基因间重复一致序列(ERIC)和BOX片段。有研究发现rep-PCR的初步分型可作为变形链球菌多位点序列分型(multilocus sequence typing,MLST)的预筛选工具,这种预筛选可以为下一步应用更高级的分子技术节约成本[14]。在对耐甲氧西林金黄色葡萄球菌(MRSA)的分型中,基于rep-PCR的DiversiLab分型的鉴别力高于spa分型,但低于PFGE分型[15]。
1.5 多位点可变数目串联重复分析(multiple-locus variable-number tandem repeat analysis, MLVA)
可变数目串联重复序列(variable number of tandem repeats, VNTR)是基因组中一类较短的序列重复、首尾相连的序列,MLVA通过PCR与电泳技术,分析多种VNTR基因座的数量进行基因分型。基于MLVA的多重PCR技术在大肠埃希菌与志贺菌快速分型方面,优于MLST和DiversiLab rep-PCR[16]。在鼠疫疫情监测中,MLVA方案能够获得与基于SNP分析基本相同的系统发育关系,在基因分型中也具有较高的判别力[17]。在产超广谱 β内酰胺酶(ESBL)的大肠埃希菌的流行病学分型中,7通用位点大肠埃希菌MLVA(GECM-7)与PFGE分型结果的相关性很好[18]。尽管MLVA是一种快速、简便、廉价且可重现的高分辨率基因分型方法,该方法也依旧存在一些缺陷,如容易受位点本身以及酶切片段长度的影响。另外,VNTR基因座在细菌基因组中并不总是常见的,这限制了MLVA的应用[7]。
1.6 高分辨率熔解分析技术(high-resolution melting analysis, HRM)
HRM是实时PCR与熔解曲线分析相结合的一种技术。与特定荧光染料结合的双链DNA在升温过程中会解链为单链,从而使监测的荧光信号发生变化。不同的DNA长度,GC含量与碱基差异都会形成不同的熔解曲线。在对MRSA的基于PCR测序鉴定的12种spa分型中,HRM识别出了其中的11种[19]。有研究人员建立了多重HRM分析方法以区分结核分枝杆菌复合体的罕见亚型,其分型结果与商用试剂盒结果达到了100%一致性[20]。
1.7 PCR-RFLP
结合了PCR技术的RFLP分析弥补了单纯RFLP的一些缺点。由于酶切完成后不进行探针杂交,而是通过PCR扩增后将特定片段进行电泳分离分析,样本DNA可直接来源于人体或环境并且操作步骤得以简化[7]。基于spa和coa基因的PCR-RFLP技术对金黄色葡萄球菌的辨别指数达到了0.83,高于单基因座的结果[21]。在肺炎链球菌的分子血清分型中,RFLP方法比其他血清学方法具有更好的预期特异性[22]。
1.8 扩增片段长度多态性(amplified fragment length polymorphism, AFLP)
AFLP的原理是基因组DNA先用限制性内切酶切割,然后将双链接头与DNA酶切片段的黏性末端相连,以接头序列和相邻酶切位点序列作为引物结合位点,加入互补引物之后进行PCR扩增与电泳分析。该方法具有多态性丰富,无需预知被测细菌的全基因组序列,不受环境影响,无复等位效应等优点。在耐万古霉素肠球菌的院内暴发感染调查中,AFLP方法的系统发育分析与全基因组测序(whole-genome sequencing, WGS)接近,在排除相关性方面更有优势[23]。该方法对接头序列设计要求较高,因为需要考虑引物和限制性内切酶序列的联系[24]。
1.9 MLST
MLST通过检测若干个管家基因的内部片段序列的核苷酸变异,分析不同样本的等位基因多样性,并将每组不同等位基因的排列组合定义为一种序列型(sequence type, ST),通过ST来对细菌进行分型,是当下最流行的基因分型方法之一[25]。MLST的主要缺点是需要知晓待测微生物的基因组序列信息,以便决定能否使用MLST以及选择管家位点。除此之外,亲缘关系密切菌株的变异也难以通过MLST检出。鉴于MLST的局限性,基于相似原理开发的其他方法如通过检测VNTR的MLVA可以获得与MLST相似的结果[26]。
1.10 成簇规则间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)
CRISPR位点包含多个直接重复(DR)序列,它们之间由可变间隔序列分隔。DR序列通常是保守的,而间隔序列则是多变的,且DR和间隔序列的数量不定,它们构成了一组独特的DNA序列,为分型提供了基础。CRISPR可用于肠炎沙门菌不同亚型的鉴别,且与全基因组序列分型结果有着良好的一致性[27]。在幽门螺杆菌的分型中,CRISPR-毒力分型结果与高辨别指数的RAPD方法相同,且可重复性好[28]。该方法仍存在一些局限性,比如部分菌株的分型仍存在争议[29],在某些目标菌种中不存在CRISPR系统,以及在某些情况下位点的低变异性,可能会影响高分辨率分型结果。
1.11 WGS
WGS是基因分型的最终方案,有着高度的可重复性和区分度。用WGS进行菌株分型和克隆亲缘关系测定,通常是基于SNP分析,与对照菌株的参考基因组进行比较,或通过与预先定义的一组核心基因(核心基因组MLST)逐个基因比较进行分型,即全基因组测序的单核苷酸多态性分型(whole genome-based single nucleotide polymorphisms,wgSNP)和核心基因组多位点序列分型(core genome multilocus sequence typing,cgMLST)[30]。目前基于wgSNP的服务PathoBacTyper已经可以实现400多种致病细菌的识别与基因分型,包括具有相同PFGE模式菌株间的分型[31]。cgMLST不同于传统MLST检测7个管家位点,它通过检测核心基因组位点的差异,比对成百上千个基因进行分型,分辨率更高[32]。由于DNA相比于蛋白质具有更稳定的性质,WGS的结果比常规分子生物学分型结果更具可重复性,且实验室间的结果比较也更易实现。目前WGS技术应用的主要阻碍在于高昂的成本和较长的分析时间。
2 质谱与振动光谱技术
2.1 MALDI-TOF MS
该方法原理是通过激光脉冲照射待测结晶混合物,使其离子化解吸并电离,随后电场将离子加速,测量离子的飞行时间,根据质荷比转化为质谱图,进而分型[33]。该技术已广泛应用于许多病原体的鉴定与分型中。MRSA的质谱分型结果准确度与PFGE和噬菌体开放阅读框分型(POT)相当[34]。鲍曼不动杆菌的分型结果与MLST基本一致,系统发育结果与rep-PCR结果显示良好的相关性[35]。不过质谱法对于某些细菌的分型结果容易出现偏差[36-37]。优化对特异性峰的识别能够提高判断准确性,提升数据库的质量也能提高鉴定成功率[38]。MALDI-TOF技术与传统方法最显著的差异体现在时间成本和消耗物成本上。尽管仪器与维护成本很高,综合其准确性、时间与成本效益,仍值得推广。
2.2 傅里叶变换红外光谱(Fourier transform infrared spectroscopy, FTIR)
微生物不同的组织成分在红外光照射下发生振动,经傅里叶变换技术处理后,根据其特定的红外吸收峰可用于分型,其中峰值强度提供了生化分子含量的定量信息,而峰值位置则提供了与细菌类型有关的定性信息[39]。在肺炎克雷伯菌的分型中,以WGS作为参考,FTIR与MALDI-TOF MS相比,表现出了更高的分型能力[40]。阴沟肠杆菌的FTIR分型结果与基于SNP的聚类分析和ST分型结果有良好的一致性[41]。在院内流行革兰阴性病原体的分型中,以MLST与PFGE为参考,FTIR能准确地将同一ST型的肺炎克雷伯菌、铜绿假单胞菌和阴沟肠杆菌的分离株聚类[42]。
FTIR主要基于细菌的表型特征进行分析,由于细菌存在广泛的种内多样性,对于待检样本需要强调标准化的预处理流程(包括菌株培养、样本制备和光谱采集等)[43]。同时因为表型多变,且每个波段缺乏相应的具有特定鉴别意义的生物标记,有研究倾向于检测相对稳定的基因组FTIR光谱[44-45],或是利用人工神经网络训练模型以提高分辨率[46]。
2.3 表面增强拉曼光谱(surface enhanced Raman spectroscopy, SERS)
光在发生散射时,有一部分强度极弱的光会发生频率的改变,即拉曼散射。拉曼光谱是基于拉曼散射效应,检测分子振动能级来对分子结构进行分析的方法。目前多采用SERS技术放大原有的光谱信号以便于分析[47]。SERS结合微流控技术对MRSA的分型结果与MLST的结果有着良好的一致性[48]。在对喹诺酮类非敏感大肠埃希菌分型方面,与MLST预分型结果相比,SERS 结合主成分分析(PCA)算法实现了高度相似光谱之间的区分[49]。
拉曼光谱是一种快速、灵敏且稳定的细菌检测技术。拉曼光谱法比红外光谱法有一些优势,例如水的干扰较小且谱带较窄等。然而,拉曼光谱也需要昂贵的设备,并且操作更为复杂。常规分型技术的比较见表1。

表1 细菌分型方法的评价
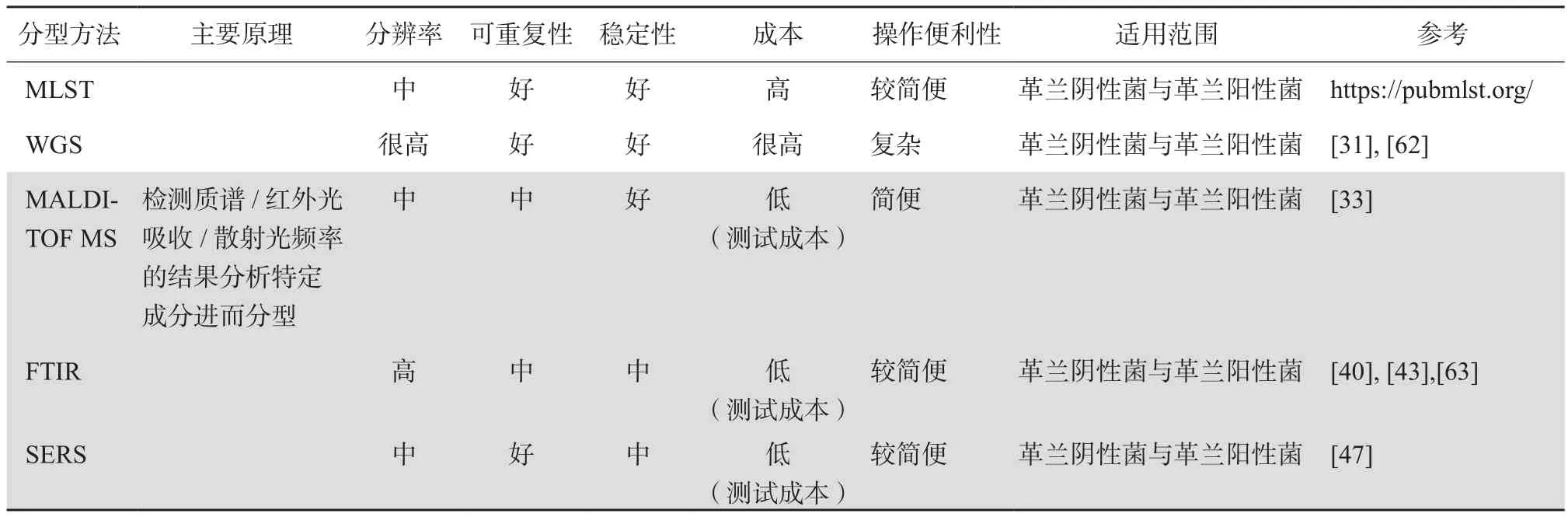
续表1
3 总结
尽管特定的分型方法可能具有较高的分辨能力和良好的可重复性,但方法的复杂性、结果的解释以及构思并使用该方法所涉及的成本有可能超出实验室的能力。因此,分型方法的选择应取决于技术水平、实验室资源以及研究的目的和范围。
随着测序技术的进步与谱学方法的拓展,新技术的应用使得与细菌有关的检测更为准确,这有助于流行病学的研究与临床实验室的工作开展,促进公共卫生与健康事业的进步。
