茶树ZF-HD转录因子基因家族的鉴定及表达分析
2021-08-02庞丹丹刘玉飞田易萍孙云南陈林波
庞丹丹 刘玉飞 田易萍 孙云南 陈林波


摘要:【目的】搜索鑒定茶树ZF-HD(Zinc finger homeodomain)转录因子基因家族成员,并对其进行生物信息学分析及在不同组织及非生物胁迫和激素处理下的表达模式,为茶树ZF-HD基因家族的功能研究及茶树遗传性状改良提供理论依据。【方法】通过隐马尔可夫模型预测和序列比对从茶树基因组中筛选鉴定出茶树ZF-HD基因家族成员,利用在线生物信息学分析软件对其基因结构、启动子元件,以及编码蛋白的理化性质、氨基酸序列、结构特征、进化关系等进行预测分析,并基于转录组测序(RNA-Seq)数据分析其在不同组织及在干旱胁迫、盐胁迫和茉莉酸甲酯(MeJA)处理下的表达模式。【结果】从茶树基因组中鉴定出17个ZF-HD基因家族成员(CsZHD1~CsZHD17),其编码区(CDS)序列长度为369~2187 bp,编码122~728个氨基酸残基,蛋白分子量13.51~80.42 kD,理论等电点为6.09~9.19,均属于亲水性不稳定蛋白,蛋白二级结构均由β-转角、延伸链、α-螺旋和无规则卷曲构成。除CsZHD2、CsZHD5和CsZHD7蛋白定位于细胞质,CsZHD9蛋白定位于细胞外,其他13个蛋白均定位于细胞核。仅CsZHD2、CsZHD7、CsZHD9、CsZHD10和CsZHD12基因含外显子和内含子,其他12个CsZHDs基因均无内含子。除CsZHD7蛋白具有2个ZF-HD_dimer结构域外,其他16个蛋白均具有1个ZF-HD_dimer结构域。CsZHD1~CsZHD17蛋白具有2~5个保守基序(motif),其中motif 1和motif 3为共有的保守基序。茶树ZF-HD家族蛋白可分为5个亚族(ZHD Ⅰ、ZHD Ⅱ、ZHD Ⅲ、ZHD Ⅳ和MIF),较拟南芥少了ZHD Ⅴ亚族。除CsZHD2、CsZHD12和CsZHD17基因在8个组织中不表达或表达量极低外,其他14个CsZHDs基因在8个组织中呈差异性表达;除CsZHD2、CsZHD12和CsZHD17基因外,其他15个CsZHDs基因在顶芽或花中表达量较高。在干旱胁迫和盐胁迫处理下,除CsZHD1、CsZHD2、CsZHD5、CsZHD12、CsZHD14和CsZHD17基因的表达量始终处于较低水平外,其他CsZHDs基因均呈不同的变化趋势;在MeJA处理下,除CsZHD2、CsZHD5和CsZHD12基因表达量极低外,多数CsZHDs基因呈下调表达的趋势。【结论】从茶树基因组中鉴定出17个ZH-HD基因家族成员,其编码蛋白具有保守的锌指结构域和同源异型盒结构域;茶树与拟南芥ZH-HD基因家族相比缺少ZHD Ⅴ亚族;CsZHDs基因的表达具有组织特异性,且大多数成员的表达受非生物胁迫和MeJA的影响。
关键词: 茶树;ZF-HD基因家族;鉴定;生物信息学;表达分析;非生物胁迫;茉莉酸甲酯(MeJA)
中图分类号: S571.103.53 文献标志码: A 文章编号:2095-1191(2021)03-0632-09
Identification and expression analysis of ZF-HD transcription factor gene family in Camellia sinensis
PANG Dan-dan, LIU Yu-fei, TIAN Yi-ping, SUN Yun-nan, CHEN Lin-bo*
(Tea Research Institute, Yunnan Academy of Agricultural Sciences/Yunnan Provincial Key Laboratory for Tea Science/Yunnan Technology Engineering Research Center of Tea Germplasm Innovation and Supporting
Cultivation, Menghai, Yunnan 666201, China)
Abstract:【Objective】Identification and analysis of ZF-HD(zinc finger homeodomain) gene family members by bioinformatics methods and detection of their expression patterns under different tissues, abiotic stresses and hormone induction were conducted, which laid theoretical basis for the functional study of tea plant ZF-HD(ZFD) family gene and improvement of tea genetic traits. 【Method】ZF-HD gene family members of tea plant were identified from tea plant genome by HMMER program prediction and sequence alignment. A variety of bioinformatics analysis tools were used to analyze gene structure, promoter elements, and the physical and chemical properties,amino acid sequences,structural characteristics, evolutionary relationships, of the encoded protein. Based on RNA-Seq data,the expression patterns of members of the ZF-HD gene family in different tissues under drought stress, salt stress, and methyl jasmonate(MeJA) were analyzed. 【Result】Seventeen ZF-HD gene family members(CsZHD1-CsZHD17) were identified from tea plant genome database. Coding region sequence(CDS) length was 369-2187 bp, encoding 122-728 amino acid residues. CsZHD encoded proteins with molecular weights ranging from 13.51 to 80.42 kD and theoretical isoelectric points of 6.09 to 9.19, which were all hydrophilic and unstable proteins. The secondary structure consisted of beta-turn, extended strand, alpha-helix and random-coil state. The results of subcellular localization showed that except CsZHD2,CsZHD5,and CsZHD7 proteins were localized in the cytoplasmic and CsZHD9 was localized in the extracellular, all other CsZHD proteins were localized in the nuclears. Only CsZHD2, CsZHD7, CsZHD9, CsZHD10 and CsZHD12 contained exons and introns, and the other 12 CsZHDs had no introns. CsZHD7 protein had two ZF-HD_dimer domains and the other 16 proteins had one ZF-HD_dimer domain. CsZHD1-CsZHD17 had 2-5 conserved motifs, and motif 1 and motif 3 were shared conserved motifs in CsZHDs. Tea ZH-HD family proteins could be divided into 5 subfamilies (ZHD Ⅰ,ZHD Ⅱ,ZHD Ⅲ,ZHD Ⅳ and MIF), but the tea plant lacked the ZHD Ⅴ subfamily compared to Arabidopsis thaliana. The expression profile of the ZHD genes using RNA-Seq data showed that except for CsZHD2,CsZHD12, and CsZHD17, which were not expressed in these eight tissues or the expression levels were low, other CsZHDs genes were differentially expressed in the 8 tissues. The expression levels of CsZHD1, CsZHD2, CsZHD5, CsZHD12, CsZHD14 and CsZHD17 were always at a low level, and other CsZHDs genes showed different expressional trends under drought and salt stresses. Under MeJA treatment, most CsZHDs genes showed a downward-regulated expression except for the extremely low expression of CsZHD2,CsZHD5 and CsZHD12. 【Conclusion】Seventeen members of the ZH-HD gene family were identified from the tea tree genome, and the encoded proteins have conserved zinc finger domain and homotype box domain. Tea trees lack ZHD Ⅴ subgroups compared to the A. thaliana ZH-HD gene family; the expression of the CsZHDs gene is tissue-specific, and most members are affected by ambiotic stress and MeJA.
Key words: tea plant(Camellia sinensis); ZF-HD gene family; identification; bioinformatics; expression analysis; abiotic stress; methyl jasmonate(MeJA)
Foundation item: National Key Research and Development Program of China(2019YFD1001601); National Tea Industry Technology System Construction Project (CARS-19)
0 引言
【研究意义】转录因子(Transcription factors,TFs)在植物生长发育的调控网络中发挥重要作用(Riechmann et al.,2000),如WRKY转录因子参与植物抗逆性反应(丁忠杰,2014),ZF-HD(Zinc finger homedomain)转录因子参与生长发育、非生物胁迫和植物激素响应等多种生物学过程(张晋玉等,2017),MYB转录因子调控果实和芽叶颜色(Wei et al.,2018),MADS-box转录因子参与花器官的发育(杨方慧等,2019)等。茶树(Camellia sinensis)是一种多年生的常绿叶用经济作物,在其生长发育和逆境适应等过程中均有多种转录因子参与调控。因此,对茶树ZF-HD基因家族进行全基因组分析,以探讨该家族蛋白在器官发育和胁迫响应中的潜在作用,对揭示茶树ZF-HD蛋白的重要生物学功能具有重要意义。【前人研究进展】同源异形盒基因(Homeobox gene,HB)是一類广泛存在于动植物中的转录因子基因,其编码的转录因子具有一个约由60个氨基酸残基组成的特征性结构域,即同源异型域(Homeodomain,HD),在生长发育过程中发挥重要作用(Gehring et al.,1990)。植物中HB是一个庞大的家族,如拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa subsp. japonica)中均含100个以上的HB基因家族成员。根据HD序列、位置及其他结构域可将HB分成6个亚家族:ZF-HD(Zinc finger motif-associated HD/Homeobox)、HD-Zip(Leucine zipper-associated HD)、WOX(WUSCHEL-related homeo-box)、Bell(Bell type HD)、PHD finger(Finger domain associated to a HD)和KNOX(Knotted-related homeobox)(Ariel et al.,2007)。其中,ZF-HD家族蛋白不仅含HD结构域,还含有一个锌指结构域(Zinc-finger domain),广泛存在于陆生植物中(Hu et al.,2008)。自ZF-HD转录因子首次在黄花菊(Flaveria trinervia)中被发现以来,陆续从拟南芥(Tan and Irish,2006)、葡萄(Wang et al.,2014)、白菜(Wang et al.,2016)和番茄(胡靖康,2018)等多种植物中分离鉴定出ZF-HD家族成员。其中,已对葡萄(Wang et al.,2014)、陆地棉(倪万潮等,2016)和白菜(Wang et al.,2016)等多种植物ZF-HD家族成员进行了系统鉴定及功能分析。研究证明,ZF-HD转录因子广泛参与到植物花和叶片的器官发育及逆境胁迫响应中(张晋玉等,2017;Shalmani et al.,2019)。其中,有关ZF-HD在花发育中的作用研究发现,拟南芥ZF-HD基因家族包括14个AtZHD基因和3个AtMIF基因,其中MIF与ZF-HD高度同源,也划分为ZF-HD家族,但缺乏HD结构域,且在拟南芥花组织中高表达,推测其参与花的发育(Tan and Irish,2006);大白菜和大麦的ZF-HD家族成员也参与花发育(Wang et al.,2016;Abu-Romman and Al-Hadid,2017)。有关ZF-HD在响应逆境中的作用研究发现,拟南芥中AtZHD1基因可被干旱、脱落酸(ABA)及盐胁迫诱导表达,可与快速应答干旱响应的脱水诱导早期应答基因(ERD1)的启动子特异性结合(Tran et al.,2007);大豆ZF-HD基因家族成员GmZHD1和GmZHD2可与病原菌胁迫的钙调蛋白家族成员GmCAM4的启动子结合,进而调控其表达水平,影响病原菌胁迫响应(Park et al.,2007);从水稻中鉴定获得4个ZF-HD转录因子,其在低温、干旱和机械损伤等抗逆中发挥重要作用(Figueiredo et al.,2012)。【本研究切入点】近年来茶树基因组已测序完成(Xia et al.,2019),为研究茶树ZF-HD基因家族打下基础,但至今鲜见有关茶树ZF-HD基因家族鉴定及表达分析的研究报道。【拟解决的关键问题】通过隐马尔可夫模型预测和序列比对从茶树基因组中筛选鉴定出茶树ZF-HD基因家族成员,利用生物信息学分析软件对其基因结构、启动子元件,以及编码蛋白的理化性质、氨基酸序列、结构特征和进化关系等进行预测分析,并基于转录组测序(RNA-Seq)数据分析其在不同组织及在干旱胁迫、盐胁迫和茉莉酸甲酯(MeJA)处理下的表达模式,为茶树ZF-HD基因家族的功能研究及茶树遗传性状改良提供理论依据。
1 材料与方法
1. 1 茶树ZF-HD基因家族鉴定
从TPIA数据库(茶树基因组数据库)(http://teaplant.org/index.html)(Xia et al.,2019)下载茶树基因组数据,然后通过以下2种方法鉴定茶树ZF-HD基因家族成员:(1)从拟南芥数据库(http://www.arabidopsis.org/)下载ZF-HD家族蛋白的氨基酸序列,利用本地BLASTp程序(E<10-5)搜索茶树总蛋白序列,从中鉴定出茶树ZF-HD家族成员;(2)从Pfam数据库(http://pfam.xfam.org/)下载PF04770(ZF-HD_dimer结构域)的隐马尔可夫模型,搜索鉴定出茶树ZF-HD家族蛋白(E<10-20)。最后利用CDD(https://www.ncbi.nlm.nih.gov/cdd)分析茶树ZF-HD家族蛋白的保守结构域,筛选出具有ZF-HD_dimer结构域的ZF-HD家族蛋白(CsZHDs)。
1. 2 茶树ZF-HD家族蛋白生物信息学分析
分别使用ProtParam(https://web.expasy.org/protparam/)、CDD(https://www.ncbi.nlm.nih.gov/cdd)、MEME(http://meme-suite.org/)、PSORT(https://wolf-psort.hgc.jp/)及SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)等在线生物信息学分析软件对茶树ZF-HD家族蛋白进行理化性质、保守结构域、保守基序、亚细胞定位和二级结构预测。利用TBtools(https://github.com/CJ-Chen/TBtools)进行保守结构域和基序的可视化作图(Chen et al.,2020)。
1. 3 茶树ZF-HD家族基因结构和进化关系分析
从茶树基因组数据中查找ZF-HD基因家族外显子和内含子分布的相关数据,利用TBtools绘制其基因结构图(Chen et al.,2020)。使用ClustalW对茶树ZF-HD家族蛋白与拟南芥ZF-HD家族蛋白进行氨基酸序列比对,然后利用MEGA 7.0的邻接法构建系统发育进化树(Bootstrap设为1000)(Hall,2013)。
1. 4 茶树ZH-HD家族基因启动子分析
从茶树基因组数据中提取ZF-HD基因家族启动子序列,即起始密码子(ATG)上游2000 bp,然后提交到PlantCARE数据库(Lescot,2002),预测启动子上的顺式作用元件,并借助TBtools对预测结果进行可视化(Chen et al.,2020)。
1. 5 茶树ZH-HD家族基因表达分析
从TPIA数据库下载茶树不同组织(根、茎、顶芽、嫩叶、成熟叶、老叶、花和果)及胁迫处理[干旱胁(25% PEG)、盐胁迫(200 mmol/L NaCl)和MeJA(0.25%)]的转录组数据(Xia et al.,2019)。利用HemI 1.0制作基因表达热图。
2 结果与分析
2. 1 茶树ZF-HD基因家族成员的鉴定及其理化性质分析结果
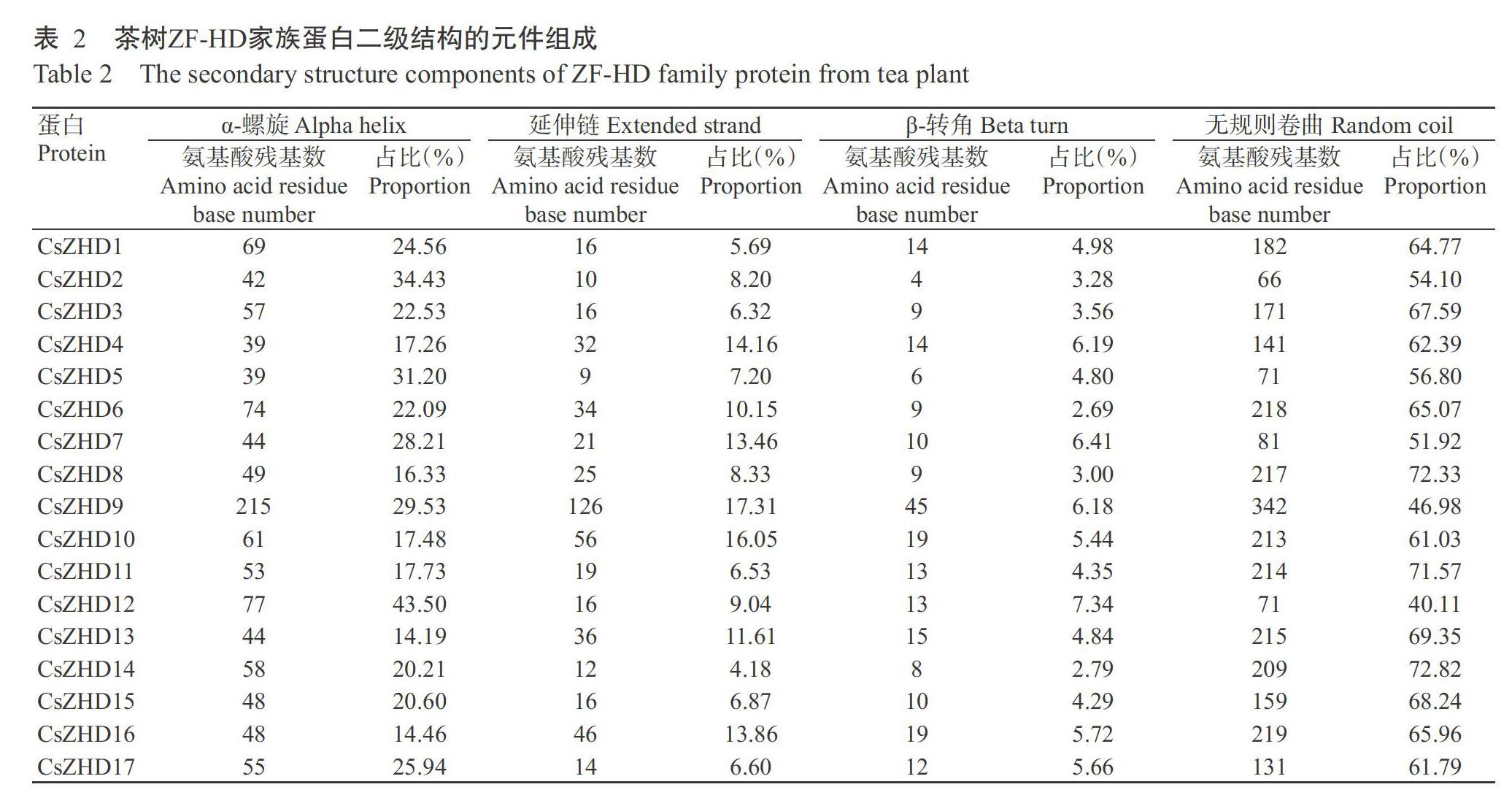
通过序列筛选分析,去除相似性低的序列和重复序列,最终获得17个茶树ZF-HD基因家族成员。根据基因ID大小进行排序,依次命名为CsZHD1~ CsZHD17(表1)。CsZHD1~CsZHD17基因编码区(CDS)序列长度为369~2187 bp,编码122~728个氨基酸残基,蛋白分子量为13.51~80.42 kD,理论等电点(pI)为6.09~9.19,均属于亲水性蛋白(GRAVY<0),不稳定系数均大于40,为不稳定蛋白。亚细胞定位分析结果显示,除CsZHD2、CsZHD5和CsZHD7定位于细胞质,CsZHD9定位于细胞外,其他13个蛋白均定位于细胞核。蛋白二级结构预测结果(表2)显示,茶树ZHD蛋白均由β-转角、延伸链、α-螺旋和无规则卷曲构成,且均以无规则卷曲占比最大,其次是α-螺旋和延伸链,β-转角占比最小。
2. 2 茶树ZF-HD家族蛋白结构域组成及保守基序分析结果
基于茶树基因组中ZF-HD基因家族成员的注释信息,绘制其基因结构图谱(图1-A)。CsZHD1~CsZHD17基因序列全长为377~15287 bp,其中以CsZHD5基因全长序列最短,以CsZHD9基因全长序列最长;仅CsZHD2、CsZHD7、CsZHD9、CsZHD10和CsZHD12基因含外显子和内含子,其他12个CsZHDs基因均无内含子。
利用CDD(Conerved domain database)对17个茶树ZF-HD家族蛋白的保守结构域ZF-HD_dimer进行分析,结果发现除CsZHD7具有2个ZF-HD_dimer结构域外,其他16个蛋白均只有1个ZF-HD_dimer结构域(表1)。17个茶树ZF-HD家族蛋白的氨基酸残基数目不同,且其ZF-HD_dimer结构域位置也各不相同(表1),基因内含子和外显子存在间隔,致使ZF-HD_dimer结构域的编码序列在基因序列上被分开(图1-A)。
利用MEME对茶树ZF-HD家族蛋白的保守基序进行分析,结果(图1-B)显示,17个茶树ZF-HD家族蛋白具有2~5个保守基序(motif),其中motif 1和motif 3为共有的保守基序,CsZHD2、CsZHD4、CsZHD5、CsZHD7和CsZHD12仅含有motif 1和motif 3,CsZHD3含有motif 1、motif 2和motif 3,其他蛋白均具有motif 1、motif 2、motif 3和motif 4,其中CsZHD9、CsZHD10、CsZHD13和CsZHD16还含有motif 5。
2. 3 茶树ZF-HD家族蛋白的系统发育进化树构建结果
依据植物ZF-HD家族进化关系及其在拟南芥中的分组方法,利用MEGA 7.0构建茶树和拟南芥ZF-HD家族蛋白的系统发育进化树,如图2所示。17个茶树ZF-HD家族蛋白可分成5个亚族,其中CsZHD3、CsZHD4和CsZHD15属于ZF-HD Ⅰ亚族;CsZHD8、CsZHD11和CsZHD17属于ZF-HD Ⅱ亚族;CsZHD1、CsZHD6和CsZHD14属于ZF-HD Ⅲ亚族;CsZHD9、CsZHD10、CsZHD12、CsZHD13和CsZHD16属于ZF-HD Ⅳ亚族;CsZHD2、CsZHD5和CsZHD7属于MIF亚族。未发现与拟南芥AtZHD13和AtZHD14同属于ZHD Ⅴ亚族的茶树ZF-HD家族蛋白。
2. 4 茶樹ZF-HD家族基因启动子顺式作用元件分析结果
由图3可知,茶树ZF-HD家族基因启动子上除具有多个TATA-box和CAAT-box類的基础顺式作用元件外,还存在大量与光响应相关的元件如ACE、ATC-motif、Box 4、GATA-motif、GT1-motif、MRE、TCCC-motif、TCT-motif、ATCT-motif、G-box、Sp1、3-AF1 binding site、AE-box和I-box等,以及多种激素响应的相关元件如生长素响应元件(AuxRR-core和TGA-element)、赤霉素响应元件(GARE-motif、P-box和TATC-box)、脱落酸响应元件(ABRE)、茉莉酸甲酯响应元件(CGTCA-motif和TGACG-motif)和水杨酸(TCA-element)。此外,茶树ZF-HD家族基因启动子序列上存在多个胁迫诱导相关的元件如厌氧胁迫响应元件(ARE和GC-motif)、干旱胁迫响应元件(MBS)、低温胁迫响应元件(LTR)、机械损伤胁迫响应元件(WUN-motif)和TC-rich repeats,以及多种与生长发育相关的顺式作用元件如参与栅栏叶肉细胞分化的调节元件(HD-Zip 1)、胚乳表达的调节元件(GCN4_motif)和分生组织表达的调控元件(CAT-box)。可见,CsZHD1~CsZHD17基因可能参与茶树的生长发育、胁迫反应和激素应答。
2. 5 茶树ZF-HD家族基因不同组织的表达分析结果
基于TPIA数据库中茶树8个代表性组织的转录组数据,对CsZHD1~CsZHD17基因的表达模式进行分析,结果(图4)表明,CsZHD12基因在8个组织中均未检测到表达量,CsZHD2和CsZHD17基因在8个组织中的表达量均很低或不表达,其他基因在8个组织中基本均有表达,但在不同组织中的表达量不同。整体来看,除CsZHD2、CsZHD5、CsZHD12和CsZHD17基因外,其他13个CsZHDs基因在顶芽中有较高的表达水平,而在根中的表达量较低;在茎、顶芽、嫩叶、成熟叶和老叶中CsZHD1~CsZHD17基因表达模式相近,尤其在茎、顶芽和嫩叶中更相似,表达水平均处于中等水平,CsZHD5在花中表达量极高。综上所述,除CsZHD2、CsZHD12和CsZHD17基因之外,其他15个CsZHDs基因可能在茶树顶芽和花的发育过程中发挥重要作用。
2. 6 茶树ZF-HD家族基因在不同非生物胁迫和激素处理下的表达分析结果
基于TPIA数据库收集茶树在干旱胁迫、盐胁迫和MeJA胁迫处理不同时间的转录组数据,对CsZHD1~CsZHD17基因的表达模式进行分析,结果(图5)显示,CsZHD1、CsZHD2、CsZHD5、CsZHD12、CsZHD14和CsZHD17基因在干旱胁迫和盐胁迫处理下的表达量始终处于较低水平,推测这些基因对干旱胁迫和盐胁迫无响应;CsZHD7和CsZHD13基因随干旱胁迫和盐胁迫处理时间的延长呈先升高后降低的变化趋势;CsZHD10基因在盐胁迫下呈先升高后降低的变化趋势,但在干旱胁迫下表现为先降低后升高;其他CsZHDs基因均随着处理时间的延长其表达量不断下降。在MeJA处理下,除表达量极低的CsZHD2、CsZHD5和CsZHD12外,其他CsZHDs基因均呈下调表达趋势。综上所述,大多数CsZHDs基因可能参与茶树非生物胁迫及MeJA处理下的调控。
3 讨论
茶树起源于我国西南部,是常绿叶用经济林木,由于其嫩梢加工而成的茶叶富含茶多酚(尤其儿茶素)、茶氨酸、茶多糖和咖啡碱等多种对人体有益的生理活性成分。随着人们对茶叶保健功能认识的不断加深,使得茶叶市场需求量不断增长。茶树生长发育和响应胁迫受多种基因调控,其中转录因子基因是重要的调控基因(Riechmann et al.,2000)。经大量研究证实,植物ZF-HD转录因子参与生长发育、非生物胁迫和植物激素反应等多种生物学过程(张晋玉等,2017)。本研究从茶树全基因组中鉴定出17个ZF-HD基因家族成员(CsZHD1~CsZHD17),与拟南芥(Tan and Irish,2006)ZF-HD基因家族成员数量相同,高于葡萄(13个)(Wang et al.,2014)、水稻(15个)(Xu et al.,2014)和卷柏(7个)(Khatun et al.,2017),低于番茄(22个)(胡靖康,2018)和甘蓝型油菜(62个)(宋敏等,2019)的ZF-HD基因家族成员数量;但从植物基因组大小相比,茶树基因组较大,约3.1 Gb,远高于拟南芥(164 Mb)、水稻(441 Mb)、甘蓝型油菜(283.8 Mb)、卷柏(212.5 Mb)和葡萄(490 Mb)等的基因组。可见,植物ZF-HD家族基因数量与基因组大小无明显相关性,究其原因可能是基因组较小的植物中发生了基因组复制事件,从而引起ZF-HD基因家族扩展(Khatun et al.,2017)。
依据植物ZF-HD家族基因进化关系及拟南芥ZF-HD家族基因的分组方法(Hu et al.,2008;Wang et al.,2016),本研究将茶树的17个ZF-HD家族蛋白分为5个亚族:MIF、ZHD Ⅰ、ZHD Ⅱ、ZHD Ⅲ和ZHD Ⅳ,与拟南芥ZF-HD家族相比,茶树缺少ZHD Ⅴ亚族,其原因可能是在进化过程中遗失。据报道,ZF-HD亚族丢失现象在水稻和玉米中均有发生(李春艳,2018)。本研究的茶树ZF-HD家族基因结构分析结果显示,仅CsZHD2、CsZHD7、CsZHD9、CsZHD10和CsZHD12基因含有外显子和内含子,其他12个CsZHDs基因仅含外显子,无内含子,说明仅含外显子的基因所占比例较高,约70.59%。该结论与其他植物ZF-HD家族基因结构相似,如拟南芥(Tan and Irish,2006)、番茄(胡靖康,2018)和椰子(孙熹微等,2020)的ZF-HD家族基因也仅含外显子。内含子的缺失会导致ZF-HD家族基因无法进行可变剪切,从而使ZF-HD家族蛋白具有较高的保守性(胡靖康,2018)。此外,本研究发现有3个茶树ZF-HD家族蛋白(CsZHD2、CsZHD5和CsZHD7)属于MIF亚族,与拟南芥MIF亚族一样均缺少HD结构域(Tan and Irish,2006;Wang et al.,2016);除CsZHD2、CsZHD5和CsZHD7外,CsZHD4和CsZHD12也缺少HD结构域,但在系统发育进化树上未聚在MIF亚族,而是分别聚在ZHD Ⅰ和ZHD Ⅳ亚族。在其他植物中未见类似的研究报道,其原因可能是CsZHD4和CsZHD12基因发生变异或重组,使得HD结构域缺失,具体原因有待进一步研究。
CsZHD1~CsZHD17基因启动子上除含有多个TATA-box和CAAT-box类的基础顺式作用元件外,还存在大量与光响应、激素响应、胁迫响应及生长发育等相关的顺式作用元件,故推测其可能参与生长发育、胁迫响应和激素应答。经表达分析发现,除CsZHD2、CsZHD12和CsZHD17基因外,其他14个CsZHDs基因在不同组织中均呈差异表达,由于基因的差异表达与其功能密切相关,故推测这些基因的生物学功能存在差异,如CsZHD4、CsZHD9、CsZHD10和CsZHD11基因等在顶芽和嫩叶中高表达,其可能参与茶树新梢芽叶的发育;CsZHD5、CsZHD9和CsZHD10在花中高表达,尤其CsZHD5基因在花中表达量极高,其可能与茶树花器官的发育密切相关。在拟南芥(Tan and Irish,2006)、水稻(Xu et al.,2014)和白菜(Wang et al.,2016)中,ZF-HD转录因子基因参与叶和花等组织发育。
经前人研究证实,植物ZF-HD家族基因参与非生物胁迫响应和激素应答(Tran et al.,2007;Figueiredo et al.,2012)。本研究发现,大多数CsZHDs基因参与茶树非生物胁迫及MeJA处理的响应调控,多数基因表现为下调表达趋势,与Tran等(2007)研究发现干旱胁迫和盐胁迫诱导AtZHD1基因上调表达的结果不一致。因此,CsZHDs基因的生物学功能仍需深入研究。
4 结论
从茶树基因组中鉴定出17个ZF-HD基因家族成员,其编码蛋白具有保守的锌指结构域和同源异型盒结构域;茶树与拟南芥ZF-HD基因家族相比缺少ZHD Ⅴ亚族;CsZHDs基因的表达具有组织特异性,且大多数成员的表达受非生物胁迫和MeJA的影响。
参考文献:
丁忠杰. 2014. 拟南芥WRKY转录因子在非生物胁迫响应中的功能研究[D]. 杭州:浙江大学. [Ding Z J. 2014. Functional study of Arabidopsis WRKY transcription factors in response to abiotic stresses[D]. Hangzhou:Zhejiang University.]
胡靖康. 2018. 番茄ZF-HD转录因子的全基因组挖掘及抗逆相关基因筛选[D]. 哈尔滨:东北农业大学. [Hu J K. 2018. Bioinformatic analysis and identification of some resistance-associated genes of ZF-HD gene family in tomato[D]. Harbin:Northeast Agricultural University.]
李春艳. 2018. 玉米ZF-HD转录因子家族耐盐、抗旱相关基因的鉴定及特性分析[D]. 重庆:西南大学. [Li C Y. 2018. Identification and characterization of drought and salt resistance-related genes of ZF-HD transcription factors fa-mily in maize[D]. Chongqing:Southwest University.]
倪万潮,徐珍珍,沈新莲. 2016. 陆地棉ZF-HD蛋白的全基因组分析[J]. 棉花学报,28(6):519-526. doi:10.11963/issn. 1002-7807.201606001. [Ni W C,Xu Z Z,Shen X L. 2016. Genome-wide analysis of the Gh ZHD protein fa-mily in upland cotton[J]. Cotton Science,28(6):519-526.]
宋敏,张瑶,王丽莹,彭向永. 2019. 甘蓝型油菜ZF-HD基因家族的鉴定与系统进化分析[J]. 植物学报,54(6):699-710. doi:10.11983/CBB19055. [Song M,Zhang Y,Wang L Y,Peng X Y. 2019. Genome-wide identification and phylogenetic analysis of zinc finger homeodomain family genes in Brassica napus[J]. Chinese Bulletin of Botany,54(6):699-710.]
孫熹微,范海阔,弓淑芳,刘蕊,纠凤凤,肖勇. 2020. 椰子ZF-HD基因家族的鉴定及生物信息学分析[J]. 热带作物学报,41(2):284-291. doi:10.3969/j.issn.1000-2561.2020. 02.011. [Sun X W,Fan H K,Gong S F,Liu R,Jiu F F,Xiao Y. 2020. Identification and bioinformatics analysis of coconut ZF-HD gene family[J]. Chinese Journal of Tro-pical Crops,41(2):284-291.]
杨方慧,夏丽飞,陈林波,田易萍,宋维希,梁名志. 2019. 茶树MADS-box家族基因AGL9的克隆及表达分析[J]. 西南农业学报,32(10):2299-2303. doi:10.16213/j.cnki.scjas. 2019.10.007. [Yang F H,Xia L F,Chen L B,Tian Y P,Song W X,Liang M Z. 2019. Cloning and expression ana-lysis on family gene AGL9 of MADS-box in tea plant[J]. Southwest China Journal of Agricultural Sciences,32(10):2299-2303.]
張晋玉,晁毛妮,杜弘杨,喻德跃,黄方. 2017. 大豆ZF-HD转录因子GmZHD1的克隆及表达分析[J]. 华北农学报,32(2):1-7. doi:CNKI:SUN:HBNB.0.2017-02-001. [Zhang J Y,Chao M N,Du H Y,Yu D H,Huang F. 2017. Clo-ning and expression analysis of ZF-HD transcription factor GmZHD1 in Glycine max[J]. Acta Agriculturae Boreali-Sinica,32(2):1-7.]
Abu-Romman S,Al-Hadid K. 2017. Novel zinc finger-homeodomain gene from barley(HvZFHD1) is differentially regulated during spike development and under hormonal treatments and abiotic stresses[J]. Notulae Botanicae Horti Agrobotanici Cluj-Napoca,45(1):89-96. doi:10.15835/nbha45110612.
Ariel F D,Manavella P A,Dezar C A,Chan R L. 2007. The true story of the HD-zip family[J]. Trends in Plant Scien-ce,12(9):419-426. doi:10.1016/j.tplants.2007.08.003.
Chen C J,Chen H,Zhang Y,Thomas H R,Frank M H,He Y H,Xia R. 2020. TBtools:An integrative toolkit develo-ped for interactive analyses of big biological data[J]. Molecular Plant,13(8):1194-1202. doi:10.1016/j.molp.2020. 06.009.
Figueiredo D D,Barros P M,Cordeiro A M,Serra T S,Louren?o T,Chander S,Oliveira M M,Saibo N J. 2012. Seven zinc-finger transcription factors are novel regulators of the stress responsive gene OsDREB1B[J]. Journal of Expe-rimental Botany,63(10):3643-3656. doi:10.1093/jxb/ers035.
Gehring W J,Müller M,Affolter M,Percival-Smith A,Bille-ter M,Qian Y Q,Otting G,Wüthrich K. 1990. The structure of the homeodomain and its functional implications[J]. Trends in Genetics,6(10):323-329. doi:10.1016/0168-9525(90)90253-3.
Hall B G. 2013. Building phylogenetic trees from molecular data with MEGA[J]. Molecular Biology and Evolution,30(5):1229-1235. doi:10.1093/molbev/mst012.
Hu W,de Pamphilis C W,Ma H. 2008. Phylogenetic analysis of the plant-specific zinc finger-homeobox and mini zinc finger gene families[J]. Journal of Integrative Plant Bio-logy,50(8):1031-1045. doi:10.1111/j.1744-7909.2008. 00681.x.
Khatun K,Nath U K,Robin A H K,Park J I,Lee D J,Kim M B,Kim C K,Lim K B,Nou I S,Chung M Y. 2017. Genome-wide analysis and expression profiling of zinc finger homeodomain(ZHD) family genes reveal likely roles in organ development and stress responses in tomato[J]. BMC Genomics,18(1):695. doi:10.1186/s12864-017-4082-y.
Lescot M. 2002. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico ana-lysis of promoter sequences[J]. Nucleic Acids Research,30(1):325-327. doi:10.1093/nar/30.1.325.
Park H C,Kim M L,Lee S M,Bahk J D,Yun D J,Lim C O,Hong J C,Lee S Y,Cho M J,Chung W S. 2007. Pathogen-induced binding of the soybean zinc finger homeodomain proteins GmZF-HD1 and GmZF-HD2 to two repeats of ATTA homeodomain binding site in the calmodulin isoform 4(GmCaM4) promoter[J]. Nucleic Acids Research,35(11):3612-3623. doi:10.1093/nar/gkm273.
Riechmann J L,Heard J,Martin G,Reuber L,Jiang C,Keddie J,Adam L,Pineda O,Ratcliffe O J,Samaha R R,Creelman R,Pilgrim M,Broun P,Zhang J Z,Ghandehari D,Sherman B K,Yu G. 2000. Arabidopsis transcription factors:Genome-wide comparative analysis among eukaryo-tes[J]. Science,290(5499):2105-2110. doi:10.1126/scien-ce.290.5499.2105.
Shalmani A,Muhammad I,Sharif R,Zhao C,Ullah U,Zhang D,Jing X Q,Amin B,Jia P,Mobeen Tahir M,Xu Z,Chen K M,An N. 2019. Zinc finger-homeodomain genes:Evolution,functional differentiation,and expression profi-ling under flowering-related treatments and abiotic stresses in plants[J]. Evolutionary Bioinformatics Online. doi:10. 1177/1176934319867930.
Tan Q K,Irish V F. 2006. The Arabidopsis zinc finger-homeo-domain genes encode proteins with unique biochemical properties that are coordinately expressed during floral development[J]. Plant Physiology,140(3):1095-1108. doi:10.1104/pp.105.070565.
Tran L S,Nakashima K,Sakuma Y,Osakabe Y,Qin F,Simpson S D,Maruyama K,Fujita Y,Shinozaki K,Yamaguchi-Shinozaki K. 2007. Co-expression of the stress-induci-ble zinc finger homeodomain ZFHD1 and NAC transcription factors enhances expression of the ERD1 gene in Arabidopsis[J]. Plant Journal for Cell and Molecular Bio-logy,49(1):46-63. doi:10.1111/j.1365-313X.2006.02932.x.
Wang H,Yin X J,Li X Q,Wang L,Zheng Y,Xu X Z,Zhang Y C,Wang X P. 2014. Genome-wide identification,evolution and expression analysis of the grape(Vitis vinifera L.) zinc finger-homeodomain gene family[J]. International Journal of Molecular Sciences,15(4):5730-5748. doi:10.3390/ijms15045730.
Wang W,Wu P,Li Y,Hou X L. 2016. Genome-wide analysis and expression patterns of ZF-HD transcription factors under different developmental tissues and abiotic stresses in Chinese cabbage[J]. Molecular Genetics and Genomi-cs,291(3):1451-1464. doi:10.1007/s00438-016-1196-x.
Wei K,Wang L Y,Zhang Y Z,Ruan L,Li H L,Wu L Y,Xu L Y,Zhang C C,Zhou X G,Cheng H,Edwards R. 2018. A coupled role for CsMYB75 and CsGSTF1 in anthocyanin hyperaccumulation in purple tea[J]. The Plant Journal,97(5):825-840. doi:10.1111/tpj.14161.
Xia E H,Li F D,Tong W,Li P H,Wu Q,Zhao H J,Ge R H,Li R P,Li Y Y,Zhang Z Z,Wei C L,Wan X C. 2019. Tea plant information archive:A comprehensive genomics and bioinformatics platform for tea plant[J]. Plant Biotechnology Journal,17(10):1938-1953. doi:10.1111/pbi. 13111.
Xu Y,Wang Y H,Long Q Z,Huang J X,Wang Y L,Zhou K N,Zheng M,Sun J,Chen H,Chen S H,Jiang L,Wang C M,Wan J M. 2014. Overexpression of OsZHD1,a zinc finger homeodomain class homeobox transcription factor,induces abaxially curled and drooping leaf in rice[J]. Planta,239(4):803-816. doi:10.1007/s00425-013-2009-7.
(責任编辑 陈 燕)
