大白猪和长白猪新复等位基因及其多态性与结合肽谱分析
2021-07-07李奇润魏孝辉
李奇润 , 魏孝辉 , 夏 春
(中国农业大学动物医学院 , 北京 海淀 100193)
猪经典主要组织相容性复合体(Major histocompatibility complex,MHC) I 类分子略写为SLA-I,包含SLA-1,SLA-2和SLA-3三个基因座[1]。迄今为止,主要组织相容性复合体数据库(IPD ; https://www.ebi.ac.uk/ipd/mhc/)共登录了SLAI/II等位基因450条[2]。猪 SLA-I分子主要与β2m和抗原多肽形成三分子复合物(pSLA-I)递呈抗原,诱发特异性细胞毒性T细胞(Cytotoxic T lymphocyte, CTL)免疫应答[3]。由于SLA-I α1和α2区的高变性导致了其复等位基因的多态性,使不同的等位基因能够产生针对特定抗原肽不同锚定残基的抗原肽结合槽[4]。
抗原递呈细胞(Antigen presenting cell, APC)递呈CTL表位是引发CTL免疫应答最关键的环节[5]。目前对猪CTL表位的研究大多集中在预测、体外能否形成pSLA-I复合体以及四聚体技术鉴定这3个层面[6-8]。常用的与猪SLA-I结合的网站有Net-MHC4.0、NetMHCpan、NetMHCstabpan等[9]。然而,由于猪等位基因的多态性,使用软件预测的结果具有明显的瑕疵[10-11]。质谱测定pMHC-I结合多肽的方法相对而言数据真实,降低了偏差[12],再联合质谱从头测序技术(MS de novo sequencing)用于MHC-I分子的体外结合抗原肽谱及基序的鉴定,由于其结果准确已被认可为测定pMHC-I CTL候补表位的方法之一[12-13]。
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome, PRRS),又称猪蓝耳病,由猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)引起 ; PRRSV抑制细胞免疫,造成细胞免疫反应延迟并减弱[14]。目前主要通过接种疫苗诱导体液免疫应答的方式控制PRRS[15],有关PRRSV CTL候补表位与细胞性免疫应答的研究极少[16]。本文对法系大白猪和长白猪SLA-I基因进行了测序及多肽性分析; 对其代表性的pSLA-I结合随机9肽谱进行鉴定与分析; 从中筛选出部分与PRRSV相关的CTL候补表位。
1 材料与方法
1.1 试验材料 本试验所使用的实验猪为法系大白猪和长白猪。SLA-I克隆引物对的合成与测序由上海英骏生物技术有限公司完成。RNA提取试剂TRIzol、SuperScript III First-Strand Synthesis System for RT-PCR试剂盒,均购自Inventrogen公司。红细胞裂解液、限制性内切酶、T4 DNA连接酶等,均购自宝生物工程(大连)有限公司。E.coliTrans5α、E.coliTransetta (DE3)感受态细胞,均购自北京全式金生物技术有限公司。随机9肽(半胱氨酸除外)由北京中科亚光生物科技有限公司合成。
1.2 试验方法
1.2.1 大白猪和长白猪SLA-I等位基因克隆 选10头原种大白猪、长白猪前腔静脉采血,按照TRIzol法提取外周血淋巴细胞总RNA。使用Nanodrop测定浓度及OD值,结合琼脂糖凝胶电泳及OD260 nm/OD280 nm判断RNA的质量。对上述RNA进行RT-PCR反应,合成cDNA链。
参照GenBank数据库中猪SLA-I基因(EU170457,EU170458,EU170459)序列,采用Oligo 6.0软件,分别设计扩增猪SLA-1、SLA-2、SLA-3等位基因座的3对特异性引物[16]。引物序列见表1。PCR扩增,产物经过凝胶电泳回收后连接至pMD-19T 载体,转化至氨苄抗性的培养皿中,挑取单个菌落送苏州金唯智生物科技有限公司进行测序。
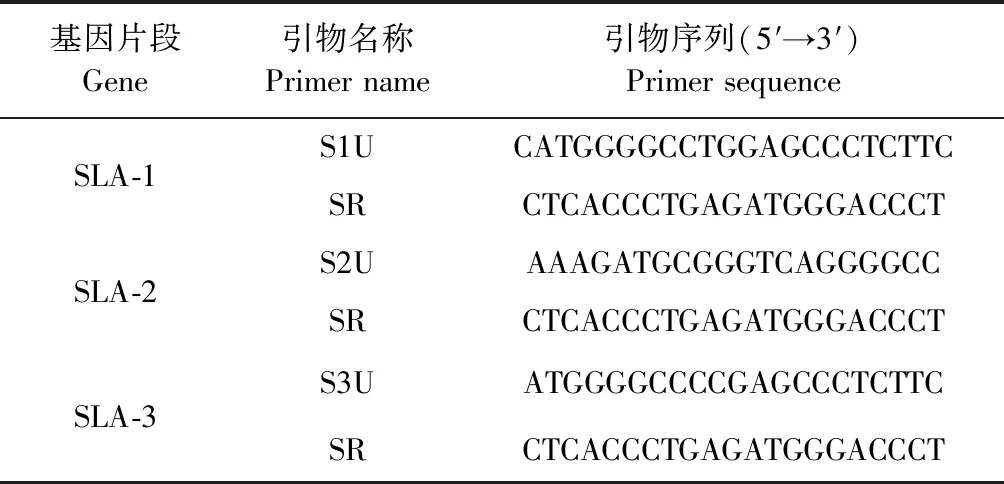
表1 克隆SLA-I引物序列Table 1 Cloning of SLA-I primer sequence
1.2.2 大白猪和长白猪多态性分析 通过对克隆的等位基因进行序列分析比较,将获得的序列上传至NCBI的GenBank并获得相应的序列编号。分别区分出信号肽(http://www.cbs.dtu.dk/services/SignalP/)、跨膜区以及胞内区 (http://www.cbs.dtu.dk/services/TMHMM/)。并通过在线软件CLUSTALW (https://www.genome.jp/tools-bin/clustalw)分析SLA-I氨基酸序列的同源性,通过PVS (http://imed.med.ucm.es/PVS/) 分析构成抗原肽结合槽(PBG)的氨基酸保守与高变异位点。
1.2.3 SLA-1*1204蛋白表达 设计特定等位基因SLA-1*1204胞外区的特异性引物,如表2 所示,PCR扩增,收集目的片段连接至pET-28a 载体,连接产物转化感受态细胞Trans5α,PCR扩增鉴定,提取阳性质粒转入Transetta (DE3),IPTG诱导表达,提取并纯化包涵体[16]。

表2 SLA-1*1204胞外区特异性引物Table 2 SLA-1*1204 extracellular region specific primers
1.2.4 随机肽结合质谱从头测序鉴定SLA-1*1204结合基序 随机9肽通过固相合成法按照每位19种氨基酸等摩尔比进行合成,纯化除盐后,通过质谱从头测序进行质检[12]。
按照SLA-1*1204∶β2m∶随机9肽为1∶1∶5比例进行体外共复性[400 mmol/L L-Arg HCl,2 mmol/L EDTA,5 mmol/L GSH,0.5 mmol/L GSSH和100 mmol/L Tris-HCl (pH 8)],通过分子筛层析[20 mmol/L Tris (pH 8)和50 mmol/L NaCl]以及离子交换层析[Buffer A:20 mmol/L Tris 396 (pH 8)和5 mmol/L NaCl;Buffer B:20 mmol/L Tris (pH 8) 和500 mmol/L NaCl]进行纯化。浓缩换液后,通过弱酸进行洗脱,除盐后经液相色谱-串联质谱(LC-MS/MS)检测,De novo sequencing解析可信的多肽序列。通过聚类分析获得SLA-1*1204抗原肽结合槽每个口袋对氨基酸的偏好性,分析主要锚定位点和结合基序,以此为基础对基序及氨基酸进行打分,并用来预测PRRSV来源的潜在表位肽[12]。
1.2.5 基序SLA-1*1204结合基序的PRRSV表位预测 将筛选过滤得到的9肽中限制性锚定残基进行计算[12,17],获得每个位置每个氨基酸的可能性,基于锚定残基加权概率(位置概率矩阵)计算限制性基序(限制性锚定残基的数量Nres)上的特定氨基酸和成分[18-19]。
2 结果
2.1 新SLA-I多态性分析 首先将克隆、测序的等位基因进行序列分析、比较,去掉100%同源序列,最终获得了22条新SLA-I基因(中插彩版图1)。随后,将克隆的SLA-I进行命名,再上传至NCBI的GenBank,获得了相应的基因序号,其中,SLA-1基因共7条:SLA-1*08:19(MN381837),SLA-1*08:20(MN813669),SLA-1*12:04(MT230602),SLA-1*20:01(MN813662),SLA-1*20:02(MN813660),SLA-1*20:03(MT230601),SLA-1*20:04(MT230598);SLA-2基因共13条:SLA-2*01:04(MN381838),SLA-2*01:06(MN813663),SLA-2*09:07(MN813654),SLA-2*10:10(MN381835),SLA-2*10:11(MN381835),SLA-2*10:12(MT230599),SLA-2*10:17(MN813655),SLA-2*10:18(MN813667),SLA-2*10:20(MN813668),SLA-2*10:21(MT230600),SLA-2*16:04(MN813666),SLA-2*16:05(MN813665),SLA-2*16:06(MN813664);SLA-3基因共2条:SLA-3*05:09(MN381839),SLA-3*05:10(MN381840)。
参照本实验室已发表的SLA-I序列[21]分析发现,所克隆的22条SLA-I分子α重链N端具有20~30个 不等长度的氨基酸构成了信号肽,C端具有60个左右的氨基酸组成了跨膜区和胞内区;胞外区约270个氨基酸,包括α1,α2和α3 三个结构域。即22条新SLA-I序列存在明显的长度变化(中插彩版图1)。
基于SLA-I的结构与序列比对发现(中插彩版图1),α1/α2/α3分别由90个氨基酸左右组成,其中,差异氨基酸主要位于α1/α2结构域中。其中,α1~α5螺旋组成抗原结合槽侧壁,而β1~β2折叠组成抗原结合槽底部并起支撑侧壁的功能。SLA-I能够结合并递呈相应的抗原,归功于组成抗原结合槽两端口袋的保守氨基酸(L5,Y7,Y59,Y84,Y123,K146,Y159,L160和Y171)与多肽末端形成的作用力网[21]。α3结构域相对较保守,与β2m作用维持SLA-I分子三维结构的稳定性,并与CD8αα形成稳定的作用力,从而保证T细胞的激活。α1/α2结构域中的Y7,F8,A11,V25,Y27,F33,F36,H93,L110,G112和Q115,D122,E128分别协助α3结构域与猪β2m/CD8αα相互作用(中插彩版图1)。保守性N87,Q88,E89位可发生糖基化,C110/C164/C203/C259形成2对二硫键从而维持SLA-I三维构象。
通过在线软件PVS (http://imed.med.ucm.es/PVS/)对抗原结合槽进行变异位点分析、Wu-Kabat法输出结果如中插彩版图2A所示。同时,槽内相对变异位点映射至三维结构(PDB:3QQ3)[20]中,如中插彩版图2B,抗原结合槽根据锚定位点可分为A~F 口袋,每个口袋均有多个氨基酸组成,这些氨基酸的变异改变相应口袋的性质,如大小、电荷及亲疏水性。第62、163、167、170位氨基酸位于A口袋上侧,第9、24、66、67、99位氨基酸位于B口袋内侧和上侧,第73、74、77、95、97、114、116、143、147位氨基酸分布于F口袋周围,这些氨基酸残基的改变能够直接影响多肽末端锚定残基的偏好性。相对而言,D口袋虽然重要性弱于A/B/F口袋,但其变异位点99、155、156位能够通过直接影响多肽P3位残基的性质而影响多肽结合。而组成C口袋的第70、73、74位氨基酸和组成E口袋的第114、152位氨基酸能够辅助影响多肽P4~P8位氨基酸的构象,从而影响TCR的识别。
2.2 SLA-1*1204的基序鉴定 随机肽法体外复性SLA-1*1204,通过分子筛层析法(中插彩版图3A)及离子交换层析法(中插彩版图3B)对pSLA-1*1204复合体进行纯化,SDS-PAGE进行纯度鉴定(中插彩版图3C)。酸洗法对高亲和力多肽进行洗脱并通过质谱从头测序法得到1 506条可信多肽(伪发现率FDR=1%),Weblogo(https://weblogo.berkeley.edu/logo.cgi) 结果进行展示,如中插彩版图3D所示。SLA-1*1204的基序为T/K/L-V/A/L-X-X-X-X-X-P/L-L/Y/F,其对多肽N端和C端具有明显的限制性,尤其是P1和P9,其次是P2和P8,相对于之前所报道的SLA-1,SLA-1*1204的P3相对缺乏限制性,而P4~P7为TCR识别的关键位点,其非限制性决定了递呈表位的多样性,从而更有效的激活更多的CD8+T淋巴细胞。正如经典MHC I,C端(P9位)为长侧链及芳香烃氨基酸,垂直插入F口袋,具有直接影响多肽亲和力的作用。P1和P2发挥N端固定多肽的作用,P8为多肽转折点,脯氨酸的存在有助于P9位转向插入F口袋。
2.3 基于SLA-1*1204结合基序的PRRSV表位预测 通过质谱获得的1 506条可信9肽,在EXCEL表中依次列为9列,统计每个锚定位点中18种氨基酸(半胱氨酸、异亮氨酸除外)出现的次数及频率。结合SLA-1*1204多肽结合基序 P1、P2、P9位各氨基酸出现频率及病毒序列(GenBank:EF641008.1)[23],通过Python对PRRSV JXwn06毒株病毒序列9肽打分 (1.2.5所述),从中筛选出10条评分最高的潜在9肽表位,如表3所示,10条高亲和力潜在表位分布于PRRSV的不同蛋白,如结构蛋白GP4、GP5,非结构蛋白NSP、RNA依赖性RNA聚合酶。

表3 基于SLA-1*1204基序的PRRSV(JXwn06)表位预测Table 3 Epitope prediction of PRRSV (JXwn06) based on SLA-1*1204 motif
3 讨论
3.1 与已报道SLA-I序列比对 本文从大白猪和长白猪中克隆获得了22条新SLA-I等位基因,与已克隆获得的21条藏香猪、28条黑山猪[21]和44条长白猪[16]序列比对发现,不同的品种间其SLA-I序列存在长度与个别氨基酸上的差异,主要表现在组成PBG的α1和α2结构域,尤其是组成A~F口袋的氨基酸;而在α3区、β2m结合区等处相对保守,整体序列同源性大于80%,来自不同品种的猪SLA-I可能处于进化树的同一基因簇上。再次验证了新SLA-I等位基因的出现可能是由于基因突变、插入和缺失、片段重组等现象[24],表明SLA-I的进化可能与猪的品种及地域环境等没有必然的联系。但是,目前主要组织相容性复合体数据库(IPD;https://www.ebi.ac.uk/ipd/mhc/)共登录了SLA-I等位基因229条[2],与人类MHC I分子已报道16 200个(http://hla.alleles.org/nomenclature/)相比,数据还较少,仍需要克隆更多的SLA-I等位基因进行大数据比对分析。
3.2 多肽预测方法 近几年,大多采用计算机方法对多肽进行预测,最多的NetMHC使用人工神经网络法对MHC 等位基因预测,通过NetMHCpan,SLA-1*0401[20]、SLA-3*hs0202[27]成功预测了流感病毒(IAV)结合多肽,SLA-1*1502[16]、SLA-1*0501[27]预测了PRRSV结合多肽,该方法更多考虑分子肽内部不同位置氨基酸之间的相互关系,预测结果数量不稳定,多的可达上百条,少的可能为0。本文通过随机肽库结合质谱从头测序方法预测SLA-1*1204结合PRRSV多肽,与NetMHC方法相比,可快速鉴定出该等位基因的结合基序,结合病毒蛋白序列计算出高结合率的PRRSV 9肽,但也存在需要合成随机9肽、质谱分析等过程较多、成本较高的缺点。当然,多肽预测只是SLA-I递呈的一个环节,能否与SLA-1结合并递呈给CTL需要进一步的验证。
3.3 锚定残基 抗原肽主要通过多肽N端和C端2~3个氨基酸锚定于PBG相应的口袋中[25]。其中,B和F(有的为B、C、F或B、D、F)在结合抗原肽上起着决定性的作用[26]。我们曾通过3D结构获得了SLA-1*1502、SLA-1*0401、SLA-3*hs0202 PBGs的锚定残基为P2、P3、P9位[20-22],本文SLA-1*1204结合的抗原肽谱显示,其PBG的锚定残基为P1、P2、P9,P9位是长侧链氨基酸能够锚定的C端,N端的P1/P2位也同时发挥了锚定氨基酸的作用。
