集胞藻PCC6803中N-乙酰鸟氨酸转氨酶的生化表征及结构分析
2021-06-23白福美李至敏王小琴胡紫微鲍玲玲李志敏
白福美 李至敏 王小琴 胡紫微 鲍玲玲 李志敏,3
(1.江西农业大学生物科学与工程学院,南昌 330045;2.江西农业大学理学院,南昌 330045;3.江西省农业微生物资源开发与利用工程实验室,南昌 330045)
蓝藻,又名蓝细菌或蓝绿藻,是地球上最早出现的光合自养微生物之一,其种类繁多,分布广泛,具有高效的光能利用率,是氧气的主要生产者和生物圈不可或缺的组成者[1]。集胞藻PCC6803是1968年从淡水湖泊中分离得到的单细胞非固氮球形蓝藻。相比于其他蓝藻,集胞藻PCC6803不仅可以光合自养,还能完全依赖外部能源进行异养[2-3]。自1996年其全基因组序列被公布以来,集胞藻PCC6803就成为了研究蓝藻的模式生物之一[4]。除此之外,集胞藻PCC6803在生物能源和环境保护上也有广泛的应用前景。如Gao等[5]运用同源重组的方法使集胞藻PCC6803中乙醇的日产量达到212 mg/L,26 d内达到5.5 g/L,使得乙醇作为生物燃料成为了可能;Aizouq等[6]揭示了在集胞藻PCC6803中能够产生甘油三酯和蜡酯,开辟了使用原核光合细胞产油的可能性;张兵等[7]研究发现集胞藻PCC6803对于砷污染的水体具有潜在的修复功能,可作为水体砷污染修复的生物材料。
三羧酸循环是需氧生物体内普遍存在的代谢途径,为细胞提供能量、还原力和合成前体。由于α-酮戊二酸脱氢酶的缺失,长期以来学术界一直认为蓝藻没有经典的三羧酸循环途径[8]。自Vermass研究组发现在集胞藻PCC6803中可以通过氧化半循环从α-酮戊二酸经由某种途径生成琥珀酸以来,已经发现3种可以使蓝藻三羧酸循环变得完整的旁路,分别为:α-酮戊二酸脱羧酶/琥珀酸半醛脱氢酶途径、乙醛酸途径以及γ-氨基丁酸途径。Vermaas研究组和Bryant研究组分别通过体内及体外实验证明了集胞藻PCC6803中slr1022基因编码的蛋白具备编-氨基丁酸转氨酶(GABA-AT)功能[9-10]。然而氨基酸序列分析表明slr1022基因编码的蛋白是N-乙酰鸟氨酸转氨酶(AcOAT)。此外,Bryant研究组于2016年证实slr1022基因编码的重组蛋白体外具备AcOAT功能[10]。但是slr1022基因编码的蛋白作为N-乙酰鸟氨酸转氨酶的生化性质以及具体的酶动力学参数还未被表征,相关内容还未报道。
N-乙酰鸟氨酸转氨酶是磷酸吡哆醛(Pyridoxal phosphate,PLP)依赖性酶[11],属于此家族的酶还有GABA-AT,鸟氨酸转氨酶(Ornithine aminotransferase OAT),2,2-二烷基甘氨酸脱羧酶等,它们大多数为二聚体或四聚体,而且其单体均由N-末端结构域、PLP结合结构域以及C-末端结构域组成[12]。该酶以PLP为辅因子催化N-乙酰鸟氨酸(N-acetylornithine,AcOrn)与N-乙酰谷氨酸半醛(N-acetylglutamate semialdehyde,NAGSA)的相互转化,是精氨酸代谢途径中的一个关键酶。因此,研究集胞藻PCC6803中AcOAT的生化性质对于理解集胞藻PCC6803的精氨酸代谢途径具有重要意义。而且,大肠杆菌来源的AcOAT还可以催化赖氨酸合成途径中N-琥珀酰基-L,L-二氨基庚二酸与N-琥珀酰基-L-2-氨基-6-氧庚二酸的相互转化[13],在细菌细胞壁的合成中起着重要作用。由于精氨酸和赖氨酸代谢在细菌中的重要作用,AcOAT也逐渐成为潜在的抑菌靶点。因此,对集胞藻PCC6803中AcOAT的生化研究也为抑菌药物的设计提供理论依据。
本研究以集胞藻PCC6803基因组DNA为模板,经PCR扩增得到slr1022基因,继而将其连接到pET-28a载体,构建得到pET28a-slr1022重组质粒。将该质粒转化大肠杆菌BL21(DE3)感受态,经IPTG诱导,亲和层析纯化后得到重组Slr1022蛋白,并对其进行酶动力学表征,同时利用生物信息学软件对Slr1022蛋白结构进行预测和模拟,旨在为进一步探索Slr1022蛋白的催化机制奠定重要基础。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒 大肠杆菌DH5α、BL21(DE3)菌株和表达载体pET-28a均来自于本实验室保藏,集胞藻PCC6803基因组DNA由中国科学院青岛生物能源与过程研究所吕雪峰研究员实验室提供。
1.1.2 主要试剂和仪器 2×Pfu DNA聚合酶、2×Taq Mix、限制性核酸内切酶Nde I、Xho I、T4 DNA连接酶、DNA Ladder、Protein Marker、质粒提取试剂盒、普通DNA纯化试剂盒、胶回收试剂盒均购于天根生化科技(北京)有限公司;定点突变试剂盒与Ni-NTA购于全式金生物技术(北京)有限公司;所用引物和测序由上海祥音生物科技有限公司合成和完成;其余主要试剂均为分析纯,均购于北京索莱宝科技有限公司。高速冷冻离心机,上海卢湘仪离心机仪器有限公司;超声波细胞粉碎机,宁波新芝生物科技股份有限公司;层析实验冷柜,北京亚星仪科科技发展有限公司;紫外-可见分光光度计,上海仪电分析仪器有限公司。
1.2 方法
1.2.1 目的基因的PCR扩增 以集胞藻PCC6803基因组DNA为扩增模板,通过PCR扩增得到slr1022基因片段。PCR反应体系:模板2 μL,上游引物(slr1022-F)和下游引物(slr1022-R)各1 μL(终浓度为 0.2 μmol/L),2×Pfu DNA 聚合酶 25 μL,超纯水21 μL。扩增反应条件:95℃预变性5 min;95℃变性1 min,53℃退火30 s,72℃延伸2 min,30个循环;72℃延伸10 min。引物序列如表1所示。

表1 PCR引物序列Table 1 PCR primer sequences
1.2.2 pET28a-slr1022重组质粒的构建与鉴定 经琼脂糖凝胶电泳后,将PCR扩增得到的slr1022基因片段进行胶回收。使用Nde I和Xho I 对slr1022基因进行双酶切并进行胶回收,用同样的方法得到pET-28a线性大片段(获得相同的黏性末端)。将酶切后的slr1022基因片段与pET-28a线性大片段(目的基因片段与载体片段的摩尔比为9∶1)借助T4 DNA连接酶于16℃水浴恒温下过夜连接。取连接液转化至大肠杆菌DH5α感受态细胞,涂布到含卡那霉素(50 μg/mL)的LB固体培养基上,置于37℃恒温过夜培养。通过菌落PCR验证,将正确的阳性克隆培养后送检测序。
1.2.3 重组Slr1022蛋白的诱导表达 将上述鉴定正确的阳性克隆单菌落培养提取质粒后,转化至大肠杆菌BL21(DE3)感受态细胞,然后涂布到含卡那霉素(50 μg/mL)的LB固体培养基,37℃恒温过夜培养。挑取单克隆菌落置于LB液体培养基中(含有50 μg/mL卡那霉素),于37℃、180 r/min的振荡培养箱中过夜培养。次日,以1%(V/V)的接种量接种到400 mL LB液体培养基(含50 μg/mL卡那霉素)中进行菌体扩大培养。待菌体生长至OD600~0.6时,冰浴30 min后,加入诱导剂IPTG(终浓度为0.2 mmol/L),于16℃、180 r/min 的振荡培养箱中诱导表达,24 h后离心收集菌体。使用20倍体积的缓冲液(20 mmol/L Tris-HCl pH 7.5)重悬菌体,超声破碎细胞后,通过SDS-PAGE电泳观察表达情况。
1.2.4 重组Slr1022蛋白的分离纯化 根据上述诱导表达条件重新诱导表达1.6 L的菌液,离心收集菌体。经20 mmol/L Tris-HCl pH 7.5缓冲液20倍体积重悬后,置于冰水中超声破碎细胞1 h(超声功率5%,超声工作时间2 s,超声间隔时间28 s)。然后将细胞破碎液于4℃、10 000 r/min冷冻离心1 h,收集上清液。利用0.45 μm的纤维素滤膜过滤上清液,将其流穿预先用缓冲液处理的Ni-NTA。随后用含有浓度为20 mmol/L-200 mmol/L的咪唑洗脱液进行梯度洗脱,分别收集咪唑洗脱液,用SDS-PAGE电泳检测目的蛋白的含量。然后合并含有高浓度高纯度的目的蛋白洗脱液,将其置于透析袋中,用聚乙二醇吸水浓缩。最后将浓缩液置于透析液(含1 mmol/L DTT,20 mmol/L Tris-HCl pH 7.5缓冲液)中透析3次,然后用Bradford方法对目的蛋白进行定量。
1.2.5 重组Slr1022蛋白的酶动力学表征 重组Slr1022蛋白的酶动力学常数在室温下测得。N-乙酰鸟氨酸转氨酶催化底物N-乙酰鸟氨酸与α-酮戊二酸转化为产物N-乙酰谷氨酸半醛和L-谷氨酸。L-谷氨酸在谷氨酸脱氢酶(GDH)的催化下生成α-酮戊二酸,此过程伴随着NAD+形成NADH,NADH可在340 nm处被检测,通过使用紫外分光光度计在340 nm处检测NADH的生成量来衡量N-乙酰鸟氨酸转氨酶的催化活性[13]。反应总体积为500 μL,反应体系中包含100 mmol/L Tris-HCl pH 8.5缓冲液,6 mmol/L NAD+,0.05 mmol/L-2 mmol/L N-乙酰鸟氨酸,1 mmol/L α-酮戊二酸,0.1 mmol/L PLP,10 U GDH,0.26 μmol/L 重组Slr1022蛋白。使用紫外分光光度计连续测定反应混合液在340 nm处吸光值A340的变化,测定不同N-乙酰鸟氨酸浓度下的初始反应速度,利用KaleidaGraph软件拟合到米氏方程中,计算出最大反应速度Vmax,米氏结合常数Km以及催化速率常数kcat。测定α-酮戊二酸与重组Slr1022蛋白的结合常数时,将N-乙酰鸟氨酸的浓度固定为2 mmol/L,α-酮戊二酸的浓度为0.01 mmol/L-1 mmol/L。实验重复3次。
1.2.6 pH对重组Slr1022蛋白催化活性的影响 采用分步法检测不同pH条件下重组Slr1022蛋白的最大初始反应速度。为了减少缓冲液种类对酶催化效率的影响,本实验用4种不同的缓冲液试剂配置8个不同的pH值。第一步:500 μL反应体系包含不同的缓冲体系(分别为:pH 6.0和6.5,100 mmol/L Bis-Tris;pH 7.0和7.5,100 mmol/L HEPES;pH 8.0和 8.5,100 mmol/L Tris-HCl;pH 9.0 和 9.5,100 mmol/L CHES),1 mmol/L α-酮 戊 二 酸,2 mmol/L N-乙酰鸟氨酸,0.1 mmol/L PLP,0.24 μmol/L 重组Slr1022蛋白。将该反应体系置于室温下反应10 min后,立即加热失活重组Slr1022蛋白,12 000 r/min离心10 min后,得到上清液。第二步:500 μL反应体系包含 100 mmol/L Tris-HCl pH 8.2,100 μL反应上清液,6 mmol/L NAD+,5 U GDH[14]。使用紫外分光光度计连续测定反应混合液在340 nm的吸光值(A340)的变化,并计算出初始反应速度。实验重复3次。
1.2.7 Slr1022蛋白结构分析 通过在线软件ESPript 3.0对Slr1022蛋白及其他同源性蛋白二级结构进行分 析(http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi),同时利用在线软件SWISS-MODEL对其三级结构进行预测(http://swissmodel.expasy.org/)。最后用Procheck方法以及在线软件Super-Pose(http://superpose.Wishartlab.com/)对建模后结果进行分析。
2 结果
2.1 pET28a-slr1022重组质粒的构建与鉴定
以集胞藻PCC6803基因组DNA作为模板,通过PCR扩增slr1022基因,经琼脂糖凝胶电泳后观察结果。结果表明,在约1.3 kb出现特异性条带(图1-A),该条带与slr1022基因理论大小(1 290 bp)相匹配。同时,在目的条带slr1022基因下方有一条明显的非特异性扩增条带(图1-A)。将双酶切后的pET-28a载体和slr1022基因片段(图1-B)通过T4 DNA连接酶连接,之后转化至大肠杆菌DH5α感受态细胞。采用pET-28a载体上的T7和T7 ter作为引物进行单克隆点菌落PCR,鉴定阳性克隆单菌落(图1-C)。由于菌落PCR扩增时包括载体上约250 bp的碱基,菌落PCR产物碱基数比目的slr1022基因碱基数高约250 bp(图1-B和1-C)。随机抽取验证正确的阳性克隆单菌落,摇瓶培养后提取质粒送检测序,测序结果表明该基因序列与NCBI数据库中slr1022基因序列完全一致。因此,本实验成功构建并得到pET28a-slr1022重组质粒。
2.2 重组Slr1022蛋白的诱导表达
将pET28a空载体与pET28a-slr1022表达质粒分别转化大肠杆菌BL21(DE3)感受态细胞,其菌液于16℃、180 r/min培养条件下经0.2 mmol/L IPTG诱导表达24 h,超声波破碎后,进行SDS-PAGE电泳,结果如图2所示。相比于泳道1中pET-28a空载体的大肠杆菌细胞破碎液,泳道4中pET28a-slr1022表达质粒在约45 kD处有大量蛋白表达。集胞藻PCC6803中slr1022基因编码一个含有430个氨基酸残基的蛋白质,其分子量为46.5 kD,重组后带有6xHis标签的蛋白理论分子量为48.9 kD,与泳道4中条带大小一致。泳道5的蛋白上清液里含有重组Slr1022蛋白,说明其在大肠杆菌BL21(DE3)中部分可溶,可进行下一步的纯化。
2.3 重组Slr1022蛋白的分离纯化
将含有重组Slr1022蛋白的上清液流穿事先处理的Ni-NTA,用含有20 mmol/L-200 mmol/L咪唑缓冲液(20 mmol/L Tris-HCl pH 7.5)进行梯度洗脱。纯化结果如图3-A所示,当咪唑浓度增加至100 mmol/L时,少量的重组Slr1022蛋白被洗脱下来。当咪唑浓度增加至200 mmol/L时,大量重组Slr1022蛋白被洗脱下来。收集10-13泳道的咪唑洗脱液,于4℃下采用透析袋浓缩透析后,获得纯度大于95%的重组Slr1022蛋白(图3-B)。用Bradford方法对浓缩透析后的目的蛋白进行浓度测定,最终得到的蛋白质浓度为20.9 g/L,蛋白收率约为4.7 mg/g菌体。
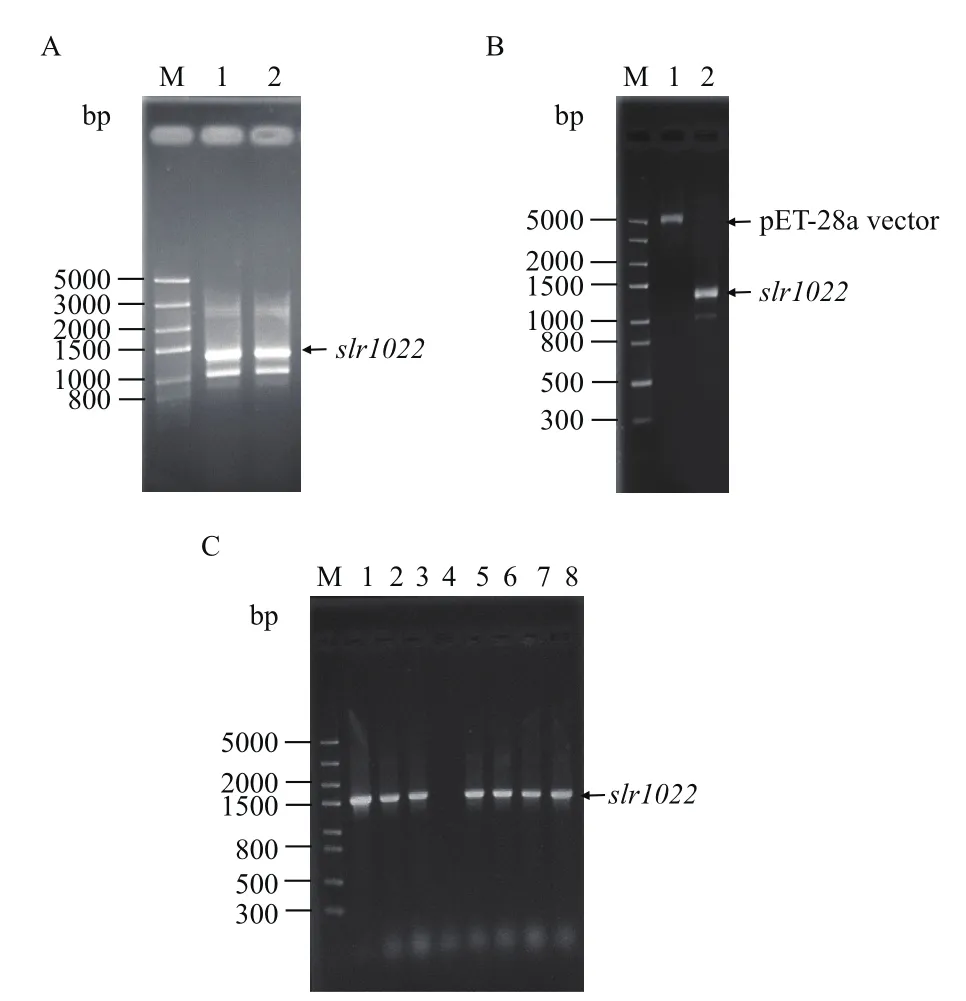
图1 集胞藻PCC6803中slr1022基因克隆及pET28aslr1022重组质粒的构建Fig.1 Cloning of slr1022 gene from Synechocystis sp.PCC6803 and construction of its recombinant plasmid pET28a-slr1022
2.4 重组Slr1022蛋白的酶动力学表征

图2 重组Slr1022蛋白的诱导表达Fig.2 Expression of recombinant Slr1022 protein

图3 重组Slr1022蛋白的亲和层析纯化结果电泳图Fig.3 SDS-PAGE of recombinant Slr1022 protein
当固定底物α-酮戊二酸浓度为1 mmo/L,改变底物N-乙酰鸟氨酸浓度时,测得的最大初始反应速度 Vmax为 0.60 ± 0.02 μmol/(L·s),重组Slr1022蛋白与底物N-乙酰鸟氨酸的结合常数Km为(0.12±0.01)mmol/L,反应体系中所用重组Slr1022蛋白浓度为0.26 μmol/L,由此可计算出重组Slr1022蛋白对底物N-乙酰鸟氨酸催化速率常数kcat为2.31 s-1,kcat/Km为 1.93×104mol/(L·s)(图4-A)。当固定底物N-乙酰鸟氨酸浓度为2 mmo/L,改变底物α-酮戊二酸浓度时,测得的最大初始反应速度Vmax为(0.65 ± 0.02)μmol/(L·s),重组 Slr1022 蛋白与底物α-酮戊二酸的结合常数Km为(0.039±0.004)mmol/L,通过计算可知重组Slr1022蛋白对底物α-酮戊二酸催化速率常数kcat为2.50 s-1,kcat/Km为6.41×104mol/(L·s)(图4-B)。由此可知,重组Slr1022蛋白与两个底物的结合能力均较强,催化效率较高。相比于底物N-乙酰鸟氨酸而言,重组Slr1022蛋白与另一底物α-酮戊二酸的亲和性和催化效率更高。
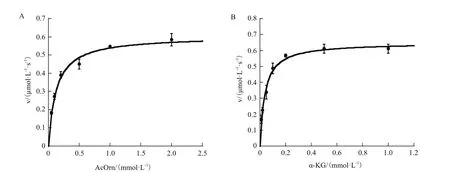
图4 重组Slr1022蛋白的酶动力学参数Fig.4 Kinetic profiles of recombinant Slr1022 protein
2.5 pH对重组Slr1022蛋白催化活性的影响
在pH范围为6.5-9.5的缓冲体系中重组Slr1022蛋白表现出不同的催化活性。当缓冲液的体系为pH 8.5,100 mmol/L Tris-HCl时,重组Slr1022蛋白催化底物N-乙酰鸟氨酸转化成产物N-乙酰谷氨酸半醛的活性最强。总体而言,重组Slr1022蛋白在碱性条件下行使催化功能较酸性条件下更高(图5)。
2.6 Slr1022蛋白的结构
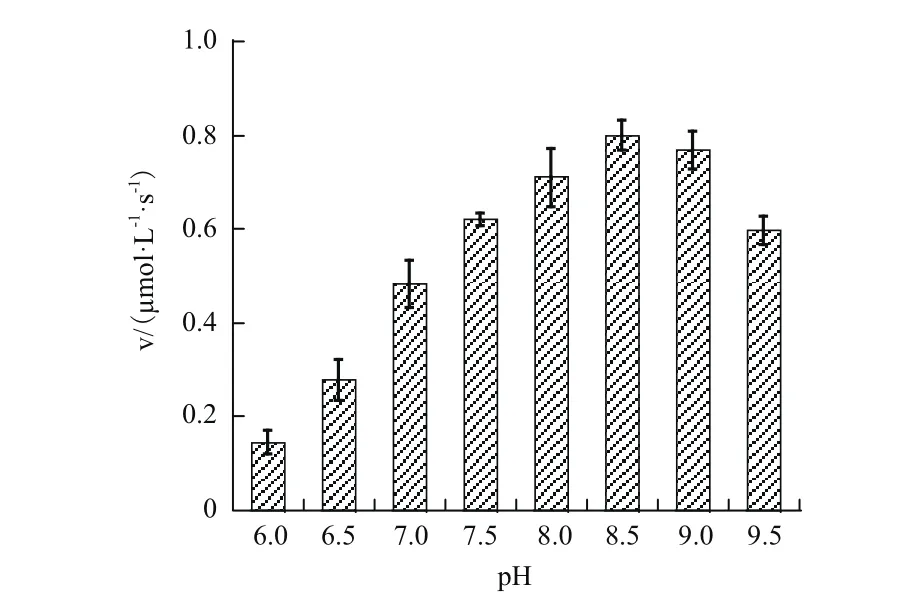
图5 pH对重组Slr1022蛋白催化活性的影响Fig.5 Effects of pH on the catalytic activity of recombinant Slr1022 protein
集胞藻PCC6803中的slr1022基因编码的蛋白含有429个氨基酸残基,在NCBI数据库中该蛋白被预测为N-乙酰鸟氨酸转氨酶(AcOAT),属于磷酸吡哆醛依赖性酶家族。Slr1022蛋白和其他4个已知晶体结构的不同来源的AcOAT的氨基酸序列比对结果,如图6所示。Slr1022蛋白与其他来源的AcOAT氨基酸序列一致性普遍较低,而其中与海栖热袍菌来源AcOAT(PDB ID:2E54)的序列一致性最高,为47.5%。虽然它们之间的氨基酸序列同源性较低,但其活性中心的氨基酸残基高度保守。用在线软件ESPript对Slr1022蛋白的二级结构进行预测,结果如图6所示,该蛋白由13个α-螺旋、17个β-折叠及多个无规则卷曲组成。
由于Slr1022蛋白和海栖热袍菌来源AcOAT的氨基酸序列同源性为47.5%,因此选择海栖热袍菌来源AcOAT的晶体结构(PDB ID:2E54)为模板,运用SWISS-MODEL在线软件(http://swiss-model.expasy.org)对Slr1022蛋白结构进行建模(图7)。模拟出Slr1022蛋白的结构为同源二聚体,具有两个相等的活性位点,与大部分磷酸吡哆醛依赖性酶相似。该酶的活性中心位于晶体结构的中心区域(图7-A),活性中心的氨基酸残基大部分位于无规卷曲或者α-螺旋,这与预测的二级结构结果是一致的(图6和图7-B)。辅因子PLP位于活性位点口袋,被紧密地包裹在活性口袋内。Slr1022蛋白与PLP结合的活性中心示意图如图7-C所示,辅因子PLP通过Schiff碱共价结合到Lys280的ε-氨基上(图中未显示),吡啶环上的氮原子和羟基分别与Asp251和Gln254有氢键作用。磷酸根上的氧二和氧三分别与主链上的Ser125、Gly126、Ala127组成的酰胺氮形成氢键,额外的氢键由第二亚单位Thr308的侧链羟基提供的。

图6 不同来源的N-乙酰鸟氨酸转氨酶的氨基酸序列比对Fig.6 Protein sequences alignment of N-acetylornithine aminotransferases from different sources
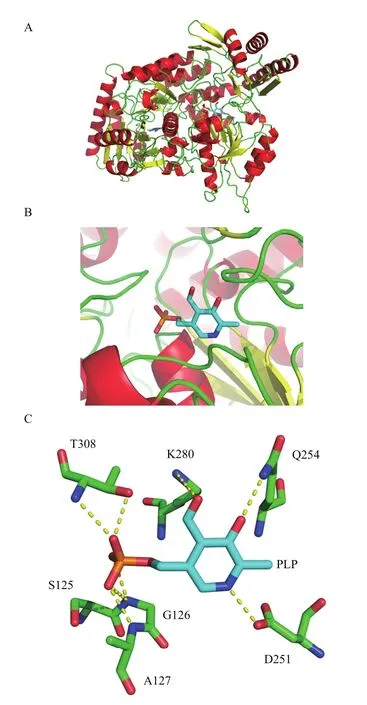
图7 Slr1022蛋白分子同源建模结构Fig.7 Homologous modeling structure of Slr1022 protein
3 讨论
集胞藻PCC6803中slr1022基因编码的N-乙酰鸟氨酸转氨酶,其具体的酶动力学参数以及生化性质此前还未报道。本研究对Slr1022蛋白酶动力学参数进行了详细表征,结果显示Slr1022蛋白与底物N-乙酰鸟氨酸的结合常数Km为(0.12 ± 0.01)mmol/L,催化效率 kcat/Km为 1.93×104mol/(L·s),说明Slr1022蛋白是N-乙酰鸟氨酸转氨酶,而且与底物N-乙酰鸟氨酸的亲和力较强,催化效率较高。与大肠杆菌和鼠伤寒沙门氏菌来源的N-乙酰鸟氨酸转氨酶相比,Slr1022蛋白对底物N-乙酰鸟氨酸的亲和力与大肠杆菌来源蛋白(0.15 mmol/L)相似,比鼠伤寒沙门氏菌来源蛋白(0.037 mmol/L)的弱,Slr1022蛋白的催化效率比大肠杆菌来源[4.0×103mol/(L·s)]高,但比鼠伤寒沙门氏菌来源[4.2×105mol/(L·s)]低[12-13]。不同的缓冲体系对 Slr1022蛋白催化活性有较大的影响。当缓冲液的体系为pH 8.5,100 mmol/L Tris-HCl时,Slr1022蛋白作为N-乙酰鸟氨酸转氨酶的催化活性最强。相对而言,Slr1022蛋白在碱性条件下的催化功能较酸性条件下更强。这一结果与来自于Corynebacterium crenatum[15]和 Klebsiella aerogenes[16]的 AcOAT 的酶学性质相似,其最适pH分别为8.0和8.7,同为碱性条件下行使最佳催化活性。
尽管Slr1022蛋白与其他来源的N-乙酰鸟氨酸转氨酶的氨基酸序列同源性不高,但是三级结构尤其是活性中心氨基酸残基极为保守。Mehta等[17]对氨基转移酶进行了序列比对发现,在所有被研究的序列中有4个已知保守的残基:赖氨酸、天冬氨酸、精氨酸和甘氨酸。其中活性位点赖氨酸通过醛亚胺键与PLP共价锚联;天冬氨酸残基与PLP吡啶环上的质子化氮形成氢键,作为锚定位点;精氨酸残基锚定在底物的羧基上,以促进催化周转;甘氨酸残基是已知的底物结合位点的屋顶,但其具体的功能研究较少。集胞藻PCC6803来源的N-乙酰鸟氨酸转氨酶中活性位点的Lys280通过醛亚胺键与PLP共价连接,Asp251与PLP吡啶环上的质子化氮形成氢键。此外,与PLP有氢键作用的氨基酸残基还有Ser125、Gly126、Ala127、Gln254以及另一单体的Thr308。近些年来,对N-乙酰鸟氨酸转氨酶的底物结合及催化功能的研究较少,但是对同家族的OAT与GABA-AT的研究较多。其中,人来源的OAT,由于Arg413与Glu235相互作用,底物鸟氨酸的α-羧酸被Arg180识别,将鸟氨酸的δ-氨基正确地定位到进行转移功能的辅因子PLP上[18]。这与猪来源的GABA-AT的γ-氨基丁酸类似,其中Glu270通过盐桥与Arg445相互作用,γ-氨基丁酸的α-羧酸通过Arg192静电保持,将γ-氨基定位到辅助因子PLP[19]。同理,集胞藻PCC6803来源的AcOAT中,也可能是由于Arg402与Glu223相互作用,底物N-乙酰鸟氨酸的α-羧酸被Arg163识别,最终将N-乙酰鸟氨酸上的δ-氨基正确地定位到PLP上。
通过不同的方法对Slr1022蛋白的结构模型质量进行检测。首先在SWISS-MODEL中,Slr1022蛋白结构模型的QMEAN(模型全局质量评估)得分为-0.24,此值接近于零,表明该模型与已知晶体结构的相似蛋白之间具有良好的一致性;其次使用Procheck方法对得到的Slr1022蛋白结构进行验证,结果显示99.4%的残基位于Ramachandran图的允许区域内,说明该模型的构象符合立体化学的规则;最后使用在线软件Super-Pose计算出Slr1022蛋白结构中Cα原子和海栖热袍菌来源AcOAT之间的RMSD(均方根偏差)为0.23 Å,说明模拟Slr1022蛋白结构与模板的结构重叠度非常高。综上所述,模拟的Slr1022蛋白结构准确性较高。
研究表明,slr1022基因编码的蛋白还具备γ-氨基丁酸转氨酶功能,而γ-氨基丁酸转氨酶是集胞藻PCC6803中三羧酸循环GABA代谢旁路中一个的关键酶,是调节细胞内GABA浓度的重要蛋白[9]。GABA是一种天然的非蛋白质氨基酸,在生物体内具有重要的生理学功能。在细菌和植物中,GABA对于维持细胞内的碳氮代谢平衡至关重要[20-21]。此外,GABA在植物的正常生长和抵抗胁迫过程中也发挥了重要作用[22-23]。在哺乳动物中,GABA是一种重要的抑制性神经递质[24]。细胞内GABA浓度过低会引起多种神经系统失调,如癫痫、帕金森症、阿尔兹海默症、舞动及可卡因上瘾等症[25]。因此,Slr1022蛋白作为γ-氨基丁酸转氨酶功能的催化动力学表征将是我们下一步的研究对象。
4 结论
本研究利用分子生物学手段成功构建pET-28aslr1022重组质粒,将其转化大肠杆菌BL21(DE3)感受态,经IPTG诱导,Ni-NTA亲和层析纯化后获得重组Slr1022蛋白。对Slr1022蛋白进行酶动力学表征,Slr1022蛋白与底物N-乙酰鸟氨酸的结合常数Km和最大反应速度Vmax分别是0.12 mmol/L和0.60 μmol/(L·s),与另一底物 α-酮戊二酸的 Km和Vmax分 别是 0.039 mmol/L 和 0.65 μmol/(L·s)。 此外,Slr1022蛋白在pH 8.5时催化活性最强。生物信息学分析表明Slr1022蛋白和其他来源的AcOAT在结构上有较高的相似性,活性中心氨基酸具有高度保守性。酶学性质和生物信息学研究表明集胞藻PCC6803来源的重组Slr1022蛋白具有N-乙酰鸟氨酸转氨酶功能。