Bacillus subtilis转录因子CtsR蛋白中clpC操纵子结合区域的NMR研究
2021-06-09陈晓雯黄碧玲黄少华赵玉芬
陈晓雯,黄碧玲,黄少华#,赵玉芬,2,3*
1. 宁波大学 新药技术研究院,浙江 宁波 315211;2. 厦门大学 化学化工学院,福建省化学生物学重点实验室,福建 厦门 361005;3. 清华大学 化学系,生命有机磷化学与化学生物学重点实验室,北京 100084
引 言
蛋白质和核酸是组成生命体的重要生物大分子,它们各自具有结构特征和特定功能,二者间的相互作用构成了诸如生长、繁殖、运动、遗传和代谢等生命现象的基础,在整个基因组表达调控的过程中发挥着重要作用.作为一种重要的动态生物调节过程,蛋白质磷酸化是生物体内最普遍、最重要的一种蛋白质翻译后修饰方式,具有非常重要的生物学意义[1].
磷酸化修饰可以改变蛋白质的构象、活性以及蛋白-蛋白间相互作用等,从而对信号传导、基因表达、细胞分裂等生物学过程发挥重要的调控功能[2].目前,构成天然蛋白质的 20种氨基酸中有 9种氨基酸的侧链可以发生磷酸化修饰,主要分为以下四种类型:O-磷酸化(以P-O键连接的磷酸化丝氨酸、苏氨酸和酪氨酸)、N-磷酸化(以P-N键连接的磷酸化组氨酸、精氨酸和赖氨酸)、COO-磷酸化(以P-(CO)O键连接的磷酸化天冬氨酸或谷氨酸),以及S-磷酸化(以P-S键连接的磷酸化半胱氨酸)[3].由于O-磷酸化比较稳定,易于检测、分析,因此当前对于蛋白质磷酸化的研究主要集中于O-磷酸化[4].近年来,O-磷酸化蛋白激酶作为药物靶点已被广泛研究,基于这些研究,目前已经有30多种新药被开发并成功上市[5].虽然相比于 O-磷酸化,N-磷酸化的研究才刚刚起步,但有研究[4]表明 N-磷酸化在生命过程中可能发挥着重要作用,因此亟需我们从多方面开展相关基础研究,从而为后续新药研发提供新的思路.
2009年,Clausen等[6]报道了枯草芽孢杆菌中的第一个也是目前唯一一个精氨酸激酶McsB.他们发现,当细菌响应热刺激时,McsB磷酸化转录因子CtsR蛋白中结合clpC操纵子区域的Arg62(R)位点,导致CtsR与clpC操纵子发生解离,从而启动clpC相关基因的转录.将R突变为天冬氨酸或谷氨酸后,突变体CtsR蛋白则不能结合clpC操纵子.由此说明位于CtsRβ转角的R位点是调控相关基因转录的关键残基.此外,他们解析了CtsR与clpC操纵子的复合物晶体结构,发现R侧链结合于clpC操纵子的小沟.蛋白质精氨酸磷酸化不仅在调控相关基因转录过程中具有重要意义,而且作为被ClpCP蛋白酶降解的标签,在蛋白质量控制中起关键作用[7].2012年,YwlE蛋白被鉴定为精氨酸磷酸酶,同时它也是McsB的互补调节蛋白[8];2014年,Clausen等在敲除ywlE基因的枯草芽孢杆菌中鉴定出包含217个精氨酸磷酸化位点的134个蛋白,这些蛋白可能在细菌蛋白质量控制、基因转录调控、核酸修饰、核糖体功能等方面发挥重要作用[9];2015年和2016年,鼠抗和兔抗的精氨酸磷酸化多克隆抗体相继被开发[10,11],尽管目前没有应用的相关案例,但是可以预见抗体的开发将会给精氨酸磷酸化的生理功能研究带来极大的便利[12].
蛋白质精氨酸磷酸化改变了精氨酸侧链的电荷性质,即精氨酸侧链由于共价连接了带有负电荷的磷酸从而由正电荷转变为负电荷;同时磷酸化改变了蛋白构象,进而影响蛋白质与 DNA的相互作用,从而发挥调控基因转录的功能.然而,精氨酸磷酸化如何影响多肽或蛋白构象仍然未知,且精氨酸磷酸化调控CtsR中clpC操纵子结合区域(KRGGGG)与clpC操纵子相互作用的机制尚未完全清楚.目前研究蛋白质磷酸化主要使用质谱技术,而质谱需要对样品进行酸化处理,这对于高能且不耐酸的P-N修饰是极其不利的,极易导致N-磷酸化的丢失.而用于核磁共振(NMR)检测的样品可保持在接近生理pH条件下,这对于不耐酸的精氨酸磷酸化的研究极为有利,而且 NMR技术可以研究动态过程,如磷酸化与去磷酸化过程等.
本文以转录因子CtsR中结合clpC操纵子的区域(KRGGGG)为研究对象(图1),通过1H NMR、1H-1H COSY、1H-1H TOCSY、1H-15N HSQC以及1H-13C HSQC谱图对该片段进行化学位移归属(化学位移的指认均对应图1中的原子编号),以期通过获得的NMR数据从原子水平为精氨酸磷酸化调控相关基因转录机制的研究提供可靠基础.

图1 KRGGGG肽段的结构Fig. 1 The structure of KRGGGG
1 实验部分
1.1 试剂
含有内标物四甲基硅烷(TMS)的氘代水(D2O,氘代率为99.9%)购于青岛腾龙微波科技有限公司;磷酸盐缓冲液(pH=6.33)由本课题组配制,分别称量0.06 g的KH2PO4、0.36 g的Na2HPO4·2H2O、2 g的NaCl和0.05 g的KCl,将其溶解在200 mL去离子水中.
1.2 肽段和DNA片段样品制备
KRGGGG肽段样品委托上海生工生物工程有限公司合成并纯化,溶于磷酸盐缓冲液中,用于后续实验.
clpC操纵子中与KRGGGG肽段结合的DNA片段委托上海生工生物工程有限公司合成,两条互补单链DNA序列分别为5′-ATTAAGGTCAAA-3′和5′-TTTGACCTTAAT-3′,两条单链DNA分别溶于去离子水,取相同体积混合至一起,在95 ℃的温度下变性5 min后,再在室温下退火2 h获得双链DNA(dsDNA)溶液,保存于-20 ℃备用.
1.3 电子圆二色谱(ECD)滴定实验
使用JASCO-1700型ECD谱仪.将190 μL浓度为63.44 μmol/L DNA溶液置于1 mm比色皿中,以浓度为3 mmol/L KRGGGG溶液滴定DNA溶液.每次向DNA溶液中滴加2μL KRGGGG溶液,直至ECD信号不再变化,即滴定达到饱和.扫描波长范围为200~320 nm,扫描速率为50 nm/min.
1.4 NMR实验
向KRGGGG溶液中加入10%(V/V)D2O,最终配制成浓度为8.38 mmol/L的肽段溶液,转移至外径为2.5 mm的微量NMR样品管中,全部NMR实验均在配备了5 mmZ梯度场反向四共振超低温探头的Bruker Ascend 600 MHz谱仪上完成,实验温度恒定在298 K,由Bruker VT-200温控器控制,控温精度为 ±0.1 K.1H、13C和15N核的工作频率分别为600.38、150.96和60.83 MHz,1H NMR谱宽为11 904.8 Hz.二维NMR实验均采用Bruker标准脉冲序列,用zgwegp脉冲序列压制溶剂水峰.其中1H-1H COSY谱脉冲序列选择为cosydfesgpph,采样数据点阵t2×t1=2 048×256,累加32次;1H-1H TOCSY谱脉冲序列选择为mlevesgpph,采样数据点阵t2×t1=2 048×256,累加32次;1H-15N HSQC谱脉冲序列选择为hsqcfpf3gpphwg,采样数据点阵t2×t1=2 048×256,累加32次;1H-13C HSQC谱脉冲序列选择为hsqcfpf3gpphwg,采样数据点阵t2×t1= 2 048×256,累加32次.对各自由感应衰减(FID)信号进行傅里叶变换,随后对1H-1H COSY和1H-1H TOCSY谱图使用窗函数QSINE进行处理;对1H-15N HSQC和1H-13C HSQC谱图使用窗函数SINE进行处理.
1.5 NMR滴定实验
向KRGGGG溶液中加入10% (V/V)的D2O,最终配置成浓度为1.07 mmol/L的肽段溶液,将200 μL KRGGGG溶液置于外径为2.5 mm的微量NMR样品管中,以浓度为5.35 mmol/L的DNA溶液在298 K温度下滴定.每次向KRGGGG溶液中滴加2 μL DNA溶液,直至信号不再变化,即滴定达到饱和.
2 结果与讨论
2.1 ECD实验
双链DNA具有特征性的CD信号,即存在206 nm和248 nm处的两个负峰以及218 nm和274 nm处的两个正峰,因此DNA的信号变化可用于表征DNA是否与其它分子存在相互作用.如图2所示,随着向DNA体系中不断加入KRGGGG,位于206 nm处CD信号逐渐减弱;而位于248 nm处的CD信号则逐渐增强,表明KRGGGG的加入导致DNA的CD信号发生变化,说明两者之间存在相互作用(后文将讨论基于NMR数据分析所得到的二者相互作用信息).因此,本文所研究的KRGGGG肽段与DNA相互作用,可作为研究转录因子与DNA相互作用模型肽.
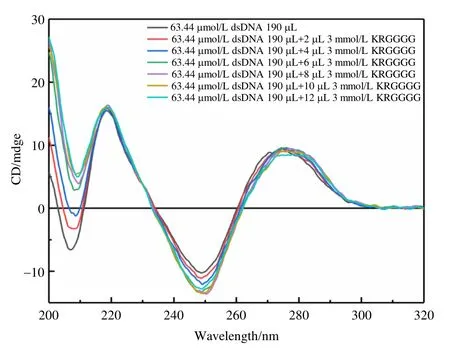
图2 KRGGGG滴定DNA的ECD光谱Fig. 2 ECD spectra of KRGGGG titration to DNA
2.2 1H核的归属
综合分析该片段(KRGGGG)的1H NMR谱(图3)和1H-1H TOCSY谱(图4),可知δH8.83归属为R2-NHα.在1H-1H TOCSY谱中:δH1.83和4.38均与R2-NHα相关,由化学位移规律,δH1.83可归属为R2-Hβ,δH4.38可归属为R2-Hα;δH1.69、δH3.22与R2-Hα相关,可归属为R2-Hγ、Hδ;δH7.18与R2-Hδ相关,可归属出R2-NHε.1H-1H COSY谱(图5)中,δH1.69与δH3.22的相关,以及δH1.83与δH1.69的相关,验证了上述归属.

图3 KRGGGG溶液的1H NMR谱Fig. 3 1H NMR spectrum of KRGGGG solution

图4 KRGGGG溶液的1H-1H TOCSY谱Fig. 4 1H-1H TOCSY spectrum of KRGGGG solution
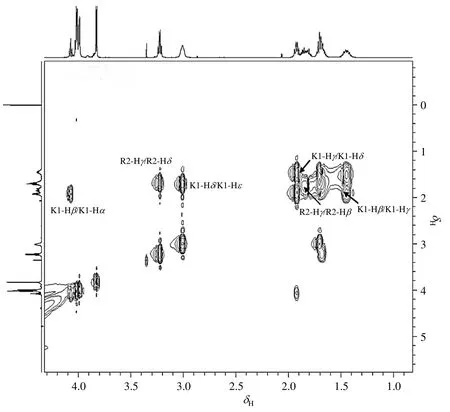
图5 KRGGGG溶液的1H-1H COSY谱Fig. 5 1H-1H COSY spectrum of KRGGGG solution
由于G4与G5化学环境相同,δH8.37/8.38可归属为G4-NHα/G5-NHα;在1H-1H TOCSY谱中:δH4.01/4.02与G4-NHα,G5-NHα相关,可分别归属为G4-Hα/G5-Hα;同时G3,G4与G5的Hα也处于相似化学环境,因此G3-Hα应与G4-Hα/G5-Hα有相近的化学位移,于是δH3.98可归属为G3-Hα,在1H-1H TOCSY谱中,δH8.66与G3-Hα相关,可归属为G3-NHα;此外也获得了δH3.83与δH8.08的相关信号,于是可以将δH3.83与δH8.08分别归属为G6-Hα和G6-NHα.
由于K1-Hα与K1-Hβ、Hγ、Hδ相关,从1H-1H TOCSY谱的局部图可将δH4.07归属为K1-Hα.在1H-1H COSY谱中:δH1.92与K1-Hα相关,归属为K1-Hβ;δH1.45与K1-Hβ相关,归属为K1-Hγ;δH1.70与K1-Hγ相关,归属为K1-Hδ;δH3.01与K1-Hδ相关,归属为K1-Hε.
2.3 15N核的归属
在1H-15N HSQC(图6)谱中:δN124.60与R2-NHα相关,归属为R2-NαH;δN111.72与G3-NHα相关,归属为G3-NαH;δN109.16/108.91与G4-NHα,G5-NHα相关信号,归属为G4-NαH,G5-NαH;δN114.16与G6-NHα相关,归属为G6-NαH;δN84.72与R2-NHε相关,归属为R2-NεH.
2.4 13C核的归属
在1H-13C HSQC谱(图 7)中:δC42.54与 G4-Hα、G5-Hα、G3-Hα相关,归属为G4-Cα、G5-Cα、G3-Cα;δC43.00与G6-Hα相关,归属为G6-Cα;δC30.65与K1-Hδ相关,归属为K1-Cβ;δC21.04与K1-Hγ相关,归属为K1-Cγ;δC26.47与K1-Hδ相关,归属为K1-Cδ;δC39.35与K1-Hε相关,归属为 K1-Cε;δC28.12与R2-Hδ相关,归属为R2-Cβ;δC24.43与R2-Hγ相关,归属为 R2-Cγ;δC40.64与R2-Hδ相关,归属为R2-Cδ;δC52.90与K1-Hα相关,归属为K1-Cα(由于信号较弱,谱图要切很低才能看到);由于R2-Hα信号在实验过程中信号被部分压制,因此未获得R2-Cα信号.
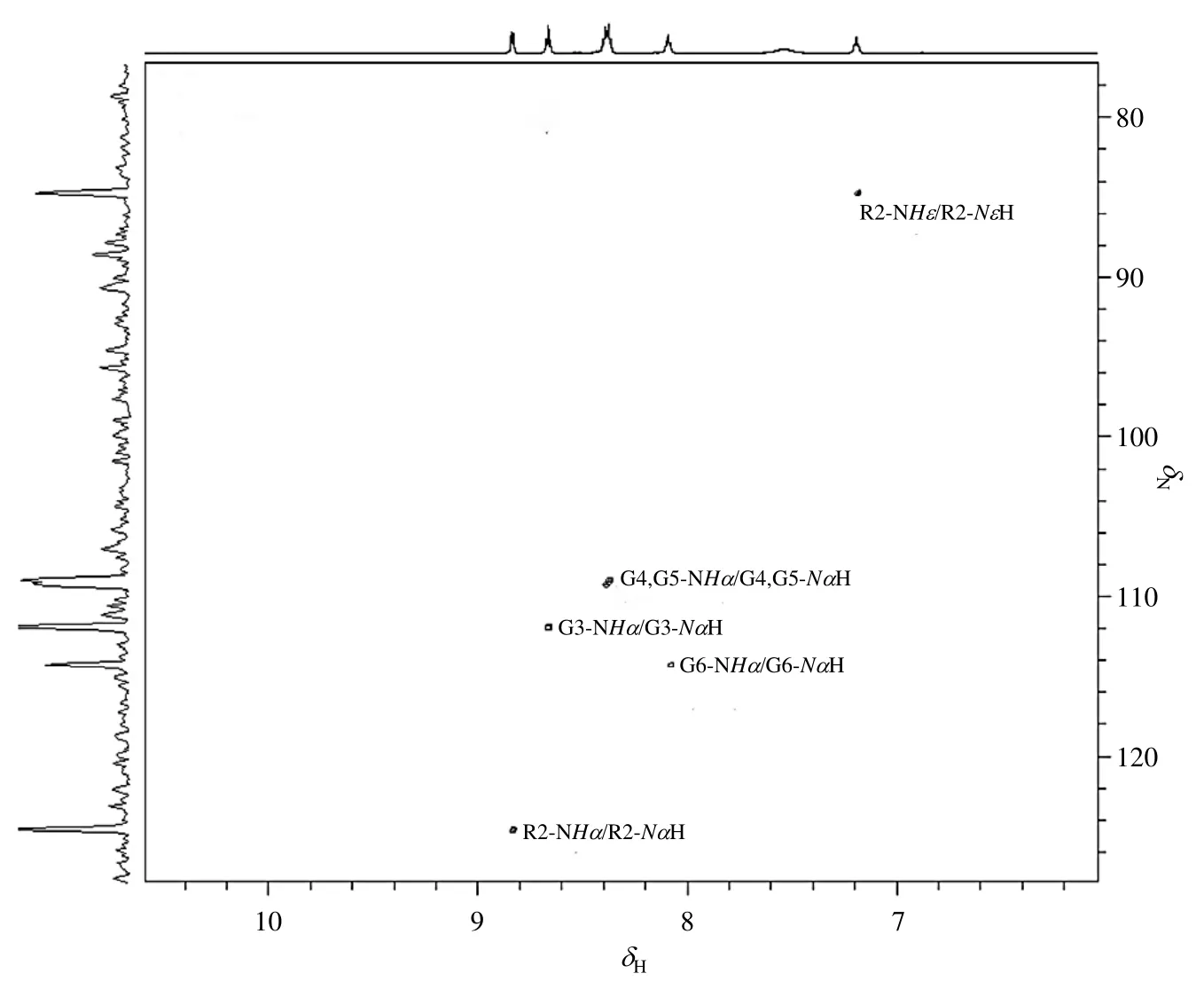
图6 KRGGGG溶液的1H-15N HSQC谱Fig. 6 1H-15N HSQC spectrum of KRGGGG solution

图7 KRGGGG溶液的1H-13C HSQC谱Fig. 7 1H-13C HSQC spectrum of KRGGGG solution
通过对以上NMR数据进行综合分析,完成了转录因子CtsR中与DNA相互作用区域KRGGGG片段的1H NMR、13C NMR以及15N NMR信号归属,其详细化学位移列于表1.

表1 KRGGGG片段的1H、13C以及15N化学位移Table 1 1H, 13C and 15N chemical shifts of KRGGGG
2.5 DNA滴定KRGGGG
为了进一步证明KRGGGG与DNA相互作用,我们进行了DNA滴定KRGGGG的NMR实验(图8).
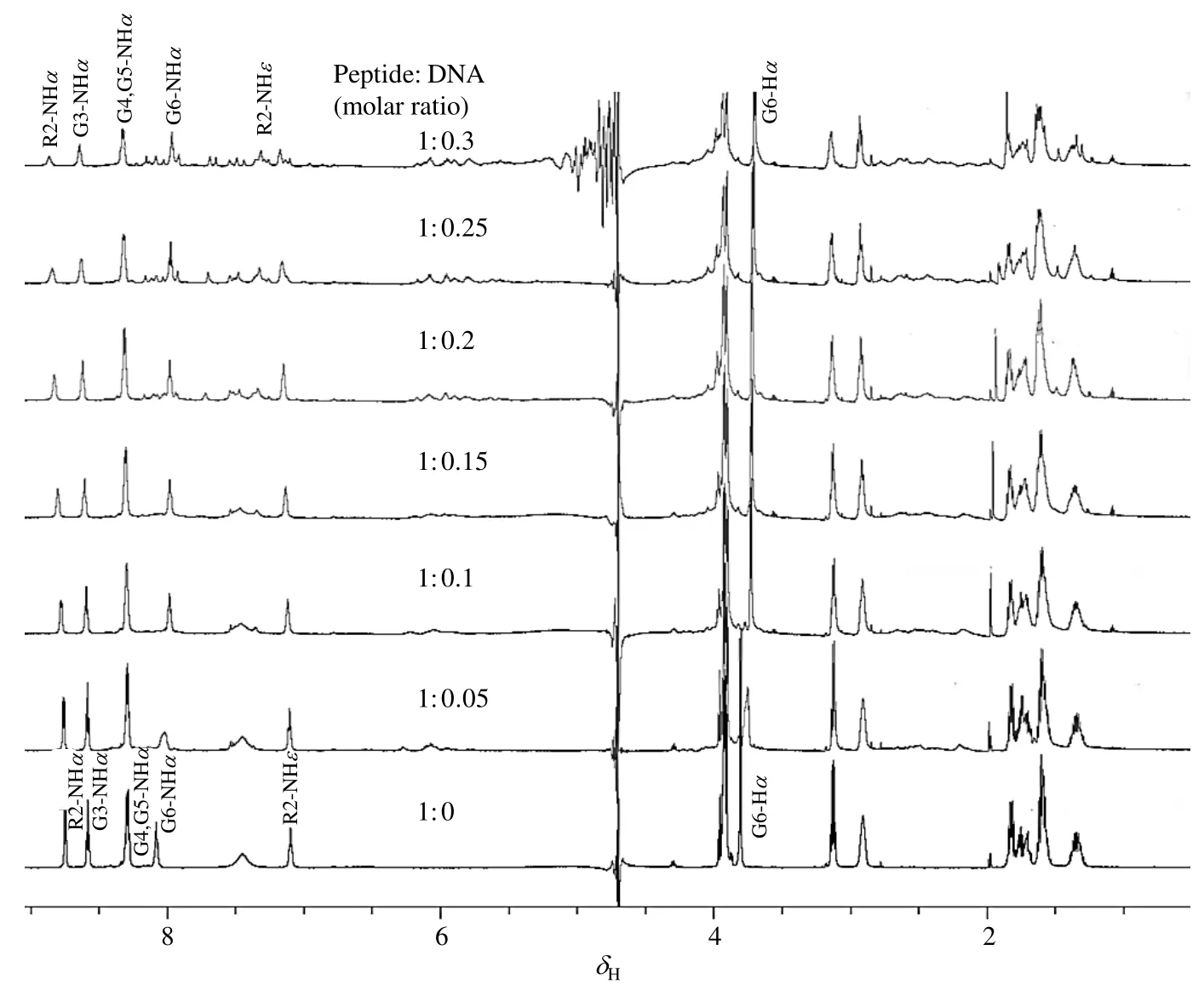
图8 DNA滴定KRGGGG的1H NMR谱图Fig. 8 1H NMR spectra of DNA titration to KRGGGG
从图中可以看出,随着DNA的不断滴加,多肽的1H NMR信号逐渐发生变化.主链R2-NHα、G3-NHα,G4-NHα、G5-NHα和侧链的 R2-NHε向低场移动;G6-NHα和侧链 G6-Hα向高场移动.说明这些原子的化学环境发生改变,而这些变化源于它们与DNA的相互作用.
3 结论
本文通过综合分析一维NMR谱图以及多种同核和异核相关二维NMR谱图,归属了枯草芽孢杆菌(Bacillus subtilis)转录因子CtsR中clpC操纵子结合区域KRGGGG的1H、13C和15N化学位移,获得了该肽段溶液结构的较为完整的NMR数据,为精氨酸磷酸化的调控机制研究提供了可靠科学的数据基础.
致谢
感谢国家自然科学基金资助项目(91856126)以及福建省化学生物学重点实验室(厦门大学)开放基金资助项目(2021001)对本文的资助.
利益冲突
无
