间隔区寡核苷酸DNA微阵列分型在结核分枝杆菌检测中的应用
2021-05-18杨建伟王少华杨卫华杨俊梅
杨建伟,李 辉,王少华,杨卫华,杨俊梅
(1.河南省儿童医院,郑州儿童医院,郑州市儿童感染与免疫重点实验室,河南 郑州 450018;2.河南省疾病预防控制中心,河南 郑州 450016;3.江苏猎阵生物科技有限公司,江苏 如东 226499)
结核分枝杆菌(Mycobacteriumtuberculosis,MTB)是引起结核病的病原菌,可侵犯全身器官,其中以肺结核最为常见。我国是结核病高发国家之一,结核病负担居全球第3位[1-2]。分子生物学技术结合流行病学调查形成了现代分子流行病学。分子分型技术是结核病分子流行病学的主要方法之一,可通过对MTB基因型鉴定而监测结核病暴发流行、传染源传播链追踪及实验室污染等,还可为结核病传播模式的建立提供数据支持[3-4]。近年来,研究者建立了多种MTB基因分型技术,IS 6110为基础的限制性片段长度多态性(IS 6110 Restriction Fragment Length Polymorphism,IS 6110-RLFP)[5]、多位点可变数目串联重复序列分析(multiple loci variable number tandem repeat analysis,MLVA)[6-7]和间隔区寡核苷酸分型(spacer oligonucleotide typing,Spoligotyping)[8-9]被广泛应用,其中Spoligotyping是公认的MTB一线分型方法。Spoligotyping是以聚合酶链反应(polymerase chain reaction,PCR)为基础,检测MTB基因组中直接重复区(direct repeat,DR)的基因分型方法,Spoligotyping通过识别间隔子的PCR产物来确定DR是否存在,在MTB基因分型中其具有效率高、识别力强和快速的特点[10]。目前,国际上已经建立了MTB Spoligotyping基因分型数据库SITVIT2(http://www.pasteur-guadeloupe.fr:8081/SITVIT_ONLINE/),该数据库包含世界各地MTB的Spoligotyping 分型结果,可供比对研究,将Spoligotyping分型结果与数据库比较就能获得不同MTB的间隔区寡核苷酸分型国际分类号(spoligotype international type,SIT)和基因型家族[11-13]。本研究旨在探讨间隔区寡核苷酸DNA微阵列法在MTB基因分型检测中的应用价值。
1 材料与方法
1.1 菌株来源97株临床MTB菌株和MTB标准菌株H37Rv、BCG菌株均为河南省疾病预防控制中心结核病参比实验室保存。
1.2 试剂与仪器含间隔区寡核苷酸的Biodyne C膜购自法国Pall Biosupport公司,Taq酶购自德国Qiagen公司,链霉亲和素-过氧化物酶连接液购自美国Sigma公司;Miniblotter转渍槽购自法国 Interchim公司,IQ5扩增仪购自美国Bio-Rad公司,杂交仪购自美国 Shellab公司,扩增引物序列均由生工生物工程(上海)股份有限公司合成。
1.3 实验方法
1.3.1 DNA提取使用Lab-Aid 824核酸提取仪(厦门致善生物科技股份有限公司)进行DNA提取,严格按照说明书操作。使用无菌接种环刮取1环MTB新鲜培养物,悬浮于300 μL DNA裂解液中,80 ℃灭活30 min,金属浴中95 ℃裂解 20 min。将裂解液加入提取试剂条第1孔中,安装磁棒套,将提取架放入仪器,选择设定程序进行提取。提取结束后,吸取试剂条最后1孔溶液即为提取的DNA溶液。
1.3.2 传统Spoligotyping法分型按照KAMERBEEK等[8]建立的Spoligotyping法对MTB进行分型。在PCR扩增仪进行提取DNA的扩增,DR上游引物序列为5′-GGTTTTGGGTCTGACGAC-3′(5′-端生物素标记),下游引物序列为5′-CCGAGACTGGGACGGAAAC-3′;扩增产物于99 ℃热变性后与Miniblotter转渍槽内的含间隔区寡核苷酸的Biodyne C膜进行杂交;杂交后的膜与链霉亲和素-过氧化物酶连接液反应;反应后的膜进行洗涤,洗涤后的膜与摄影底片一起曝光;底片显影后43个间隔区的每个区均会产生1条暗带(表明存在间隔区)或无暗带(表明不存在间隔区)。
1.3.3 间隔区寡核苷酸DNA微阵列方法分型间隔区寡核苷酸DNA微阵列法所用的探针和引物序列见表1,杂交步骤参照结核分枝杆菌基因分型试剂盒(江苏猎阵生物科技有限公司)说明书。具体如下:(1)PCR扩增:将2 μL提取的 DNA加入到18 μL预混好的PCR反应液,50 ℃、2 min,96 ℃、4 min;96 ℃、30 s,55 ℃、45 s,72 ℃、20 s,共30个循环;96 ℃、30 s,68 ℃、45 s,72 ℃、20 s,共20个循环;72 ℃、10 min。(2)杂交:在62 ℃恒温振荡器中进行杂交;向反应槽中分别加入600 μL反应液Ⅰ和20 μL PCR扩增产物,1 500 r·min-1反应10 min;洗涤液充分洗涤后加入600 μL预混合液(599 μL反应液Ⅰ+1 μL反应液Ⅱ)进行酶连,将晶格放入恒温振荡仪中,1 500 r·min-1反应3 min。(3)显色:洗涤后向每个反应槽内加入500 μL反应液Ⅲ,1 500 r·min-1显色反应3 min;每个反应槽内加入2~3 mL无菌水,终止反应,记录每个芯片上的反应结果。(4)结果判读和质量控制:间隔区寡核苷酸DNA微阵列43个探针的排序见图1,其中右下角为杂交质控(hybridization control,HC)点,杂交后,当HC点显色时,认定本次结果有效;然后将各分型探针的显色结果与下图猎阵芯片探针排布图进行比对,间隔区存在以“1”表示,间隔区缺失以“0”表示。阴性对照品的膜芯片除HC点有显色外,其他位置均不显色,有显色则表明很有可能出现污染情况。
1.3.4 灵敏度检测挑取1环生长3周的H37Rv菌落,制备1 麦氏浓度(每毫升含细菌3×108个)标准菌悬液,将此菌悬液做梯度稀释,获得每毫升含107、106、105、104、103、102个细菌的菌悬液,菌悬液80 ℃灭活30 min,95 ℃裂解10 min,用于间隔区寡核苷酸DNA微阵列法的灵敏度检测,配制的PCR体系内对应含菌量为每毫升106、105、104、103、102、101个。
1.4 统计学处理应用SPSS 11.0软件统计分析杂交结果,Spoligotyping法中的43个间隔区(Spac-er)中的每个间隔区均使用二进制数字“1”和“0”表示,“1”代表该间隔区存在,“0”表示不存在。43个数字即表示MTB的基因型别,八进制由相邻的3个点连在一起构成,3个点存在杂交点时分别计为4、2、1,合并后记为7。将各个菌株的数字代码输入Spoligotype的国际数据库SITVIT[5],以获得该菌株的SIT和基因型家族;并对2种检测方法的一致性进行分析。

表1 间隔区寡核苷酸DNA微阵列法探针和引物序列
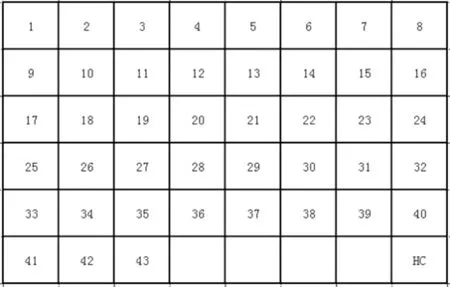
图1 间隔区寡核苷酸DNA微阵列43个探针排布示意图
2 结果
2.1 传统Spoligotyping法分型结果结果见表2。依据传统Spoligotyping法,在数据库SITVIT2中MTB标准菌株H37Rv的国际类型编号为451,基因型为T-H37Rv;BCG的国际类型编号为482,基因型为BOV_1。通过与SITVIT2数据库比对,97株临床MTB Spoligotyping指纹图谱表现出29种基因型,其中北京基因型家族(1~34间隔区缺失)61株(62.89%,61/97),包括典型北京基因型58株、非典型北京基因型(35~43间隔区也有缺失)3株;T基因型家族25株(25.77%,25/97),包括T1型15株、T2型6株、T3型4株;Manu2型2株;LAM9型1株;新发现未在数据库内的8株(8.2%,8/97)。
2.2 间隔区寡核苷酸DNA微阵列法分型结果97株临床MTB以及H37Rv和BCG菌株的寡核苷酸DNA微阵列法分型结果见表3。MTB标准菌株H37Rv和BCG的国际类型编号为451和482,基因型为T-H37Rv和BOV_1。97株临床MTB分出29种基因型,其中北京基因型家族61株(62.89%),包括58株典型北京基因型、3株非典型北京基因型;T基因型家族25株(25.77%),包括15株T1型、6株T2型、4株T3型;Manu2型2株;LAM9型1株;新发现8株(8.2%)。
2.3 间隔区寡核苷酸DNA微阵列法与Spoligotyping法的一致性检验97株临床MTB及H37Rv、BCG菌株的间隔区寡核苷酸DNA微阵列法分型结果与传统Spoligotyping法杂交结果比较显示,阳性探针的数目、位置与传统膜杂交完全一致,经与SITVIT2数据库比对,获得的SIT和基因型完全一样,2种方法的符合率为100.00%。
2.4 间隔区寡核苷酸DNA微阵列法的灵敏度将1麦氏浓度的H37Rv按照10倍梯度稀释后采用间隔区寡核苷酸DNA微阵列法检测,当检测体系含菌量降至每毫升10个时仍可以出现杂交图谱,只是杂交点清晰度降低,表明该方法有较好的检测效能。
3 讨论
DR是MTB复合群特有的基因序列,DR由保守的36 bp直接重复序列和35~41 bp的非重复序列组成,非重复序列作为间隔区将直接重复序列隔开,且所有菌株的间隔区序列不同。针对该序列的多态性,KAMERBEEK等[8]开发了Spoligotyping法,即通过在保守的36 bp直接重复序列设计通用引物扩增样本DNA,可以获得不同菌株的间隔区DNA片段,这些扩增片段与固定在固相膜上根据间隔区序列设计的探针进行杂交,通过曝光、显影、定影等,获得菌株的Spoligotyping指纹图谱。目前,国际上已经建立了Spoligotyping基因分型数据库SITVIT2,该数据库包含141个国家的近4万株MTB的Spoligotyping结果,可供研究人员进行比对[13]。
相对于传统的固相膜杂交技术,本研究采用间隔区寡核苷酸DNA微阵列法,使用不对称PCR扩增方式,扩增产物可以直接用于杂交和显色,不需要传统方法的曝光、显影、定影等操作,减少了操作步骤,缩短了操作时间,使得操作更简便。通过对97株临床MTB及H37Rv和BCG菌株进行检测,其检测结果与传统固相膜杂交结果完全一致;经标准菌株梯度稀释检测,结果显示该方法有较好的灵敏度。
传统的Spoligotyping杂交过程中扩增产物DNA直接暴露在开放环境中极容易扩散而污染检测环境,而间隔区寡核苷酸DNA微阵列法将杂交膜和DNA封闭在反应盒内,降低了DNA污染环境的概率。间隔区寡核苷酸DNA微阵列法的显色结果可以在配套的结果自动扫描仪上进行扫描和判读,提高了检测效率和准确性。但2种方法均需要经历DNA扩增、变性、杂交、洗脱和显色等过程,整个实验过程相对复杂,手工操作较多,自动化程度较低,需要进一步优化检测步骤,提高自动化程度。此外,北京基因型家族菌株在全球流行的MTB菌株中占有主导地位,但Spoligotyping作为一线分型方法对北京基因型家族菌株的分辨率不足[14],需要使用更高分辨率的方法对北京基因型家族菌株进一步分型。
本研究显示间隔区寡核苷酸DNA微阵列杂交方法较传统Spoligotyping法更操作更简便快速,可以用于结核病溯源和分子流行病学调查研究。
