基于转录组学的不明原因复发性流产关键基因及机制分析
2021-04-19肖润颖肖建华王比男李寒梅郭海春莫鸿英黎小弟任懂平柳红艳
肖润颖,肖建华,王比男,李寒梅,郭海春,莫鸿英,黎小弟,任懂平,付 枭,柳红艳,黎 丽,
(1.南华大学衡阳医学院病原生物学研究所,湖南省衡阳市 421001;2.长沙市妇幼保健院,湖南省长沙市 410007;3.优生贝(北京)生物技术有限公司,北京市 102206)
复发性流产(recurrent spontaneous abortion,RSA)是指和同一性伴侣存在连续3次及以上、主要发生在孕28周之前的自然流产(spontaneous abortion,SA)[1]。RSA病因复杂,其潜在病因包括生殖系统解剖结构异常、内分泌代谢疾病、母体凝血功能异常、自身抗体的产生及感染等[2-3]。但40%~60%的RSA患者不属于以上任何原因,确切原因尚不清楚,故称不明原因复发性流产(unexplained recurrent spontaneous abortion,URSA)[4]。有研究表明,成功的妊娠需要母体和胚胎之间保持动态的免疫平衡,一旦平衡状态被打破,则可能导致母体对胎儿组织的排斥反应,引起流产或者胚胎停止发育[5]。
探究URSA相关的基因表达谱和关键信号通路有助于临床寻找新的治疗靶点或标记物,为此研究者们进行了许多尝试。Strug等[6]通过对子宫内膜修复部位组织的转录组测序(RNA-sequencing,RNA-seq),发现URSA的发生与子宫内膜γ干扰素(interferon γ,IFN-γ)等炎症信号通路上调影响内膜的修复有关,且转录因子RBPJ可能是其关键调节因子。Söber等[7]对早孕胎盘绒毛组织进行转录组测序,发现差异表达基因(differentially expressed genes,DEGs)主要参与DNA的复制、RNA的转录及加工、线粒体功能等,最关键的是大部分DEGs都受E2F/DP1转录因子的调节,且与不良妊娠结局发展或胎盘功能相关。Gu等[8]和Zhang等[9]发现了白细胞介素-10(interleukin-10,IL-10)和白细胞介素-1β(interleukin-1β,IL-1β)基因启动子区相关位点存在多态性变异与URSA的发生相关。然而,目前对这些机制的认识仍有许多疑问。
本研究采用RNA-seq对URSA患者与正常妊娠蜕膜组织转录组基因进行分析,运用高密度单核苷酸多态性(single nucleotide polymorphisms,SNP)芯片检测关键基因的位点多态性变异,从而探索母胎界面微环境的基因表达谱改变及与URSA发生相关的关键基因及遗传因素,为URSA的分子免疫机制研究和临床预防、诊断及治疗提供一定参考依据。
1 资料和方法
1.1 一般资料
本研究纳入长沙市妇幼保健院2018年4月—2020年10月的15例患者;其中6例不明原因复发性流产孕妇纳入URSA组,9例既往无流产史且无其他异常的人工流产孕妇纳入对照组。URSA组入组标准:年龄21~40岁,体质量指数(body mass index,BMI)19~24 kg/m2,孕周≤12周,连续自然流产次数≥3次。排除标准:生殖道解剖结构异常,内分泌、血栓史、感染、抗心磷脂抗体、染色体异常,孕期已接受黄体酮等药物治疗。对照组入组标准:年龄21~40岁,BMI 19~24 kg/m2,孕周≤12周,妊娠状况良好,既往无胚胎停育史,有一次以上正常生产史且子女健康者[3]。本研究经长沙妇幼保健院医学伦理学委员会批准,并获得患者知情同意。
1.2 样本采集和预处理
选取URSA组4例及对照组3例孕妇,获取其蜕膜组织,放入RNA组织保存液中充分渗透后(康为世纪,货号:CWY042)迅速冷冻,并储存在液氮中,以备后续RNA-seq检测。URSA组及对照组所有患者各取2 mL外周血于一根EDTANa2一次性负压采血管中,上下轻柔混匀,防止血液凝固,然后转移到样本冻存管中,-20 ℃冻存,待DNA提取和高密度SNP芯片检测用。
1.3 RNA测序
提取1.2中蜕膜组织总RNA。对所提取的RNA完成质量检测(其中对照组1例RNA样本不合格,弃去),质量检测合格后进行下一步的mRNA富集、片段化及cDNA合成。然后通过末端修复、加接头及扩增等步骤,完成文库的构建。文库质量检测合格后上机进行测序,测序及生物信息分析由优生贝(北京)生物技术有限公司完成。
1.4 测序数据分析
原始下机数据经过过滤及质量控制后得到Clean Reads,然后通过软件HISAT2(网址http://ccb.jhu.edu/software/hisat2/index.shtml)比对至人类参考基因组(版本:GRCh38),且需满足对比到人类参考基因组的唯一比对率均超过90%的标准。根据|log2(Fold Change)|>1,P<0.05的原则筛选出DEGs进行GO和KEGG分析。并将DEGs比对到STRING蛋白质相互作用数据库,构建蛋白相互作用网络(protein-protein interaction network,PPI network),找出关键节点基因并分析其可能的生物学功能。利用GSEA 3.0软件(网址http://www.gsea-msigdb.org/gsea/index.jsp)进行基因集富集分析(gene set enrichment analysis,GSEA),富集标准为:|NES|>1,NOM p-val<0.05,FDR q-val<0.25;NES的正负分别指示基因集的升高和降低(NES:标准化富集分数;FDR q-val:假阳性率;NOM p-val:标准化P值)。
1.5 高通量SNP芯片测序
采集URSA组和对照组外周血共15例,提取外周血DNA,使用高密度SNP芯片(ASA-CHIA,illumine,USA)对DNA样本进行单核苷酸多态性基因型检测,使用iScan平台对SNP芯片进行扫描分析,采用Bioconductor 软件(版本号:v3.12,网址http://www.bioconductor.org/)对获取的下机数据进行分析。操作简述为:将质检合格的DNA样品移入MSA3平板上,经片段化处理、恒温扩增、沉淀及重悬后与芯片孵育杂交,清洗后进行单碱基延伸及染色操作后上机分析。使用iScan(Illumina,美国)对芯片进行扫描及分析。所有实验操作流程均严格按照厂家标准操作说明书进行。芯片数据分析软件为 GenomeStudio(Illumina,美国,网址https://support.illumina.com/array/array_software/genomestudio/downloads.html)。
1.6 统计学方法

2 结 果
2.1 两组患者临床特征比较
两组患者年龄、身高、体质量、BMI、孕周、入院HCG及入院孕酮水平比较,差异均无统计学意义。
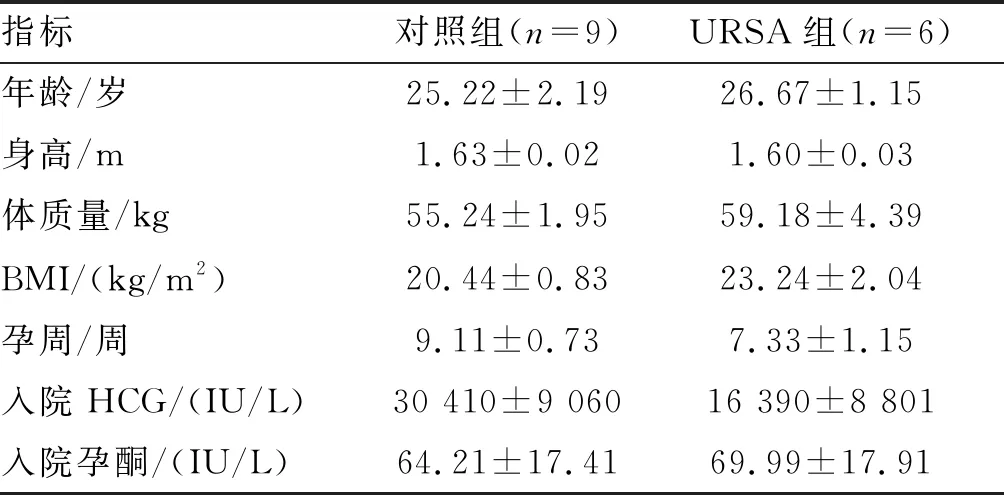
表1 两组患者临床特征比较
2.2 相关性分析与差异表达基因的筛选
两组数据的主成分分析(principal component analysis,PCA)结果如图1所示,URSA组(n=4)和对照组(n=2)在转录水平能得到了较好的线性分离。以|log2(Fold Change)|> 2,P<0.05为筛选标准,共获得1 057个差异基因。与对照组相比,URSA蜕膜组织中上调表达基因469个,下调表达基因588个。由此构建URSA患者基因差异表达热图(图2)。
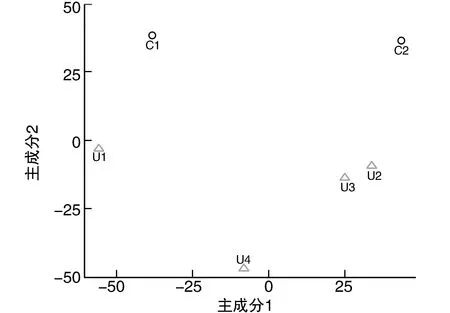
图1 蜕膜组织RNA测序数据主成分分析U1~U4为URSA组4例样本;C1~C2为对照组2例样本。
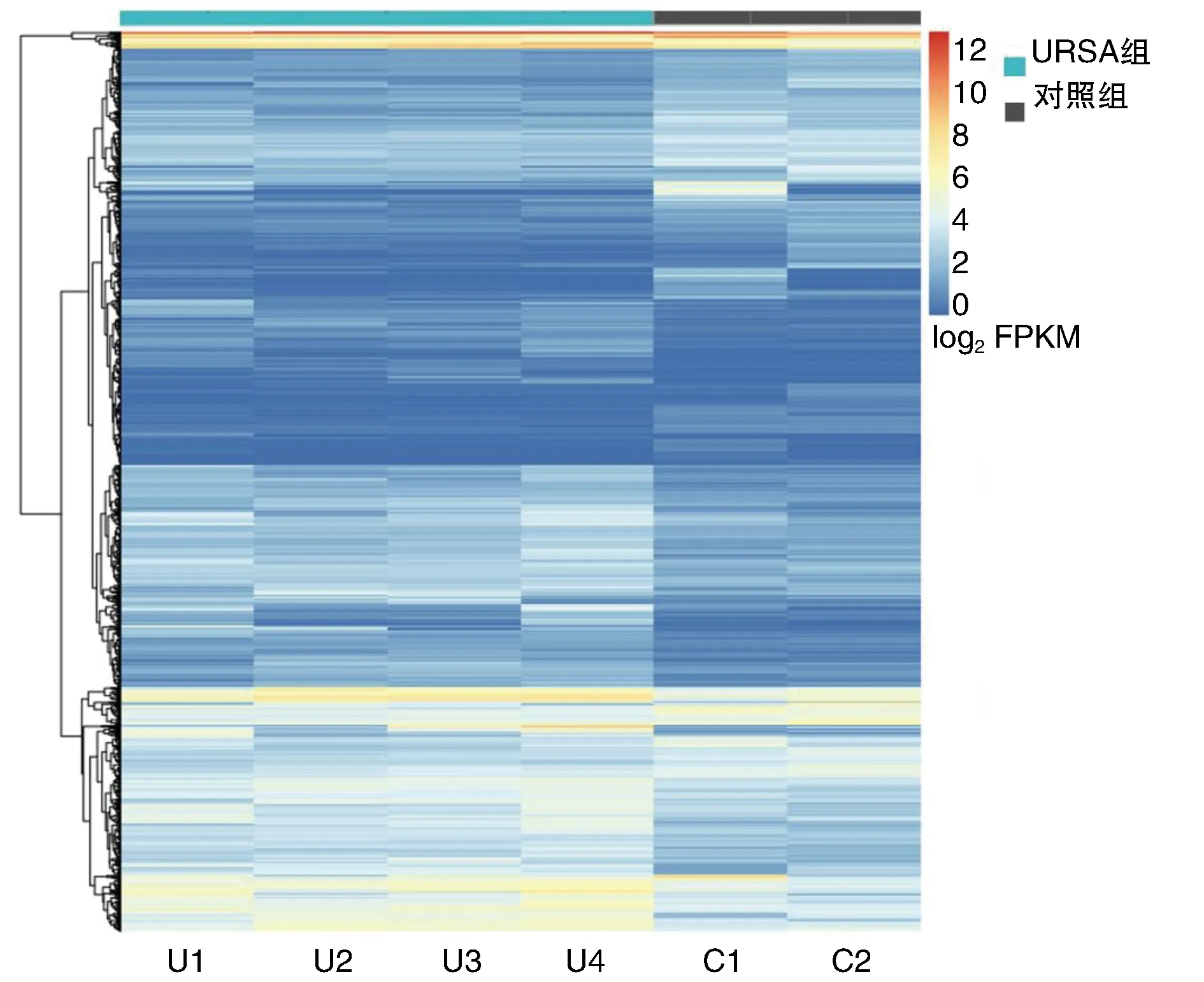
图2 DEGs等级聚类图红色表示基因表达量高,蓝色表示基因表达量低。U1~U4为USRA组4例样本;C1和C2为对照组2例样本。
2.3 差异表达基因富集分析
GO富集分析结果如图3所示,DEGs在生物过程中主要富集于T细胞活化与分化、白细胞趋化性、免疫应答过程的负调控及Toll样受体信号通路等(P<0.05);在分子功能中主要富集于受体配体活性、抗原肽与主要组织相容性复合体Ⅱ类分子(major histocompatibility complex class Ⅱ,MHC-Ⅱ)结合活性等(P<0.05);在细胞成分中主要富集于细胞外基质、MHC蛋白复合物、MHC-Ⅱ类蛋白复合物、分泌性颗粒膜等(P<0.05)。KEGG富集分析结果如图4所示,DEGs主要富集于吞噬体、金黄色葡萄球菌感染、抗原加工和递呈、自身免疫病、同种异体移植排斥反应、造血细胞谱系、补体和凝血级联反应及Toll样受体信号通路等(P<0.05)。这些DEGs主要与炎症反应、异常免疫应答、母体高凝状态有关,不利于母胎免疫耐受的维持。
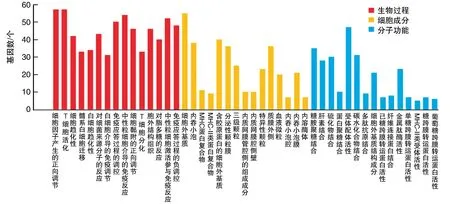
图3 差异基因GO富集分析
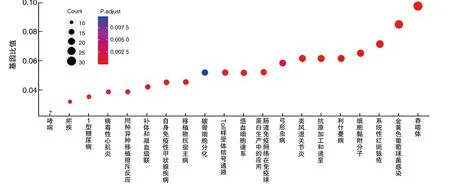
图4 差异基因KEGG富集分析圆形面积代表该信号通路富集的DEGs数量,面积越大DEGs数量越多;P.adjust由蓝到红色的过度色表示P校正值由大到小。
RNA测序数据进行GSEA通路富集分析结果如图5所示,富集基因集参与的主要通路有IL-2/STAT5通路、PI3K/Akt通路、IL-10抗炎信号通路、丝裂原活化蛋白激酶(mitogen activated protein kinase,MAPK)通路的负反馈调节、转化生长因子-β1(transforming growth factor β1,TGF-β1)通路、FOXP3信号通路的激活等,且这些信号通路均与Treg细胞活化及免疫抑制功能的发挥密切相关。以上信号通路的下调可能会影响Treg细胞的活化及免疫抑制功能发挥,使得URSA患者母胎界面的免疫平衡被打破,造成不良妊娠结果。
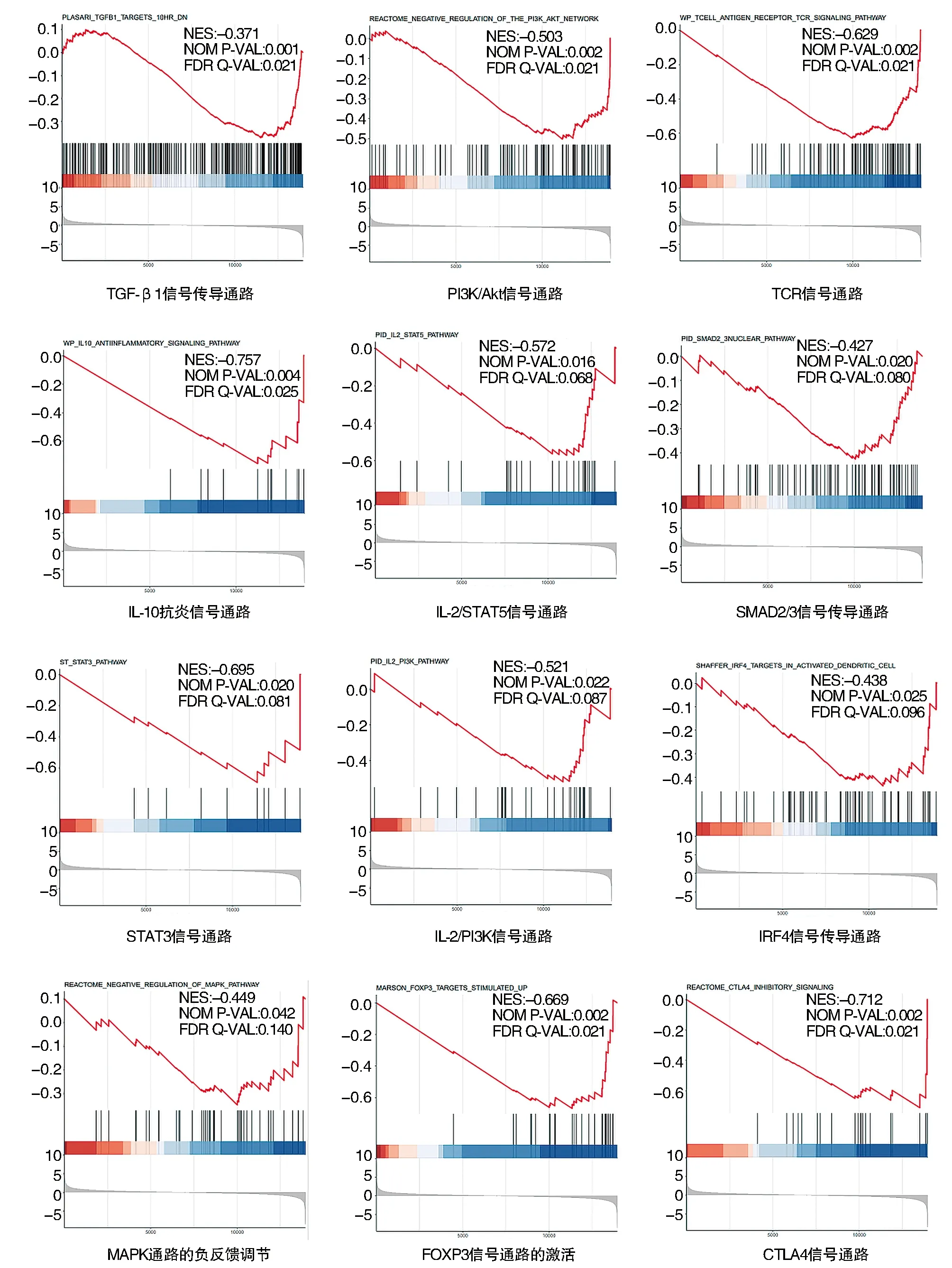
图5 GSEA基因集富集分析图
2.4 PPI网络分析
其中贡献度排名前24的节点基因有CGB8、CGB5、MUC5AC、GPR183、SLC5A8、FOXS1、CD180、ALPP、HLA-DRB5、CXCL9、MMP27、UST、TUBA3D、SIGLEC14、STX11、BAMBI、HLA-DRB1、SLC2A12、PPP1R3C、SCGB3A1、SHISA3、SLC10A6、CD300C、XCL1等(图6)。
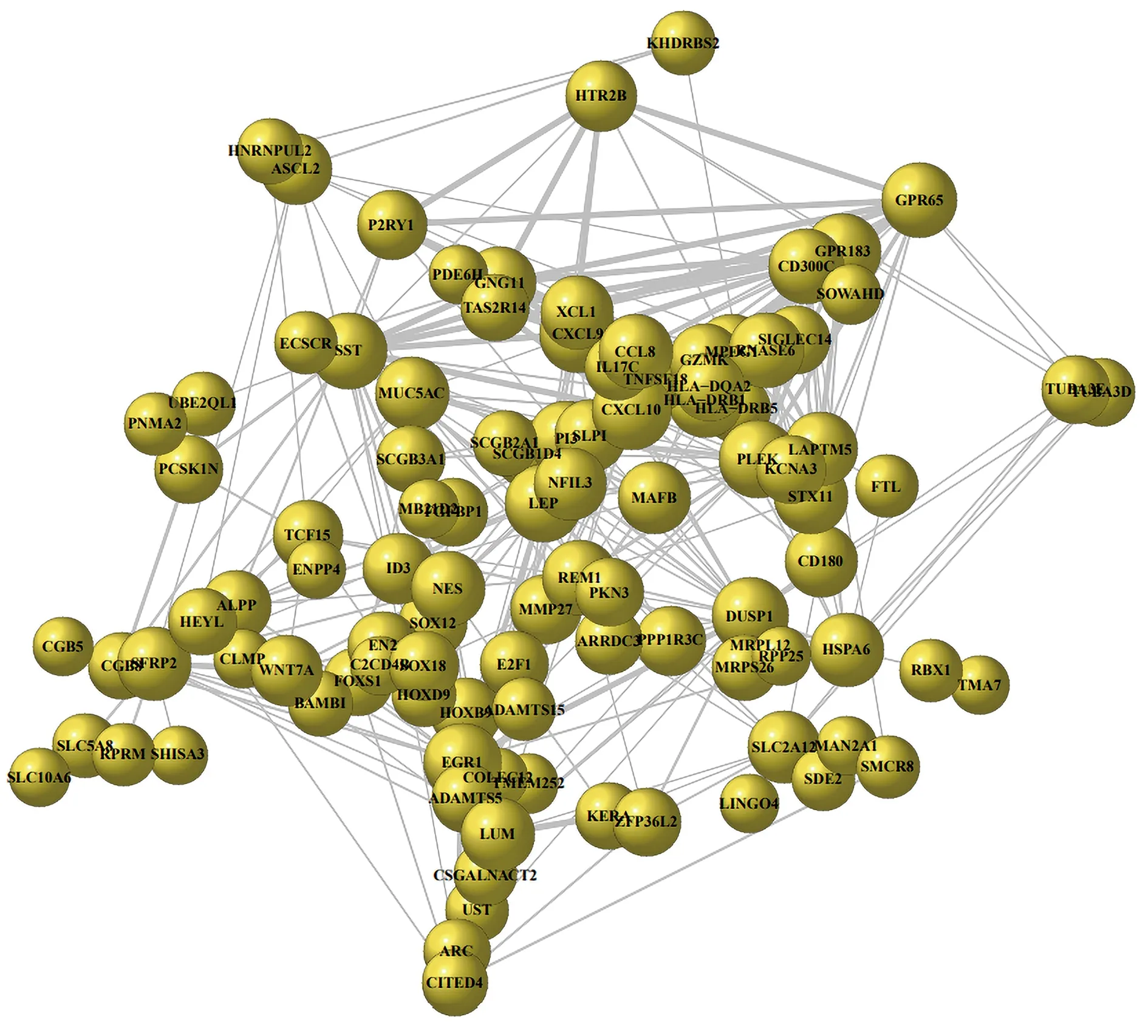
图6 PPI网络分析图圆代表蛋白质节点,直线代表蛋白质之间相互作用,线段粗细代表相互作用强弱。
2.5 SNP芯片检测结果
使用高密度SNP芯片对URSA组(n=6)和对照组(n=9)关键差异基因HLA-DPA1、IL-2R、CD80、PTPRC、IRF4、CTLA4、XCL1、PDCD1、DUSP1、MAFB、TIGIT、IL-10、TRIB1的位点多态性变异进行检测。结果显示仅HLA-DPA1 基因多态性位点(rs1042866、rs2567279)和IL-2RA基因多态性位点(rs2025346)的等位基因频率在两组间的分布差异有统计学意义(表2,P<0.05)。且HLA-DPA1 rs1042866位点C→T的突变可能减低URSA的发病风险,而HLA-DPA1 rs2567279位点T→C的突变及IL-2RA rs2025346位点G→A的突变则可能提高URSA的发病风险。其余关键基因SNP的差异无统计学意义(P>0.05)。
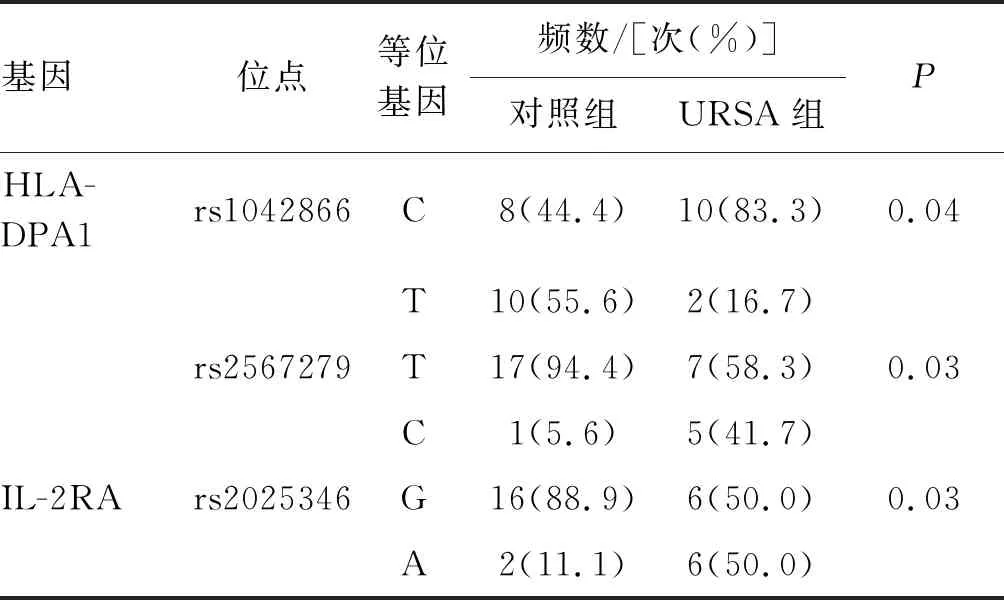
表2 SNP基因芯片筛选出的关键基因位点分布
3 讨 论
RSA为常见的产科病症,其患者及伴侣均承受了很大的身体、心理压力。其中URSA的具体机制尚不明确,临床的各种治疗手段也无法取得让人满意的疗效[10],因此探究URSA病因,找到行之有效的诊疗方法已经成为目前妇产及生殖医学上急需解决的难题之一。目前认为URSA发生主要与免疫因素相关,但其内在的免疫调控机制及关键因素尚未明确[11]。
本研究采用RNA-seq对URSA与正常妊娠蜕膜组织的基因表达谱进行检测,结果显示:与对照组比较,URSA蜕膜组织中有1 057个差异表达基因(DEGs),其中上调469个,下调588个。对DEGs进行GO、KEGG、PPI及GSEA等生物信息学分析。GO分析显示,DEGs主要富集于T细胞分化与活化、白细胞趋化性、抗原肽与MHC-Ⅱ类分子结合活性及免疫应答过程的负调控。KEGG分析显示,DEGs主要与自身免疫病、补体和凝血级联反应及Toll样受体信号通路等相关,这提示URSA的发生可能与炎症信号通路的激活、母体对胚胎免疫耐受状态的打破以及母体处于高凝状态相关。为进一步明确URSA患者蜕膜局部功能改变的机制,对DEGs进行GSEA通路富集分析,发现URSA患者蜕膜组织的IL-2/STAT5通路、PI3K/Akt通路、IL-10抗炎信号通路、MAPK通路、TGF-β1通路、FOXP3蛋白激活通路等富集且表达下调。上述信号通路均与Treg细胞密切相关,这些通路的下调可能影响Treg细胞的活化及免疫抑制功能。Treg细胞是一群免疫抑制性T细胞,对妊娠的维持及母体耐受均起重要作用。其主要功能包括消耗IL-2,抑制NK细胞或杀伤性T细胞的杀伤作用,以及表达FOXP3、TGF-β、IL-10等免疫抑制性分子[12-13]。
综合PPI及GSEA分析挑选出的核心DEGs有HLA-DPA1、IL-2RA、CD80、CTLA4、PDCD1、IL-10、PTPRC、IRF4、XCL1、DUSP1、MAFB、TIGIT及TRIB1,这些基因可能影响Treg细胞的功能,与URSA发生有关。HLA-DPA1属于HLA-Ⅱ类α链旁系同源基因。能够递呈外源性抗原肽,在免疫应答中发挥重要作用[14]。Shima等[15]发现,小鼠在孕前几个小时内就可以在阴道黏液中发现父系抗原和母系MHC-Ⅱ类细胞,可诱导CD4+CD25+FoxP3+Tregs的增殖,促进对父系同种抗原的耐受。IL-2RA基因(即CD25)的突变与白细胞介素-2受体α缺乏有关。CD25为Treg细胞表面的关键分子标志,IL-2是启动和维持Treg细胞增殖和分化的重要细胞因子,而Treg细胞表面的CD25分子能通过高效结合IL-2从而耗尽免疫微环境中的IL-2,维持母胎免疫耐受[12]。CD80(即B7)通过与T细胞表明的CD28或CTLA-4的结合而促进或抑制T细胞活化及细胞因子产生。PDCD1(即PD-1)是活化的T细胞表达的一种免疫抑制受体,能抑制T细胞的活化,还可以促进CD4+T细胞向Treg细胞分化,PD-1基因的下调与自身免疫病有关[16]。IL-10是Treg细胞、Th2细胞分泌的能调节免疫反应的最为重要的妊娠保护性细胞因子之一,该分子下调则不利于母胎界面免疫耐受的维持。PTPRC基因编码的蛋白是T细胞和B细胞抗原受体信号转导的调节因子,能够抑制JAK激酶从而抑制IL-2/STAT5信号通路[17]。IRF4基因编码蛋白是Ⅰ型干扰素基因表达所必需的转录因子之一[18]。XCL1是趋化因子超家族成员,对T细胞具有特异性趋化作用[19]。DUSP1编码的蛋白能使MAPK1/ERK2激酶去磷酸化,对细胞的增殖起负调控作用[20]。MAFB是一种转录因子,在调节特异性造血细胞谱系中起重要作用。TIGIT能辅助调节T细胞对B细胞的应答[21]。TRIB1主要参与脂质代谢,影响M2型巨噬细胞及Treg的分化等[22]。综上,共筛到的13个关键基因均与Treg细胞密切相关,这些基因的表达下调可能影响了Treg细胞分化及免疫抑制功能的发挥。
运用高密度SNP芯片检测两组DNA样本的基因位点多态性变异,发现除HLA-DPA1及IL-2RA外,其他的关键DEGs位点多态性在两组间的差异无统计学意义(P>0.05)。HLA-DPA1 rs1042866、HLA-DPA1 rs2567279及IL-2RA rs2025346等位基因频率分布间差异有统计学意义(P<0.05)。HLA-DPA1 rs1042866位点C→T的突变可能减低URSA的发病风险,而HLA-DPA1 rs2567279位点T→C的突变及IL-2RA rs2025346位点G→A的突变则可能提高URSA的发病风险。本研究为URSA发病机制的探究提供了新的思路和新靶点基因。
综上所述,本研究通过比较URSA组及正常妊娠组蜕膜组织的差异表达基因,发现了1057个差异表达基因。在这些差异基因中有下调Treg细胞活化与功能的相关基因及促进炎症反应和免疫应答的相关基因,它们可能使URSA患者母胎界面的免疫平衡被打破,造成不良妊娠结果。在蛋白质相互作用的关键节点基因中,发现了3个新的与URSA相关的关键基因DUSP1、MAFB、TIGIT,它们的表达下调可能影响Treg细胞的活化及功能发挥。SNP芯片检测显示在URSA患者中存在HLA-DPA1 rs1042866、rs2567279及IL-2RA rs2025346基因位点多态性变异,可能影响URSA的发生。
