DNA条形码基因ITS和LSU在红曲菌分类鉴定中的比较分析
2021-02-22李志强吴丽云
李志强,林 风,吴丽云*
(福建省微生物研究所 福建省红曲微生物技术开发应用工程研究中心 福建省新药(微生物)筛选重点实验室,福建 福州 350007)
红曲菌(Monascus)为一类小型、腐生的丝状真菌,虽然该属于1884年才建立[1],但红曲菌的应用已有上千年的历史。我国红曲(红曲菌发酵米饭制成的紫红色曲米)制作的历史最悠久,其可信记录最早可追溯至公元740年[2]。用红曲菌制作的最有名的产品当为红曲,在东亚和东南亚国家被广泛用作食品着色剂、鱼肉防腐剂、醋、酒发酵剂以及心血管病治疗药,因此,红曲菌具有重要的经济价值[3-5]。现代研究表明,红曲菌可产生多种具有生物活性的次级代谢产物,如莫纳可林类、γ-氨基丁酸、麦角甾醇、黄酮类及食用色素等[5-6],具有降血脂、降血压、降血糖、抑菌、抗氧化和提高免疫力等多种功效[5-8]。我国工业化生产的红曲产品主要有3类,即酿造用红曲、色曲和功能性红曲。
进行准确的物种鉴定和分类在红曲菌的基础研究和应用研究中均具有重要意义,是开展其他相关生物学科学研究的基础条件。现阶段红曲菌的鉴定和分类主要依靠形态特征[1]。传统的形态学鉴定是开展物种鉴定和系统分类工作的重要手段,但需要较高的专业技术知识和丰富的经验,程序繁琐、周期长、标准化程度低、受环境因素影响大,存在主观性强、稳定性和重复性差等方面的局限。脱氧核糖核酸条形码技术(deoxyribonucleic acid(DNA)barcoding)作为一种新兴的物种鉴定方法,以其简便、准确、高效、客观和标准化程度高的优势,一经提出就在动物、植物和真菌中得到了广泛应用[9-10]。生物条形码协会将其定义为:通过聚合酶链式反应(polymerase chain reaction,PCR)扩增和测序来获得一段标准同源序列(500~800碱基对(base pairs,bp)),再将序列进行多重序列比对和聚类分析,从而实现对物种的快速、准确和自动鉴定[9]。该技术近年来发展迅速并形成了标准化,《中国药典》2015年版已收录“中药材DNA条形码分子鉴定法指导原则”作为中药材质量控制的新方法[11],提出需确定不同物种、不同条形码序列的种内变异阈值。红曲为药食两用的传统中药材,但红曲菌的不同条形码序列的种内及种间变异阈值鲜有报道。核糖体内转录间隔区(internal transcribed spacer,ITS)和核糖体大亚基(ribosomal large subunit,LSU)被广泛用于红曲菌属的系统发育分析[12-15]。
本研究从美国国家生物技术信息中心(national center for biotechnology information,NCBI)的GenBank数据库中下载不同红曲菌的ITS和LSU基因序列,结合系统发育和遗传距离的分析方法,比较这两个基因的鉴定和聚类效果,确定不同红曲菌的ITS和LSU基因的种间变异阈值及部分红曲菌的种内变异阈值,并用本研究所保存的部分红曲菌株的基因序列进行验证,以期为在生产实践中选择红曲菌的条形码基因提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 序列及菌株来源
通过查阅文献及在NCBI 的GenBank 数据库中以“Monascus&28S ribosomal RNA gene”、“Monascus&large subunit ribosomal RNA gene”或“Monascus &internal transcribed spacer”为关键字进行检索,从GenBank数据库中下载各种红曲菌不同菌株(具有不同的菌株编号)的ITS和LSU序列。理论上每株红曲菌的相同基因片段应该只有一个GenBank序列号,由于不同的研究者可能对同一株红曲菌(特别是模式菌株)的同一基因片段进行了测序,因此同一个菌株的相同基因片段可能有多个GenBank序列号,在这种情况下选择质量高、序列长的一条序列作为该菌株的代表序列。
曾描述记载的Monascus种名有30多个[1,14,16-17],但许多种类被视为同种异名[12-14,16]。结合形态特征和多基因系统发育分析,现阶段红曲菌属共包括10种红曲菌[14-15],因此在本研究中主要参考BARBOSA R N等[14]的分类系统。
用于验证的红曲菌株从本研究所的红曲菌种资源库中选取。利用NCBI中基本局部比对搜索工具(basic local alignment search tool,BLAST)对这些菌株的ITS和LSU序列与GenBank数据库已收录的红曲菌属同一基因序列进行比较,若有差异,则提交至GenBank数据库。
1.1.2 主要试剂
TaqDNA聚合酶、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)、DNA分子质量标准C、琼脂糖和Ezup柱式真菌基因组DNA提取试剂盒:生工生物工程(上海)股份有限公司产品;其他化学试剂均为国产分析纯。
1.2 仪器与设备
DYY-6C稳流稳压电泳仪:北京六一仪器厂;UVI Fire Reader凝胶成像系统:英国UVItec公司;S1000 PCR仪:美国Bio-Rad公司。
1.3 方法
1.3.1 菌株的培养
菌株的液体培养参照文献[18]。
1.3.2 DNA的提取
菌株培养完成后收集菌丝体,加入液氮研磨成粉末,然后用Ezup柱式真菌基因组DNA提取试剂盒提取DNA。
1.3.3 PCR扩增和测序
PCR扩增ITS基因采用引物ITS5(5'-TCCTCCGCTTATTGATATGC-3')和ITS4(5'-GGAAGTAAAAGTCGTAACAAGG-3'),PCR扩增LSU基因采用引物LR0R(5'-ACCCGCTGAACTTAAGC-3')和LR5(5'-TCCTGAGGGAAACTTCG-3')[19]。PCR扩增体系(50 μL):10×PCR反应缓冲液5 μL,MgCl2(25 mmol/L)3 μL,脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphates,dNTPs)(10 mmol/L)0.75 μL,10 μmol/L的正向引物和反向引物各1 μL,TaqDNA聚合酶(2 U/μL)0.5 μL,DNA模板2 μL,加无菌蒸馏水至50 μL。PCR扩增程序:94 ℃预变性3 min;94 ℃变性30 s,52 ℃(LSU退火温度为48 ℃)退火30 s,72 ℃延伸90 s,共34个循环;72 ℃再延伸8 min。每次反应均设立不含DNA模板的空白对照组。用1%的琼脂糖凝胶电泳检测扩增产物,扩增阳性样本送铂尚生物技术(上海)有限公司进行双向测序。
1.3.4 数据分析
拼接本研究中获得的序列,并根据测序图谱人工校正碱基。采用Clustal X软件对下载的序列进行排序,去除5'和3'端非对位排列区。利用MEGA 6.0[20]软件基于木村双参数(Kimura 2-parameter,K2P)模型计算下载的各种红曲菌的ITS和LSU基因序列的种内和种间遗传距离;并用MEGA 6.0软件基于K2P模型的邻接(neighbour-joining,NJ)法构建系统发育树,自展1 000次计算各分支的支持率。构建系统发育树所用的外类群为黄曲霉(Aspergillusflavus,ITS:DQ683124,LSU:HQ395773)、寄生曲霉(Aspergillus parasiticus,ITS:EF661568,LSU:JN938934)和烟曲霉(Aspergillus fumigatus,ITS:HQ026746,LSU:FM179606)。
2 结果与分析
2.1 基于系统发育分析的结果
下载的各种红曲菌的ITS和LSU基因序列的信息见表1,共包括了10种红曲菌的60条ITS基因序列和96条LSU基因序列,且均包含了10种红曲菌的模式菌株(见图1)。每种红曲菌下载的序列数目在1~36之间。下载的红曲菌ITS和LSU基因序列经排序和编辑后的长度(包括插入和缺失)分别为677 bp和567 bp,可变位点分别为164 bp(24.2%)和43 bp(7.6%),简约信息位点分别为112 bp(16.5%)和36 bp(6.3%)。
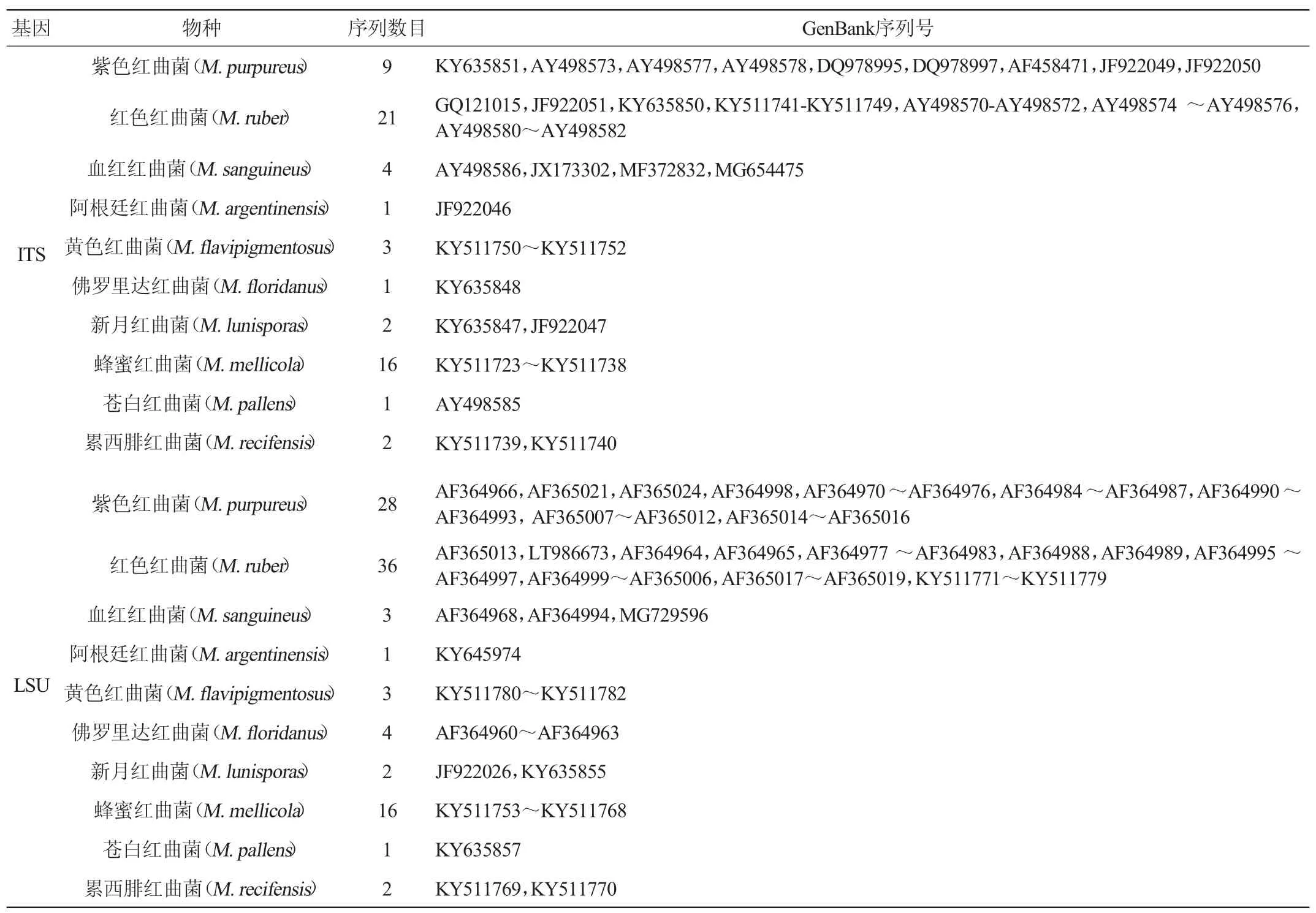
表1 10种红曲菌的ITS和LSU基因序列信息Table 1 Sequence information of ITS and LSU genes of 10 Monascus species
基于系统发育的分析方法(一般采用邻接法)是条形码研究中常用的方法,其进行物种鉴定的原则是同一物种的所有个体在系统发育树中形成单系。利用下载的10种红曲菌和3种曲霉的ITS和LSU基因序列基于K2P模型进行1 000次自展检验构建50%多数一致性系统发育树,结果见图1。
由图1可知,用ITS基因序列构建的系统发育树中,同种红曲菌所有菌株构成的单系的支持率为60%~100%,均大于50%,能明显被区分开。只有M.purpureus、M.floridanus和M.mellicola的同种菌株在利用LSU基因序列构建的系统发育树中分别聚成单系,且支持率均大于50%。3株M.flavipigmentosus和2株M.recifensis的LSU基因序列分别没有种内变异。M.lunisporasCBS142230和M.lunisporasCBS 113675的LSU基因序列有2 bp的差异,且分别插入到M.recifensis和M.flavipigmentosus的分支中,导致这3种红曲菌种内的菌株没有按物种分别形成单系。M.lunisporasCBS 142230和2株M.recifensis的LSU基因序列相同。M.sanguineusATCC 200613和M.sanguineusATCC 16436的LSU基因序列完全相同,均与M.purpureus的LSU基因序列只有2 bp的差异,M.sanguineusSICC 3.292与上述2株M.sanguineus在这两个位点的碱基相同。M.sanguineusSICC 3.292与另外2株M.sanguineus相比,前者具有1 bp缺失和1 bp转换。
从本研究所的红曲菌种资源库中选取了52株红曲菌,BLAST分析显示这些菌株的ITS和LSU基因序列与GenBank数据库已收录的红曲菌属同一基因的序列没有差异。
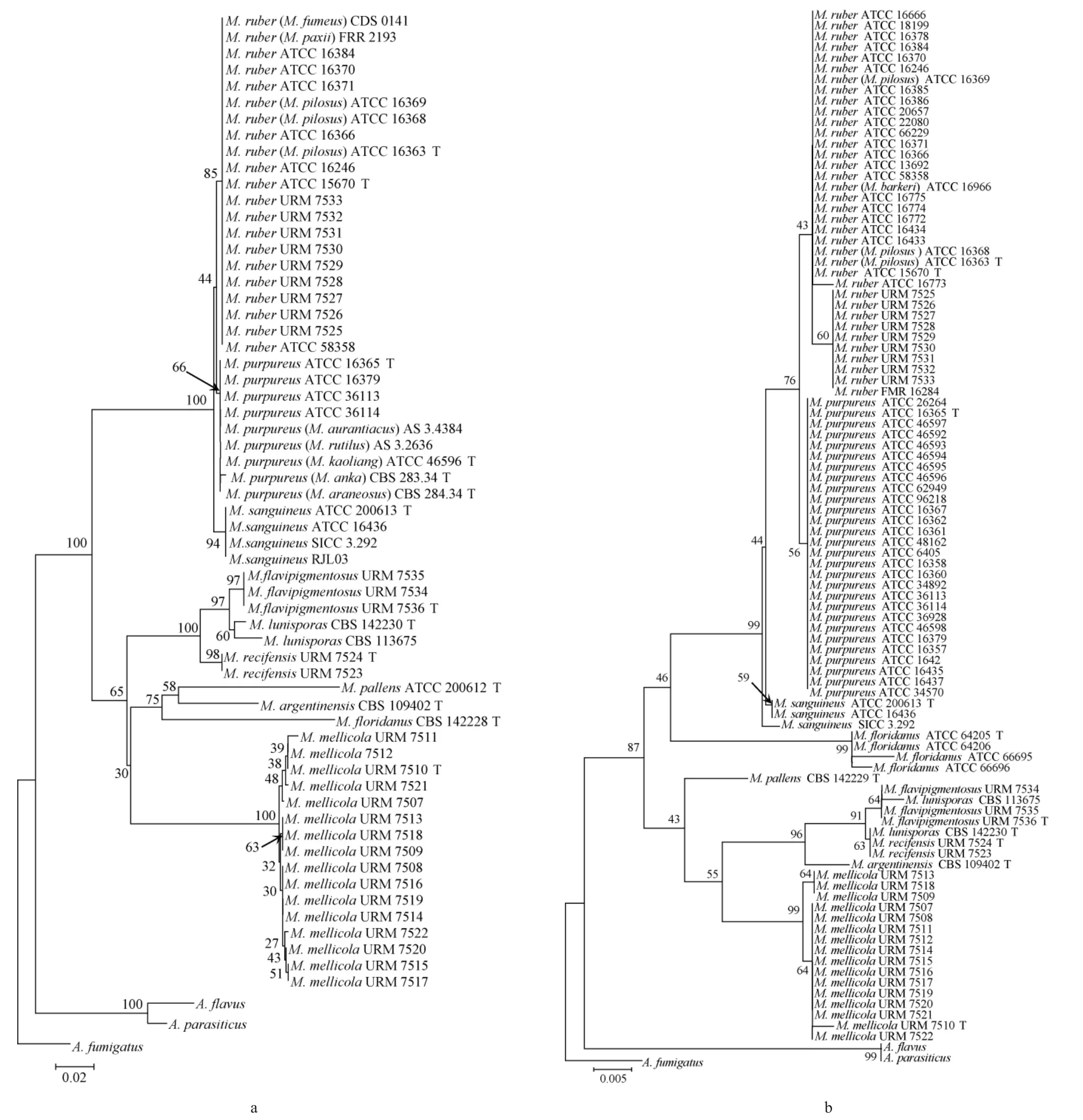
图1 基于ITS(a)及LSU(b)基因序列构建的系统发育树Fig.1 Phylogenetic trees constructed based on ITS (a) and LSU (b) gene sequences
2.2 基于K2P距离法的分析结果
各种红曲菌的ITS和LSU基因序列基于K2P模型的种内遗传距离范围、种内平均遗传距离及种间遗传距离范围见表2。由表2可知,对于ITS基因序列,M.argentinensis、M.floridanus和M.pallens无种内遗传距离的计算值,其他红曲菌种内遗传距离范围为0~0.014 0,种内平均遗传距离范围为0~0.014 0;10种红曲菌种间遗传距离范围为0.003 9~0.146 7。对于LSU基因序列而言,M.argentinensis和M.pallens无种内遗传距离的计算值,其他红曲菌种内遗传距离范围为0~0.005 3,种内平均遗传距离范围为0~0.003 5;10种红曲菌种间遗传距离范围为0.001 8~0.051 0。M.lunisporas的ITS基因序列的种内遗传距离明显高于其他红曲菌,但M.lunisporas与其他红曲菌LSU基因序列的种内遗传距离差异并不大。

表2 10种红曲菌ITS和LSU基因的K2P种间及种内遗传距离Table 2 K2P inter-and intra-specific genetic distances of ITS and LSU genes of 10 Monascus species
10种红曲菌ITS和LSU基因序列的K2P遗传距离的比较见表3。由表3可知,M.lunisporas的ITS基因序列最小种间遗传距离和最大种内遗传距离的比值为1.04,条形码间隙不明显;M.purpureus、M.ruber、M.sanguineus、M.flavipigmentosus、M.mellicola和M.recifensis的ITS基因序列的最小种间遗传距离均明显大于各自ITS基因序列的最大种内遗传距离,条形码间隙明显。对LSU基因序列而言,M.ruber和M.lunisporas的最小种间遗传距离均小于各自的最大种内遗传距离;M.recifensis的最小种间遗传距离为0.001 8,最大种内遗传距离为0,但2株M.recifensis与M.lunisporasCBS 142230的LSU基因序列相同。因此这3种红曲菌LSU基因序列的条形码间隙不明显。M.purpureus、M.sanguineus、M.floridanus、M.flavipigmentosus和M.mellicola的LSU基因序列的最大种内遗传距离均明显小于各自LSU基因序列的最小种间遗传距离,条形码间隙明显。
分别比较了同种红曲菌的ITS和LSU基因序列的最小种间遗传距离和最大种间遗传距离结果见表3。由表3可知,每种红曲菌ITS基因序列的最小种间遗传距离均明显大于同种红曲菌LSU基因序列的最小种间遗传距离,其比值范围为1.39~14.11;每种红曲菌ITS基因序列的最大种间遗传距离均明显大于同种红曲菌LSU基因序列的最大种间遗传距离,其比值范围为2.32~4.78。最小及最大种间遗传距离的比较表明不同红曲菌ITS基因的种间差异均大于该种红曲菌LSU基因的种间差异。一段DNA序列需满足以下两个条件才有可能被作为DNA条形码:用通用引物易在大多数或全部目标类群中扩增;种间变异高,种内变异低,存在条形码间隙[21]。用通用引物均易在红曲菌中扩增ITS和LSU基因。遗传距离的比较表明,除M.lunisporas的ITS基因外,其他红曲菌的ITS和LSU基因的种内变异低(最大种内遗传距离均低于0.7%);同种红曲菌ITS基因的种间变异均高于其LSU基因的种间变异。
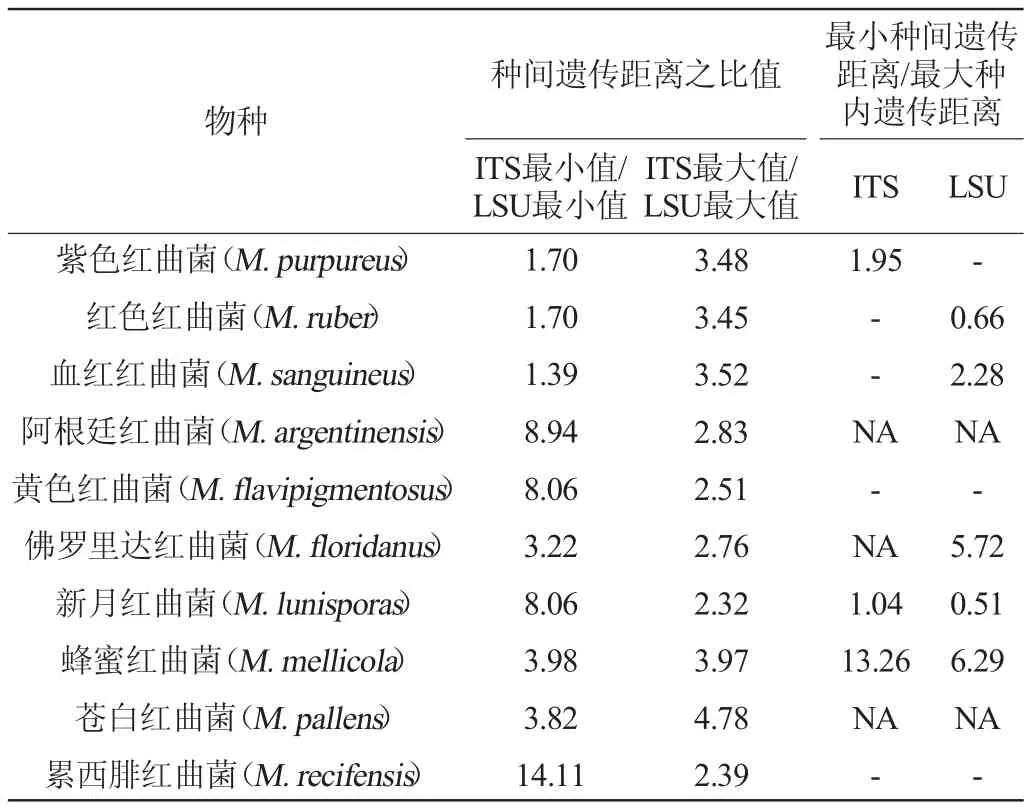
表3 10种红曲菌ITS和LSU基因的K2P遗传距离的比较Table 3 Comparison of K2P genetic distances of ITS and LSU genes of 10 Monascus species
DNA条形码研究除了采用系统发育的分析方法,也常用基于K2P模型的遗传距离法,其进行物种鉴定的准则是“遗传距离阈值”与“条形码间隙”。HEBERT P D N等[9,22]在基于细胞色素氧化酶C亚基I(cytochrome C oxidase subunit I,COI)的研究中将2%作为遗传距离阈值,并建议种间平均遗传距离至少是种内平均遗传距离的10倍以上。但部分物种COI的遗传距离并不符合2%的阈值标准[23]。对真菌的ITS基因而言,一般采用3%作为遗传距离阈值,但该阈值并不合适所有真菌[10,24-25],如同属子囊菌门(Ascomycota)的烟曲霉(Aspergillus fumigatus)和鹿角炭角菌(Xylaria hypoxylon)的ITS基因的种内遗传距离分别为0.2%和24.2%[24],差异极大。本研究表明,M.lunisporas的ITS基因的种内遗传距离明显高于其他红曲菌。因为不同的基因具有不同的进化速率,不同物种的进化有快慢,对所有物种的所有基因采用某一固定的遗传距离阈值是不尽合理的。对条形码间隙而言,有学者认为采用遗传距离的平均值来构建条形码间隙是不合理的,因为平均值是对整个研究群体遗传距离的折中,应比较最小种间遗传距离与最大种内遗传距离是否形成明显的条形码间隙[23]。只要该物种的最小种间遗传距离明显大于其最大种内遗传距离即可,前者不一定要为后者的10倍以上[21,26]。分布广泛的物种若采样的地理范围不完备,则可能低估种内遗传距离;若未包含某一研究类群中尽可能多的不同物种,则会高估种间遗传距离(真正的最小种间遗传距离需依据真正的姐妹群的相同基因序列来计算)[26]。本研究包括了红曲菌属现阶段公认的所有物种(从而可以得到真正的最小种间遗传距离),并包括了同一种红曲菌的多个菌株,在此基础上比较不同红曲菌ITS和LSU基因的最小种间遗传距离与最大种内遗传距离。
3 结论
同种红曲菌同一基因的最小种间遗传距离与最大种内遗传距离的比较表明,具有ITS基因种内遗传距离的红曲菌,除M.lunisporas外,其他红曲菌ITS基因的条形码间隙明显;具有LSU基因种内遗传距离的红曲菌,除M.ruber、M.lunisporas和M.recifensis外,其他红曲菌LSU基因的条形码间隙明显。利用ITS序列构建的系统发育树能明显区分各种红曲菌。只有M.purpureus、M.floridanus和M.mellicola的同种菌株在利用LSU序列构建的邻接树中分别聚成单系,且支持率均大于50%。因此ITS基因在红曲菌的分类鉴定中优于LSU基因,LSU基因在红曲菌物种鉴定中的分辨力有限。
