基于高通量测序技术的竹䇲鱼微卫星标记开发与评价
2021-01-11孔啸兰陈作志许友伟江艳娥
孔啸兰,李 敏,陈作志,张 俊,许友伟,江艳娥
(1.农业农村部东海渔业资源开发利用重点实验室,上海 200090;2.中国水产科学研究院南海水产研究所,农业农村部外海渔业开发重点实验室,广州 510300)
竹䇲鱼(Trachurus japonicus),隶属于鲈形目(Perciformes),鲹科(Carangidae),竹䇲鱼属,因其体呈纺锤形,侧线上均具高而强的棱鳞,形如用竹板编制的组合隆起䇲,由此得名,主要分布于我国渤海、黄海、东海、南海,以及日本海域、朝鲜半岛海域、西北太平洋温暖水域,是我国近海海域灯光围网和拖网的主要捕捞对象,南海近海是竹䇲鱼的主要渔场之一,年捕捞量约为2.5×104t,占全国总量的68%[1-4]。研究表明,东海及福建海域竹䇲鱼资源早已衰退,至今难以恢复[4-7];南海海域竹䇲鱼虽然产量仍较为稳定,但是也已经被充分利用[4,8]。为了进一步科学管理和合理利用南海区竹䇲鱼资源,急需开展竹䇲鱼资源评估及种群遗传特性的研究。目前,国内关于竹䇲鱼遗传方面的研究主要有SONG等[9]、ZHAO等[10]、张艳丽等[11]、牛素芬等[12]采用线粒体和扩增片段长度多态性(AFLP)标记对日本海域、北部湾和福建海域竹䇲鱼遗传特性的研究,涉及南海海域较少,且未见有关于竹䇲鱼微卫星标记的研究。
微卫星分子标记是共显性标记,具有多态性高、变异性强、数据易统计等突出优点[13],被广泛应用于海洋生物遗传结构及遗传多样性分析[14-15]。但是,由于微卫星标记通用性较差,常常具有极强的种属特异性。因此,应用微卫星标记开展种群遗传分析常常需要针对特定物种开发微卫星标记。CHANG 等[16]早期采用构建(CA)n文库的方法开发出10个竹䇲鱼微卫星标记。文库构建方法开发微卫星标记工作量大,效率低,难以满足现今遗传评估、图谱构建等需要大批和多样化的微卫星标记。
因此,本研究通过基于酶切位点的简化基因组测序技术(restriction-site associated DNA sequence,RAD-seq)开发竹䇲鱼2、3核苷酸微卫星分子标记并对测试群体进行多样性分析,旨在为竹䇲鱼种群遗传评估提供技术基础,以促进其种群资源的评估和管理。
1 材料与方法
1.1 样品采集与基因组DNA提取
竹䇲鱼样品采集于湛江东部海域(111°30′E、20°18′N),共32尾。剪取背部肌肉样品加入无水乙醇保存。每个样品剪取少量肌肉组织,使用 “海洋动物组织基因组DNA提取试剂盒”(北京天根生化科技有限公司)提取基因组DNA,0.8%的琼脂糖凝胶电泳检测DNA提取质量,之后置于-20℃保存备用。
1.2 高通量测序与引物合成
使用Hiseq2000高通量测序仪(Illumina,USA)对竹䇲鱼基因组DNA进行RAD-seq高通量测序(测序服务由广州基迪奥生物科技有限公司提供),经生物信息学搜索出微卫星位点[17]。使用Premier5.0软件在重复单元侧翼序列上选择性设计出89条引物,主要参数为:GC含量为40% ~60%,引物长度为18~25 bp,退火温度为45~60℃,预期扩增长度150~350 bp。送上海英潍捷基贸易有限公司合成引物。
1.3 引物筛选与分型检测
选取3个样本混合成的基因组DNA为模板,优化PCR反应条件,对引物进行首轮筛选。PCR反应体系为15μL,其中包括10×PCR Buffer 1.5 μL,2.5 mmol·L-1MgCl21.2μL,2 mmol·L-1dNTPs 2μL,正反向引物(10μmol·L-1)各0.8 μL,Taq酶(5 U· μL-1)0.15μL,DNA模版1 μL,加双蒸水至15μL。PCR扩增程序为:94℃预变性5 min;94℃变性45 s,55~60℃退火45 s,72℃延伸45 s,35个循环,72℃延伸10 min。琼脂糖电泳检测是否能扩增出稳定且均一目的片段。之后分别选取8尾个体的基因组DNA作为模板,对琼脂糖电泳有稳定且均一目的条带的引物PCR扩增后聚丙烯酰胺凝胶电泳和硝酸银染色检测扩增产物多态性。
多态且条带清晰的引物使用三引物法[18],利用M13荧光接头引物进行PCR扩增,并送华大基因公司进行毛细管电泳分型检测。PCR反应体系为15μL,其中包括10×PCR Buffer 1.5μL,2.5 mmol· L-1MgCl21.2μL,2 mmol· L-1dNTPs 2μL,M13正向引物(10μmol·L-1)0.2 μL,M13反向引物(10μmol·L-1)0.6μL,M13通用荧光引物(10μmol·L-1)0.5μL,Taq酶(5 U·μL-1)0.15μL,DNA模版1μL,加双蒸水至15μL。PCR扩增程序为:94℃预变性5 min;94℃变性45 s,55~60℃退火45 s,72℃延伸45 s,35个循环;94℃变性45 s,53℃退火45 s,72℃延伸45 s,8个循环;72℃延伸30 min。
1.4 群体遗传学评价
使用32尾竹䇲鱼个体的基因组DNA为模板,对通过筛选的微卫星标记的种群遗传学特征进行评价。PCR反应体系和条件、等位基因分型方法如上。使用软件Genepop4.0[19]对每个标记的种群遗传学特征值进行计算,包括等位基因数(Na)、表观杂合度(Ho)和期望杂合度(He),进行“哈迪-温伯格平衡” 检验和连锁不平衡检测,并对P值进行Bonferroni校正。使用Cervus3.0.7[20]软件计算多态信息含量(PIC)。
2 结果与分析
2.1 高通量测序结果与微卫星位点分析
RAD-seq高通量测序共获得竹䇲鱼基因组原始数据1.26 G,GC含量为45.79%,Q20为95.33%,说明测序结果质量较好,可用于后续分析。搜索后共获得微卫星序列19 920条,2~6核苷酸重复微卫星位点21 912个,其中2核苷酸重复微卫星位点最多,有12 478个,占总数的56.95%,具体见表1。说明2核苷酸重复为主要的微卫星重复类型,其次为3核苷酸重复。
2.2 PCR引物设计和筛选
选取89条2、3核苷酸重复序列设计引物,其中2核苷酸重复为66条,3核苷酸重复为23条。经过聚丙烯酰胺凝胶电泳筛选后,共有27对引物通过筛选(表2),27对引物扩增的序列中21个位点为2核苷酸重复,重复次数为10~13次;6个位点为3核苷酸重复,重复次数为7~9次。

表1 竹䇲鱼基因组中不同类型SSR统计Tab.1 Different types of SSR statistics in T.japonicus genome
2.3 微卫星标记的种群遗传学评价
使用采集于湛江东部海域的竹䇲鱼群体对筛选合格的微卫星标记进行种群遗传学评价。如表3,所有27个标记在测试群体中共检测到265个等位基因,等位基因数分布范围为3~19,表观杂合度分布范围为0.344~0.938,平均为0.654;期望杂合度分布范围为0.336~0.880,平均为0.729。多态信息含量(PIC)分布范围为0.318~0.882,平均为0.707,表明开发的微卫星位点具有较高的杂合度。除2个标记外,其他标记等位基因频率均符合“哈迪-温伯格”平衡(HWE)。连锁不平衡检测表明各位点间无连锁不平衡现象。
3 讨论
3.1 高通量测序发掘微卫星序列的技术优势
传统微卫星标记开发方法耗时长、花费高、技术难度大。以磁珠富集法为例,标记开发过程中基因组DNA浓度、接头连接效率、富集过程中的杂交温度以及洗脱条件的控制等因素都会影响微卫星筛选的效率[21-22],且最终获得的有效微卫星序列仅几百条[23-24]。相比较而言,高通量测序技术开发微卫星标记,省略了建库、克隆、筛选等,只需提取基因组DNA测序,利用生物信息学手段可直接获取微卫星序列,通常是传统方法获得微卫星序列数目的几百倍[25-27],具有高效、便捷、准确的特点,能够满足于短时间内大批量微卫星位点的开发需求,比如连锁图谱构建、数量性状基因座(quantitative trait locus,QTL)定位等[28-29]。同时,传统的微卫星开发方法获得的微卫星重复类型基本为2核苷酸重复,而高通量测序技术的发展,更加方便了研究者获得3、4甚至更多核苷酸重复的微卫星引物[26]。可见,高通量测序技术较传统微卫星开发的方法更为快速、高效。
本次RAD-seq高通量测序共获得竹䇲鱼基因组原始数据1.26 G,GC含量45.79%,测序质量Q20为95.33%;共获得微卫星序列19 920条。说明测序质量稳定高效,并获得了数量庞大、类型丰富的微卫星序列,可用于后续竹䇲鱼微卫星标记大规模开发和相关遗传学研究。
3.2 不同核苷酸重复微卫星位点比较
本次高通量测序结果表明在竹䇲鱼微卫星位点中2核苷酸重复为主要重复类型,其中AC/TG类重复数量最为丰富,占41.6%,GC/CG类重复较为少见。此结果与其他水产动物微卫星位点研究结果相一致[30-32]。例如,熊良伟等[31]对中华鳑鲏(Rhodeus sinensis)微卫星的分析中,2核苷酸占总微卫星位点的53.59%,2核苷酸重复中AC/TG类占60.63%;GC/CG类仅占0.32%。在裸体异鳔鳅鮀(Xenophysogobio nudicorpa)中[30],2核苷酸重复占总微卫星位点比例高达83.15%,AC/GT类重复占49.36%,GC/CG类重复仅有4个。
尽管研究表明开发的微卫星标记以2核苷酸重复为主,仍有学者对3、4核苷酸重复的微卫星标记感兴趣。对人类基因组的研究中发现,3核苷酸重复序列与遗传疾病的发生有关,并且具有较高的多态性和遗传稳定性[33]。在对水产动物3、4核苷酸重复微卫星标记的筛选中,部分学者的研究结果存在差异。例如,房祖业等[26]对大刺鳅(Mastacembelus armatus)2、3、4核苷酸重复微卫星标记的筛选发现,2核苷酸重复较3、4核苷酸重复具有更高的筛选效率和多态性;而鲁翠云等[34]、谭照君等[35]和李文升等[36]的研究认为3、4核苷酸具有更高的多态性和分型效果。本文考虑高通量测序结果中2、3核苷酸重复微卫星位点数已占82%,因此,仅选取2、3核苷酸重复位点进行筛选,结果显示,2、3核苷酸重复微卫星位点的多态性差异并不明显。
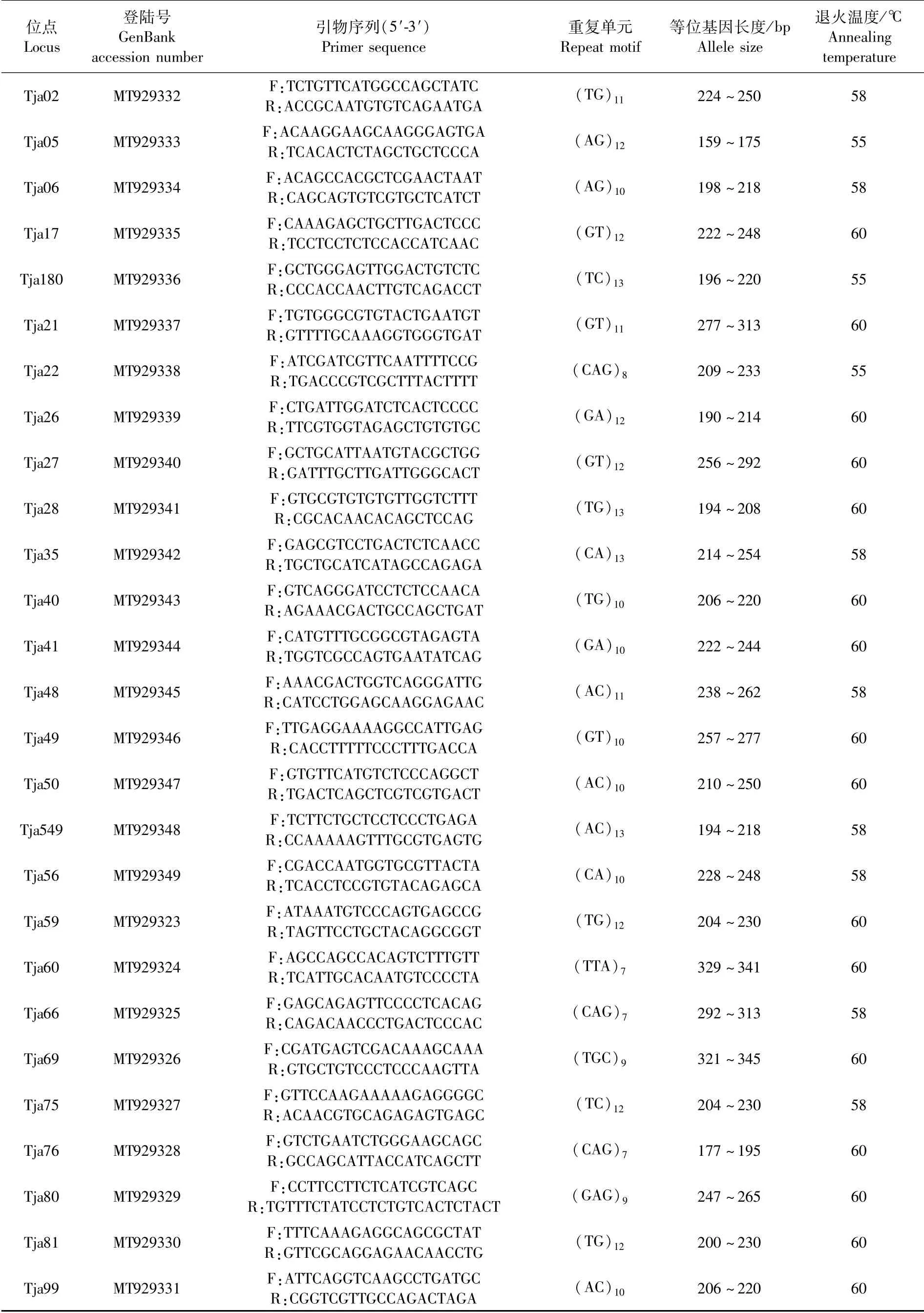
表2 27对竹䇲鱼微卫星引物信息Tab.2 Information of 27 pairs of primers in T.japonicus
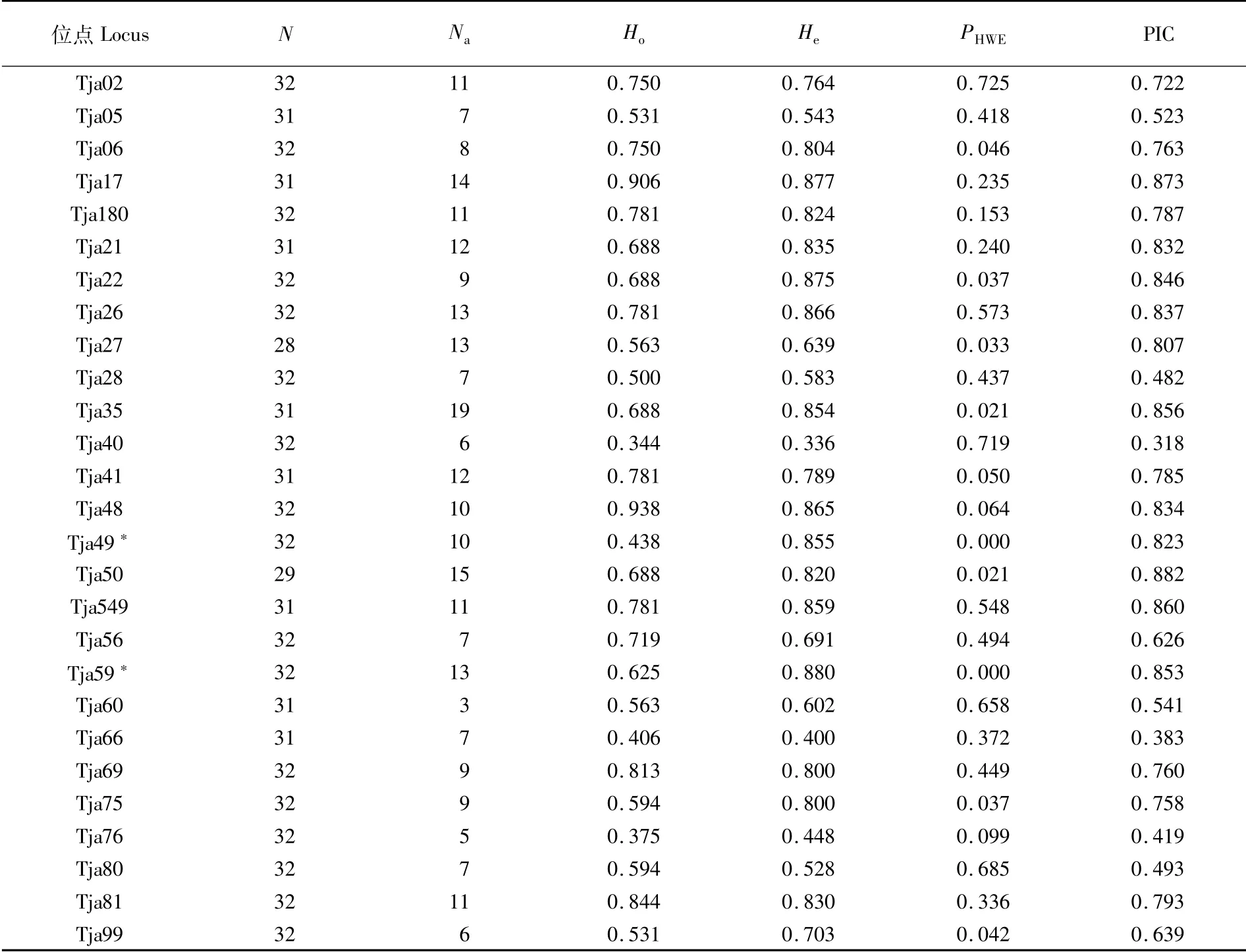
表3 竹䇲鱼微卫星标记的种群遗传学特征Tab.3 Characteristics of microsatellite loci in T.japonicus
3.3 微卫星标记的种群遗传学特征
ELLEGREN[37]提出真核生物微卫星位点重复大部分在30次以下,2核苷酸重复以15~19次为主。基于WEBER[38]的研究结果,重复次数高的微卫星在种群中表现出的多态性较高。龚小玲等[39]对澳洲鳗鲡(Anguilla australis)进行标记开发时发现,微卫星重复序列的重复次数过高会影响PCR效果,选择居中的重复次数为宜。因此,本研究选择重复次数较为适中的位点,分别为2核苷酸重复10~15次、3核苷酸重复7~9次。
群体杂合度的高低反映了群体在多个基因座上的遗传变异及群体遗传多样性丰富度[36]。本研究中竹䇲鱼群体的平均表观杂合度为0.654,平均期望杂合度为0.729,说明该群体的遗传多样性较高。平均表观杂合度和期望杂合度存在差异,说明存在杂合子缺失或者纯合子过剩的情况。多态信息含量(PIC)也是衡量群体遗传多样性的重要指数,研究者BOTSTEIN等[40]认为当基因标记PIC>0.5时,为高度多态位点;当0.25<PIC<0.5时,为中度多态位点;当PIC<0.25时,为低度多态性位点,通常不作为遗传多样性分析。本文中竹䇲鱼位点有5个为中度多态位点,其他均为高度多态位点。表明开发所得的竹䇲鱼微卫星标记在该群体中具有较好的遗传稳定性和丰富的遗传多样性。
在所有27个位点中有2个位点偏离了 “哈迪-温伯格”平衡(HWE),近亲杂交、无效等位基因、种群退化和自然选择等因素皆可能导致微卫星位点偏离HWE[25]。另外,有学者研究发现,样本量小于25时,对平均等位基因数、有效等位基因数及表观杂合度具有较明显的影响[41]。本研究中,有效样本量≥28,表明位点的多样性参数结果较为可靠。
