基于线粒体COI基因序列的智利竹䇲鱼遗传多样性分析
2021-01-11魏鸿擎梁述章蒋科技黄洪亮马春艳张凤英陈雪忠马凌波
刁 乐,宋 炜,魏鸿擎,梁述章,蒋科技,黄洪亮,马春艳,张凤英,陈雪忠,马凌波
(1.中国水产科学研究院东海水产研究所,农业农村部远洋与极地渔业创新重点实验室,上海 200090;2.上海海洋大学水产与生命学院,上海 201306)
智利竹䇲鱼(Trachurus murphyi)属脊索动物门, 硬 骨 鱼 纲 (Osteichthyes), 鲈 形 目(Perciformes),鲹科(Carangidae),竹䇲鱼属,为暖水性中上层鱼类,是典型的大洋性中上层聚群鱼类[1]。智利竹䇲鱼广泛分布于秘鲁和智利沿海水域以及智利专属经济区以外的公海海域[2]。其生长快、生产力高且捕捞产量多,是世界上主要的海洋经济鱼类之一[3]。研究者曾利用声呐模型[4]和捕食营养学[5]等研究智利竹䇲鱼的资源量和分布范围,20世纪90年代智利竹䇲鱼的资源量可高达440万t[6],但随着高强度商业捕捞的发展,智利竹䇲鱼种群结构遭到严重破坏,南太平洋区域渔业管理组织强调,相关机构迫切需要对竹䇲鱼种群结构和资源保护展开更多的追踪研究[7],以保证竹䇲鱼良好的种质资源。
迄今为止,对智利竹䇲鱼的研究多集中在资源调查[8]、时空分布变化[9]、摄食营养[10]和生长发育[11]等方面,对其遗传多样性和遗传分化方面的研究较少。张敏等[12]以细胞色素b(Cytb)基因作为分子标记,对智利沿岸(33°S、72°W)、太平洋区域(33°S~35°S、93°W ~95°W)等的智利竹䇲鱼群体做了遗传关系初步研究。张伟等[13]利用RAPD技术对东南太平洋海域(34°S~37°S、90°W ~110°W)智利竹䇲鱼种群结构进行了研究。CÁRDENAS等[14]运用微卫星技术对新西兰区域(37°S、178°E)、智利沿岸(20°S~39°S、70°W ~75°W)和太平洋区域(32°S、91°W)等的智利竹䇲鱼进行了遗传关系分析研究。这些研究的采样区域多集中在智利沿岸和太平洋区域,缺少对智利外海(78°W ~81°W、40°S~44°S)区域智利竹䇲鱼群体的研究。此外,目前利用细胞色素氧化酶亚基I(COI)作为分子标记对智利竹䇲鱼的遗传多样性进行分析的研究较少,这使得本研究具有重要意义。
鱼类线粒体基因为共价闭合环状双链DNA分子,结构简单且遵循母系遗传,无组织特异性和遗传重组,因此常被用作分子标记,并广泛应用于种群遗传结构和系统进化的研究中[15-17]。COI作为线粒体13个蛋白编码基因的一员,在分子标记上是研究最为清楚的线粒体基因之一,也有学者认为相比其他线粒体基因,COI是更为理想和常用的分子标记[18]。本研究选用COI为分子标记,检测COI基因的序列变异,分析不同群体智利竹䇲鱼的遗传结构,以期为智利竹䇲鱼资源遗传多样性和渔业管理提供科学评价和理论指导。
1 材料与方法
1. 1 样本采集和基因组DNA提取
本研究样本2014年4—6月采集于智利外海(78°40′W ~81°45′W、40°18′S~44°25′S)水域内,具体分为A、B、C、D、E、F等6个不同采样点,每个采样点均采集36个样本,其采样站点和地理分布见图1。采集样本后取其肌肉组织,利用海洋动物组织基因组DNA提取试剂盒(北京天根生化科技有限公司)提取样本总DNA,经琼脂糖凝胶电泳检测基因组的完整性,并用分光光度法测定DNA的浓度和纯度,于-20℃保存备用。
1. 2 智利竹䇲鱼mtDNA COI序列扩增及测定
对智利竹䇲鱼mtDNA COI序列采用通用引物[19](由上海生工生物技术服务有限公司合成),CFF:(TCRACYAAYCAYAAAGAYATYGGCA C)和CFR:(ACTTCWGGGTGRCCRAAGAATCA)进行PCR扩增。PCR反应体系为50μL,其中包括10×PCR buffer(Mg2+)5.0μL,2.5 mmol·L-1dNTP 4.0 μL,10 μmol· L-1上 下 游 引 物 各2.0μL,5 U· μL-1Taq DNA 聚合酶0.5μL,50 ng·μL-1DNA模板2μL,最后加双蒸水至总体积为50μL。样品在Eppendorf PCR仪上进行PCR扩增,反应程序为:94℃预变性5 min;94℃变性30 s,52℃退火30 s,72℃延伸1 min,共35个循环;循环结束后进行72℃延伸8 min。PCR产物用1.2%的琼脂糖凝胶电泳检测,经成像系统拍照记录,选择目的条带清晰的PCR产物送至上海杰李生物技术有限公司进行双向测序后拼接,测序引物同为PCR扩增引物。

图1 采样站点地理分布图Fig.1 Geographical distribution of sampling sites
1. 3 数据分析
运用Clustal X对测序所得的216个样本的COI序列进行拼接及人工校正,将双向拼接的全序列在NCBI中进行BLASTn同源性分析。利用DnaSP 5.1软件分析样本单倍型数目、单倍型多样性、多态位点数和核苷酸多样性。使用MEGA 5.1软件构建单倍型邻接关系树,关系树的可靠性采用1 000次重抽样进行评估。
使用MEGA 5.1软件计算其变异位点、简约信息位点数和碱基含量;分组计算群体内遗传距离及两两群体间遗传距离,根据Kimura-2-Parameter(K-2-P)模型构建智利竹䇲鱼群体间的UPGMA树。采用简化的中介网络法(medianjoining)[20-21]构建单倍型网络关系图来探讨单倍型的谱系结构。
利用Arlequin 3.1软件中的AMOVA计算两两群体间的遗传分化指数、群体内及群体间的遗传变异分布、遗传变异指数Fst值,利用1 000次重抽样分析来检验统计学显著性。关于群体历史动态研究,利用Arlequin 3.1软件完成Tajima’ s D检验和Fu’s Fs检验,来检测中性假说是否成立,Tajima’s D[22]和Fu’s Fs[23]中性检验如果是负值并且显著偏离中性,则可能是由于群体扩张引起的。利用DnaSP 5进行核苷酸错配分布(mismatch distribution)分析,检验是否存在群体扩张。
2 结果与分析
2. 1 序列特征分析
智利竹䇲鱼所有个体的线粒体COI序列均可被稳定扩增,PCR扩增产物经凝胶电泳,均显示为一明亮清晰的条带,大小为650 bp左右,PCR扩增产物经试剂盒纯化回收后测序。将测序所得到的序列通过BLAST比对,发现其与GenBank数据库中的智利竹䇲鱼COI序列(GenBank登录JQ354519.1)NC_002813.1相似度为99%,经比对和校正,最后用于群体数据分析的COI序列长为624 bp。COI基因序列中,变异位点13个,占总位点数的2.08%;其中简约性信息位点4个,单变异位点9个;平均碱基含量(%)分别为A(23.4)、T(31.1)、G(18.0)、C(27.5),A+T的含量(54.5)略高于G+C的含量(45.5)。
2. 2 单倍型在群体中的分布及遗传关系
在216尾智利竹䇲鱼样本中共检测到14个单倍型,各样本的单倍型分布见图2。9个单倍型为单个群体所独有,而群体间共享单倍型为5个,占单倍型总数的35.7%,其中Hap1和Hap3为6个群体所共享。Hap1拥有最高的共享率,共135个个体拥有此单倍型,占总数的62.5%。其次为Hap3,其被49个个体共享,共享率为22.7%。Hap10被5个群体的7个个体共享,Hap2被4个群体的11个个体共享。在独享单倍型中,群体A拥有最多的独享单倍型,共计4个,分别为Hap4、Hap5、Hap6和Hap8,且为1个个体所拥有。运用所有单倍型构建的邻接关系树显示(图3),其节点分支支持率普遍偏低,没有呈现明显的地理谱系结构。

图2 智利竹䇲鱼线粒体COI基因序列变异位点Fig.2 Trachurus murphyi mitochondrial COI gene sequence variation sites注:A:腺嘌呤,G:鸟嘌呤,C:胞嘧啶,T:胸腺嘧啶Note:A:adenine,G:guanine,C:cytosine,T:thymine
2. 3 群体遗传多样性和遗传结构
智利竹䇲鱼6个群体的遗传多样性分析见表1。从整体水平来看,216个样本的平均单倍型多样性为0.550±0.033,平均核苷酸多样性为0.001 10,单倍型多样性和核苷酸多样性水平均比较低。从单倍型多样性来看,群体A和E的单倍型多样性较高,群体B、C、D和F的单倍型多样性较低,其中群体F仅为0.375±0.090。在核苷酸多样性中,6个群体的核苷酸多样性均比较低,群体F最低为0.000 63。综合单倍型多样性和核苷酸多样性可见,6个群体普遍拥有较低的遗传多样性,其中群体A遗传多样性最高,而群体F的遗传多样性最低。
分析智利外海6个智利竹䇲鱼群体的遗传距离,群体内的平均遗传距离为0.000 63~0.001 70,其中群体F群体内遗传距离最小(0.000 63);群体间的平均遗传距离为0.000 77~0.001 42,群体B和群体F群体间遗传距离最小(0.000 77)(表2)。由以上结果可见,群体内和群体间的遗传距离均较小。用Arlequin3.11软件分析两两群体间的遗传分化指数Fst,结果显示,两两群体间的遗传分化系数在-0.019 50~0.041 75,统计检验均不显著(P>0.05),这表明群体间存在高度的遗传同质性。另外,从基于K-2-P群体间遗传距离构建的UPGMA树看,6个群体两两先后聚为一支,没有明显的遗传隔离(图4)。
AMOVA分子方差分析结果见表3,将所有群体作为一个组群分析,群体间的遗传变异为0.3%,群体内遗传 变异为99.7%,Fst值为0.002 62,变异主要来源于群体内,且群体间无分化。这些分析表明6个群体之间不存在显著的遗传分化。

图3 智利竹䇲鱼14种单倍型在6个群体中的分布及其分子系统树Fig.3 Distribution and molecular phylogenetic tree of 14 kinds of haplotypes in 6 Trachurus murphyi populations
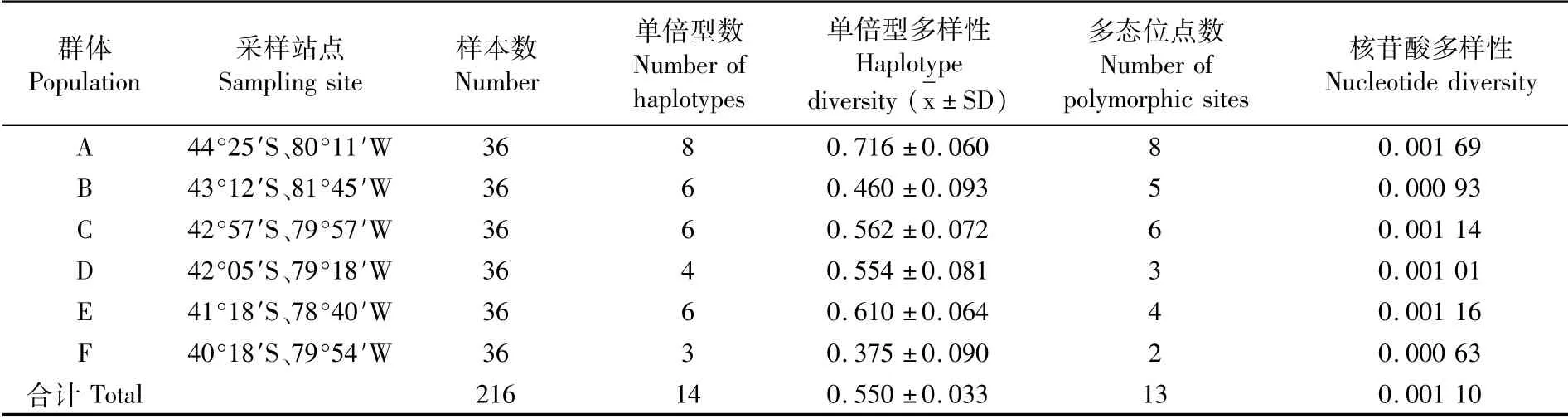
表1 不同群体智利竹䇲鱼的遗传多样性参数Tab.1 Genetic diversity of different populations of Trachurus murphyi
2. 4 群体历史动态分析
依据Tajima’s D检验和Fu’s Fs检验方法对6个智利竹䇲鱼群体线粒体COI基因DNA多样性信息进行中性检验,检验结果见表4。Tajima’s D检验结果和Fu’s Fs检验结果均为负值,Tajima’s D值呈显著差异,Fu’s Fs检验结果呈极显著差异,推测智利竹䇲鱼在智利外海范围内经历过种群扩张事件。

表2 智利竹䇲鱼群体间遗传距离(对角线上)和遗传分化系数(F st)(对角线下)Tab.2 Pairwise genetic distances(above diagonal),fixation index(F st)(below diagonal)between every two populations of Trachurus murphyi

图4 基于K-2-P群体间遗传距离构建的智利竹䇲鱼6个群体的UPGMA树Fig.4 UPGMA tree of 6 Trachurus murphyi populations constructed by K-2-P genetic distance
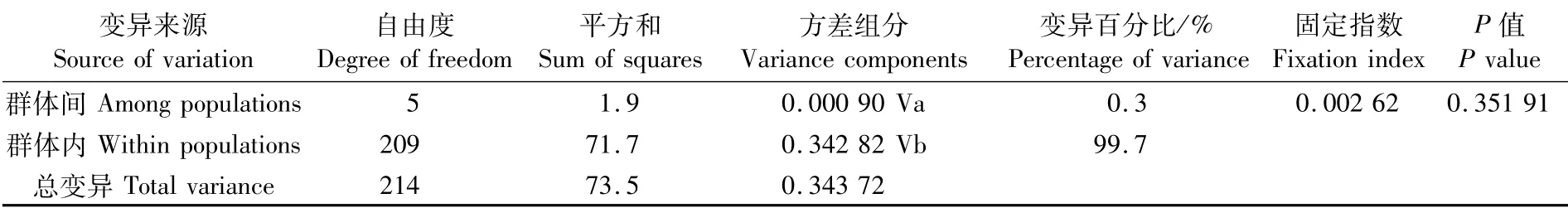
表3 智利竹䇲鱼群体遗传差异的AMOVA分析Tab.3 Analysis of molecular variance(AMOVA)among populations of Trachurus murphyi

表4 智利竹䇲鱼线粒体COI的中性检验和错配分布Tab.4 Neutrality tests and mismatch distribution of mitochondrial COI gene of Trachurus murphyi
3 讨论
3. 1 智利竹䇲鱼群体的遗传多样性
遗传多样性是生物多样性的核心基础,其多样性的高低水平对物种的环境适应力、进化潜能和生存能力至关重要,遗传多样性的丰富程度越大表明其对环境的适应能力越强,同样进化潜能和生存能力也相对较强。相反,遗传多样性的下降会导致物种对环境的适应力降低[24-25]。
在对智利竹䇲鱼为数不多的遗传多样性研究中,研究者们采用的线粒体基因、微卫星等方法分析得出,智利竹䇲鱼普遍具有较低的遗传多样性,并推断智利竹䇲鱼在南太平洋具有广阔的分布带,可视为一个遗传群体[12-14]。本研究共获得14个单倍型,群体间共享单倍型为5个。Hap1拥有最高的共享率,共135个个体拥有此单倍型,占总数的62.5%。种群共享及个体共享单倍型占比大,说明智利外海竹䇲鱼间基因交流频繁。 根 据 LAN 和 SHI[26]及 GRANT 和BOWEN[27]对群体遗传多样性的研究表明,当单倍型多样性小于0.5、核苷酸多样性小于0.005时,其群体的遗传多样性为较低水平。智利外海竹䇲鱼在整个分布范围内皆拥有较低的单倍型多样性(0.375~0.716)和核苷酸多样性(0.000 63~0.001 69),表明其近期出现过或正经历种群瓶颈效应,或种群由单一、少数系群所发生的奠基者效应,导致其遗传多样性偏低,这一结果也验证了可把该竹䇲鱼分布带视为一个群体的推断。GARDENASA等[14]将在南太平洋区域的竹䇲鱼视为一个种群,张敏等[12]认为智利经济区内、外3个群体不存在显著遗传分化,本研究将该智利竹䇲鱼分布带视为一个群体的推断可与上述研究相互印证和补充。
一般单倍型多样性与群体大小和环境相关联,较大的群体和较大差异的环境会导致群体单倍型多样性偏高[28]。本研究中群体A出现较高单倍型多样性,这可能是由智利竹䇲鱼不同季节的空间分布所致,智利竹䇲鱼在冬春季节受西风漂流带来的冷水势力影响,整体向北移动[29],环境因子的变化可能造成遗传多样性的变化。同时,智利竹䇲鱼是高度洄游物种,SERRA[30]认为智利竹䇲鱼的洄游轨迹是一个环形的迁移。样本的随机采集和样本数量的限制,也可能会造成结果偏差,因此,需要研究者们对智利竹䇲鱼群体不断的追踪调查,以检测其多样性的实时变化。
3. 2 智利竹䇲鱼的群体遗传结构
单倍型构建的邻接关系树表明(图3),单倍型的分支与不同采样站点的群体之间没有明显关联,AMOVA分析结果显示,群体间的遗传变异(0.26%)远远小于群体内遗传变异(99.74%),6个智利竹䇲鱼群体没有呈现出显著的系统地理格局。从遗传距离来看,群体内遗传距离和群体间遗传距离都比较小,遗传分化系数Fst所有数值均小于0.05,且大部分为负值。根据Fst划分的遗传分化水平,当Fst<0.05时,说明群体间无分化;当0.05<Fst<0.15时,说明群体间呈低度分化;当Fst>0.15时,说明群体间呈高度分化[31]。分析结果表明,本研究中6个智利竹䇲鱼群体遗传分化主要来源于群体内,且群体间无明显分化,这与张伟等[13]的研究结果一致,即东南太平洋智利竹䇲鱼群体间不存在显著遗传分化。
智利外海竹䇲鱼具有季节性洄游的特点,有较强的扩散能力,因此智利竹䇲鱼的分布范围较广,同时其在较大的水域内可随机交配,这导致其产卵区域和仔鱼分布也十分广泛,本研究所采样本站点属东南太平洋水域,各群体间无明显分化,可将其视为一个较大的智利竹䇲鱼群体。何宗会[32]研究表明由于捕捞船队的大量捕捞和环境因素的变化,造成南太平洋智利竹䇲鱼渔业资源量呈逐渐下降的趋势。同时受经济区内智利竹䇲鱼养殖群体等因素影响,可能导致自然选择多样性被破坏,使得整个智利竹䇲鱼群体的遗传多样性水平较低。为了长远的维持智利竹䇲鱼种质资源和遗传丰富度,应科学规范管理、适度适量捕捞,响应南太平洋区域渔业管理组织的积极号召,以保证智利竹䇲鱼群体的生态平衡。
