基于转录组西施舌微卫星标记开发及隐种鉴定
2022-03-20王雨吉孟学平易乐飞
王雨吉,孟学平,易乐飞
( 江苏海洋大学 海洋科学与水产学院,江苏 连云港 222005 )
西施舌(Coelomactraantiquata)俗称“海蚌”,是瓣鳃纲、蛤蜊科的一种双壳贝类。其足大如舌,肉质饱满肥嫩,与鲍、海参媲美,是国宴上的珍馐美馔。西施舌最初由Spengler于1802年在南海发现并命名[1],长期以来,分布于太平洋西部(特别是我国沿海)的西施舌被视为同一个物种[2]。但是近十多年的研究发现,分布于我国北方和南方的西施舌形态不完全相似,而且彼此间存在较大遗传差异。形态学多元统计分析发现,福建与江苏的西施舌群体在5个数量性状上均表现出显著与极显著差异[3]。生化水平等位酶的研究表明,福建、江苏两地西施舌有一定遗传分化[4];大量DNA水平分析表明,南、北西施舌遗传差异已经达到了种间差异水平[5-9]。比较分析发现,南、北西施舌间的差异甚至达到了四角蛤蜊(Mactraveneriformis)和中国蛤蜊(M.chinensis)的种间差异水平[7]。因此建议将我国西施舌划分为2个物种或隐种,即分布于江苏、山东的北方西施舌和分布于福建的南方西施舌[8]。物种的重新划分迫切需要对2种西施舌的种质资源进行重新评估,为后续的开发、利用和保护提供理论基础。2种西施舌的形态差异不大,不易区分。贝类幼体形态特征通常不明显,成体形态特征易受环境影响,这也增加了鉴别难度。如何准确鉴别2种西施舌也是一个迫切需要解决的问题。
微卫星序列,又称简单重复序列(SSR),广泛存在于真核生物基因组内,具有多态性高、保守性高、共显性遗传、检测方便等特点[10],已广泛应用于遗传图谱构建、数量性状位点定位、群体遗传学、进化遗传学、种质鉴定与辅助育种等研究领域[11-12]。在物种鉴定方面,微卫星已经成功应用于四大家鱼[13]、黄颡鱼属(Pelteobagrus)[14]和扇贝[15]等水产生物的物种鉴别。来自转录组的微卫星标记不仅具备上述特点,还与基因功能直接关联,相应的微卫星引物保守性高、通用性强[16]。所以基于转录组开发2种西施舌微卫星分子标记有助于解决上述问题。
目前西施舌微卫星分子标记的相关报道不多[17-18],因此笔者以西施舌转录组数据为基础,综合利用多种生物信息学工具,挖掘西施舌微卫星序列,并统计转录组中微卫星序列的发生频率、重复基元以及物种特异性微卫星分子标记,为2种西施舌的鉴别,种质资源评估、开发、利用和保护提供理论基础。
1 材料与方法
1.1 原始数据
西施舌转录组原始测序数据下载自NCBI的SRA数据库(https:∥www.ncbi.nlm.nih.gov/sra)和GSA数据库(https:∥bigd.big.ac.cn/gsa)。其中,南方西施舌个体取自福建福州(SRR7699535、SRR7700826)和福建长乐(SRR3107264),北方西施舌个体取自江苏启东(SRR3107263)和山东日照(CRX028172、CRX 025974)。取每个西施舌个体的外套膜、闭壳肌和斧足组织,混合后用于RNA抽提、建库和测序。为准确地获得直系同源基因,从SRA数据库中还下载了中国蛤蜊、四角蛤蜊和薄壳马珂蛤(Mactrotomafragilis)转录组原始测序数据(SRR-1263980、SRR7876669、SRR7876670、SRR4431559、SRR4431558、SRR8217819)。
1.2 转录组组装
转录组原始数据的预处理和从头组装参考文献[8]的方法进行。首先使用Trimmomatic程序[19]去除原始数据中的低质量reads(Q≤20)、接头以及小于50 nt的reads,然后使用Karaken程序[20]去除可能的污染序列,接着使用Trinity程序[21]软件进行转录组的从头组装,最后使用TGICL程序[22]去除组装产物中的冗余序列,从而获得非冗余的转录本。
1.3 直系同源基因筛选
将中国蛤蜊、四角蛤蜊和薄壳马珂蛤转录组与2种西施舌的转录组合并。使用OrthoFinder[23]从中搜索得到直系同源基因组。使用iqtree程序[24]在每个直系同源基因组内利用最大似然法构建系统进化树,然后使用PhyloTreePruner程序[25]对树进行解析,最后挑选出一对一关系的直系同源基因。
1.4 微卫星序列筛选
使用MISA程序[26]进行微卫星位点搜索。筛选标准为单碱基、二碱基、三碱基、四碱基、五碱基、六碱基重复,且各类型重复次数分别不小于10次、7次、5次、5次、5次、5次。若100 bp内出现2个或2个以上的微卫星,则视为复合型微卫星。将搜索结果导入到Excel软件中,对微卫星的基本信息进行统计分析。
1.5 试验材料与DNA抽提
试验材料共21个个体,其中南方西施舌样本共10个,采集自福建漳州,北方西施舌样本共11个,采集自山东日照。取闭壳肌,采用Ezup柱式动物组织基因组DNA抽提试剂盒[生工生物工程(上海)股份有限公司]抽提21个个体的基因组DNA,-20 ℃保存备用。
1.6 微卫星引物设计与筛选
随机选取14组含有不同微卫星序列的直系同源基因,采用Primer Premier 5程序在直系同源基因的保守区设计引物。从南方、北方西施舌中各随机选择2个个体的总DNA进行引物初筛。从中筛选出扩增条带清晰且稳定的4对引物(表1)用于后续样品扩增,荧光标记引物由生工生物工程(上海)股份有限公司合成。
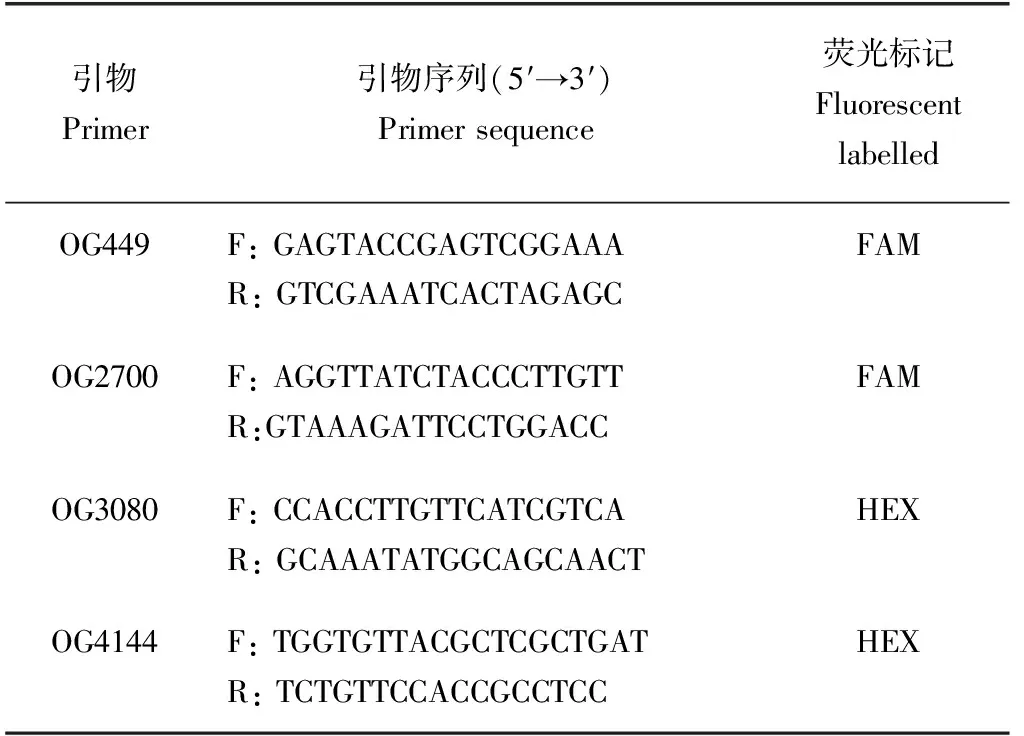
表1 微卫星引物信息Tab.1 SSR primers information
1.7 荧光PCR扩增及毛细管电泳检测
PCR反应在25 μL体系中进行,体系中包含20~50 ng模板DNA,10 pmol正向、反向引物,10 pmol dNTP,1 U Taq DNA聚合酶[生工生物工程(上海)股份有限公司]。荧光PCR扩增采用两步PCR扩增法。首先95 ℃预变性3 min;然后进行第1步PCR扩增:94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,共10个循环;最后进行第2步PCR扩增:94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共35个循环。PCR产物送生工生物工程(上海)股份有限公司进行毛细管电泳检测(3730XL,美国ABI公司),利用GeneMarker分析软件对原始数据进行分析,以确定PCR产物片段大小。
2 结 果
2.1 微卫星数量及在转录组中的分布
原始数据拼接后,在北方西施舌转录组中得到272 074个unigene,N50为869 bp,从中检测到18 611个微卫星,复合型微卫星为674个,分布于16 348条unigene中,平均每16.6个unigene中含有1个微卫星,平均跨度为9.3 kb,出现频率(含有微卫星位点的unigene数量/unigene总数量)为6.0%,发生频率(微卫星个数/unigene总数量)为6.8%,有1758条unigene含有1个以上微卫星。在南方西施舌转录组中得到287 887个unigene,N50为1297 bp,从中检测到27 553个微卫星位点,复合型微卫星为1143个,分布于23 614条unigene中,平均每10.5个unigene中含有1个微卫星,平均跨度为8.4 kb,出现频率为8.2%,发生频率为9.6%,有3189条unigene含有1个以上微卫星。从分布情况来看,南方西施舌转录组中的微卫星比北方西施舌更密集。
2.2 碱基重复类型
重复基元种类、数量以及优势重复基元在2种西施舌的转录组中存在差异(表2)。在北方西施舌的18 611个完美型微卫星中共有63种重复基元,二至五碱基重复基元分别有3、10、24、24种;在南方西施舌的27 553个完美型微卫星中共有89种重复基元,多于北方种,其二至五碱基重复基元分别为3、10、28、43种,此外,还发现了3种六碱基重复基元。在北方西施舌和南方西施舌转录组中二碱基重复中的优势基元均为AT/AT,分别有1119和2176个,占完美型微卫星总数的6.01%和7.9%。三碱基重复中的优势基元不一致,分别为AAC/GTT和ATC/ATG,数量为1765个和2408个,占微卫星总数的9.5%和8.7%。四碱基重复中的优势基元分别为AATC/ATTG和AAAC/GTTT,数量为137个和376个,占微卫星总数的0.7%和1.4%。
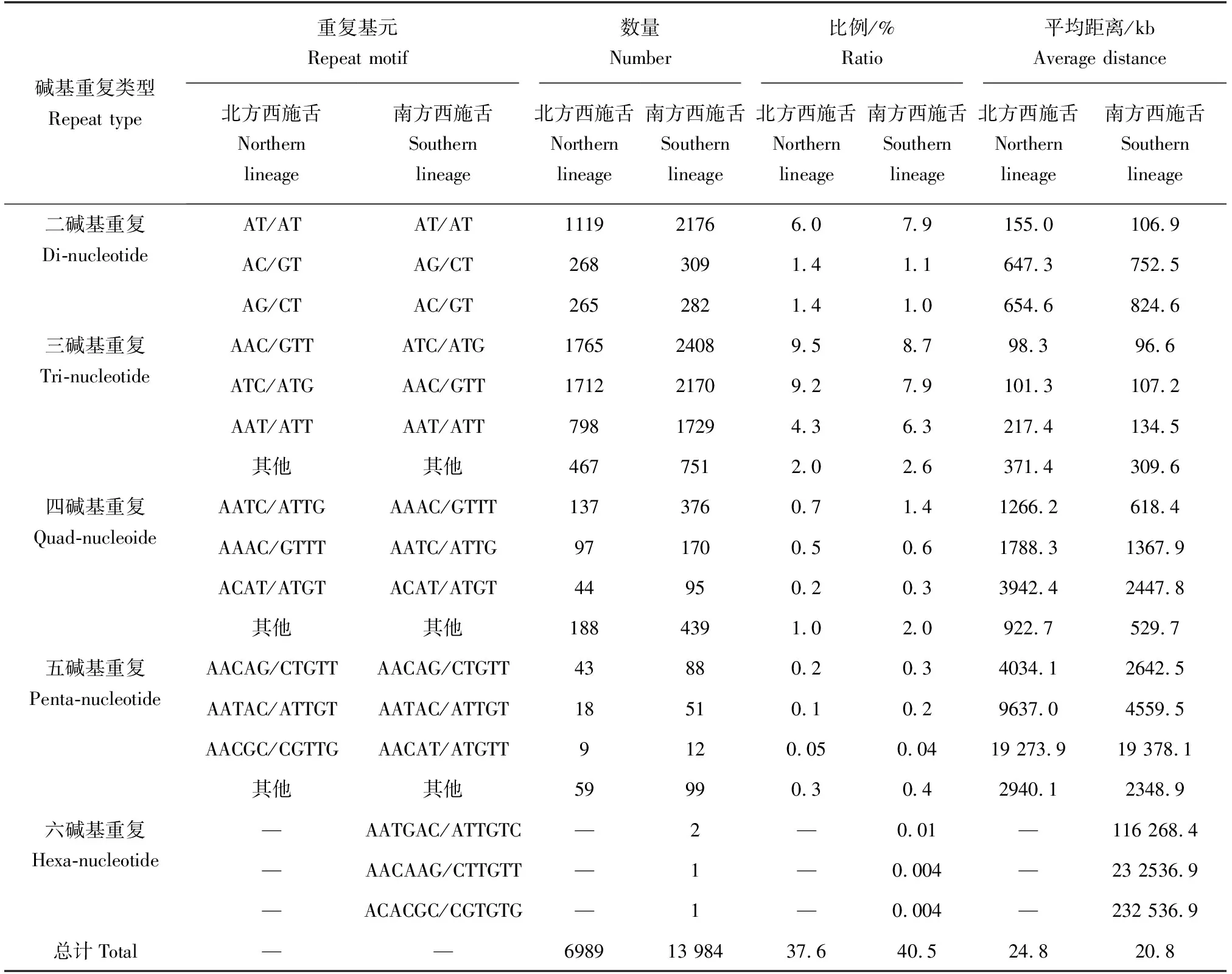
表2 微卫星重复基元出现情况Tab.2 The occurrence of different motifs of SRRs
2.3 碱基重复拷贝次数
在北方西施舌和南方西施舌转录组中,微卫星重复拷贝次数最高为31和40次,最低均为5次。当微卫星重复拷贝次数为10次时,微卫星数量最多,丰度最大。含5~10次重复的微卫星在2个种中最多,这些微卫星在北方西施舌中有14 087个,占总微卫星的75.7%,在南方西施舌中有20 139个,占总微卫星的73.1%。
2.4 重复基元长度
在北方西施舌和南方西施舌转录组中微卫星重复基元长度区间分别为10~75 bp和10~116 bp,其中最长的基元分别为五碱基的15次重复(75 bp)和四碱基的29次重复(116 bp)。在2个种中除了单碱基重复外,最常见的微卫星长15 bp,分别有3552个和4581个,分别占完美型微卫星总数的19.09%和16.6%,均为三碱基的5次重复。微卫星的长度与发生频率呈负相关。11~15 bp的短微卫星数量最多,发生频率为5.58%和7.35%;其次为16~20 bp的微卫星,发生频率为0.96%和1.52%;21~25 bp的微卫星的发生频率为0.26%和0.57%;大于26 bp的微卫星的发生频率0.04%和0.13%。
Temnykh等[27]按照碱基长度差异将微卫星分成2类:当微卫星长度≥20 bp时具有高度多态性(Ⅰ型);微卫星长度>12 bp且<20 bp时具有中等多态性(Ⅱ型);而≤12 bp的微卫星多态性较低,但具有突变潜能。本试验中,在北方西施舌中,7240个微卫星(38.90%)为Ⅰ型微卫星,1293个微卫星(6.95%)为Ⅱ型微卫星;在南方西施舌中,10 867个微卫星(39.44%)为Ⅰ型微卫星,2718个微卫星(9.86%)为Ⅱ型微卫星。
2.5 含微卫星序列的直系同源基因
在2个种的转录组中一共检索出4916组一对一关系的直系同源基因。这些微卫星被分为3类:第1类为两者均无微卫星的直系同源基因组,有3695组;第2类为只有一方有微卫星的直系同源基因组,有911组;第3类为两者均有微卫星的直系同源基因组,有310组。第3类直系同源基因中,有252组在2个种中含有相同的微卫星序列,不能用于区分西施舌的2个种。其余58组在2个种中含有不同的微卫星重复次数。对这58组直系同源基因和第2类直系同源基因(合计969组)进行进一步的筛选,剔除单碱基重复微卫星、旁侧序列长度不足20 bp的直系同源基因后,余下409组。在这409组直系同源基因中,2个种间的微卫星序列长度不同,但是微卫星序列两侧序列相似,这种含微卫星序列的直系同源基因有利于设计通用引物,使用这些通用引物可在2种西施舌中扩增出不同大小的条带,进而可以作为潜在的物种特异性分子标记来区分2种西施舌。
2.6 直系同源基因中微卫星位点的验证
利用4对荧光标记微卫星引物对21个西施舌个体进行PCR扩增,扩增产物经毛细管电泳检测后,每对引物在每个个体中均获得了清晰、明确的DNA片段,且无杂峰(图1显示了部分结果),4个微卫星位点在21个个体上的基因型见表3。引物OG449和OG4144多态性较高,分别检测到了10个和8个等位基因;引物OG2700和OG3080多态性较低,分别检测到了5个和3个等位基因(表3)。在这4对微卫星引物中,没有1对微卫星引物能单独区分南方、北方西施舌,但是使用引物组合还是可以初步区分南方、北方西施舌。引物OG4144在北方西施舌中扩增产物大于380 bp,在南方西施舌中小于380 bp;引物OG3080在北方西施舌中扩增一定会出现489 bp的产物(图1a),在南方西施舌中一定会出现492 bp的产物(图1b)。
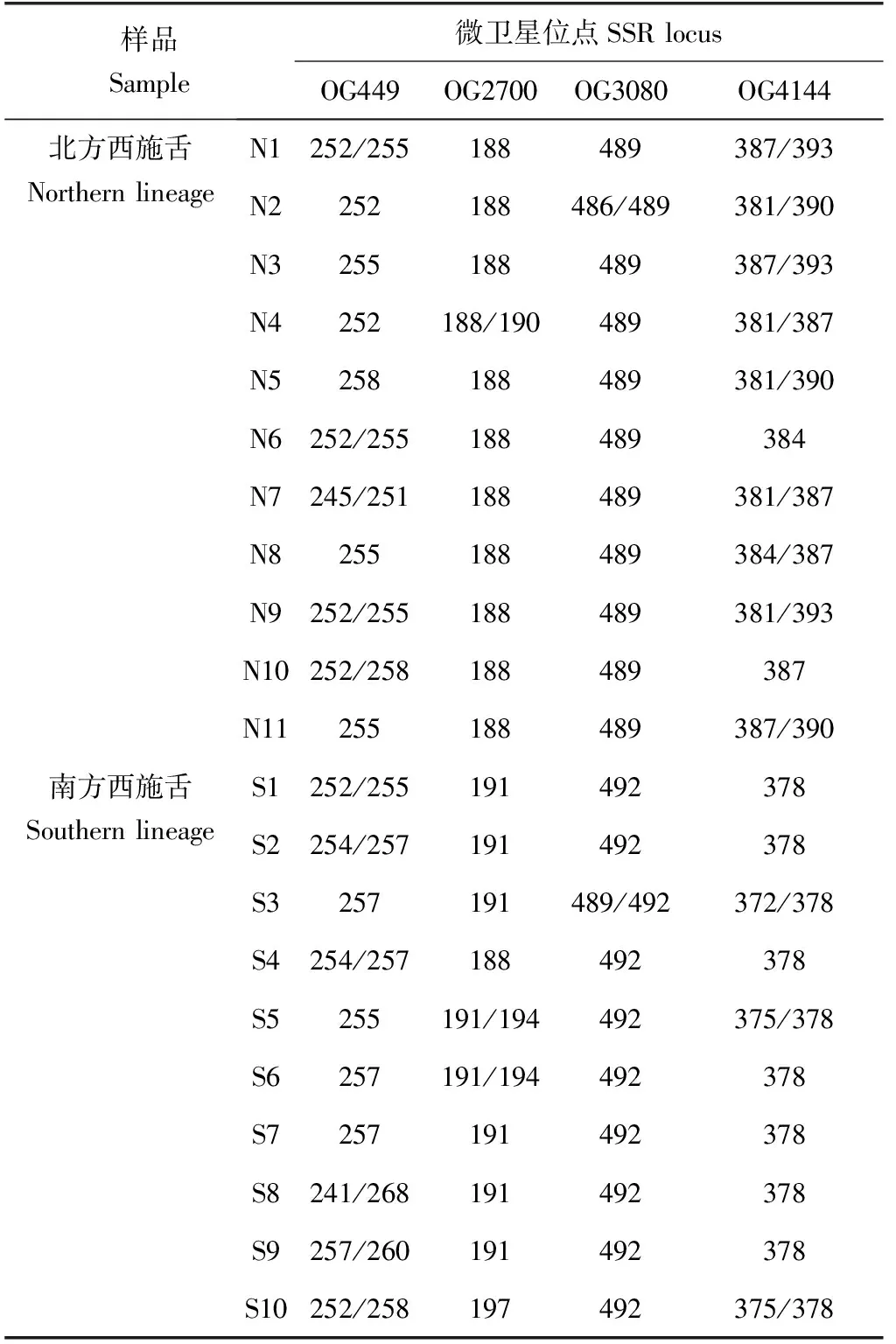
表3 4个微卫星位点基因型Tab.3 The genotypes at 4 SSRs loci
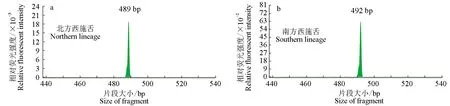
图1 引物OG3080对南方、北方西施舌扩增产物的带型Fig.1 Amplification bands in northern and southern lineage of surf clam C. antiquata using primer OG3080
3 讨 论
3.1 西施舌微卫星的发生频率
笔者对2种西施舌转录组进行了微卫星挖掘,北方西施舌每隔9.3 kb出现1个微卫星,出现频率为6.0%,南方西施舌每隔8.4 kb出现1个微卫星,出现频率为8.2%。与其他贝类相比,它们的微卫星的出现频率均高于马氏珠母贝(Pinctadafucatamartensii)心脏(5.98%)[28],均低于马氏珠母贝血细胞(11.0%)[29]、“渤海红”扇贝(A.irradians)(11.0%)、墨西哥湾扇贝(A.irradiansconcentricus)(9.0%)[30]、织锦巴非蛤(Paphiatextile)(10.0%)[31]转录组微卫星出现频率。这表明不同物种之间或同物种之间,甚至是同一物种不同组织间,因为物种和组织差异导致表达的mRNA种类、序列和丰度不同,因此挖掘出的微卫星分布情况也随之不同。
3.2 西施舌微卫星的优势基元
大多数贝类的微卫星主要以二碱基和三碱基重复为主要类型。缢蛏(Sinonovaculaconstricta)、“渤海红”扇贝和墨西哥湾扇贝转录组微卫星的优势基元为三碱基重复,分别占各自微卫星位点总数的37.13%、46.66%和45.61%[30,32];而虾夷扇贝(Patinopectenyessoensis)的优势重复类型为二碱基重复,占40.54%[33]。2种西施舌的优势重复类型均为三碱基重复,分别占各自微卫星位点总数的25.5%和25.6%,占比少于上述物种。不同贝类的优势微卫星重复类型有所不同,产生这种差异的原因可能是多样的。这种差异可能是物种固有特性的体现,也可能是由分析方法和组学数据不同所致。转录组可以看作基因组的一个子集,所以基于不同的组学数据必然导致不同的结果;不同分析方法的算法基础不同,所以基于不同的分析软件也会导致不同的结果。
3.3 西施舌微卫星标记与隐种鉴定
当近缘种形态差异不明显时,微卫星标记可有效鉴别近缘物种。这种鉴别技术往往依赖于通用性强且特异性高的几对PCR引物。引物的通用性要求引物的结合位点必须位于基因的保守区,引物的特异性要求扩增产物必须位于基因的高变区。如果引物和被扩增片段都位于保守区,那么不能有效地区分物种;如果引物和被扩增片段均位于高变区,那么只能鉴别出物种内部的不同组群。在含有微卫星的直系同源基因上设计引物能够很好地实现引物的通用性和特异性。虽然使用非同源位置的微卫星标记也可以鉴别近缘物种,但是这种扩增产物并不都是同源产物,扩增产物大小与物种间的亲缘关系无明显的相关性。非同源产物和扩增产物大小异源同型会严重干扰系统发育、群体遗传学和进化研究[34],而使用直系同源基因内的微卫星标记可以有效避免此类问题。将引物设计在直系同源基因的微卫星侧翼的保守序列上,仅扩增高变的微卫星位点,可以有效鉴别这2种西施舌。有409组直系同源基因在2种西施舌中呈现了多态性的微卫星序列,这些序列的发现为通用引物设计和西施舌物种鉴别奠定了基础。
贝类微卫星的挖掘传统上采用磁珠杂交筛选、建立cDNA文库的方式检索微卫星位点。李晶晶[17]用磁珠杂交筛选和表达序列标签(EST)数据库检索2种方法从西施舌闭壳肌中鉴定出51条含微卫星的序列,包含12种重复基元。朱立静等[18]用生物素—磁珠吸附法从西施舌中筛选出38个微卫星位点,包含5种重复基元。但是这些方法效率较低,信息获取度也较低。随着高通量测序的飞速发展,基于转录组数据检索微卫星的方法得到广泛应用,这种方法获得的微卫星不仅具有一般分子标记的特点,而且信息量大、通用性好、快速高效[35]。因此,本试验中,使用基于转录组的方法得到了很多微卫星,显著多于上述研究。这些微卫星将为2种西施舌的鉴别,种质资源评估、开发、利用和保护奠定理论基础。
4 结 论
在北方西施舌转录组中微卫星出现频率为6.0%,含63种重复基元;南方西施舌的微卫星出现频率为8.2%,含89种重复基元。在2个隐种的转录组中,除了单碱基重复外,优势重复类型均为三碱基重复,三碱基重复的优势基元分别为AAC/GTT和ATC/ATG,最常见的微卫星长度均为15 bp。微卫星的重复次数主要集中在5~10次。有409组直系同源基因在2个隐种中含有不同的微卫星序列,可以作为潜在的物种特异性分子标记。
