厚壳贻贝足丝超氧化物歧化酶的重组表达及功能
2020-12-26
(浙江海洋大学海洋科学与技术学院,海洋生物蛋白质工程研究室,浙江舟山 316022)
贻贝足丝是贻贝通过足腺分泌的一种丝状非细胞组织,其端部为圆盘状,即足丝盘[1]。利用足丝盘,贻贝可在水环境中黏附于各种物质表面,因而足丝成为新型生物粘附剂研究的重要对象[2]。贻贝足丝的主要成分为各种足丝蛋白(Mussel Foot Protein,MFP),目前已报到的贻贝足丝蛋白有十多种,多具有黏附功能[3-4]。已鉴定的贻贝足丝蛋白可根据其功能以及在足丝形成和黏附过程的作用主要分为五类,第一类是作为足丝的外套,负责足丝的整体保护,这一类代表为MFP-1,该蛋白主要覆盖于足丝表面构成足丝的保护外套,以防止海水的腐蚀及微生物的降解[5-6]。第二类是负责形成足丝纤维的核心结构,主要包括前胶原蛋白分子(Pre-collagen,包括D 型、P 型和NG 型),纤维基质蛋白(Thread matrix protein,TMP)等形成足丝的纤维状内核,赋予足丝纤维韧性和弹性[7-9];第三类是产生交联并负责足丝的结构稳定,包括MFP-2 和MFP-4 等,这些分子或者形成交联结构将其他足丝蛋白分子结合在一起,或者形成用以包裹其他足丝蛋白的基质,最终使得足丝形成一个稳定而致密的结构[10-11];第四类是直接行使黏附功能的蛋白,包括MFP-3 和MFP-5,均为高多巴含量的分子,依赖多巴转换为多巴醌并通过氢键与外界基质形成牢固的连接[12-13]。第五类则是辅助性足丝蛋白,主要负责维持足丝的氧化-还原平衡,使得足丝的黏附性能处于可控状态,目前仅报道了MFP-6,是一种抗氧化足丝蛋白[14-15]。
厚壳贻贝是一种研究足丝蛋白的极好材料,在前期研究中,采用蛋白质组学研究策略,从其足丝中鉴定一种具有超氧化物歧化酶(Superoxide Dismutase,SOD)结构域的新型足丝蛋白,被命名为Byssal Superoxide Dismutase (BSOD)[16]。这也是首次从贻贝足丝中发现SOD 类的蛋白。SOD 是一类抗氧化蛋白,广泛分布于生物体内,对细胞防止氧化损伤具有极为重要的作用[17-18]。SOD 是一种金属结合酶,根据其结合的金属离子不同,可以将SOD 分子分为四类,包括Fe-SOD,Mn-SOD,Cu/Zn-SOD 和Ni-SOD。其中Cu/Zn-SOD分布最为广泛,包括各种生物的细胞内或者细胞外基质。贻贝足丝分泌后,在其固化并发挥黏附功能之前,需要保持一定的还原态,以防止被海水中各种因子的氧化而导致黏附失败[14]。尽管足丝蛋白MFP-6 已被证明是一种抗氧化蛋白并对足丝的黏附具有重要作用[15],但厚壳贻贝足丝中BSOD 的发现则提示了在贻贝足丝中不止MFP-6 一种抗氧化蛋白,也表明贻贝足丝具有一套复杂的抗氧化体系。同时,BSOD 是一种具有信号肽的分泌蛋白,因此相比胞内SOD 具有更强的结构稳定性,在医学、药学、食品等领域具有潜在的应用价值。
为深入研究贻贝BSOD 的结构与功能,采取了密码子优化策略,根据大肠杆菌偏爱密码子重新设计和优化厚壳贻贝BSOD 基因序列,在此基础上,进一步通过原核重组表达和蛋白质纯化手段,获得了重组BSOD 蛋白并开展了功能分析。为进一步研究BSOD 的结构与功能关系,探讨BSOD 在贻贝足丝黏附过程中的分子机制奠定了基础,也为BSOD 的后续蛋白质工程研究提供了科学依据。
1 材料与方法
1.1 厚壳贻贝BSOD 的序列分析
BSOD 序列已上传数据库(GenBank 编号AKI87983.1)。通过生物信息学手段对其开展了序列分析。其中,开放阅读框以Lasergene 软件(版本7.0)分析;序列比对采用DNAman 软件进行;蛋白质同源搜索以NCBI 网站的BLAST 工具在线(https://blast.ncbi.nlm.nih.gov/Blast.cgi#)进行;多序列比对和进化树分析采用软件DNAMAN(版本7.0)进行;结构域预测采用SMART 软件在线(http://smart.embl-heidelberg.de/)进行;信号肽预测采用SignalP4.1 软件在线进行(http://www.cbs.dtu.dk/services/SignalP/)。二级结构预测采用Jpred4进行(http://www.compbio.dundee.ac.uk/jpred4);三级结构预测采用SWISS MODEL 服务器(https://swissmodel.expasy.org/)进行。
1.2 目标基因的密码子优化、设计与表达载体的构建
对厚壳贻贝BSOD 目标基因(495 bp)开展密码子优化以便适于大肠杆菌表达体系。采用OptimumGeneTM技术平台(南京金斯瑞公司,http://www.genscript.com/)将目标基因中的部分密码子替换为大肠杆菌偏爱密码子;并分别在目标基因的5’端和3’端添加Nco I 酶切位点(CCATGG)和Xho I 酶切位点(CTCGAG);此外,在目标基因5’端添加保护碱基(CA)和多聚组氨酸标签(CATCATCATCATCATCAC);最终设计出的目标基因全长为526 bp 并由南京金斯瑞公司化学合成并测序验证。
目标基因与表达载体(pET28a)均采用Nco I 和Xho I 双酶切;之后采用T4-DNA 连接酶将目标基因与pET28a 质粒的酶切片段进行连接;连接产物(BSOD/pET28a)转化至大肠杆菌DH5α 感受态细胞并涂布于含卡那霉素的LB 平板上,37 ℃过夜培养后挑取阳性单克隆进行测序验证。
1.3 重组足丝BSOD 蛋白的表达、复性及分离纯化
将BSOD/pET28a 重组表达质粒转化进表达菌株(E.coli,BL21)并涂布于含卡纳霉素的LB 平板培养基中,37 ℃培养过夜。挑取单克隆菌落,于含卡纳霉素的LB 液体培养基中培养过夜。菌液按照1:100 比例扩大培养。设置37 ℃和16 ℃两种培养温度分别培养,转速为200 r·min-1。在菌液OD630值为0.6~0.8 时加入IPTG(1 mmol·L-1)进行诱导。37 ℃培养的菌株IPTG 诱导时间为4 h;16 ℃培养的菌株IPTG 诱导时间为16 h。
IPTG 诱导结束后,收集菌液离心(1 000×g,4 ℃,10 min)收集菌体,并以菌体裂解液重悬,冰浴超声破碎,离心(8 000×g,4 ℃,15 min)分别收集上清和沉淀(包涵体)。用包涵体溶解液溶解包涵体,之后离心(8 000×g,4 ℃,20 min)并收集上清液;上清液经0.45 μm 微孔膜过滤后上样镍柱并洗脱和收集目的蛋白。
参照文献[19]方法,对镍柱纯化后的目的蛋白进行稀释复性。分别配置不同尿素浓度(1~6 mmol·L-1)的复性液(含0.1 mmol·L-1氧化型谷胱甘肽,0.9 mmol·L-1还原型谷胱甘肽,20 mmol·L-1Tris-HCl,pH 8.0)4 ℃条件下,将蛋白样品从高浓度尿素到低浓度尿素依次进行透析。
复性后的目标蛋白采用高效液相色谱进行进一步纯化。高效液相色谱仪为Waters600 E 型(Waters 公司,美国),检测器为Waters 2487 双波长紫外检测器(Waters 公司,美国);色谱柱为C8 反相色谱柱(Vydac,218TP);采用线性梯度洗脱,即B 液(含0.1%三氟乙酸的乙腈)在30 min 内,其比例由20%上升至50%。A液为含0.1%三氟乙酸的纯水;流速为1 mL·min-1;检测波长为280 nm。
1.4 SDS-PAGE 和Western 分析
SDS-PAGE 采用4%浓缩胶结合12%分离胶,样品事先以2×电泳上样缓冲液溶解;以牛血清白蛋白作为定量分析对照,上样体积为15 μL。恒压模式电泳。电泳结束后以考马斯亮蓝R250 进行胶染色并观察。
将电泳分离后的蛋白胶转印至PVDF 膜。因所表达的目标蛋白含有多聚组氨酸标签(His-tag),因此,膜经洗涤和封闭后,采用鼠抗His-tag 抗体(南京金斯瑞公司,货号A00186)进行孵育过夜,之后再加入辣根过氧化物酶标记的羊抗鼠二抗(南京金斯瑞公司,货号A00160)孵育60 min,以ECL 化学发光试剂进行显影。
1.5 重组足丝BSOD 的质谱鉴定
利用质谱(Voyager DETMSTR,美国Applied Biosystems 公司)进行重组蛋白的精确分子量鉴定;基质为α-氰基-4-羟基-肉桂酸(CCA),通过以下方式制备样品:取2 μL 样品液加入到8 μL CCA(溶于50%乙腈溶液,含0.1%三氟乙酸)的饱和溶液,混匀后取1 μL 点样,室温干燥后测定,采用内标法进行分子量校正。
对重组蛋白开展串联质谱(5700 Proteomics Analyzer TOF/TOFTM,美国Applied Biosystems 公司)分析所表达的蛋白肽段序列;蛋白样品经还原烷基化以及胰蛋白酶酶解后上样质谱仪;质谱分析工作电压为20 kV,采用正离子模式,自动模式采集数据。PMF 质量扫描范围为700~3 600 Da,以强度最大的8 个峰进行二级质谱分析;谱图用myoglobin 酶解肽段进行外标校正。根据肽段的理论序列生成理论胰酶酶切肽段质量与肽指纹图进行比对,质量相符的肽段进一步进行串联质谱谱图分析,获得其对应的氨基酸序列并与理论氨基酸序列进行比较。
1.6 重组BSOD 的功能测试
对重组并复性成功的BSOD 开展功能分析,其中重组BSOD 溶液按摩尔比1:2 加入预先配置的硫酸铜和氯化锌溶液进行Cu/Zn 离子鳌合,并比较鳌合前后重组BSOD 的功能。酶活采用总SOD 活性检测试剂盒(WST-8 法)(货号:S0101,碧云天公司)并参照说明书进行;SOD 标准品为牛红细胞Cu/Zn-SOD(上海生工公司,货号:A003540);重组BSOD 的自由基清除率采用DPPH 法测定[20]。取重组蛋白溶于0.02%DPPH 的无水乙醇溶液,样品终浓度为0.015 mmol·L-1;采用无水乙醇作参比,混匀后在室温下避光反应30 min,于517 nm 波长下测吸光度。平行测定3 次取平均值。采用维生素C(Vc)为阳性对照。根据下列公式计算样品对DPPH 自由基的清除率:清除率=(A0+A1-A2)/A0*100%;式中,A0 为无水乙醇加水加DPPH 溶液的吸光度;A1 为含蛋白样品的无水乙醇溶液的吸光度;A2 为含蛋白样品的DPPH 溶液的吸光度。
2 结果与分析
2.1 厚壳贻贝SOD 序列分析结果
厚壳贻贝BSOD 的cDNA 基因(GeneBank 编号AKI87983.1)全长851 bp,其开放阅读框长度为546 bp,编码一个由181 个氨基酸残基组成的前体蛋白,其核苷酸-氨基酸序列比对见图1。
经NCBI 网站的BLAST 搜索,发现厚壳贻贝足丝BSOD 分子与数据库中其他物种的SOD,特别是Cu/Zn-SOD 具有较高的序列相似性,其氨基酸一致性在40%~60%之间,序列覆盖率在70%~96%之间。在BLAST 结果中,选择有确定注释名称且氨基酸一致性在50%以上的同源序列共计38 条进行多序列比对分析,结果如图2。由图2 可见,各序列在SOD_Cu 结构域肽段具有较高的序列相似性,特别是在第60-140号残基所处肽段,涉及铜和锌离子结合位点的关键氨基酸残基,如第70、73、75、90、98 和107 号组氨酸在各序列中完全保守(图1 方框和图2*号标注);此外,84 和175 号半胱氨酸残基(图1 圆框和图2#号标注)在各序列中也完全一致。此外,我们注意到BSOD 序列与其他物种SOD 序列相比,在30 号和176 号额外多出两个半胱氨酸残基(图1 圆框和图2▼号标注),表明BSOD 可能比其他同源SOD 多出一对二硫键。

图2 厚壳贻贝BSOD(方框所示)与同源序列的多序列比对Fig.2 Multiple sequence alignment of BSOD with 38 homologys form other species
采用临近法(Neighbor-joining) 绘制厚壳贻贝BSOD 与38 条同源序列的进化树,结果如图3。厚壳贻贝BSOD 与其他物种的SOD 相比,其进化地位较为独特,且与来自水生甲壳类(如虾、蟹等)的胞外Cu/Zn-SOD 具有较近的亲缘关系。
对BSOD 的二级结构预测结果表明,其序列中含有7%的a-螺旋和38%的β-折叠,其中a-螺旋主要位于信号肽区域(图4A);SMART 结构域分析结果显示,厚壳贻贝BSOD 前体序列中N 端17 个氨基酸残基(MNMISIFLLSVVALSTA)为信号肽,进一步的SignalP 软件对信号肽预测结果证实了SMART 软件的计算结果,表明信号肽预测可靠;此外,序列中第34-178 号肽段为SOD_Cu 结构域(pfam 编号为PF00080)。图4B和C 分别展示了BSOD 三级结构预测的二聚体和四聚体模型,可见BSOD 成熟肽区主要包含7 段反平行β-折叠,与二级结构预测基本一致。
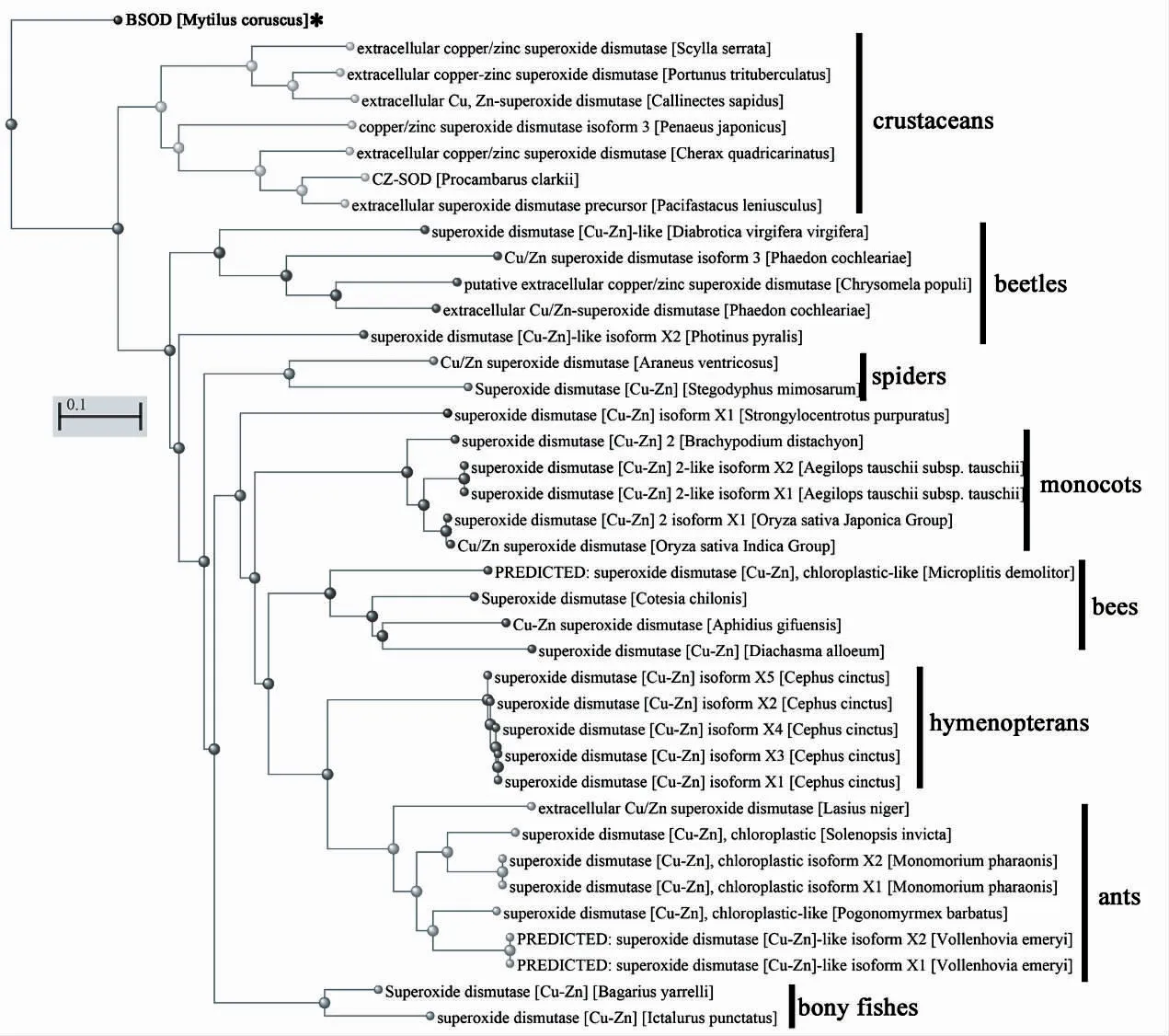
图3 采用临近法构建系统树对厚壳贻贝BSOD 与38 条同源序列的进化树分析结果Fig.3 Phylogenetic analysis of M.coruscus BSOD with 38 homologous from other species using the Neighbor-joining approach
2.2 BSOD 基因的设计和密码子优化
天然BSOD 成熟肽区域的基因长度为495 bp,经重新设计和优化,目标基因长度为526 bp,设计后的目标基因编码一条长度为173 个氨基酸残基组成的重组蛋白(图5A),与天然BSOD 相比,重组BSOD 在N端多出一段MAHHHHHH 肽段,其余序列与天然BSOD 一致,两者的序列比对如图5B。重新设计的目标基因与天然BSOD 序列的一致性为68%。
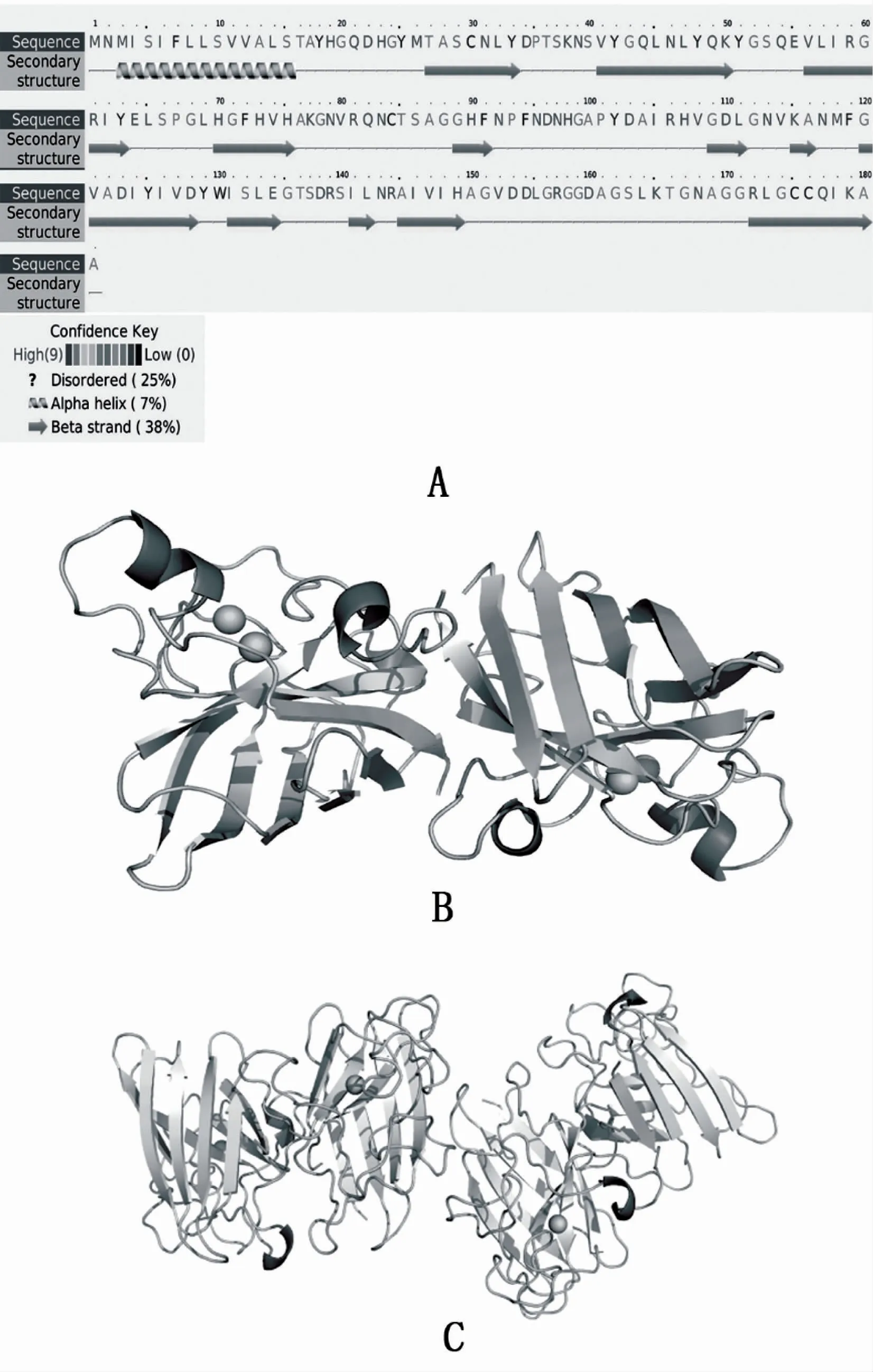
图4 厚壳贻贝BSOD 空间结构预测Fig.4 The structural features of M.coruscus BSOD
对优化后的目标基因开展了密码子适应指数(Codon Adaptation Index,CAI)分析,结果如图6。BSOD 基因在优化前,其CAI 值经OptimumGeneTM软件在线计算(http://www.genscript.com.cn/technology-support/online-tools),为0.62;经优化后,其CAI 值达到了0.97。CAI 值大于0.8 则意味着该目标基因在大肠杆菌中的表达效率较高[21]。
2.3 重组BSOD 的表达、纯化与鉴定
BSOD 重组菌经IPTG 诱导表达后,收集菌体经超声破碎和离心后,上清与沉淀部分分别以SDSPAGE 进行检测,结果如图7。由图7A 可见,目标蛋白主要表达于沉淀部分,呈包涵体表达特征。目标蛋白条带约为19 kDa,与重组BSOD 的预期分子量(18.7 kDa)接近。进一步经western blot 检测,所表达的目标条带能与抗组氨酸抗体结合并产生特异性亮带,表明所表达的重组BSOD 带有多聚组氨酸标签。同时,在Westernblot 检测中,也发现有分子量约36,55 和72 kDa 的条带,推测为BSOD 分别形成二聚体,三聚体和四聚体所致(图7B)。

图5 BSOD 基因优化后的核苷酸-氨基酸序列比对图Fig.5 The deduced amino acid sequence of recombinant BSOD gene (A) and sequence alignment of BSOD gene before and after OptimumGeneTM optimization (B)
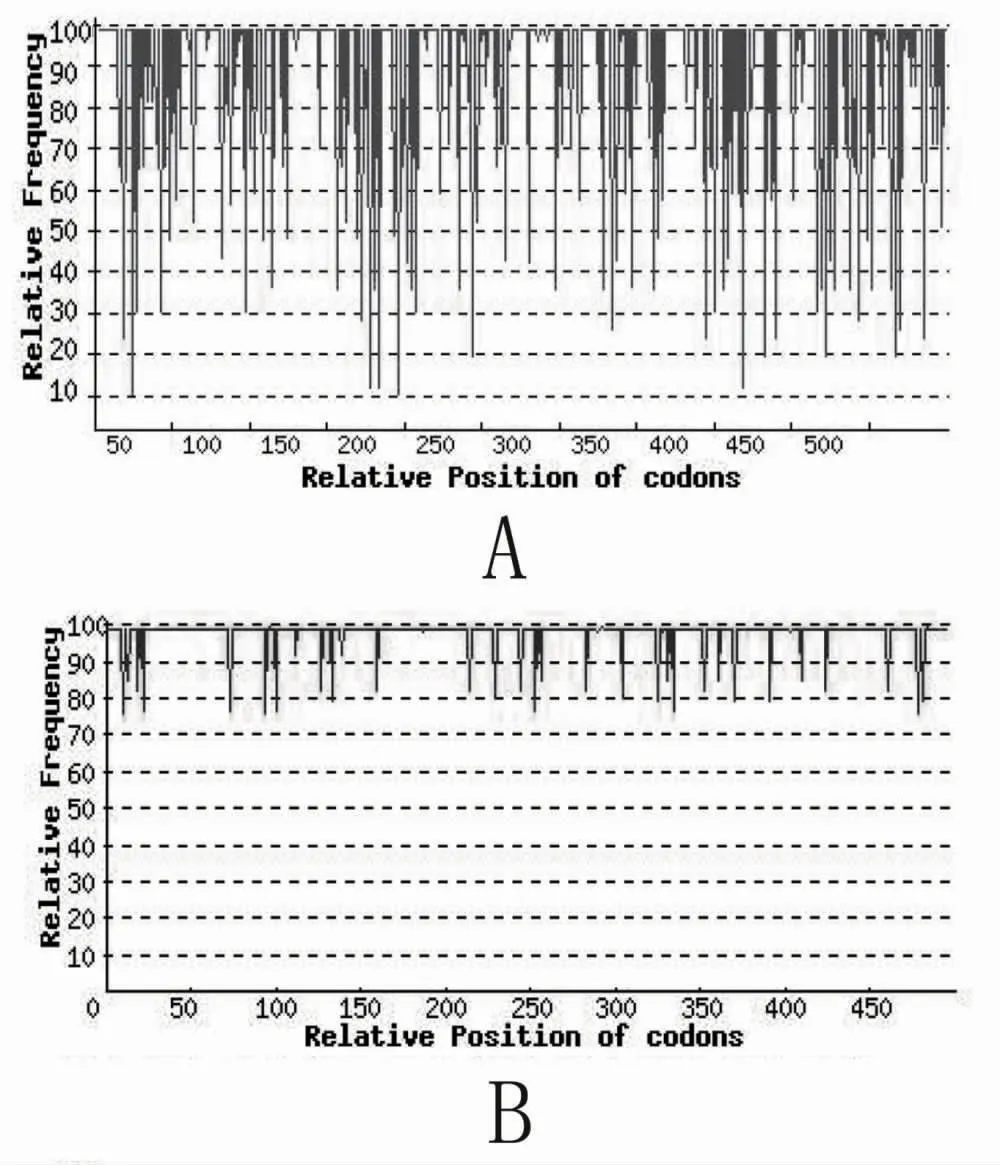
图6 BSOD 基因经优化前后的密码子使用偏好性分布比较图Fig.6 The distribution of codon usage frequency along the gene sequence of BSOD before (A) and after (B)OptimumGeneTM optimization
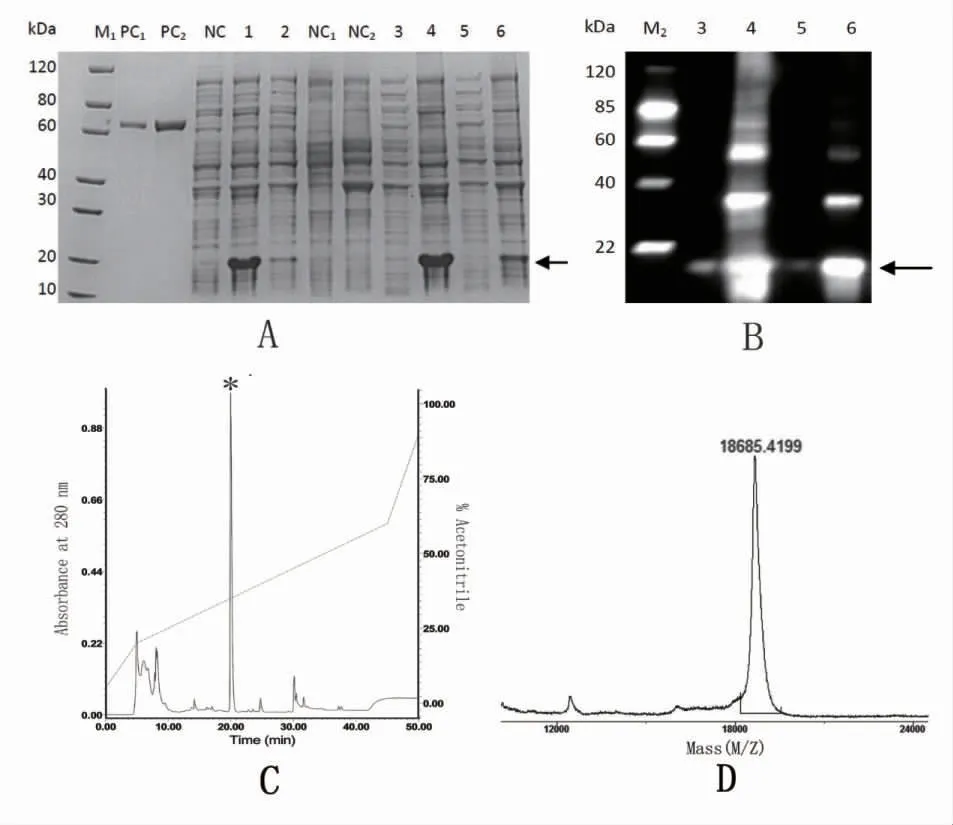
图7 重组BSOD 蛋白的表达及纯化Fig.7 Recombinant expression and purification of BSOD
重组BSOD 经镍柱纯化后采用稀释法进行复性,以期完成正确折叠。复性后的重组BSOD 采用反相高效液相色谱进行分离,结果如图7C。由图可见,复性后重组BSOD 经HPLC 分离并在35%乙腈浓度出峰,为单峰,表明纯度较高。目标峰进一步开展质谱分析。结果见图7D,可见重组BSOD 的精确分子量为18 685.42 Da;与理论分子量18 707.83 Da 相比,相差仅22 Da。
进一步对重组BSOD 酶解后开展串联质谱分析,共分析三个肽段,分别为1 064.486 7、1 335.648 7 和1426.6383;3 个肽段的序列分析结果分别为“-YGSQEVLIR-”、“-AIVIHAGVDDLGR-”和“-NSVYGQLNLYQK-”(图8),分别对应重组BSOD 分子序列中第42~50、136~148 和30~41 肽段(图1);表明重组蛋白的序列正确,表达成功。
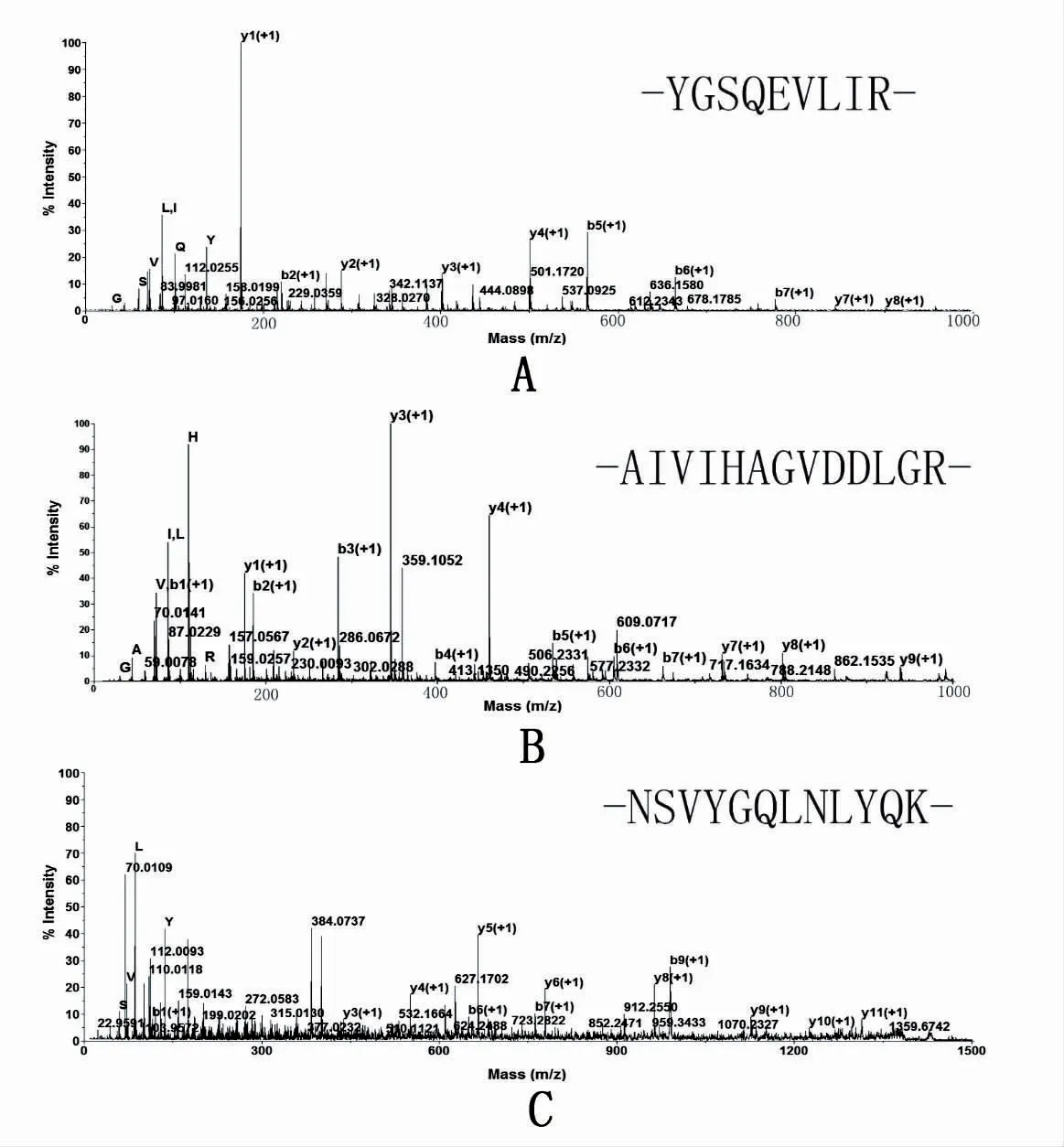
图8 重组BSODLC-MS/MS 质谱分析图Fig.8 MS/MS spectrum of recombinant BSOD
2.4 重组BSOD 的功能分析
采用总SOD 活性检测试剂盒(WST-8 法)(货号S0101,碧云天公司)鉴定重组BSOD 的酶活性。分别检测重组BSOD、鳌合Cu/Zn 离子后的重组BSOD 及SOD 标准品的酶活力,并进行比较(结果如图9A),可见经Cu/Zn 离子鳌合后的重组BSOD,与重组SOD 和SOD 标准品相比,具有更强的酶活力。进一步采用DPPH 法鉴定重组BSOD 的自由基清除率,结果如图9B。可见重组BSOD 具有较明显的自由基清除能力,但其清除率明显低于维生素C(P<0.01,n=3);而经Cu/Zn 离子鳌合后,重组BSOD 的自由基清除率明显上升且强于维生素C(P<0.01,n=3)。测试不同温度下重组BSOD 的酶活力变化,由图9C 可见,重组SOD 以及Cu/Zn 离子鳌合后的重组BSOD 的最佳酶活力温度均在30 ℃左右;而在70 ℃条件下,重组BSOD 以及Cu/Zn 离子鳌合后的重组BSOD 处仍然保留约50%的酶活力,表明BSOD 具有较好的热稳定性。测试重组BSOD 在不同pH 条件下的酶活变化,发现重组BSOD 以及Cu/Zn 离子鳌合后的重组BSOD 酶活力的最佳pH 在4 左右(图9D)。
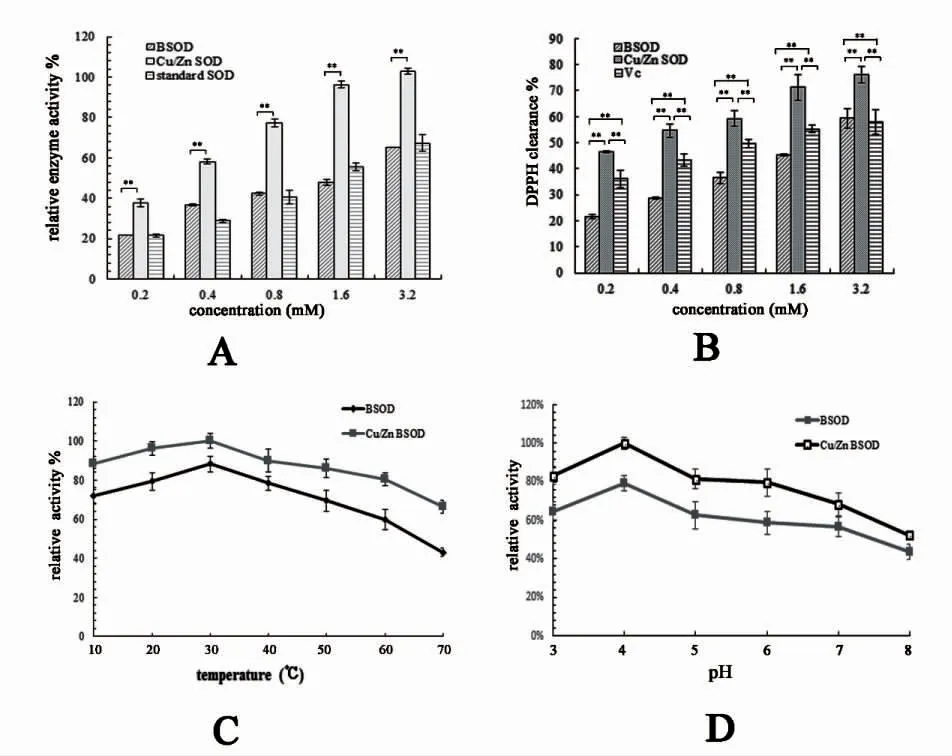
图9 重组BSOD 的功能分析Fig.9 Functional analysis of recombinant BSOD
3 讨论
SOD 作为一种广泛分布于生物体内的抗氧化剂,也是生物用以对抗活性氧伤害的最重要防线。目前,已报到的SOD 中,Cu/Zn-SOD 主要存在于真核生物,其活性形式为同源二聚体[22-23]。早在1975 年,第一种Cu/Zn-SOD(来自小牛血清)的空间结构被解析。Cu/Zn-SOD 的空间结构包含一段由8 个beta-折叠片组装成希腊钥匙(Greek Key)型折叠桶结构,其铜和锌离子的结合位点由6 个组氨酸残基和一个天冬氨酸残基所组成[23]。Mn/Fe-SOD 主要存在于原核生物以及真核生物的线粒体和叶绿体内[24],其中,Fe-SOD 主要以二聚体形式发挥功能,而Mn-SOD 则以二聚体或者四聚体形式发挥功能;而Ni-SOD 则主要存在于原核细胞,其空间结构中含有一个由右手螺旋组成的六聚体结构,每一个右手螺旋的N 端可以鳌合一个镍离子,其鳌合镍离子的活性位点由His-Cys-X-X-Pro-Cys-Gly-X-Tyr 肽段所组成[25]。厚壳贻贝BSOD 与其他物种的Cu/Zn-SOD 具有较高的序列相似性,但其氨基酸一致性最高也仅为61%,表明厚壳贻贝BSOD 与其他物种的Cu/Zn-SOD 仍具有较大的氨基酸差异,推测与厚壳贻贝BSOD 需适应贻贝足丝的所处环境和黏附性能有关。从进化树分析结果来看,厚壳贻贝BSOD 主要与来自甲壳类的红螯螯虾Cherax quadricarinatus 和信号小龙虾Pacifastacus leniusculus 的细胞外Cu/Zn-SOD,以及克氏原螯虾Procambarus clarkii 的Cu/Zn-SOD 亲缘关系较近,而与贝类已报到的Cu/Zn-SOD 的亲缘距离反而较远(图5),可能是由于目前数据库中尚无其他贝类的足丝SOD 数据有关,同时也暗示着,厚壳贻贝BSOD 与贝类的其他SOD 分子可能在进化上发生了较大的偏移。
厚壳贻贝BSOD 分子序列中含有4 个半胱氨酸,与其他物种的SOD 相比,额外多出2 个半胱氨酸(图2),推测厚壳贻贝BSOD 可能形成2 对二硫键,因而与其他物种的Cu/Zn-SOD 相比具有更好的稳定性。考虑到足丝本身是一种负责黏附的非细胞组织,其所处环境又是直接暴露于富含各种氧化因素,如溶解氧和高价位金属离子的海水中,因此,BSOD 面临的是比细胞内更为复杂和严苛的环境,因而在结构和功能方面发生了适应性进化,且可能在二硫键分布以及空间结构上与其他贝类已报道的Cu/Zn-SOD 存在差异。此外,厚壳贻贝BSOD 分子成熟肽序列中含有较为丰富的组氨酸残基。根据厚壳贻贝BSOD 与其他物种Cu/Zn-SOD 的多序列比对结果(图4),我们发现BSOD 的活性位点中,6 个组氨酸以及3 个天冬氨酸与其他物种的Cu/Zn-SOD 具有较高的保守性,考虑到Cu/Zn-SOD 与Cu/Zn 离子的结合位点主要由组氨酸和天冬氨酸组成[23],因此,厚壳贻贝BSOD 也可能具有Cu/Zn 离子的螯合能力。这一点在重组BSOD 的功能分析中被初步证实(图9)。与已知的贻贝足丝抗氧化蛋白MFP-6 相比,BSOD 与其在序列上差异较大。MFP-6 是一种富含半胱氨酸的足丝蛋白,且其半胱氨酸多数未形成二硫键,因此其侧链巯基处于游离状态,因此,推测MFP-6 的抗氧化活性主要来自于其半胱氨酸残基的游离巯基[14-15],而BSOD 的抗氧化活性则可能来自其SOD 结构域及其螯合的二价金属离子。
通过密码子优化结合大肠杆菌原核重组表达体系,成功表达出厚壳贻贝BSOD 蛋白分子。采用密码子优化策略能改善目标蛋白在大肠杆菌表达体系中的表达效率[26]。从表达结果来看,重组BSOD 可能形成二聚体和三聚体(图7B),这与其他物种的Cu/Zn-SOD 类似[23]。根据western blot 检测结果以及质谱测序,发现所表达的重组BSOD 序列中带有多聚组氨酸标签,且质谱测得的肽片段序列与表达目标蛋白的序列一致(图8),表明表达成功。但重组BSOD 在大肠杆菌中主要以包涵体形式表达,且低温(16 ℃)诱导表达并未改善表达产物的可溶性(图7)。包涵体表达虽然有助于防止蛋白降解并有助于后续的纯化,但是由于包涵体表达的蛋白是变性蛋白,因此,在测试其活性前,必须采取氧化复性的形式以便获得折叠正确的蛋白质分子。为此,采取稀释复性法完成对重组BSOD 的复性。复性后的重组BSOD 表现出较强的酶活力和自由基清除能力,且该活性在鳌合了Cu/Zn 离子后,明显增强。表明BSOD 在发挥活性时需要二价金属离子的辅助。同时,重组BSOD 具有较好的热稳定性,在70 ℃条件下,重组BSOD 仍保留约50%的酶活(图10B)。
4 结论
厚壳贻贝BSOD 是一种新型的SOD 分子。通过原核重组表达体系结合密码子优化策略,成功获得BSOD 表达产物,重组BSOD 在复性以及经Cu/Zn 离子鳌合后,具有较强的酶活性和DPPH 清除能力,同时也具有较强的热稳定性,上述研究结果表明,厚壳贻贝BSOD 分子是一种Cu/Zn-SOD,其较强的酶活力和较好的热稳定性为后续基于厚壳贻贝BSOD 的蛋白质工程改造奠定了基础。
