基于线粒体DNA D-Loop 区分析我国沿海厚壳贻贝群体遗传结构
2020-12-26
(浙江海洋大学海洋科学与技术学院,国家海洋设施养殖工程中心,浙江舟山 316022)
厚壳贻贝Mytilus coruscus 隶属软体动物门Mollusca、双壳纲Bivalvia、贻贝目Mytioida、贻贝科Mytilidae、贻贝属Mytilus[1],主要分布于日本北海道海域、韩国济州岛海域、中国的渤海、黄海、东海以及台湾海域,厚壳贻贝壳顶尖细,壳面常呈棕褐色。厚壳贻贝生活在低潮线以下盐分高、海流大的浅海中,其垂直分布深度较深,达20 m 左右,以10 m 左右密度最大,幼贝分布较浅[2]。厚壳贻贝苗种人工繁育已经成功进行,其过程主要包括:亲本贝在适宜水温的条件下完成受精,受精卵开始进行分裂分化等过程,最后变态发育成稚贝,其胚胎发育为成体的全过程历时约35 d[3]。厚壳贻贝与海洋中许多无脊椎动物相类似,发育中的幼虫会自行选择适宜生存的栖息地点并完成它的附着过程[4]。厚壳贻贝个体大、生长快,肉味鲜美、营养丰富,且具有滋补保健作用,在国内外市场深受欢迎,价格远高于贻贝和紫贻贝,为出口创汇产品[5]。我国厚壳贻贝主要养殖区在浙江舟山,特别在嵊泗县是最主要海水养殖品种,其养殖生产已有十余年历史,目前每年养殖数量达50 亿~60 亿粒以上[6]。厚壳贻贝作为我国重要的水产养殖贝类品种之一,种质资源和野生遗传结构的研究分析对提升种源的品质和养殖贝的质量具有一定的参考价值,也可帮助当地的渔业水产了解厚壳贻贝的野生资源情况,有效保护野生种质资源,提升厚壳贻贝养殖产量。
线粒体DNA(mitochondrial DNA,简称mtDNA)相对于核DNA 具有结构简单、母系遗传、进化速率快、几乎不发生重组、富含系统发育信号等特点,是作为种属进化研究的良好标记[7]。其中,线粒体DNA DLoop 区序列由于不编码蛋白质而不受自然选择的影响,因而进化速率最快,积累了较多的突变,如碱基替换、插入、缺失,以及众多的串联重复序列等,所以在种群遗传学分析中得到普遍应用[8-9]。
本研究对我国沿海6 个厚壳贻贝野生群体的mtDNA D-Loop序列进行PCR 扩增和测序,开展关于群体间遗传变异和系统分化等方面研究,旨在为我国厚壳贻贝野生种质资源的合理开发和保护提供分子生物学依据,并为制定合理的育种方案提供数据参考。
1 材料与方法
1.1 样品采集和DNA 提取
实验所用的厚壳贻贝于2016 年3 月至12 月之间采集于青岛(QD)、舟山(ZS)、玉环(YH)、台山(TS)、平潭(PT)、厦门(XM),每个地点随机采样活体解剖取闭壳肌(图1、表1),保存于无水乙醇中,用于DNA 提取。采用改良盐析法提取基因组DNA[10],并溶解于TE缓冲液中,琼脂糖凝胶电泳检验其纯度后-20 ℃保存备用。
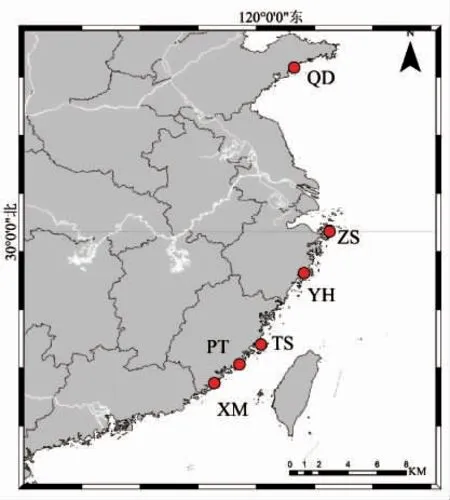
图1 我国沿海6 个地区野生厚壳贻贝样本采样地点Fig.1 Map showing six sampling locations of wild M.coruscus along the China Sea
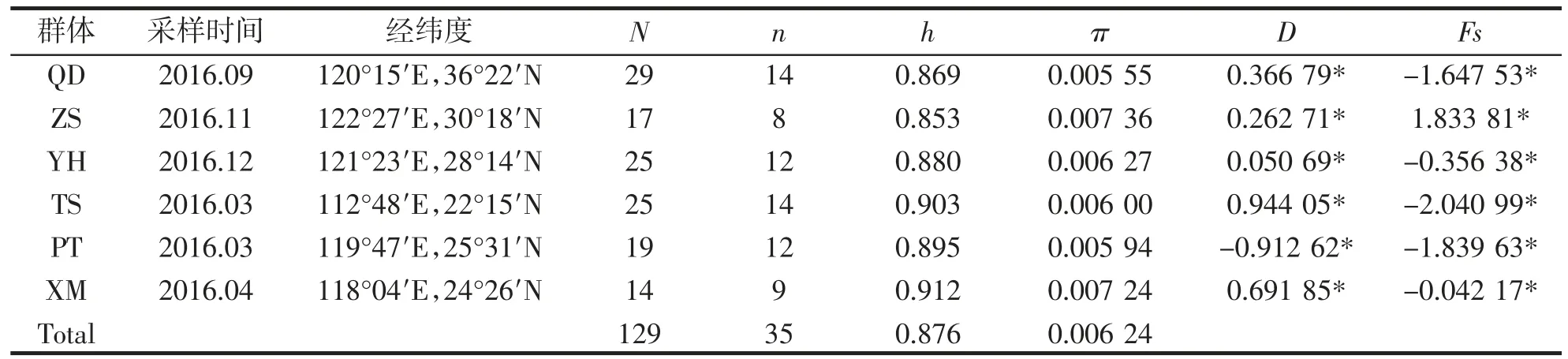
表1 厚壳贻贝的样品细节及核苷酸组成以及参数指数Tab.1 Sample details,nucleotide composition and parameter indices for M.coruscus
1.2 目的片段的扩增与测序
利用NCBI 数据库下载厚壳贻贝线粒体全基因组数据(GenBank 登录号:KJ577549)的D-Loop 序列全长,并使用Premer-primer6.0[11]设计引物,D-Loop-F:CGGTAGGAAGAAGTAGAGT;D-Loop-R:CTTGCTCACCAGGAACAT,引物合成由杭州擎科梓熙生物技术有限公司完成。PCR 反应体系为25 μL,包括:12.5 μL ExTaq (Takara),1 μL 前后引物(浓度0.1 OD),0.5 μL 模板DNA 以及10 μL 去离子水。PCR 扩增程序:94 ℃预变性2 min;94 ℃变性45 s,52 ℃退火45 s,72 ℃延伸3 min,33 个循环;72 ℃终延伸10 min,4 ℃保存。将PCR 产物置于1.5%琼脂糖凝胶中电泳检测产物质量,确认条带大概长度。将符合目的片段长度的PCR 产物冷冻封存后,由杭州擎科梓熙生物技术有限公司对PCR 产物进行双向测序,获取产物序列信息。
1.3 数据处理与分析
通过ClustalX 2.0[12]对测序获得的线粒体DNA D-Loop 区序列进行比对矫正,并对齐全部序列片段。为鉴定物种和所得片段的准确位置,使用NCBI 数据库中在线BLAST 工具(https://blast.ncbi.nlm.nih.gov/Blast.cgi)对所有片段进行在线比对。将比对矫正后的序列以FASTA 格式导入软件MEGA 7.0[13]进行序列比对及变异分析,计算群体间的遗传距离并使用邻接法构建系统发育树。使用软件DnaSP 6.0[14]对所有数据根据其群体进行分组,并计算各类遗传多样性参数,如:单倍型个数(n)、变异位点、单倍型多样性(h)和核苷酸多样性(π)。使用软件Arlequin 3.5[15]进行分子方差分析(AMOVA)、计算遗传分化系数(FST)、基于Tajima’s D 与Fu’s Fs 模型下,对数据进行中性检测。将所有单倍型在软件MEGA7.0[13]中进行计算并构建系统发育树。使用软件Network5.1[16]基于中介邻接法(median-joining method)构建各分子标记的单倍型网络图。
2 结果
2.1 群体内遗传变异
在对129 个样本D-Loop 区序列进行测序分析后,获得了长度为1 113 bp 的片段,其中变异位点31个(表2),序列组成显示A、T、G、C 碱基含量分别为A:32.8%、T:31.4%、C:13.4%、G:22.4%,A+T 含量(64.2%)显著高于G+T 含量(35.8%),具有明显的碱基组成偏倚性。共得35 个单倍型(GenBank 登录号:MG888576-MG888610)(表3)。单倍型Hap3、Hap5 和Hap11 为6 个群体共享,其中Hap11 和Hap5 为优势单倍型,分别占个体总数的30.23%和13.95%。单倍型Hap1、Hap15 和Hap18 为4 个群体共享,单倍型Hap9 为3 个群体共享,单倍型Hap4、Hap10、Hap13、Hap14、Hap17 和Hap28 为2 个群体共享,其余都为单个群体所特有单倍型。厚壳贻贝4 个地理群体的遗传学参数见表1,单倍型多样性指数为0.853(ZS)~0.912(XM),群体总体的单倍型多样性指数为0.876;核苷酸多样性指数为0.005 55 (QD)~0.007 36(ZS),群体总体的核苷酸多样性指数为0.006 24,遗传多样性处于较高水平。
2.2 群体遗传结构研究
分子方差分析(表4)结果显示,100.15%的变异都来自于群体内部。群体间的遗传分化系数FST值在在-0.021 79(QD-PT)至0.076 3(PT-XM)之间,15 个FST值中由9 个均为负值,其余的除(PT-XM)外,也都小于0.05,这表明各个群体之间几乎没有发生遗传分化,存在高度的遗传同质性。基因流范围在3.026 54~32.429 74 之间,说明各群体之间基因交流频繁(表5)。中性检验结果显示,Tajima’s D 值以及Fu’s Fs 值的P 值均不显著,表明厚壳贻贝未偏离中性选择,群体没有经历过群体扩张(表1)。
用MEGA5.0[13]构建6 个群体单倍型系统发育树如图2,从图中看出35 个单倍型样本聚类为两支,6 个群体的单倍型在两支均有出现,并未发现存在群体分化的差异现象。
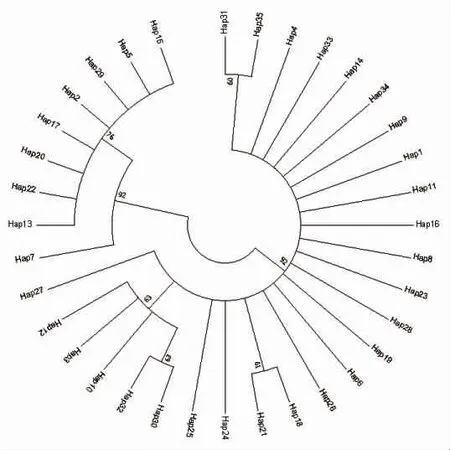
图2 自检举值为1 000 次的单倍型邻近系统发育树(phylogenetic trees)Fig.2 The NJ tree of each haplotypes based on Nei's standard genetic distance with 1 000 bootstraps
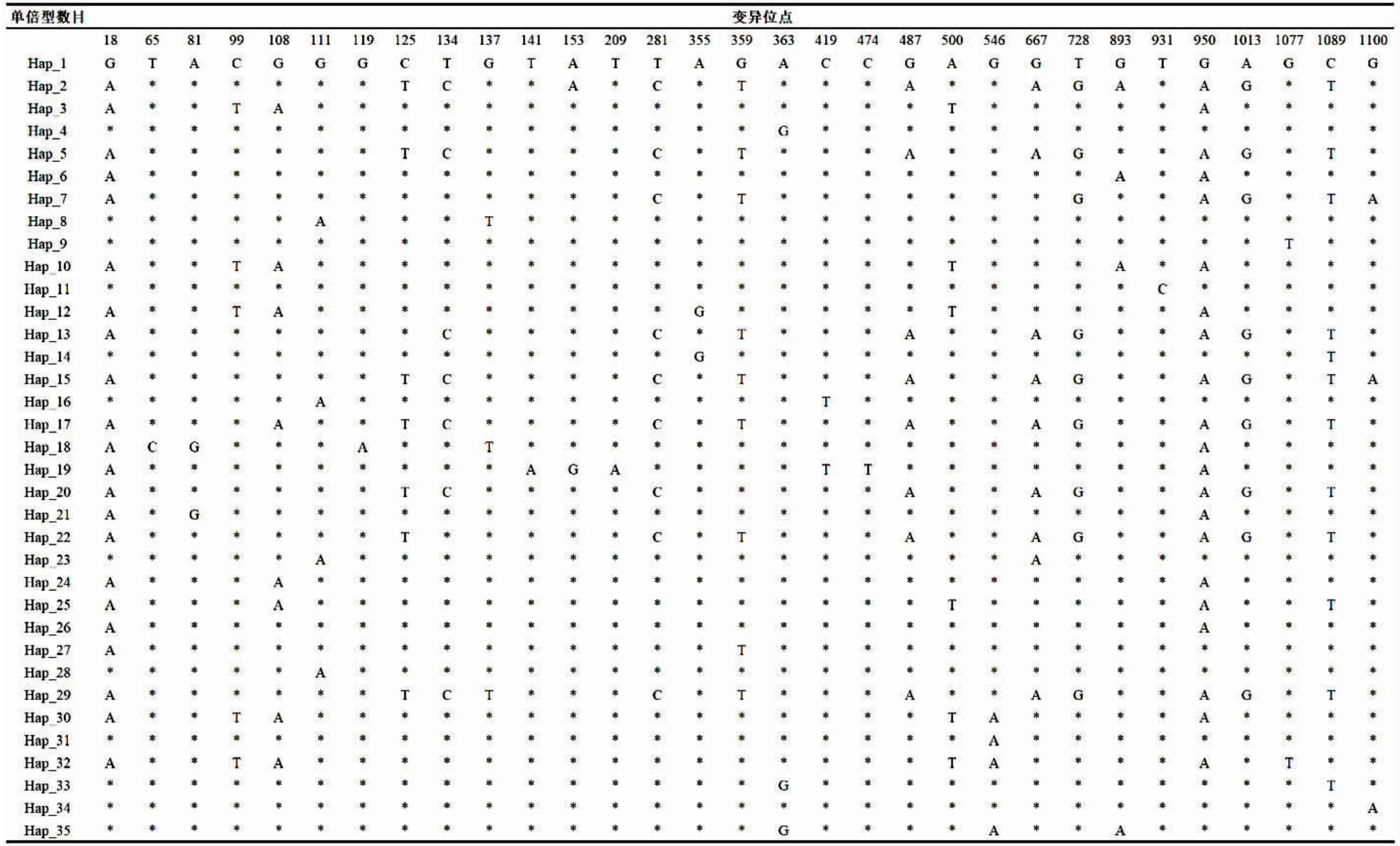
表2 所有样本的单倍型数目以及每个单倍型的变异位点Tab.2 The details of haplotypes,including the numbers of haplotypes and mutation sites
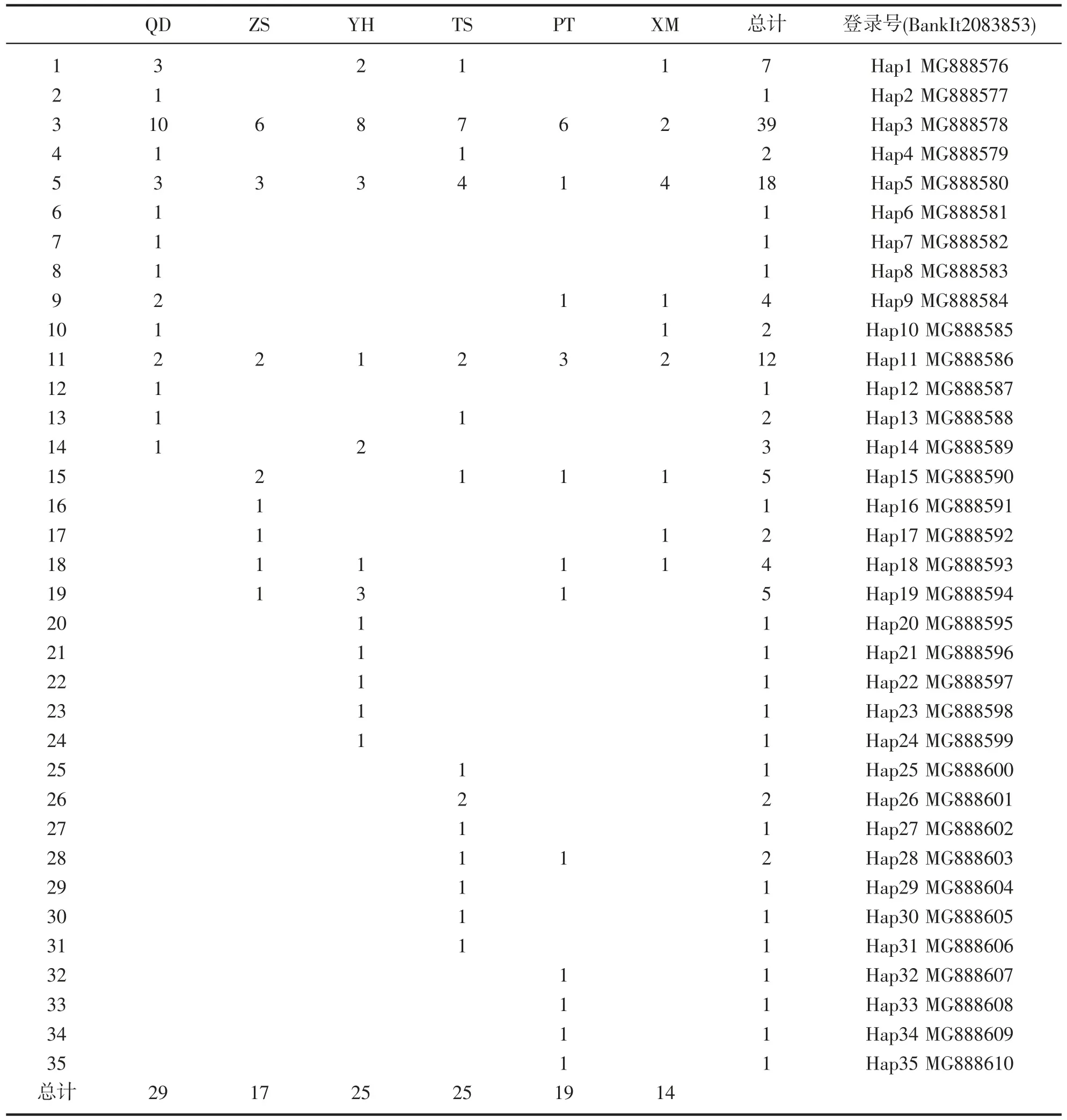
表3 厚壳贻贝群体的单倍型数量及登录号Tab.3 Haplotype and accession numbers of M.coruscus populations
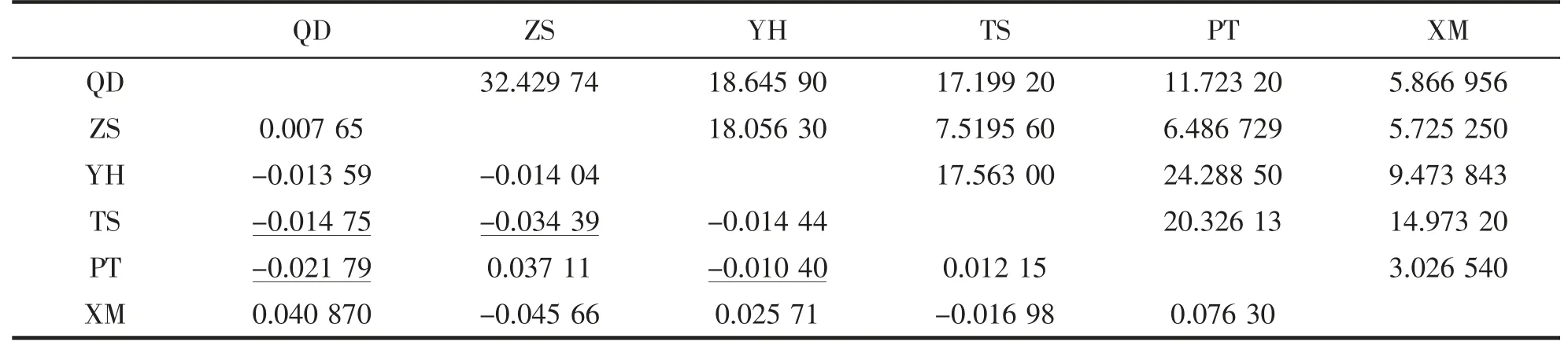
表4 基于线粒体DNA D-Loop 区域的厚壳贻贝样品之间的成对FST 值与Nm 值Tab.4 Pairwise FST and Nm values among samples of M.coruscus using mtDNA D-Loop region
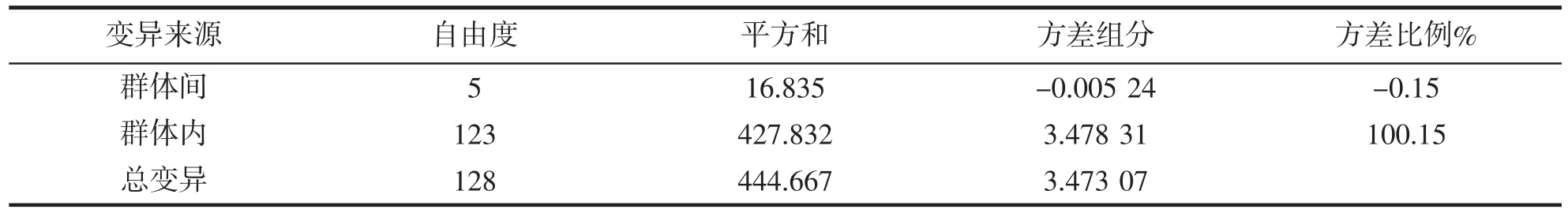
表5 基于线粒体DNA D-Loop 区域的厚壳贻贝样品之间的AMOVA 分析Tab.5 AMOVA among samples of M.coruscus based on mtDNA D-Loop region
构建单倍型网络图如图3,结果显示单倍型网络图Hap3 和Hap5 是最主要的单倍型,单倍型聚类并未完全展示地域性特色,各地理群体的单倍型具有聚类现象,显示出较近的亲缘关系,结果与单倍型系统发育树(图2)情况相符。
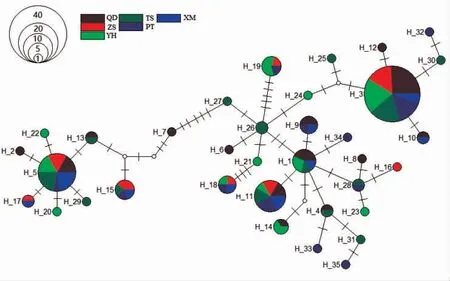
图3 6 个地区样本的线粒体DNA D-Loop 区单倍型进化网络图Fig.3 The haplotypes network of six populations of M.coruscus
3 讨论
遗传多样性参数是衡量群体遗传多样性程度的重要指标之一[17]。本研究中6 个厚壳贻贝群体的遗传多样性均表现出高的单倍型多样性和低的核苷酸多样性的特点,显示出了较高水平的遗传多样性。这与前人利用线粒体COI[18]、COIII[19]及16sRNA[20]研究我国东南沿海厚壳贻贝遗传多样性的结果一致。厚壳贻贝体外受精、大规模的繁殖能力以及广泛的幼虫传播方式可能是导致厚壳贻贝资源拥有高遗传多样性的原因。
我国沿海厚壳贻贝整体遗传结构呈现单一薄弱的情况,6 个群体间的遗传分化程度不明显,各样本的遗传没有发现明显的遗传分化现象,结果与单倍型网络图的结果相吻合,从AMOVA 中可以发现,大部分遗传变异来自于样本内部,样本间的变异程度极小,这也是导致样本,不具有明显遗传分化现象的因素之一。单倍型构建的进化树虽然聚类为2 个主要分支,但分支内各单倍型不具有地理分布相吻合的特点,呈现混杂,分支呈现嵌套模式,说明单倍型的遗传歧化不明显。
一般而言,海洋生物与陆生生物相比,其种群遗传结构和遗传分化相对匮乏,这种匮乏缘于海洋环境缺乏像陆地环境一样阻止生物扩散和交流的有效屏障[21]。大多数海洋双壳类都拥有一段或长或短的浮游期,在此期间浮游幼虫可以被动地随着海洋潮流进行有效扩散传播[22-24]。浮游期过后,这些贝类大都营固着生活(如贻贝、牡蛎等)或进行小范围活动(如蚶、蛏、蛤等滩涂性贝类)。因此,海洋双壳类的遗传结构主要形成于其浮游期。一般而言,拥有较长浮游期的物种能够在海洋潮流的推动下漂移较远的距离,更易产生遗传交流,造成单一的遗传结构[25]。我国沿海厚壳贻贝整体遗传结构呈现单一薄弱的情况,7 个群体间的遗传分化程度不明显,各样本的遗传没有发现明显的遗传分化现象,结果与单倍型网络图的结果相吻合,从AMOVA 中可以发现,大部分遗传变异来自于样本内部,样本间的变异程度极小,这也是导致样本,不具有明显遗传分化现象的因素之一。单倍型构建的进化树虽然聚类为两个主要分支,但分支内各单倍型不具有地理分布相吻合的特点,呈现混杂,分支呈现嵌套模式,说明单倍型的遗传歧化不明显。这可能是由于厚壳贻贝与大多数的海洋双壳动物一样,在其生命发育周期中会有以浮游方式生存的幼虫阶段,大约需要约35 d[3],在较长的浮游期内,幼体可以跟随洋流漂浮到远离其出生地的环境,甚至是其他群体的栖息地,这也就增加不同地区群体间的基因交流,降低不同群体之间的遗传差异,缩小了遗传分化的现象。
除此之外,我国沿海洋流构成十分复杂,除了数条沿岸流外,还有对本地区有重要影响的长江冲淡水、黑潮的支流、黄海的中央冷水团、东海的局部涡流等[26],东海海区历来是我国厚壳贻贝的主要产区,自然种质资源丰富,在每年冬季的繁殖季节会有大量幼虫被散播在海区中;而冬季海区沿岸流主要沿北向南流动,黑潮及其支流暖流在外围由南向北隔离了边缘海与大洋间的交流,冬季同样是我国沿岸江河的枯水期,从河口流入边缘海的冲淡水大幅减少,这也进一步减弱对沿岸流的影响,而各海区内存在的涡流也促进了海区内环流的循环[27]。作为一种经济海洋养殖贝类,厚壳贻贝的养殖市场广阔,在人工育苗技术相对成熟的情况下,养殖从业者一般会从主产区中购买苗种后,运输到养殖地进行养殖,而目前主要养殖方式是在自然海区利用附着基进行附苗养殖,开放的养殖方式会导致在繁殖季节,大量养殖样本的幼虫会被散播在海区内,与自然样本的幼虫混合后随洋流传播[28]。养殖样本亲本本身也是来自于单一的自然样本,而大量的养殖样本幼虫的混入会导致遗传多样性的降低和样本间遗传结构的减弱,而目前育苗技术相对原始,选苗资源单一,也导致了养殖样本近亲交配情况凸显。在养殖业缺乏对自然群体保护的管理下,大量养殖样本的幼虫势必会冲淡自然样本的遗传多样性,进而对自然样本的遗传结构产生冲击。
4 小结
厚壳贻贝的群体遗传学研究是更深入了解和揭示其自然环境下遗传现状,是识别厚壳贻贝遗传管理单元合理利用其资源的必要途径。本研究采用线粒体DNA D-Loop 区序列对采自我国沿海的7 个厚壳贻贝群体进行遗传多样性和遗传结构分析,结果显示出较高的遗传多样性水平和单一薄弱的遗传结构。通过本研究的相关的实验和分析,已经积累了相当的厚壳贻贝遗传数据。而通过这些厚壳贻贝遗传数据的分析,可以为我们在某些研究方面提供一些见解,比如遗传管理单元的识别,种质资源的有效利用,也有助于我们理解在同一地区下,具有相似浮游幼虫期的海洋贝类的遗传结构。
