多环境下大豆全生育期的QTL定位
2020-12-10张佳南王艳殊许世超李文霞宁海龙
张佳南,王艳殊,许世超,田 雨,李文霞,宁海龙
(东北农业大学 农学院,大豆生物学教育部重点实验室,农业部东北大豆生物学与遗传育种重点实验室,黑龙江 哈尔滨 150030)
【研究意义】大豆的生育期性状是决定生产的重要性状之一,对产量、品质至关重要。育种实践也表明,改变大豆生育期结构,能够增加产量。一般来说,生育期较长的大豆品种,产量较高,所以,研究大豆生育期的遗传规律有助于更加清楚地了解产量形成的机制。大豆生育期也是确定大豆生态适应性的重要性状,了解其遗传规律可以指导品种的合理选择和不同积温区的生态布局。为加快育种进程,分子标记技术被广泛应用于大豆生育期QTL定位育种研究中[1-2],利用分子标记构建的遗传连锁图谱对大豆生育期相关性状进行QTL分析,可以在分子水平上阐述控制大豆生长发育相关基因系统的表达规律,大大促进了数量性状的遗传研究。【前人研究进展】近年来,国内外关于大豆生育期QTL定位的报道已有很多。截至2015年,共定位482个生育期相关的QTL(http://soybase.org)。但是以往研究有3方面的不足。一是国内外定位生育期相关的QTL数量虽多,但多是运用单一环境进行研究。由于大豆生育期性状遗传为数量遗传,受多基因控制,很容易受到光照长度和温度等环境的影响,因此应通过多环境联合分析,可定位到多环境共表达的稳定QTL和某一环境特意表达的QTL。二是大多数研究只将整个生育期分为前、中、后3个生育阶段,很少将发育进程细分小阶段后进行分析;三是大多数研究群体是由两亲本衍生的,两个亲本的杂交后代在进行连锁分析时,一个位点涉及只两个等位基因,检测效率低。而四向重组自交系群体是由4个亲本衍生的群体,多态性标记数量以及遗传图谱标记密度都会增加,分子标记多态性也更丰富,最主要的是基于一个基因位点同时分析4个复等位基因效应,从而提高QTL检测效率。为满足自花授粉作物的需要,四向重组自交系群体被提出并应用于遗传分析[3-4]。【本研究的切入点】本研究应用前期构建的四向重组自交系群体(FW-RIL),利用SSR分子标记构建遗传图谱,应用3个年份6个不同播期下的生育期结构数据进行QTL定位分析,检测不同年份、不同播期下稳定表达的全生育期QTL,并应用四向重组自交系群体多等位基因的优势,探寻有利于生育期性状改良的优异等位基因,进一步了解大豆生育期性状的遗传控制和环境调控规律。【拟解决的关键问题】为大豆育种中生育期性状的定量设计提供理论依据。
1 材料与方法
1.1 四向重组自交系群体的构建
应用生育期性状存在差异的4个大豆亲本,垦丰14(生育期120 d)、垦丰15(生育期116 d)、黑农48(生育期118 d)和垦丰19(生育期112 d)于2008年配制双交组合(垦丰14×垦丰15)×(黑农48×垦丰19),获得FW-F1世代,将FW-F1在哈尔滨和海南省三亚市崖州区连续自交6代,采用单粒传法获得FW-F2∶8四向重组自交系群体,包含160个株系,连同其4个亲本组成本试验材料。
1.2 田间试验与性状调查
田间试验采用随机区组设计,3行区。小区行长5 m,垄距65 cm,株距10 cm,3次重复,田间管理同一般大田栽培。2014年5月10日在哈尔滨香坊农场播种第1播期(14S1),同年5月20日进行第2播期播种(14S2);2015年5月10日在哈尔滨东北农业大学农院试验田进行第1播期种植(15S1),第2播期于5月20日进行播种(15S2);2016年5月7日在哈尔滨东北农业大学农院试验田进行第1播期种植(16S1),5月17日播种第2播期(16S2)。
田间记录生育期按Fehr和Carviness的大豆生育时期划分标准记载[4],调查各株系及亲本出苗后及时记录子叶期时间,之后进入始花期,始花期后每2~3 d到田间观察大豆群体发育动态,并及时记录生育期表型数据直到群体达到完全成熟阶段,数据记录完成。根据数据及生育期图谱进行QTL定位。调查各株系及亲本的出苗期(VE)、始花期(ER1)、盛花期(ER2)、始荚期(ER3)、盛荚期(ER4)、始粒期(ER5)、鼓粒期(ER6)、始熟期(ER7)和完熟期(ER8),并计算出本实验用于QTL定位分析的生育期各阶段的数值:盛花-始荚(R23)、始荚-盛荚(R34)、盛荚-始粒(R45)、始粒-鼓粒(R56)、鼓粒-始熟(R67)、始熟-完熟(R78)、始花-完熟(R18)的天数。
1.3 SSR标记分析
参照Gregan[5]等发表的大豆公共遗传图谱挑选引物,本研究初步挑选了638对SSR引物在4个亲本之间进行多态性筛选,其中有275对引物在4个亲本之间表现出多态性。根据Soybase(http://soybase.org)网站提供的大豆SSR序列合成引物并对4个亲本及FW-RIL群体进行PCR扩增。
PCR扩增体系:3 μl总DNA(50 ng/μl)+3 μl引物(100 nmol/μl)(包括上游引物和下游引物)+0.3 μl dNTP(10 nmol/μl)+2 μl10×缓冲液+0.2 μlTap酶(5 U/μl),用超纯水定容至20 μl。
PCR扩增条件:94 ℃预变性10 min,进入循环:94 ℃变性30 s;50 ℃复性30 s;72 ℃延伸30 s;循环38次后在72 ℃延伸5 min,于4 ℃保存。
电泳方法:每个PCR反应体系加上8 μl Loading Buffer,置PCR仪中变性10 min,然后放入冰上冷却。PCR产物在6 %的聚丙烯酰胺凝胶上分离,在1500W恒功率下电泳约1.5 h。
银染方法:在20 mL酒精(95 %)+10 mL冰乙酸+3 mL AgNO3+1500 mL蒸馏水中染色10 min,清水漂洗30 s后放入30 g NaOH+6 mL甲醛中显色+1500 mL蒸馏水显色5~10 min。
1.4 连锁图谱的构建
遗传图谱由东北农业大学教授宁海龙等[6]以4个亲本杂交衍生的160个FW-RIL群体(F8代)为材料,构建了一个基于四向群体的大豆遗传图谱。图谱包含275个SSR遗传标记,20个连锁群,在大豆基因组上总遗传距离3636.26 cM,平均图距15.47 cM。每个连锁群包含遗传标记6~0个,图距在49.36~319.02 cM。
1.5 数据分析方法
对不同环境条件下全生育期表型数据进行描述性分析,并使用SAS9.1软件进行方差分析对不同环境下的表型观测值进行联合方差分析,估算相应的方差分量,通过环境方差、基因型方差、基因与环境互作以及误差方差和总体方差进行遗传率的估计,遗传率估计的计算方法如下:
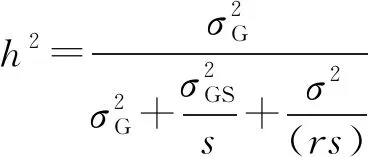

用QTL(GAPL V1.2)软件进行QTL定位,采用完备区间作图法、简单区间作图法和排列法进行加性效应分析,对每个性状分别进行1000次排列测验(permutation test),以确定每个性状的LOD临界值阈值(显著水平0.05)。
2 结果与分析
2.1 表型数据分析
FW-RIL群体各生育期性状的平均值、标准差、变幅、偏度及峰度列于(表1)。FW-RIL群体的各个生育期阶段存在较大的变异,不同年份和播期下各阶段差异同样显著、偏度及峰度均小于1,表明各阶段生育期性状的表型值基本符合正态分布,表示存在多数微效基因,因此,可用于多年多环境下生育期性状QTL的定位。
2.2 FW-RIL生育期阶段的方差分析和遗传率估计
方差分析结果表明(表2)FW-RIL群体的生育期性状在播期、家系以及播期与基因互作间存在极显著差异,表明生育期性状受播期影响、且与播期显著互作。各阶段生育期性状在不同年份下遗传率变现不同,表明可能存在年份与基因互作。
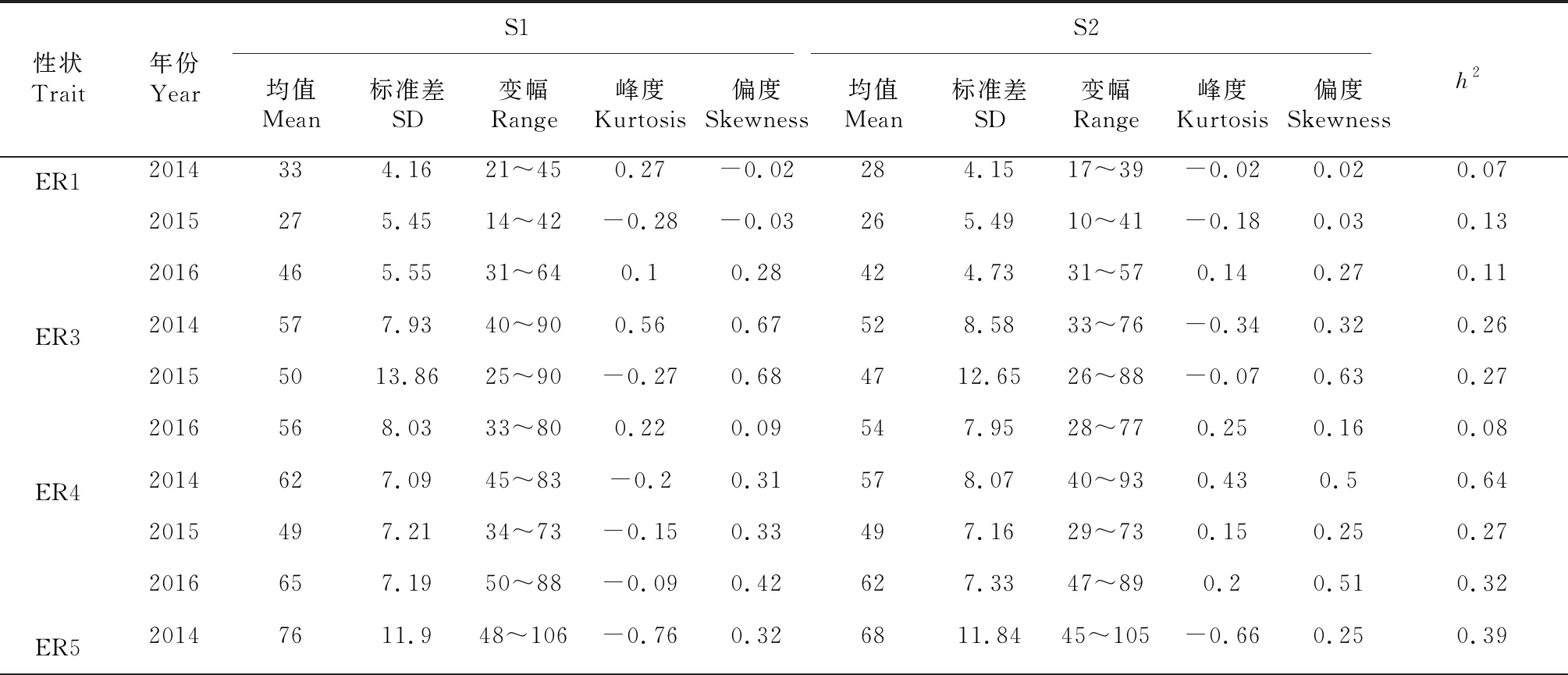
表1 不同年份下FW-RIL生育期各阶段的描述性分析
2.3 同一年份不同播期下生育期QTL定位
在本研究中,同一年份不同播期下重复检测到的生育期位点有18个(表3),分布在A1、C2、D2、F、K、O、G、B1、D1b、I、M、N,连锁群上。LOD值变化范围2.51~46.30。PVE变化范围0.43 %~11.33 %,多数为微效基因,来自亲本KF14正向加性效应等位基因型分别有3个,负向加性效应等位基因型分别有15个;来自亲本KF15正向加性效应等位基因型分别有6个,负向加性效应等位基因型分别有12个;来自亲本HN48正向加性效应等位基因型分别有6个,负向加性效应等位基因型分别有12个;来自亲本KF19正向加性效应等位基因型分别有7,负向加性效应等位基因型分别有11个。
2.4 不同年份下生育期QTL定位
在本研究中,不同年份下重复检测的生育期位点有14个QTL(图1),分布在AJ、N、C1、D1b、D2、K、L、O、A2连锁群上。LOD值变化范围2.60~10.84。PVE变化范围0.47 %~4.56 %,多数为微效基因,来自亲本KF14正向加性效应等位基因型分别有3个,负向加性效应等位基因型分别有11个;来自亲本KF15正向加性效应等位基因型分别有5个,负向加性效应等位基因型分别有9个;来自亲本HN48正向加性效应等位基因型分别有3个,负向加性效应等位基因型分别有11个;来自亲本KF19正向加性效应等位基因型分别有9,负向加性效应等位基因型分别有4个。
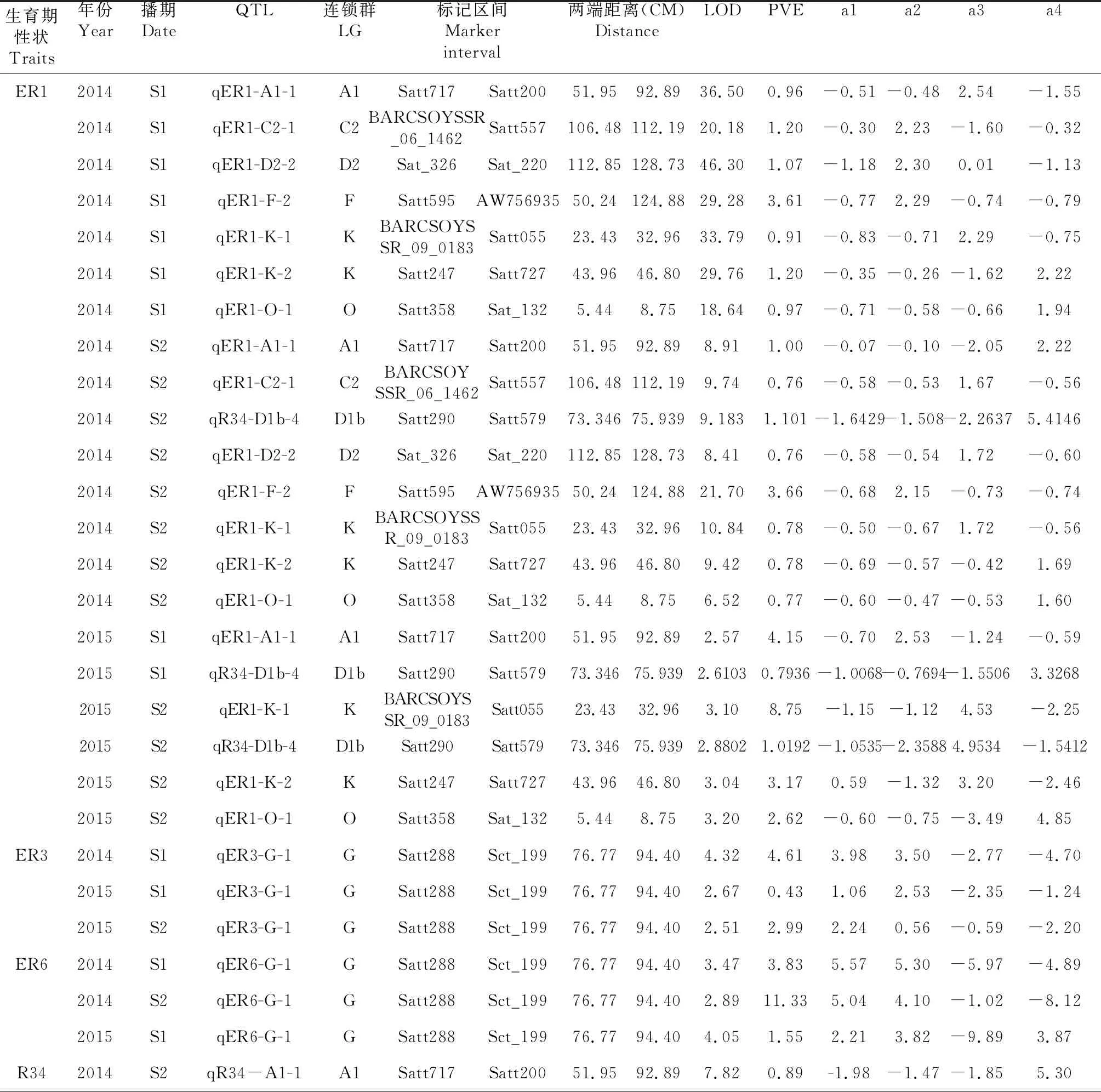
表3 相同年份不同播期下FW-RIL生育期各阶段QTL的重复定位
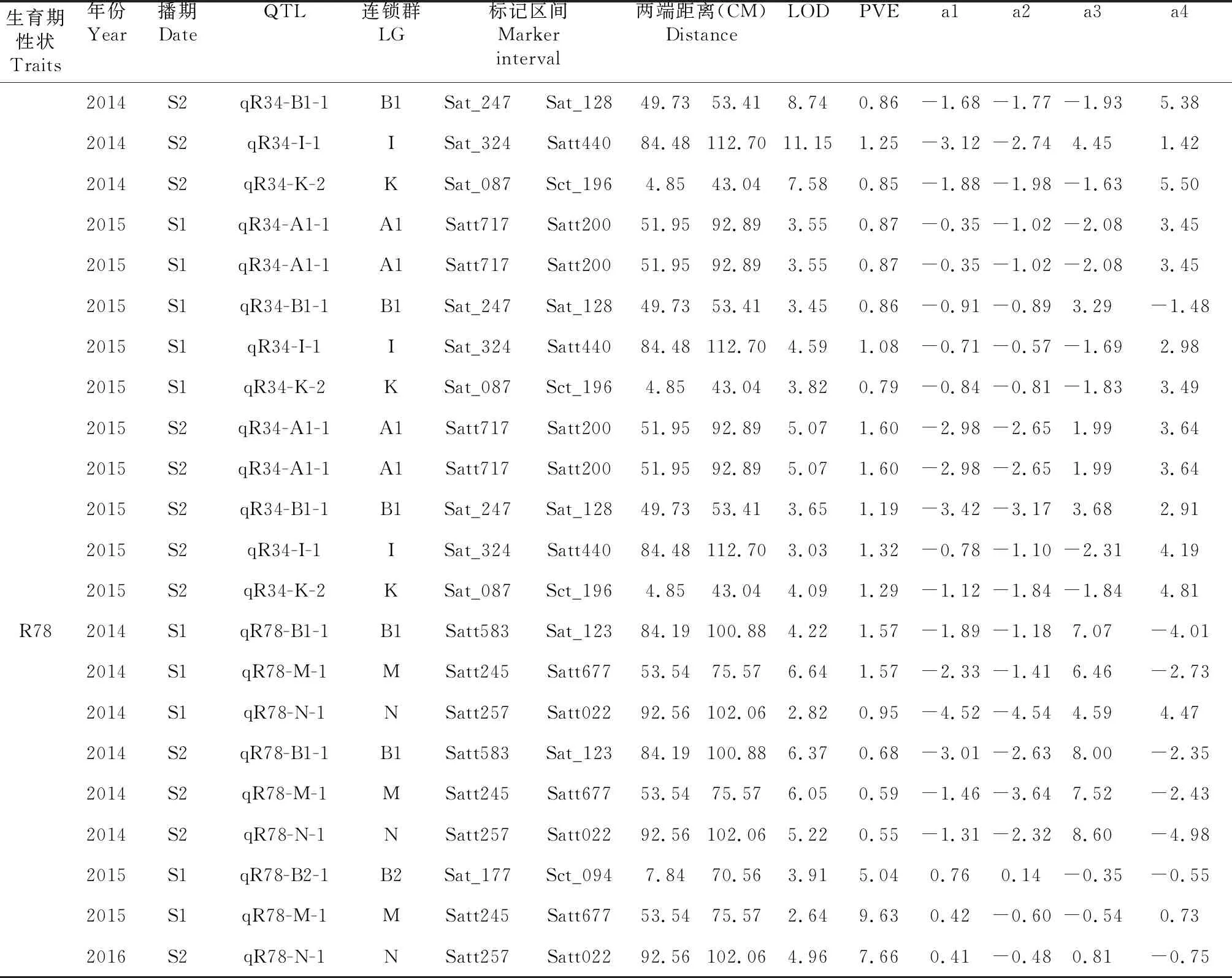
续表3 Continued table 3
3 讨 论
3.1 光周期顿感QTLs
本研究有18个在相同年份不同播期下重复检测位点(即光周期顿感QTLs),比较公共图谱发现有15个与前人发现QTL基因组相互重叠,其中4个QTL定位区间与已定位的光周期顿感QTLs区间相互重叠。qER1-C2-1在公共图谱的基因组位置为106.478(BARCSOYSSR_06_1462)~112.189 cM(Satt557),所在基因组区域与已经定位的6个成熟期、14个始花期、1个出苗期、1个营养期和1个生殖期和2个光周期顿感QTL[10-11,13-14,17,21,27-28,30,32,37]相互重叠;qER1-F-2在公共图谱的基因组位置为50.242(Satt595)~124.877 cM(AW756935),所在基因组区域与已经定位的1个成熟期、1个出苗期、2个生殖期和1个光周期顿感QTL[13,29,36]相互重叠;qER1-K-1在公共图谱的基因组位置为23.428(BARCSOYSSR_09_0183)~32.955 cM(Satt055),所在基因组区域与已经定位的2个成熟期、1个始花期1个光周期顿感QTL[17,25,29]相互重叠;qR34-K-2在公共图谱的基因组位置为4.853(Sat_087)~43.042 cM(Sct_196),所在基因组区域与已经定3个成熟期、2个始花期和1个光周期顿感QTL[17,25,29,35]相互重叠。这些位点在相同年份不同播期下重复表达,且定位区间与已定位的光周期顿感QTL区间相互重合,说明受播期环境影响较小,即为光周期顿感QTLs。
qER1-O-1在公共图谱的基因组位置为5.44(Satt358)~8.748 cM(Sat_132),所在基因组区域与已经定位的1个成熟期阶段QTL[35]相互重叠;qR34-B1-1在公共图谱的基因组位置为49.731(Sat_247)~53.412cM(Sat_128),所在基因组区域与已经定位的1个成熟期QTL[7]相互重叠;qER6-G-1、qR13-G-1、qER3-G-1标记区间相同,在公共图谱的基因组位置为76.77(Satt288)~94.403 cM(Sct_199),所在基因组区域与已经定位的8个成熟期和1个生殖期QTL[7-9,15]相互重叠;qR34-I-1在公共图谱的基因组位置为84.481(Sat_324)~112.7 cM(Satt440),所在基因组区域与已经定位的1个成熟期和个1出苗期QTL[48]相互重叠。qR78-B1-1在公共图谱的基因组位置为84.189(Satt583)~100.877 cM(Sat_123),所在基因组区域与已经定位的1个成熟期和1个生殖器QTL[10,35]相互重叠;qR78-N-1在公共图谱的基因组位置为92.558(Satt257)~102.055 cM(Satt022),所在基因组区域与已经定位的2个成熟期QTL相互重叠[15];qR78-M-1在公共图谱的基因组位置为53.538(Satt245)~75.571 cM(Satt677),所在基因组区域与已经定位的2个成熟期、1个始花期、1个营养期和1个生殖期QTL[9-10,13,19]相互重叠。qER1-K-2在公共图谱的基因组位置为43.955(Satt247)~46.796 cM(Satt727),所在基因组区域与已经定位的1个成熟期和1个营养期QTL相互重叠[10,24];qR34-D1b-4在公共图谱的基因组位置为73.346(Satt290)~75.939 cM(Satt579),所在基因组区域与已经定位的1个成熟期QTL[11,25]相互重叠。这些QTL虽然没有与已发现的光周期顿感QTL区间相互重叠,但与生育期QTL区间相互重叠,为光周期顿感QTL的发现提供理论依据与材料。
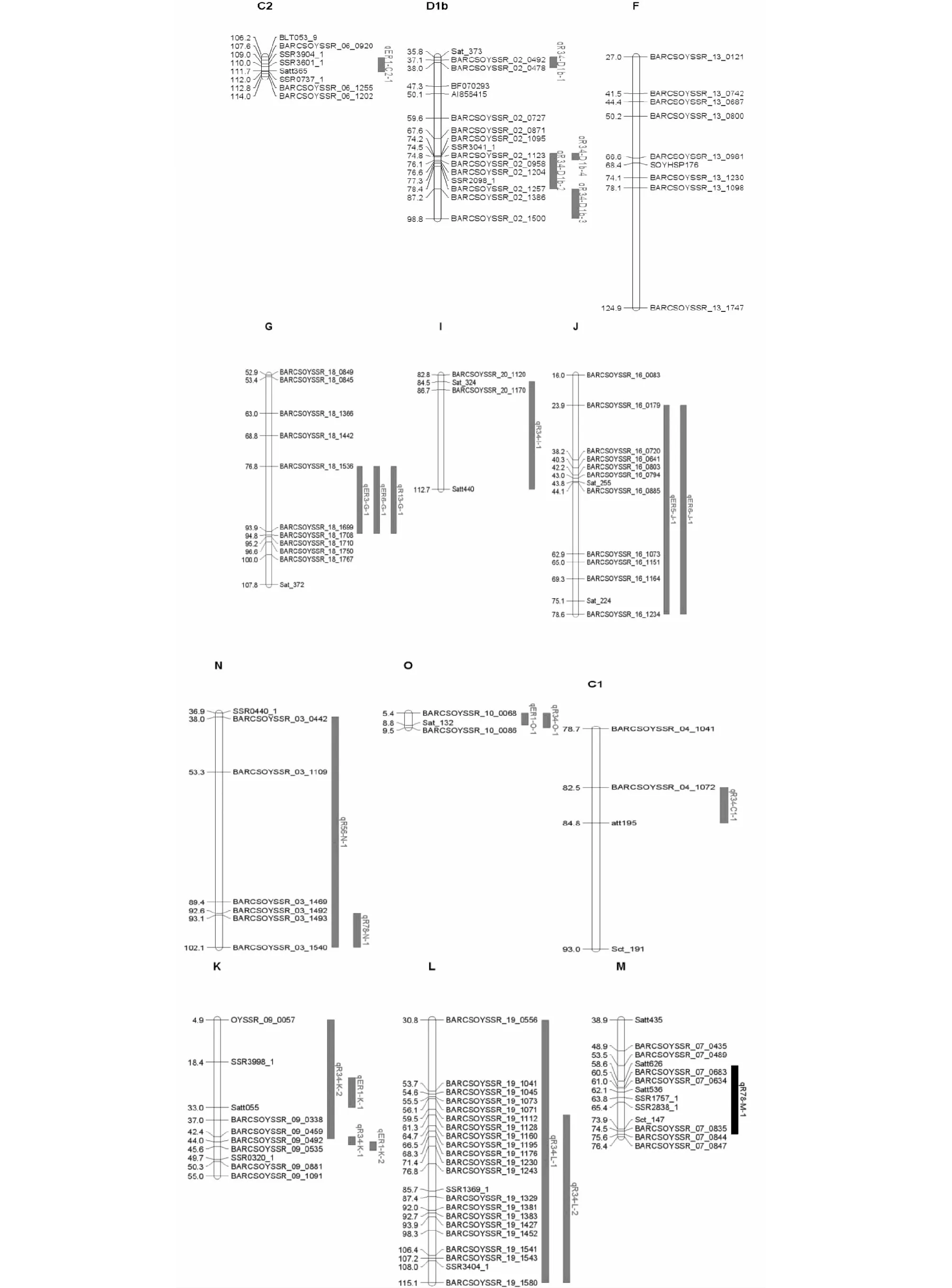
图1 不同年份下生育期QTL定位图Fig.1 QTL location of growth period in different years
3.2 不同年份重复定位QTLs分析
在不同(2年或2年以上)年份重复定位QTL(表)有14个,比较公共图谱发现有11个与前人发现QTL基因组相互重叠,qR34-C1-1在公共图谱的基因组位置为82.506(Sat_042)~84.809 cM(Satt195),所在基因组区域与已经定位的2个成熟期QTL[33]相互重叠;qR34-D1b-1在公共图谱的基因组位置为35.745(Sat_373)~40.041cM(Satt701),所在基因组区域与已经定位的1个成熟期QTL[35]相互重叠;qR34-D1b-2在公共图谱的基因组位置为73.346(Satt290)~87.204 cM(Satt546),所在基因组区域与已经定位的1个成熟期和3个始花期QTL[11,22,25]相互重叠;qR34-D1b-3在公共图谱的基因组位置为87.204(Satt546)~98.745cM(Satt703),所在基因组区域与已经定位的1个始花期QTL[34]相互重叠;qER5-J-1和qER6-J-1在公共图谱的基因组位置相同为73.346(Satt290)~87.204 cM(Satt546),所在基因组区域与已经定位的1个生殖期、1个成熟期和3个始花期QTL[14,39]相互重叠;qR34-K-1在公共图谱的基因组位置相同为42.392(Satt349)~45.053 cM(BARCSOYSSR_09_0849),所在基因组区域与已经定位的1个始花期、1个成熟期和1个营养期QTL[10,24,35]相互重叠;qR34-L-1在公共图谱的基因组位置相同为30.891(Sat_187)~115.072 cM(Sat_245),所在基因组区域与已经定位的9个始花期个、12个成熟期、7个生殖期和1个出苗期QTL[9,12-13,16-19,21-23,25-27,31,34,37]相互重叠;qR34-L-2在公共图谱的基因组位置相同为61.349(Satt076)~102.055 cM(Sat_245),所在基因组区域与已经定位的8个始花期个、9个成熟期、6个生殖期和1个出苗期QTL[12-13,18-19,26-27,31,37]相互重叠;qR56-N-1在公共图谱的基因组位置相同为37.976(Satt584)~115.072 cM(Satt022),所在基因组区域与已经定位的3个成熟期QTL[15,24]相互重叠;qR34-O-1在公共图谱的基因组位置相同为5.396(BARCSOYSSR_10_0066)~9.526 cM(Satt487),所在基因组区域与已经定位的1个成熟期QTL[35]相互重叠。表明这些QTLs在不同的年份、地区均可稳定表达,受环境的影响相对较小。
3.3 一因多效位点QTL分析
以往研究中一些研究者在定位开花期和成熟期基因时发现,很多群体中与开花期相连锁的标记位点同时与成熟期基因连锁说明控制开花期和成熟期的遗传机制相互联系。本试验细致的对生育期每个进程QTL进行定位,讨论不同生育阶段遗传机制关联性。在于前人研究一致的位点中,qR78-B1-1所在基因组包含qR34-B1-1;qER3-G-1、qER6-G-1、qR13-G-1位于同一连锁群,且标记区间相同;qER5-J-1、qER6-J-1位于同一连锁群,且标记区间相同;qR34-K-2与qER1-K-1、qR34-K-1与qER1-K-2所在基因组区域相互重叠。qR56-N-1、qR78-N-1所在基因组区域相互重叠;qR34-O-1、qER1-O-1所在基因组区域相互重叠。这些QTLs位于同一连锁群,且基因组区域紧密联系,说明这些QTLs可能为同一基因或基因紧密连锁,其中R34与ER1生育期阶段共有3对QTLs紧密联系,进一步佐证了前人关于大豆开花期与成熟区的遗传机制相互联系的观点,同时为揭示大豆各生育期阶段的遗传关系提供重要的参考。
本试验重复检测位点中qR45-A2-1、qR34-D2-1qER6-N-1、qR34-O-1,qER1-D2-2、qR34-I-1、qR78-B1-1,未与前人一致,这些位点在不同环境下被重复定位,在不同的生育期阶段发挥作用,说明这些位点的遗传具有稳定性,可以连续表达,它们的作用具有连续性,有些位点在全生育期阶段没有被检测到,这可能是后期不同位点之间的相互作用或基因效应的消减作用等掩盖了该位点的表达效应。
4 结 论
本研究重复定位到32个与生育期各阶段相关的QTL,其中18个同一年份不同播期下被重复定位,14个在不同年份下被重复定位。32个在多环境下被重复定位的位点中26个位点与前人研究一致,6个生育阶段QTL位点为新发现位点。
