致病菌抵抗溶菌酶机制的研究进展
2020-09-21张新帅刘武康顾丽红郭爱玲
张新帅,阮 瑶,刘武康,陈 倩,顾丽红,郭爱玲,2,*
(1.华中农业大学食品科学技术学院,湖北 武汉 430070;2.国家蛋品加工技术研发分中心,湖北 武汉 430070)
溶菌酶因其溶菌作用而得名,是一种序列保守、无毒无副作用的抗菌蛋白,在生物体的天然免疫系统中起重要作用。该酶广泛存在于高等动植物组织和分泌物中,在微生物和噬菌体中也有分布,蛋清中溶菌酶含量最为丰富。鸡蛋清溶菌酶是C-型溶菌酶的典型代表,也是研究溶菌酶结构和功能的基本模型,并且已投入商品化生产[1]。100多年前,英国细菌学家亚历山大·弗莱明第一个观察到溶菌酶的溶菌作用[2]。溶菌酶的溶菌性质主要归因于其能够有效水解细菌细胞壁的肽聚糖,其水解位点是N-乙酰胞壁酸(N-acetylmuramic acid,NAM)和N-乙酰葡糖胺(N-acetylglucosamine,NAG)间的β-1,4-糖苷键。现已证明,溶菌酶的活性位点通过6 个子位点(A~F)与6 个连续糖单体结合,之后结合到D子位点的催化基团谷氨酸(Glu)35,并和E位点的天冬氨酸(Asp)52通过一个双取代反应水解β-1,4-糖苷键[3-4](图1)。肽聚糖几乎是所有细菌细胞壁的组分,它赋予细胞壁一定的机械强度,使细胞能够抵抗细胞质与外部环境之间组成差异造成的渗透压,并维持细菌的特定形状(球形、棒形、螺旋形等)[5],一旦肽聚糖层受到破坏,细菌就会因处于高渗环境而快速裂解、死亡。另外,细胞壁肽聚糖的合成和精细结构是多样的,同时肽聚糖也是高度动态更新的高分子,这个过程需要相关酶和蛋白质的精确协调和控制[6-7]。
革兰氏阳性菌和革兰氏阴性菌的细胞壁肽聚糖含量存在很大差异,革兰氏阳性菌的细胞壁由多达40 层的肽聚糖以及磷壁酸(wall teichoic acid,WTA)组成,而革兰氏阴性菌通常只有不含WTA的单层肽聚糖,并夹在内膜和含脂多糖的外膜之间,这些差异解释了革兰氏阳性菌通常对溶菌酶敏感,而非革兰氏阴性菌[5,7]。然而,这种屏障可被动物的先天免疫系统的组分破坏,例如乳铁蛋白、防御素和导管素,其都能使革兰氏阴性菌的外膜透化[1]。溶菌酶基于酶活性的裂解机制已经被广泛接受,但越来越多的研究表明溶菌酶的胞壁酶活性不是体外或体内细菌杀灭所必需的,溶菌酶作为一种阳离子抗菌蛋白,可以在带负电荷的细菌细胞膜上穿孔而形成有规则的离子孔道,从而引起胞内大量的K+和内容物外流,最终导致细菌死亡[8-10]。因此,溶菌酶的酶活性和阳离子特性都与抗菌活性有关。
目前,溶菌酶由于其独特的生物学作用,在食品工业、生物工程、抗菌药物、医疗诊断等方面有着广泛的应用前景。在食品工业,溶菌酶主要用于水产类制品、肉类、乳制品、果蔬及饮料的防腐保鲜,其有效性已被广泛证明[11-12],同时被很多国家批准使用,我国已经颁布溶菌酶作为食品添加剂的食品安全国家标准GB 1886.257—2016《食品安全国家标准 食品添加剂 溶菌酶》。溶菌酶虽然有良好的抗菌性,但其抑菌谱和抑菌效力具有局限性,因此目前的研究集中在将不同的生物防腐剂与溶菌酶进行不同比例的复配。Marianna等[13]将溶菌酶、Nisin和乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA)进行复配,有效抑制了单核细胞增生李斯特菌(Listeria monocytogenes)和大部分肉类腐败菌的生长。陈赛等[14]通过溶菌酶、壳聚糖和茶多酚复合保鲜液成功延缓了冷藏过程中草鱼肌肉质量的降低。另外,研究表明将溶菌酶固定到银纳米粒子,协同提高了对大肠杆菌(Escherichia coli)的抗菌活性[15]。开发抗菌作用强、抑菌谱广的复合生物防腐剂以及结合其他防腐保鲜措施将是未来的研究趋势。
鉴于溶菌酶的广泛分布和有效抗菌活性,致病菌已经通过突变、变异等进化出逃避或破坏溶菌酶杀灭的机制,一旦逃脱,致病菌将长期存活并可能造成感染,这将对食品安全、疾病治疗和人体健康造成一定的威胁。目前,国内对溶菌酶的生物活性和保鲜应用研究较多,但对致病菌抵抗溶菌酶的研究较少。因此,本文对致病菌抵抗溶菌酶的机制和抗性基因的转录调节进行了阐述,以期为相关研究、新型食品抗菌剂的开发以及临床治疗提供一定的理论依据。
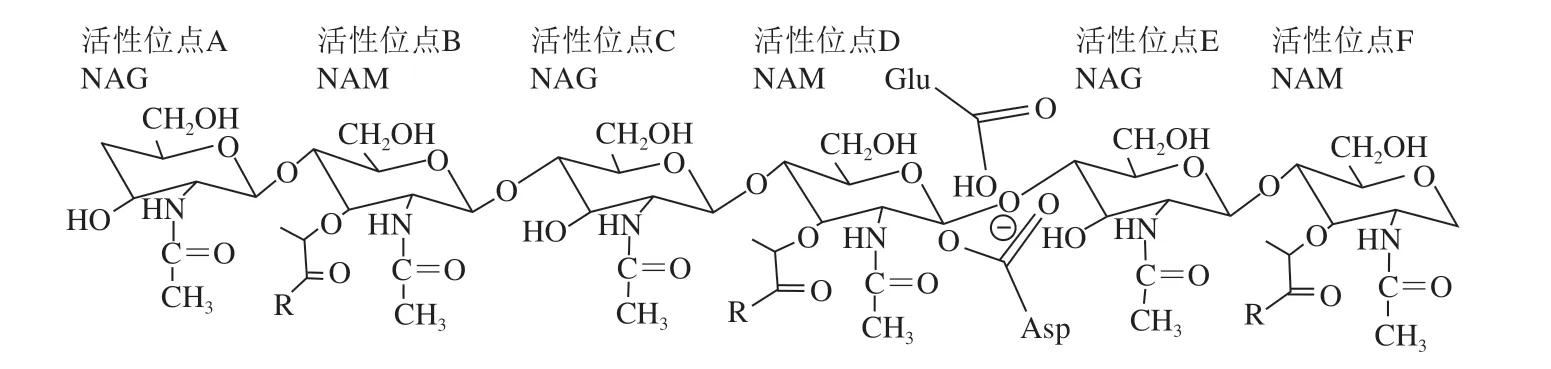
图 1 溶菌酶的作用机制Fig. 1 Mechanism of action of lysozyme
1 致病菌抵抗溶菌酶的机制
具有引发疾病潜力的细菌已经进化出多种机制来逃避溶菌酶的杀灭,主要通过肽聚糖修饰和产生溶菌酶抑制剂两种途径,如图2所示。
1.1 肽聚糖修饰
1.1.1N-脱乙酰化
溶菌酶的活性位点与肽聚糖主链上乙酰基之间的相互作用促进了有效的水解活性,为限制这些相互作用,致病菌进化出对肽聚糖单体残基进行脱乙酰化修饰的基因,以降低对溶菌酶的敏感度。肽聚糖的N-脱乙酰化是指在酶的作用下使NAM或NAG的C2残基脱去乙酰化(图2B),该反应在60多年前被Roseman[16]首次证明。该修饰主要存在革兰氏阳性菌中,不同细菌中脱乙酰化比例不同,目前,鉴定到含有N-脱乙酰化肽聚糖的革兰氏阴性菌只有幽门螺杆菌(Helicobacter pylori)和福氏志贺菌(Shigella flexneri)[17-18]。
肽聚糖去乙酰化的基因首先在肺炎链球菌(Streptococcus pneumoniae)中被鉴定出来,被命名为肽聚糖N-脱乙酰酶(peptidoglycanN-acetylglucosamine deacetylase A,PgdA),PgdA属于碳水化合物酯酶家族CE-4,其作用于NAG的C2残基使其脱乙酰化,有助于提高溶菌酶抗性,同时发现pgdA基因敲除体对溶菌酶体外杀伤更敏感,体内毒力较小,基于此,pgdA被鉴定为毒力因子基因[19]。动物病原体猪链球菌(Streptococcus suis)也有类似的表型,pgdA基因敲除体与野生型猪链球菌相比,有着更低的去乙酰化水平,对溶菌酶更加敏感,更容易被中性粒细胞介导的免疫反应清除,其体内毒性更小[20]。随后在其他细菌中也鉴定到pgdA同源物,包括粪肠球菌(Enterococcus faecalis)[21]、幽门螺杆菌[18]、单核细胞增生李斯特菌[22]、艰难梭菌(Clostridium difficile)(pdaV)[23]、结核分枝杆菌(Mycobacterium tuberculosis)(Rv1096)[24]、海豚链球菌(Streptococcus iniae)(pdi)[25]、蜡样芽孢杆菌(Bacillus cereus)(Bc1974)[26]、枯草芽孢杆菌(Bacillus subtilis)(pgdA、pdaC)[27],pdaV、Rv1096等毒力因子基因都能增强细菌自身对溶菌酶的体外抗性或增加细菌体内存活率。而革兰氏阴性菌幽门螺杆菌和福氏志贺菌由于外膜的存在,pgdA敲除体在体外仅在添加膜破坏剂(如乳铁蛋白)时对溶菌酶敏感,而在宿主体内其存活率也降低[16-17]。鉴于它们在影响细菌毒力中的重要性,肽聚糖修饰酶开发新抗菌药物的研发将是一个重要的发展方向,开发肺炎链球菌PgdA抑制剂的可行性已经得到证明[28-29]。
枯草芽孢杆菌PgdA是第一个被测定生化活性和结构的细菌肽聚糖脱乙酰酶,该酶的NodB同源结构域为(β/α)8桶状折叠结构,含有大部分CE-4家族保守残基,两个保守的组氨酸(His)聚集在活性位点的底部[30]。随后,又有相关研究报道了肺炎链球菌PgdA的晶体结构,其含有3 个独立的结构域,作为一种金属酶,在不同金属离子中酶的活性大小不同,EDTA能使其失活。该酶使用His-His-Asp锌结合三联体,以3 个连续的NAG(GlcNAc3)作为底物,邻近保守的Asp和His分别催化碱和酸反应,去除中间NAG的N-乙酰基[31]。Bhattacharjee等[29]通过分子对接和分子动力学模拟等技术手段研究幽门螺杆菌脱乙酰基酶从模型底物NAG去除乙酰基的反应机理。最近,Giastas等[26]首次通过X射线衍射报道了N-脱乙酰化Bc1974-(GlcNAc)3配合物的晶体结构,揭示了NodB结构域中活性位点环(MT4)存在两种不同的构象,同时提出Bc1974的催化机制(水或氢氧根离子对N-乙酰基的C=O的碳原子进行亲核攻击)。
1.1.2O-乙酰化
肽聚糖的O-乙酰化是指向大多数细菌中NAM的C6羟基上添加乙酰基(图2C),通过空间位阻阻止溶菌酶与肽聚糖的结合[32],一些乳杆菌和芽孢杆菌中也存在NAG的乙酰化修饰[33-34]。NAM残基的O-乙酰化比例随菌株、培养时间和生长条件而变化,通常在20%~70%[35]。
在过去的10 年中,已经在多种致病菌中鉴定出负责乙酰化修饰的基因。其中第一个鉴定到的是革兰氏阳性菌金黄色葡萄球菌(Staphylococcus aureus)中的O-乙酰转移酶(O-acetyltransferase A,OatA),其在C6位置乙酰化NAM,O-乙酰化的肽聚糖稳定性更高,同时提高了体外溶菌酶的抗性和体内细菌的存活率[32]。后续又在其他几种革兰氏阳性菌中发现了oatA的同源物,单核细胞增生李斯特菌(oatA)[22]、肺炎链球菌(adr)[36]以及炭疽芽孢杆菌(Bacillus anthracis)(oatB)[33]中O-乙酰转移酶的丧失使得这些细菌对溶菌酶敏感,但前提是它们也缺乏pgdA或具有完全N-乙酰化肽聚糖。粪肠球菌就需要多种溶菌酶抗性因子的缺失才能观察到溶菌酶敏感性,包括pgdA和oatA[21]。在分子水平上对OatA还知之甚少,预测oatA基因编码假定的跨膜整合蛋白,由N末端跨膜整合结构域连接C末端胞质外结构域组成,N末端结构域从细胞质中获取乙酰基,然后将其转移穿过内膜,胞质外C末端结构域接受乙酰基并催化乙酰基转移用以修饰NAM的C6羟基,C末端结构域中富含赖氨酸(Lys)的区域被假定为催化活性位点,对预测的氨基酸序列进行更密切的分析表明它具有SGNH/GDSL水解酶的折叠结构,使用Asp-His-丝氨酸(Ser)残基形成的催化三联体转移乙酰基,但至今其晶体结构仍然未知[35,37]。
革兰氏阴性菌不编码OatA或OatB的同源物,其肽聚糖的O-乙酰化需要一种双组分蛋白(peptidoglycanO-acetyltransferases A,PatA)和(peptidoglycanO-acetyltransferases B,PatB),预测PatA像OatA或OatB一样发挥作用,转移乙酰基穿过细胞质膜,之后周质中的PatB转移乙酰基至NAM的C6羟基,进而修饰产生溶菌酶抗性[38]。该蛋白最初在病原体脑膜炎奈瑟球菌(Neisseria meningitidis)和淋病奈瑟球菌(Neisseria gonorrhoeae)中被鉴定到,但最近研究发现patA基因的缺失不影响淋病奈瑟菌对溶菌酶的敏感性,除非细胞膜完整性也受到影响,而且并未增加其对人体中性粒细胞杀伤的敏感性[39]。在幽门螺杆菌和空肠弯曲杆菌(Campylobacter jejuni)中,在乳铁蛋白存在下patA基因的缺失增加了细菌对溶菌酶的敏感性,而且空肠弯曲杆菌patA突变体对体外巨噬细胞的杀伤更敏感,体内定植肠道的能力降低[40-41]。
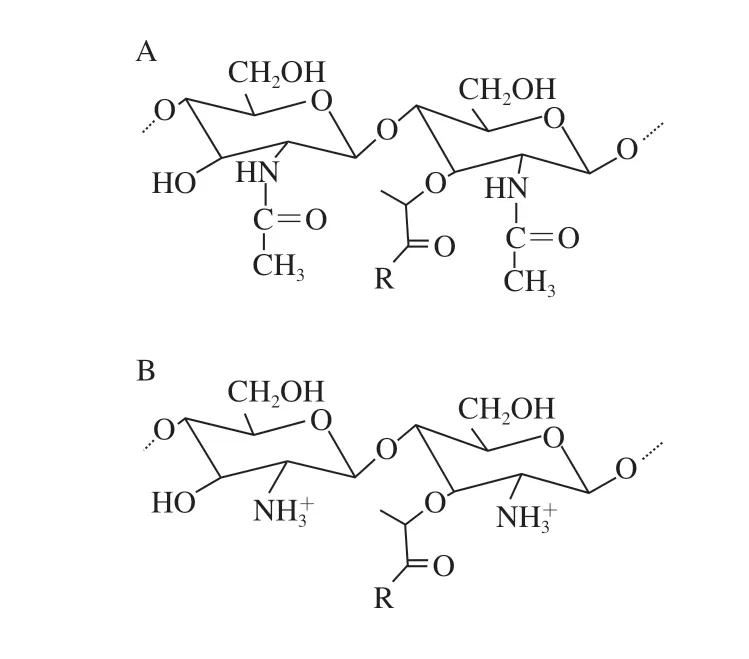

图 2 肽聚糖的不同修饰Fig. 2 Different modifications on peptidoglycan
目前,提取纯化肽聚糖和液质联用分析是肽聚糖N-脱乙酰化和O-乙酰化研究中的重要手段,但有时从细菌中分离的肽聚糖纯度无法得到保证,这使得研究具有挑战性,无论是肽聚糖的结构与功能分析还是具体抗性机制的研究,肽聚糖片段的体外化学合成研究将对此领域产生巨大推动作用[37]。另外,由于技术手段和提取修饰酶的难度和纯度的限制,只有少数酶的晶体结构已知。更多致病菌中的肽聚糖修饰基因及其结构、N-脱乙酰化和O-乙酰化的具体催化途径等问题都有待深入研究,同时,肽聚糖修饰酶的细菌生理学功能目前仍不明确。
1.1.3 其他修饰
WTA对溶菌酶抗性也有一定的贡献。金黄色葡萄球菌中WTA与NAM的C6羟基共价偶联(图2D),类似于O-乙酰化,形成的空间位阻有助于抵抗溶菌酶的水解活性,但更进一步的研究表明每9 个NAM残基仅连接一个WTA,而O-乙酰化比例较高,因此WTA仅充当一个辅助功能,当O-乙酰化修饰完全缺失时,虽然存在WTA修饰,但由于丰度低,溶菌酶仍然可以水解肽聚糖[42]。另一项研究发现WTA通过调节青霉素结合蛋白(penicillin binding proteins,PBP)4定位来控制肽聚糖交联水平,进而影响溶菌酶对其底物的可利用性来增加溶菌酶抗性[43]。同样肺炎链球菌中murMN操纵子敲除体具有较低的肽聚糖交联水平,并且对溶菌酶以及非酶促阳离子抗菌肽更敏感[44]。最近,在新月柄杆菌(Caulobacter crescentus)中发现茎杆细胞肽聚糖具有更多的LD-转肽作用(m-DAP与m-DAP交联),相比DD-交联(m-DAP与D-Ala交联)的细胞体肽聚糖具有更高的溶菌酶抗性[45]。
阳离子抗菌蛋白质高度结合细菌带负电荷的细胞膜,这种相互作用对于有效杀灭细菌很重要。因此,将净负电荷降低至中性电荷的修饰,可减少溶菌酶以及其他阳离子抗菌蛋白的结合。在金黄色葡萄球菌中,MurT/GatD酰胺基转移酶复合物在肽聚糖肽链的第二个位置将谷氨酸酰胺化为谷氨酰胺(图2E),降低了肽聚糖的净负电荷,增加完整细菌和纯化肽聚糖对溶菌酶的抗性[46]。最近,肺炎链球菌的MurT/GatD复合物的结构和具体的酰胺化反应机制已经被证明[47]。dlt操纵子对WTA的D-丙氨酰化也降低了细胞膜的净负电荷[7]。在其他致病菌也有发现WTA的D-丙氨酰化修饰[48-51]。
1.2 溶菌酶抑制因子
一些革兰氏阴性菌(铜绿假单胞菌(Pseudomonas aeruginosa)和大肠杆菌)对溶菌酶具有内在抗性,但这些细菌缺乏肽聚糖修饰。Monchois等[52]在2001年在研究大肠杆菌ykfE基因表达和编码蛋白的功能特征时发现ykfE基因产物是同源二聚体,位于周质空间,是有效的C-型溶菌酶抑制剂(Ki≈1 nm),之后将此基因更名为ivy(inhibitor of vertebrate lysozyme)。之后,在肠炎沙门氏菌(Salmonella enteritidis)、大肠杆菌和铜绿假单胞菌中鉴定出了与Ivy无关的新型周质和膜结合溶菌酶抑制剂,将其命名为MliC/PliC(membrane bound/periplasmic lysozyme inhibitors of c-type lysozyme)[53],
还鉴定出命名为PliG(periplasmic lysozyme inhibitors of g-type lysozyme)和PliI(periplasmic lysozyme inhibitors of g-type lysozyme)的G-型和I-型溶菌酶的周质抑制剂[54-55]。此外,在卡他莫拉菌(Moraxella catarrhalis)中发现了溶菌酶抑制剂LipA和LipB[56]。最近,在奈瑟氏菌属(Neisseria)中鉴定出黏附素复合蛋白(adhesin complex protein,ACP)以及在淋病奈瑟球菌中鉴定出一种由开放阅读框ngo1063编码的新型表面暴露C-型溶菌酶抑制剂,将其命名为SliC(surface-exposed lysozyme inhibitor of C-type lysozyme)[57-59]。
目前的溶菌酶抑制剂家族仅在革兰氏阴性菌中被发现,主要在变形菌门中,但不能排除革兰氏阳性溶菌酶抑制剂家族的存在。不同家族的溶菌酶抑制剂在一级序列水平上显示出很低的相似性,两种MliC和PliC仅有32%和27%,SliC与不同细菌中MliC的同一性最高仅为27.27%。但它们具有相同的特异性并具有特定的序列基序和相似的结构拓扑,MliC和PliC都含有相对保守的COG3895结构域,SliC含有假定的MliC结构域以及与溶菌酶作用的保守残基S89和K103[57]。目前一些抑制剂家族的3D结构以及一些抑制剂-溶菌酶复合物的3D结构已经通过X射线晶体学或核磁共振(nuclear magnetic resonance,NMR)揭示。Ivy中心部分由5 个反平行β-折叠片和4 个α-螺旋构成,抑制作用是通过Ivy中一个突出保守的CKPHDC环来实现的,该环中心的His残基(H60)与负责溶菌酶酶活性的3 个残基中的两个(D52和E35)形成氢键,通过这样的锁键机制阻断溶菌酶的活性位点[60]。PliC和MliC家族都折叠成一个八股反平行的β桶状结构,复合物晶体结构显示MliC的关键保守区(COG3895结构域残基86-92和97-104)插入溶菌酶的活性位点裂缝中,具体通过残基Ser89和溶菌酶Asp52之间的氢键以及残基Lys103和溶菌酶Asp52与Glu35之间的离子键来紧密结合,并通过丙氨酸(Ala)取代证实了这一结果[61]。而PliI由两个β-折叠片和一个α-螺旋构成,定点诱变表明PliI的抑制作用是通过将含有保守SGxY基因序列的环插入I-型溶菌酶的活性中心实现的[62]。PliG家族由两个β-折叠片构成,与MliC/PliC和PliI家族共享高度保守的SG(x)xY序列基序,但发现该基序不参与G-型溶菌酶的抑制[63]。
溶菌酶抑制剂最直接的功能是保护细菌免受动物宿主中溶菌酶的影响,因此对细菌定植或毒力有着重要作用。迟缓爱德华氏菌(Edwardsiella tarda)中ivy和mliC是宿主感染所必需的毒力因子基因,鼠伤寒沙门氏菌(Salmonella typhimurium)mliC基因的敲除减少了在巨噬细胞中的存活率,而淋病奈瑟球菌SliC在雌性淋病小鼠模型感染过程中对生殖道黏膜的定植起着关键作用[53,57,64]。细菌溶菌酶抑制剂另一个可能的功能是控制内源性裂解性转糖基酶,假单胞菌属中表达的两种Ivy蛋白已被证明不抑制溶菌酶活性,而是内源性裂解性转糖基酶的有效抑制剂。目前,该酶在肽聚糖代谢中的作用已经确立,但它们的调节机制尚无定论[65]。
未来,溶菌酶抑制剂可能成为抗菌药物开发的一个有吸引力的新靶点,但目前仍存在一些尚未解决的问题。是否存在革兰氏阳性菌溶菌酶抑制剂家族;溶菌酶抑制剂是否在细菌-宿主相互作用中存在其他功能;动物中不同类型溶菌酶抑制剂和不同类型溶菌酶的表达复杂性以及是否存在特异性。这些问题的解决将为相关研究和潜在应用产生积极的促进作用。
2 溶菌酶抗性基因的转录调节
虽然肽聚糖的修饰以及抑制剂的分泌等可以增加溶菌酶的耐受性,但也可能降低致病菌本身的环境适应性,如肺炎链球菌就存在此种情况[42]。因此,细菌常常使用多种机制来调控抗性因子基因的表达水平,以更好地在溶菌酶等不良环境中存活,例如,粪肠球菌仅在溶菌酶环境中发生NAG的去乙酰化修饰[66]。
2.1 胞质外功能性sigma因子
胞质外功能性σ因子(extracytoplasmic function sigma factors,ECFs)在细菌对各种环境应激的应答中起重要作用,是细菌信号转导的“第三支柱”。已知模式生物枯草芽孢杆菌中编码7 种已知的ECFs(σM、σW、σV、σX、σY、σZ、σYlaC),其中σV被溶菌酶强烈且特异性地诱导,σV的活化是通过溶菌酶与抗σ因子RsiV的结合,进而信号肽酶启动蛋白水解级联反应切割降解RsiV和激活σV[67],σM、σX和σV三者协同调控溶菌酶抗性,其中σV调控多个操纵子,通过激活两种细胞壁修饰途径赋予溶菌酶抗性:OatA催化肽聚糖的O-乙酰化和dltABCDE操纵子对WTA的D-丙氨酰化,另外PbpX可能对枯草杆菌的溶菌酶抗性也有贡献,这是目前枯草芽孢杆菌中鉴定到的3 种机制[49,68]。
其他革兰氏阳性菌中也鉴定到σV系统。艰难梭菌至少编码3 种σ因子,σT、σU和σV,编码这些σ因子基因的表达是由细胞外应激而激活的,包括抗菌肽或溶菌酶。在艰难梭菌中溶菌酶诱导csfV的表达,其编码σV是溶菌酶抗性所必需的,进一步通过微阵列和实时荧光定量聚合酶链式反应分析,发现σV使编码脱乙酰酶pdaV基因的表达上调了17 倍,进一步增加了NAG的去乙酰化比例,进而增加溶菌酶的耐受性,同时发现csfV敲除体在仓鼠感染模型中毒性严重减弱。另一项研究还发现艰难梭菌σV也调控dlt操纵子,并且该途径同样影响体内毒力,这些都表明σV是艰难梭菌的重要毒力因子[23,50]。在粪肠球菌中σV作为转录调节因子,由溶菌酶和其他细胞壁应激物诱导表达,仅显著正调节pgdA,不直接调控oatA或dlt操纵子,但sigV/oatA/dltA三基因敲除体显示出最低的溶菌酶抗性,表明三者在粪肠球菌的溶菌酶抗性中有着未知的协同作用,在小鼠模型中sigV敲除的粪肠球菌定植和维持感染能力受到显著影响[69]。可以看出,这3 种不同的革兰氏阳性菌通过不同的策略来响应溶菌酶。
2.2 双组分信号转导系统
双组分信号转导系统是细菌应对各种环境变化的重要机制,其基本结构为一个组氨酸蛋白激酶和一个反应调节蛋白。双组分信号转导系统GraRS诱导金黄色葡萄球菌中dlt操纵子的表达,并协同oatA增强细菌对溶菌酶的抗性,GraRS由特定的阳离子抗菌肽激活,但这种激活的机制仍有待研究[8]。许多其他细菌也具有类似的双组分系统,单核细胞增生李斯特菌中VirR正向调节dlt操纵子,并且virR基因敲除体在小鼠体内的毒力减弱,故virR鉴定为prfA调节子后的第二个关键毒力调节子[70]。卡他莫拉菌中响应调节器mesR基因的敲除上调了溶菌酶抑制剂编码基因lipA和lipB的表达[56]。猪链球菌中vraSR基因敲除体对氧化剂和溶菌酶的敏感性更高,小鼠感染模型证实菌株毒力也大大降低[71]。
2.3 最新转录调节因子
最近研究还发现一些新型转录调节因子来调控溶菌酶耐受性。单核细胞增生李斯特菌的degU和rli31基因敲除体对溶菌酶极其敏感,并且在体内感染期间毒性减弱,pgdA和pbpX基因在degU和rli31基因敲除体中mRNA丰度降低,而pgdA的上调恢复了rli31敲除体的溶菌酶抗性,但rli31和degU如何调节pgdA和pbpX还有待阐明[48]。SpoVG是一种保守的RNA结合蛋白,与非编码RNA Rli31相互作用,通过不确定的机制负调节单核细胞增生李斯特菌的溶菌酶抗性、毒力和群集运动能力[72]。另一项研究发现细胞分裂蛋白GpsB影响单核细胞增生李斯特菌的溶菌酶抗性,gpsB基因敲除体的溶菌酶抗性增加,生化实验结果表明溶菌酶抗性增加是依赖于PgdA而非OatA,GpsB通过双功能PBP A1对PgdA发挥作用[73]。Gogos等[74]在化脓性链球菌(Streptococcus pyogenes)中发现Rgg2/3群体感应系统在所有测序分离株中都是保守的,其诱导带正电荷的分泌蛋白StcA表达,该系统的激活将导致生物膜的形成和溶菌酶抗性的增加,体外实验结果表明StcA蛋白可能与细胞表面的负电荷结构域发生离子缔合以帮助细菌逃避溶菌酶杀伤,这是有关溶菌酶抗性的一种新机制。来自肠外致病性大肠杆菌的O-特异性多糖是脂多糖介导抑制溶菌酶杀菌活性所必需的,并且单独的O-特异性多糖也能够通过与溶菌酶直接相互作用来抑制溶菌酶的水解活性[75]。
3 结 语
溶菌酶作为宿主防御外源微生物的重要物质,一直是生命科学研究的热点,溶菌酶和入侵微生物之间的不断竞争,使致病菌进化出多种机制来阻止溶菌酶的杀灭。溶菌酶作为一种有效抑菌的天然生物防腐剂,在食品工业中得到了广泛应用,食品一旦污染了具有溶菌酶抗性的致病菌,由于溶菌酶的抑菌作用减弱,致病菌将大量生长繁殖,这将造成重大食品安全隐患,因此抗溶菌酶机制的研究对更有效、更全面食品防腐剂的开发有着积极作用。
溶菌酶抗性明显代表一种致病菌发病机理的常见机制,因为溶菌酶敏感突变体的毒力在多种感染模型中严重减弱,溶菌酶抗性基因也常被鉴定为毒力因子,因此这可以成为未来抗菌剂开发和临床治疗的方向。其中肽聚糖修饰位点可以成为有利靶点,部分肽聚糖修饰酶抑制剂的可行性已经得到证明,即使这些酶的抑制剂体外活性较低,它们也可以通过弱化肽聚糖使其易受宿主攻击而提供与先天免疫系统协同作用的机会。另外,可以通过干扰溶菌酶-抑制剂相互作用的抑制分子来增加细菌的溶菌酶敏感性,并因此降低其致病性,因而对不同抑制剂与同源溶菌酶的分子相互作用机制的研究将是开发有效抑制分子的先决条件。
多年来,国内外相关研究取得了不错的进展,但仍然存在很多空白。更多物种肽聚糖修饰酶的鉴定、结构、催化机制以及溶菌酶抗性之外可能的生理功能,仍待认识的溶菌酶抑制剂、现有抑制剂与溶菌酶之间的作用模式以及可能的生物学功能,调节溶菌酶抗性因子所涉及的因素以及具体调控机制等,都将是未来研究的方向。高通量测序技术的发展也将为相关研究提供新思路,新物种的全基因组测序序列可以通过比对工具BLAST等鉴定与已知溶菌酶抗性基因序列或蛋白质序列相似度较高的序列并进行功能预测,溶菌酶处理下的转录组测序也可以发现未知的调节基因,之后进行特定基因的功能验证,这些为更多未知功能基因的挖掘提供了一个全新的途径。
