基于mtCOI基因序列的中国葱斑潜蝇不同地理种群遗传多样性分析
2020-08-06钟裕俊杜素洁潘立婷王玉生王福莲刘万学
钟裕俊, 杜素洁, 潘立婷, 王玉生, 王福莲, 刘万学,*
(1. 长江大学农学院, 长江大学昆虫研究所, 湖北荆州 434025; 2. 中国农业科学院植物保护研究所, 植物病虫害生物学国家重点实验室, 北京 100193)
葱斑潜蝇Liriomyzachinensis隶属双翅目(Diptera)潜蝇科(Agromyzidae)斑潜蝇属Liriomyza,是一种非常重要的潜叶蝇类害虫,主要为害大葱Alliumfistulosum、韭菜A.tuberosum、洋葱A.cepa等百合科(Liliaceae)葱属Allium植物,以大葱受害最重(Spencer, 1973; 康乐, 1996)。其幼虫、成虫均可为害,主要以幼虫潜食叶肉为主,叶肉被取食后,仅剩两层表皮,呈现白色线状条斑,形成潜道,严重影响植物正常的光合作用;并且雌成虫的产卵器可刺破叶片,由此造成的机械损伤为病原物侵入提供了重要途径(潘秀美和姜官恒, 2001; 魏国先等, 2001)。葱斑潜蝇广泛分布于东南亚和太平洋地区,在中国、日本、韩国、印度尼西亚、新加坡、朝鲜、越南等国家危害严重(Tran and Takagi, 2005; Tranetal., 2007),近年来该虫也在欧洲部分地区如法国、德国等地暴发成灾(Martynovetal., 2016)。该虫在我国大部分地区均具有较高的适应性(梁超峰等, 2018),主要分布于黑龙江、内蒙古、辽宁、沈阳、山东、河北、河南、山西、福建、台湾等省区(王永卫等, 1992; 王进忠等, 2000; Shiao, 2004)。葱斑潜蝇的温度适应范围广,单雌产卵量大,在我国各地一年发生4~7代不等,如东北4~5代,山东6~7代,且世代重叠发生(王立霞等, 2000; 潘秀美和姜官恒, 2001),有春季和秋季两个发生高峰期,高峰期易在大葱种植区暴发成灾(潘秀美和姜官恒, 2000; 王立霞等, 2000; Tranetal., 2007)。
大葱作为葱斑潜蝇的主要寄主植物,其大规模化面积种植一定程度上有利于葱斑潜蝇的严重发生。据2014年农业农村部种植业管理司的统计结果,我国大葱种植面积已逾57.3万hm2,主产区位于秦岭淮河以北的广大地区,涵盖华北平原、东北平原和黄土高原三大主产区,其中华北平原的山东、河南和河北三省为我国大葱的第一大产区,总栽培面积占全国的36%,总产量占全国的51%(彭帅等, 2017)。山东省作为我国葱蒜类蔬菜种植面积最大的省份,2001年葱蒜类蔬菜种植面积约26.7万hm2,其80%的面积遭受葱斑潜蝇的危害,被害株率常达40%以上(潘秀美和姜官恒, 2001)。傅苏友和苏保乐(2011)在调查山东潍坊市的葱斑潜蝇发生情况时,发现其在大葱上为害严重,一株葱上幼虫平均为7头,被害株高达70%~80%,严重的达到100%。
目前有关葱斑潜蝇的研究多局限于生物学特性、发生规律等基础性研究,而有关葱斑潜蝇的种群遗传多样性、种群扩张等方面研究还暂未见报道。葱斑潜蝇作为一种本地害虫,相对于斑潜蝇属的其他入侵种,如南美斑潜蝇L.huidobrensis、美洲斑潜蝇L.sativae、三叶草斑潜蝇L.trifolii等,其长期累积的遗传变异是否与其严重危害存在联系,还尚不清楚。分子标记技术的快速发展,很大程度加快了国内外学者对斑潜蝇属害虫遗传分化的研究。Morgan等(2000)首先对美国加利福尼亚中部和南部的南美斑潜蝇种群间进行RAPD-PCR检测,发现南美斑潜蝇种群间的遗传变异主要来源于地理分布。王莉萍等(2007)利用rDNA-ITS1标记对我国8个不同地理的美洲斑潜蝇种群进行分析,得出各地理种群之间的遗传分化很小,且遗传分化很有可能与地理分布有关。本研究选用线粒体COI基因作为分子标记,以我国8省12个不同地区的葱斑潜蝇种群为研究对象,对不同地理种群的遗传多样性、遗传分化程度、基因交流等进行了分析,以揭示其种群系统发育地理格局与演变机制。研究结果对葱斑潜蝇的种群遗传动态分析、种群扩张机制探讨具有一定的指导意义,同时也为制定葱斑潜蝇的防治策略提供理论依据。
1 材料与方法
1.1 供试虫源
葱斑潜蝇标本于2016-2018年采集自中国8省12个地点(表1),采用五点取样法,采集露天种植的优势寄主植物大葱A.fistulosum和小葱A.ascalonicum,采集虫态为幼虫或蛹,带回实验室饲养。 待虫源羽化后,浸于无水乙醇中,-40℃保存备用。
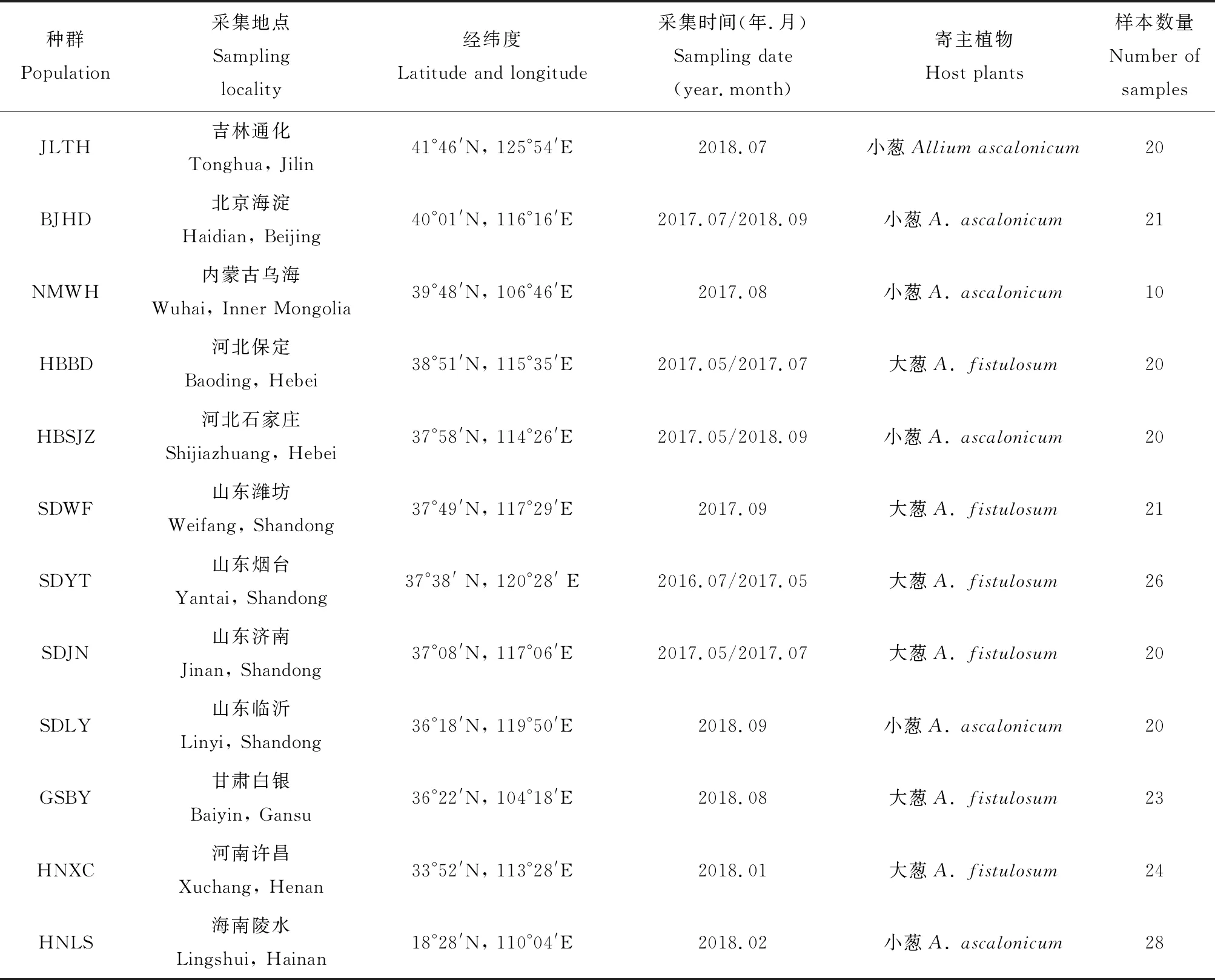
表1 葱斑潜蝇不同地理种群的采集信息Table 1 Collecting data of different geographical populations of Liriomyza chinensis
1.2 基因组DNA提取
参照De Barro和Driver(1997)的快速研磨法,并稍作改动。具体步骤如下:从标本管中取单头葱斑潜蝇雌成虫,置于干净滤纸上,待无水乙醇挥发后,吸取10 μL裂解液(0.05 mol/L Tris-HCl, pH 8.4, 50 mmol/L KCl, 0.45% Tween-20, 0.45% NP 40, 0.2% Gelatin, 80 μg/mL蛋白酶K)滴于封口膜上,用灭菌过的0.2 mL PCR管管底充分研磨虫体,将提取液吸入0.2 mL PCR管中。吸取20 μL裂解液清洗PCR管底和封口膜2次,最后将清洗液吸入0.2 mL PCR管中。将提取液进行金属浴,程序: 65℃下30 min, 25℃ 下2 min, 96℃ 下10 min,灭活蛋白酶K,最终获得50 μL的葱斑潜蝇DNA,保存在-20℃冰箱中备用。
1.3 mtCOI基因序列的扩增
采用引物组合C1-J-2183 (5′-CAACATTTATTTT GATTTTTTGG-3′)和TL2-N-3014 (5′-TCCATTGCAC TAATCTGCCATATTA-3′)(Simonetal., 1994)扩增葱斑潜蝇mtCOI基因的目的片段。引物由上海生工生物工程股份有限公司合成。
PCR反应体系(25 μL): 10×buffer(含Mg2+)2.5 μL, dNTPs(2.5 mmol/L)0.4 μL, 正反引物(10 pmol/L)各0.4 μL,Taq酶(2.5 U/μL) 0.2 μL, 模板DNA 1 μL, ddH2O 20.1 μL。PCR反应程序: 95℃预变性4 min; 95℃变性1 min, 52.5℃退火50 s, 72℃延伸90 s, 35个循环;继续72℃延伸10 min。取20 μL扩增产物在含有GoldViewⅡ的1%琼脂糖凝胶上以110 V电泳分离30 min,然后以凝胶成像系统分析电泳结果并拍照保存。
1.4 PCR产物的纯化及克隆测序
由于部分虫源直接测序时出现重叠峰,为了确保测序的准确性,对存在重叠峰的样品进行了克隆测序。在紫外灯下切下含有目的片段的明亮条带,使用AxyPrep DNA凝胶回收试剂盒将PCR产物回收纯化。将纯化产物与克隆质粒pEASY-T3 Cloning Vector连接,转化到Trans1-T1感受态细胞内,筛选阳性单克隆进行菌落PCR检测,之后送到上海生工生物工程股份有限公司进行双向测序。
1.5 序列整理及数据分析
首先将正反向引物测得的序列导入MEGA 7.0软件(Kumaretal., 2016)中,结合峰图识别软件Chromas 2.6校正序列,再进行多序列比对。利用NCBI数据库对获得的序列进行BLAST比对分析,以确定扩增的基因序列来自线粒体COI基因。
利用MEGA 7.0软件(Kumaretal., 2016)对葱斑潜蝇mtCOI基因序列进行变异分析,计算变异位点数、简约信息位点数、碱基组成等信息。同时,基于Kimura-2-Parameter(K2P)模型,以美洲斑潜蝇L.sativae、南美斑潜蝇L.huidobrensis、三叶草斑潜蝇L.trifolii、番茄斑潜蝇L.bryoniae和豌豆彩潜蝇Chromatomyiahorticola的相应序列为外群,采用非加权组平均(UPGMA)法构建单倍型系统发育树,系统树各分支的置信度(bootstrap)均进行1 000次重复检验。利用DnaSP 6.1软件(Rozasetal., 2017)对不同地理种群的葱斑潜蝇进行单倍型多样性(Hd)、核苷酸多样性(Pi)和Tajima’sD中性检验等分析。应用Network 4.6基于Median-joining算法构建单倍型间的网络关系图。
应用Arlequin 3.5软件(Excoffier and Lischer, 2010)计算种群间的遗传分化系数(FST)(Hudsonetal., 1992)、遗传距离[FST/(1-FST)]、基因流(Nm)等参数;并进行分子方差分析(AMOVA),计算遗传变异在种群间和种群内的方差贡献率。
根据采集地经纬度计算出成对种群间的地理距离,同时结合遗传分化系数(FST),用TFPGA软件(Miller, 1997)中的Mantel算法检测不同种群间的遗传距离和地理距离的相关性,进行10 000次置换,并做显著性检验。
2 结果
2.1 葱斑潜蝇线粒体COI基因的碱基组成及单倍型分布
将中国葱斑潜蝇12个地理种群共253条mtCOI基因序列比对、剪切后得到759 bp的片段,分析表明,无碱基的插入或缺失,具明显的A/T碱基偏倚性,A+T平均含量为70.3%;序列共有20个变异位点,占碱基总数的2.64%,包括13个单变异多态性位点和7个简约信息位点。
单倍型的数量及其分布和频次的结果显示(表2),供试种群中共发现13种单倍型Hap1-Hap13 (GenBank登录号: MN700917-MN700929),包括7个共享单倍型(Hap1, Hap3, Hap6, Hap8-Hap11),其余6个单倍型为相应地理种群独享单倍型,其中Hap2属于北京海淀种群(BJHD), Hap4和Hap5属于甘肃白银种群(GSBY), Hap7属于海南陵水种群(HNLS), Hap12和Hap13属于吉林通化种群(JLTH)。Hap1出现频率最高,占总样本的81.82%,且分布最广,为所有地理种群共有,其余12种单倍型出现频率均较低(≤3.95%)。各地理种群中,吉林通化种群(JLTH)的单倍型种类最多(5种),其次为河北石家庄种群(HBSJZ)、河南许昌种群(HNXC)和山东济南种群(SDJN)有4种,而河北保定种群(HBBD)与内蒙乌海种群(NMWH)仅有1种单倍型。
2.2 葱斑潜蝇种群的遗传多样性及中性检验
葱斑潜蝇各种群mtCOI基因单倍型多样性(Hd)、核苷酸多样性(Pi)、核苷酸平均差异数(K)、Tajima’sD中性检测分析结果显示(表2),总群体单倍型多样性(Hd)为0.327,核苷酸多样性(Pi)为0.00159,核苷酸平均差异数(K)为1.21011,中性检验结果不显著(Tajima’sD=-1.64799,P>0.05),说明供试葱斑潜蝇总群体在过去较近的一段历史时期内未经历明显的群体扩张和持续增长模式,群体大小保持相对稳定状态。各地理种群单倍型多样性(Hd)在0~0.763间,核苷酸多样性(Pi)在0~0.00716间,核苷酸平均差异数在0.09524~5.43684间,吉林通化种群(JLTH)的单倍型多样性最高(Hd=0.763),其次是河南许昌种群(HNXC,Hd=0.562)和河北石家庄种群(HBSJZ,Hd=0.489),而河北保定种群(HBBD,Hd=0)和内蒙乌海种群(NMWH,Hd=0)遗传多样性最低。各种群间的Tajima’sD中性检测分析结果表明,甘肃白银种群(GSBY)、河北石家庄种群(HBSJZ)和河南许昌种群(HNXC)表现显著,山东济南种群(SDJN)、山东临沂种群(SDLY)和山东烟台种群(SDYT)表现极显著,说明这6个地区的葱斑潜蝇种群在较近的历史时期经历了种群扩张,而其他地区的群体表现均不显著。
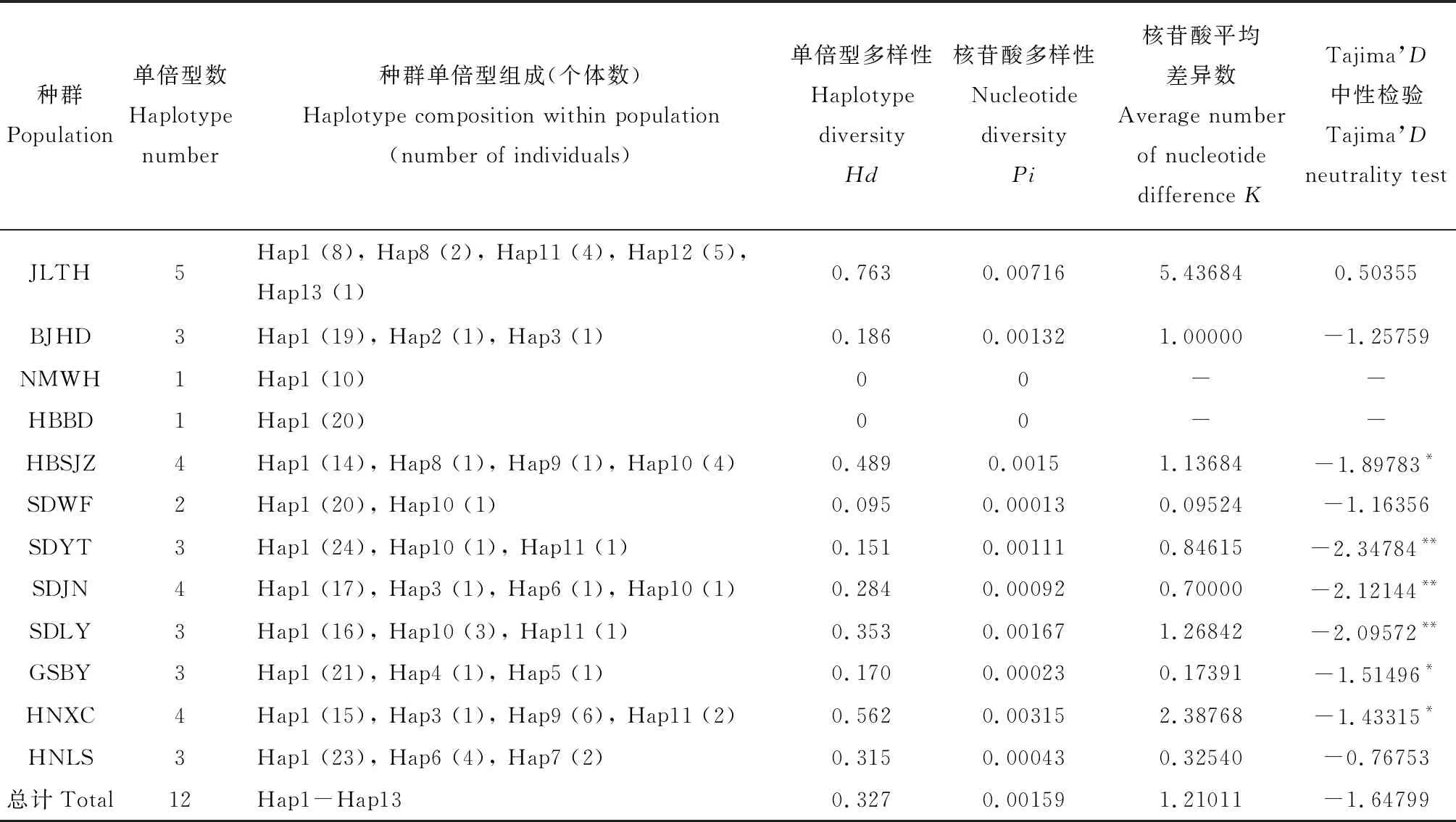
表2 葱斑潜蝇12个不同地理种群基于mtCOI基因序列的遗传多样性分析及中性检验Table 2 Genetic diversity and neutrality test of Liriomyza chinensis among 12 geographical populations based on mtCOI gene sequence
2.3 葱斑潜蝇mtCOI基因单倍型遗传距离及系统进化分析
遗传分化的结果显示(表3),各单倍型间的K2P遗传距离范围为0.001~0.020,单倍型Hap11与其他单倍型遗传距离最大,遗传距离达到了0.012~0.020;其次是单倍型Hap2, Hap3, Hap8 和Hap13,这4个单倍型与其他单倍型之间的遗传距离在0.003~0.019之间;其余7个单倍型之间遗传距离均很接近(≤0.003)。
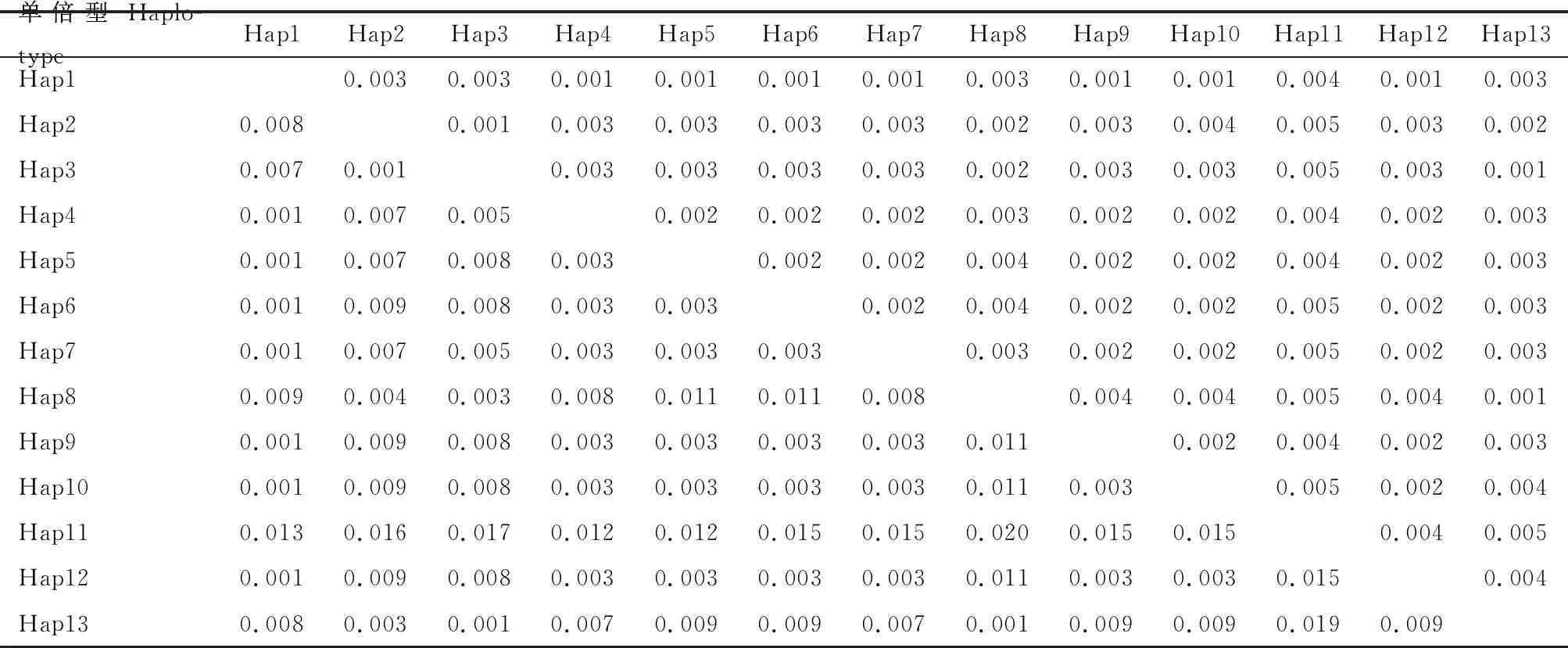
表3 基于葱斑潜蝇地理种群mtCOI基因序列不同单倍型的遗传距离Table 3 Genetic distances of different haplotypes of Liriomyza chinensis geographical populations based on mtCOI gene sequence
从葱斑潜蝇mtCOI基因单倍型系统发生树可见(图1),葱斑潜蝇13种单倍型中,Hap11单独形成一支系,其他12种单倍型聚为另一支系。这12种单倍型聚成的大支又可分为两小支系,第一小支包含Hap2, Hap3, Hap8和Hap13共4个单倍型,第二小支系包含Hap1, Hap4-Hap7, Hap9-10及Hap12共8种单倍型。

图1 UPGMA法构建的基于mtCOI基因序列的葱斑潜蝇地理种群 及近缘种的单倍型系统发育树Fig. 1 Phylogenetic tree of haplotypes of Liriomyza chinensis geographical populations and related species constructed by UPGMA method based on mtCOI gene sequence Hap1-Hap13: 单倍型Haplotypes.
为了进一步直观体现出葱斑潜蝇mtCOI基因单倍型之间的遗传分化及在种群中的分布频率,构建13种单倍型的Network网络关系图(图2)。结果显示,以单倍型Hap1为中心,其他单倍型均围绕这一单倍型呈星形辐射分布状,据此推测分布广泛且分布频次最高的Hap1为祖先单倍型。
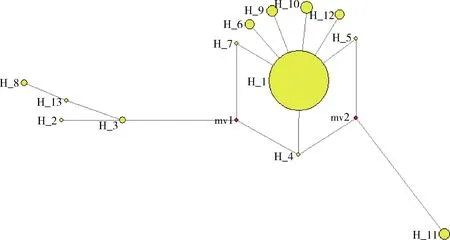
图2 葱斑潜蝇地理种群mtCOI基因单倍型中介网络关系图Fig. 2 Median-joining haplotype network of Liriomyza chinensis geographical populations based on mtCOI gene sequence 每个圆形表示一种单倍型,圆形面积反映出其单倍型在总群体中的出现频率,H_1-H_13分别与单倍型Hap1-Hap13相对应,mv1和mv2表示可能的突变位点。Each circle indicates one haplotype, and the circle size represents the frequency of haplotypes appearing in all populations tested. H_1-H_13 correspond to haplotype Hap1-Hap13, respectively, and mv1 and mv2 indicate possible mutation sites.
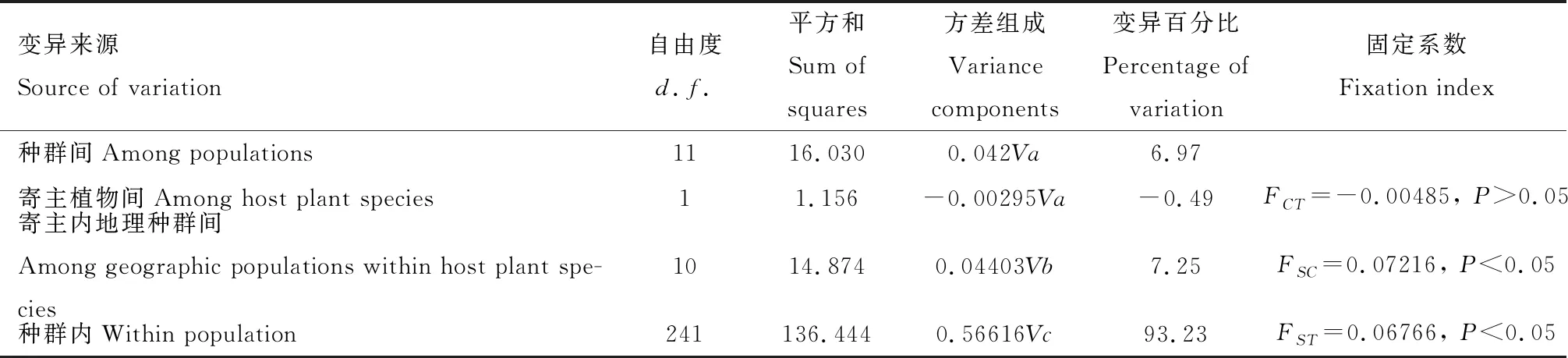
2.4 不同地理种群间的遗传分化、基因交流和分子方差分析
葱斑潜蝇总群体的基因流(Nm)为3.33629, 遗传分化系数(FST)为0.06971, 符合1≤Nm<4, 0.05 表4 葱斑潜蝇成对种群间遗传分化系数(FST, 下三角)与种群间的基因流(Nm, 上三角)Table 4 Pairwise genetic differentiation coefficient (FST, below the diagonal) and gene flow (Nm, above the diagonal) between geographical populations of Liriomyza chinensis 对供试葱斑潜蝇种群进行的分子方差分析(AMOVA)结果(表5)表明,葱斑潜蝇种群内遗传变异显著(P<0.05),占总变异的93.23%,说明遗传变异主要来自于种群内部;种群间遗传变异水平低,仅占总变异的6.97%;不同寄主间(FCT=-0.00485<0.05)表现出较低的遗传分化水平,各寄主植物间的遗传变异不明显(P>0.05);同一寄主不同地理种群间(FSC=0.07216)表现出中等程度的遗传分化,遗传变异显著(P<0.05)。 表5 基于mtCOI基因的葱斑潜蝇种群间分子方差分析Table 5 Analysis of molecular variance (AMOVA) within and among populations of Liriomyza chinensis based on mtCOI gene 对所有种群进行Mantel检验,发现遗传距离与地理距离之间不存在显著的相关性(r=0.3820,P>0.05)(图3)。 图3 葱斑潜蝇12个地理种群间遗传距离与地理距离的相关性Fig. 3 Correlation between the genetic distance and the geographical distance for 12 geographical populations of Liriomyza chinensis FST/(1-FST)表示遗传距离。The genetic distance is expressed as FST/(1-FST). Va,Vb,Vc: 分别表示方差组分的数量Number of variance components;FCT: 寄主种群间变异Variation among host populations;FSC: 寄主种群内变异Variation within host populations;FST: 种群内变异Variation within a population. 线粒体DNA(mtDNA)是常用的分子遗传标记,其中的线粒体细胞色素氧化酶亚基I(COI)基因在昆虫种类鉴定、隐存种的发现、种群遗传多样性、物种的系统进化等研究方面均得到广泛应用(DeSalleetal., 1987; 成新跃等, 2000; Hebertetal., 2003; 何恒果, 2008; Norgateetal., 2009; Yeetal., 2018)。基于mtCOI基因,本研究对我国8个省/直辖市的12个不同地区的葱斑潜蝇种群的遗传多样性、遗传分化程度、基因交流等进行了深入分析。 本研究结果表明,葱斑潜蝇mtDNA COI基因序列片段的碱基组成表现较强的A/T 偏倚性,与典型的昆虫线粒体碱基组成特点(Simonetal., 1994; Wangetal., 2014; Chenetal., 2018)相符。在12个地理种群中共定义了13个单倍型,包括7个共享单倍型和6个独享单倍型,说明不同地理区域的葱斑潜蝇在保持着稳定遗传变异的同时,也产生了适应特定地区环境的遗传变异。在7个共享单倍型中,单倍型Hap1在所有地理种群中均有分布且出现频率最高,同时结合单倍型网络结构图来看,可认为单倍型Hap1为葱斑潜蝇群体中适应性较强并广泛分布的原始单倍型。 本研究中葱斑潜蝇总群体的单倍型多样性较低(表2)。有研究报道,地理距离、寄主植物种类以及生境等是影响昆虫的种群遗传多样性和遗传结构的重要因素(Morganetal., 2000; Scheffer and Lewis, 2006; Tudaetal., 2014)。而本研究Mantel检验结果显示,地理距离与葱斑潜蝇的遗传距离间没有相关性,即地理距离并不影响葱斑潜蝇的遗传分化(图3)。从寄主植物的角度来讲,葱斑潜蝇是寡食性昆虫,仅取食百合科葱属,且主要寄主植物为大葱、小葱,在其他植物上少见发生,种类单一的寄主植物可能在一定程度上限制了葱斑潜蝇种群的遗传分化。在12个地理种群中,仅吉林通化种群的单倍型多样性表现较高(Hd=0.763)(表2)。结合采样地特征分析其原因,吉林通化的葱斑潜蝇为农家自留地采集,与商业化大规模种植的葱相比,化学农药的使用量较少,较低的药剂选择压力可能增加了葱斑潜蝇的遗传变异。总的来说,作者认为葱斑潜蝇在吉林通化可能受到人工选择压力较小以及其他原因,造成了其较高的遗传多样性。 遗传分化系数(FST)和基因流(Nm)是研究种群遗传分化的重要参数,可以解释影响种群发生遗传分化的因素(Rousset, 1997)。Tajima’sD检验可揭示物种在一定历史时期内的种群动态等情况,通过检验值来判断种群是否存在扩张及瓶颈效应(Tajima, 1989)。在本研究中,葱斑潜蝇总群体的遗传分化系数(FST)为0.06971(0.05 本研究仅以mtDNA COI基因长为759 bp的单个片段作为分子遗传标记,为研究葱斑潜蝇的系统进化等提供了基础性遗传资料。本研究发现葱斑潜蝇的遗传多样性水平低,后续还需要增加不同生境类型、寄主植物种类、虫源采集量等数据,才能更深层次地系统阐析不同地理区域葱斑潜蝇的遗传分化。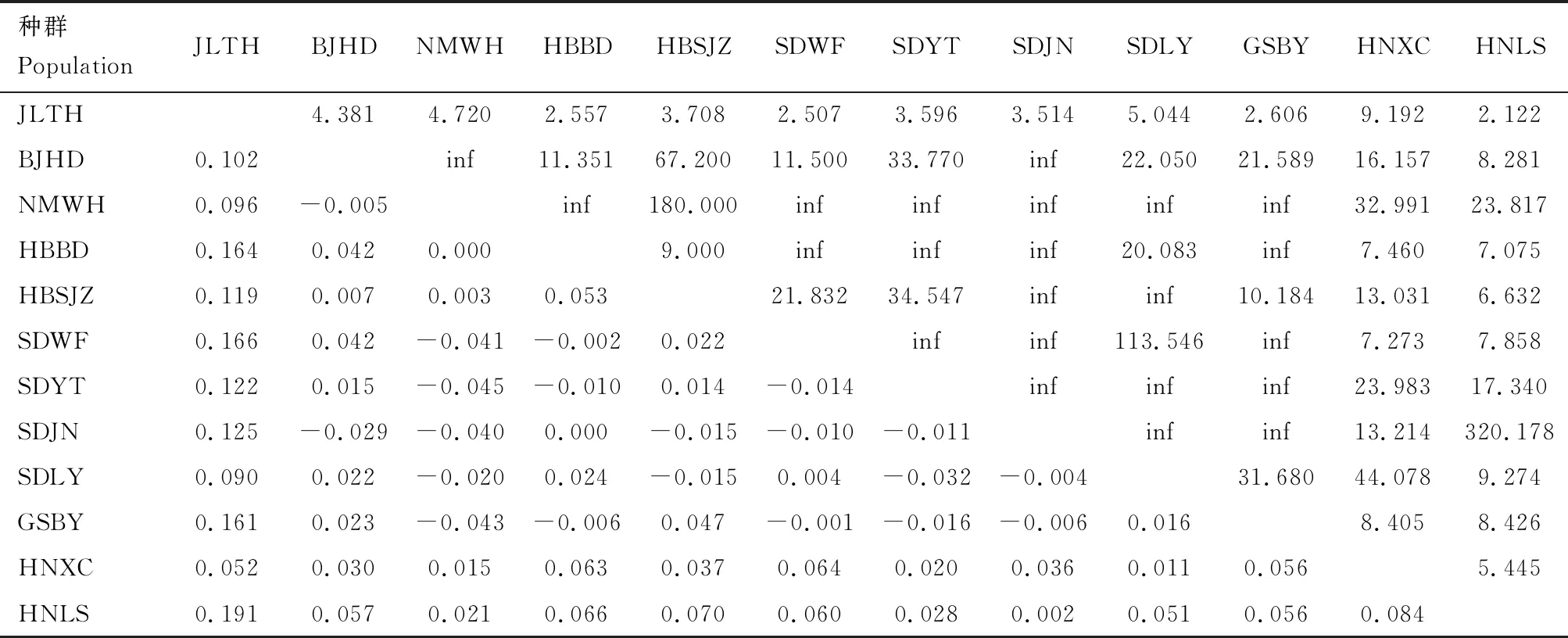

3 讨论
