长链非编码RNA作为食管癌潜在的预后生物标志物的系统评价及荟萃分析△
2020-07-13杨俊强李文耀张策杨义平高社干
杨俊强,李文耀,张策,杨义平,高社干
河南科技大学临床医学院/河南科技大学第一附属医院肿瘤内科/河南科技大学肿瘤研究所/河南省肿瘤表观遗传重点实验室,河南 洛阳4710030
食管癌的发病率居全部恶性肿瘤的第8位,病死率居全部恶性肿瘤的第6位[1]。食管癌的病理类型主要包括鳞状细胞癌和腺癌,中国约90%的食管癌为食管鳞状细胞癌[2]。目前主要通过临床分期和病理分级判断食管癌患者的预后,但相同临床病理特征的食管癌患者也可能出现不同的预后[3]。因此寻找食管癌潜在的预后生物标志物显得尤为重要。长链非编码RNA(long noncoding RNA,lncRNA)是一类转录本超过200个碱基的RNA分子,且不能编码蛋白质[4]。最初lncRNA被当作转录过程中的“噪声”未受到关注。随着研究的不断深入,研究者们发现lncRNA参与了肿瘤细胞的增殖、凋亡、侵袭和转移等生物学过程[5]。因此lncRNA可能在恶性肿瘤的发生发展过程中发挥着重要作用[6]。尽管目前不少研究表明lncRNA可能作为食管癌潜在的预后生物标志物,但这些研究存在样本量小、数据离散等局限性。本研究采用系统评价和荟萃分析的方法,评估lncRNA表达情况与食管癌患者预后的关系,现报道如下。
1 资料与方法
1.1 检索策略
在PubMed、Embase、Web of Science、中国知网和万方数据库中检索相关文献。检索日期截至2019年9月10日。检索关键词为“lncRNA”“食管癌”。
1.2 纳入和排除标准
纳入标准:①病理诊断为食管癌;②lncRNA的检测样本来源于组织或血清;③研究分析了lncRNA表达水平与食管癌患者预后的关系;④具有生存分析相关的风险比(hazard ratio,HR)及95%置信区间(confidence interval,CI)。排除标准:①综述、评论、信件;②不同研究但入组患者相同。
1.3 文献筛选和质量评估
两位研究者根据纳入和排除标准完成文献筛选。Meta分析参考流行病学中观察性研究的Meta分析指南(Meta-analysis of observational studies in epidemiology,MOOSE)。纳入研究的质量评估包括以下6点:①描述入组患者的人种和国家;②具有清晰的研究设计;③具有完整的生存分析;④描述lncRNA的检测方法;⑤报道lncRNA的cut-off值。未满足上述条件的文献将被剔除。
1.4 数据提取
两位研究者进行数据提取工作。提取的数据包括作者姓名、国家、lncRNA名称、患者人种、病例数、病理类型、样本类型、lncRNA检测方法、HR值及95%CI、P值。
1.5 统计学方法
采用Stata 15.0软件对数据进行统计分析。采用Cochran’sQ检验和HigginsI2检验分析异质性。基于合并效应量的异质性,选择固定效应模型或随机效应模型。当异质性明显(I2>50%或P<0.05)时采用随机效应模型,反之则采用固定效应模型。HR值及95%CI用于分析lncRNA表达情况与食管癌患者预后的关系。合并的HR值大于1表示高表达的lncRNA与较差的预后有关。
2 结果
2.1 文献筛选结果
根据上述检索策略,共检出1217篇文献。删除重复文献后剩余606篇文献。
通过浏览标题、摘要和全文,最终筛选出25篇满足纳入、排除标准和质量评估的文献用于后续的系统评价[7-31]。此外,5篇文献用于荟萃分析[7-9,12,18]。(图1)
2.2 lncRNA表达情况与食管癌患者预后的关系

图1 文献筛选流程
纳入的文献共包含3830例食管癌患者,报道了22个与食管癌患者预后相关的lncRNA。其中在食管癌中高表达的为 HOTAIR[7-9]、SPRY4-IT1[11]、MALAT1[12,18]、NEAT1[13]、PCAT-1[14]、ZEB1-AS1[15]、uc002yug.2[16]、 CCAT2[17]、 TUG1[19]、 BANCR[20]、BC200[21]、AFAP1-AS1[22]、ATB[23]、FOXD2-AS1[24]、LINC01296[25]、XIST[26]和 NORAD[27],低 表 达 的 为LOC285194[10]、LINC00675[28]、LINC01133[29]、MEG3[30]和GAS5[31]。在研究文献中,实时定量聚合酶链反应(quantitative real-time polymerase chain reaction,qRT-PCR)用于检测lncRNA的表达水平。大部分文献中,研究者在食管癌与癌旁组织中均检测了lncRNA的表达,1篇文献中,研究者检测了食管癌组织、癌旁组织和血浆中的lncRNA的表达。根据总生存期的HR值及95%CI,食管癌患者中过表达的HOTAIR、SPRY4-IT1、MALAT1、NEAT1、PCAT-1、ZEB1-AS1、uc002yug.2、CCAT2、TUG1、BANCR、BC200、 AFAP1-AS1、 ATB、FOXD2-AS1、LINC01296、XIST和 NORAD预示着较差的生存期。然而,低表达的LOC285194、LINC00675、LINC01133、MEG3和 GAS5预示着较差的生存期。(表 1)
2.3 HOTAIR、MALAT 1表达情况与食管癌患者预后的关系
在纳入的文献中,共有5篇文献多次报道了食管癌患者中HOTAIR、MALAT1的表达情况及患者预后。其中3篇文献报道了食管癌患者中HOTAIR的表达情况及患者的生存情况,Cochran’sQ和HigginsI2异质性检验结果显示,各个研究间无明显异质性(I2=0%,P=0.606)。采用固定效应模型进行荟萃分析,结果显示,HOTAIR高表达与食管癌患者较短的生存期有关(HR=2.404,95%CI:1.661~3.479,P<0.01)(图 2)。2篇文献报道了食管癌患者中MALAT1的表达情况及患者的生存情况,异质性检验结果显示,2个研究间存在异质性(I2=84.1%,P=0.012)。采用随机效应模型进行荟萃分析,结果表明,MALAT1表达水平与食管癌患者的预后无明显相关性(HR=3.384,95%CI:0.948~12.082,P=0.060)(图3)。由于荟萃分析的文献数少于10篇,因此无法评估发表偏倚。
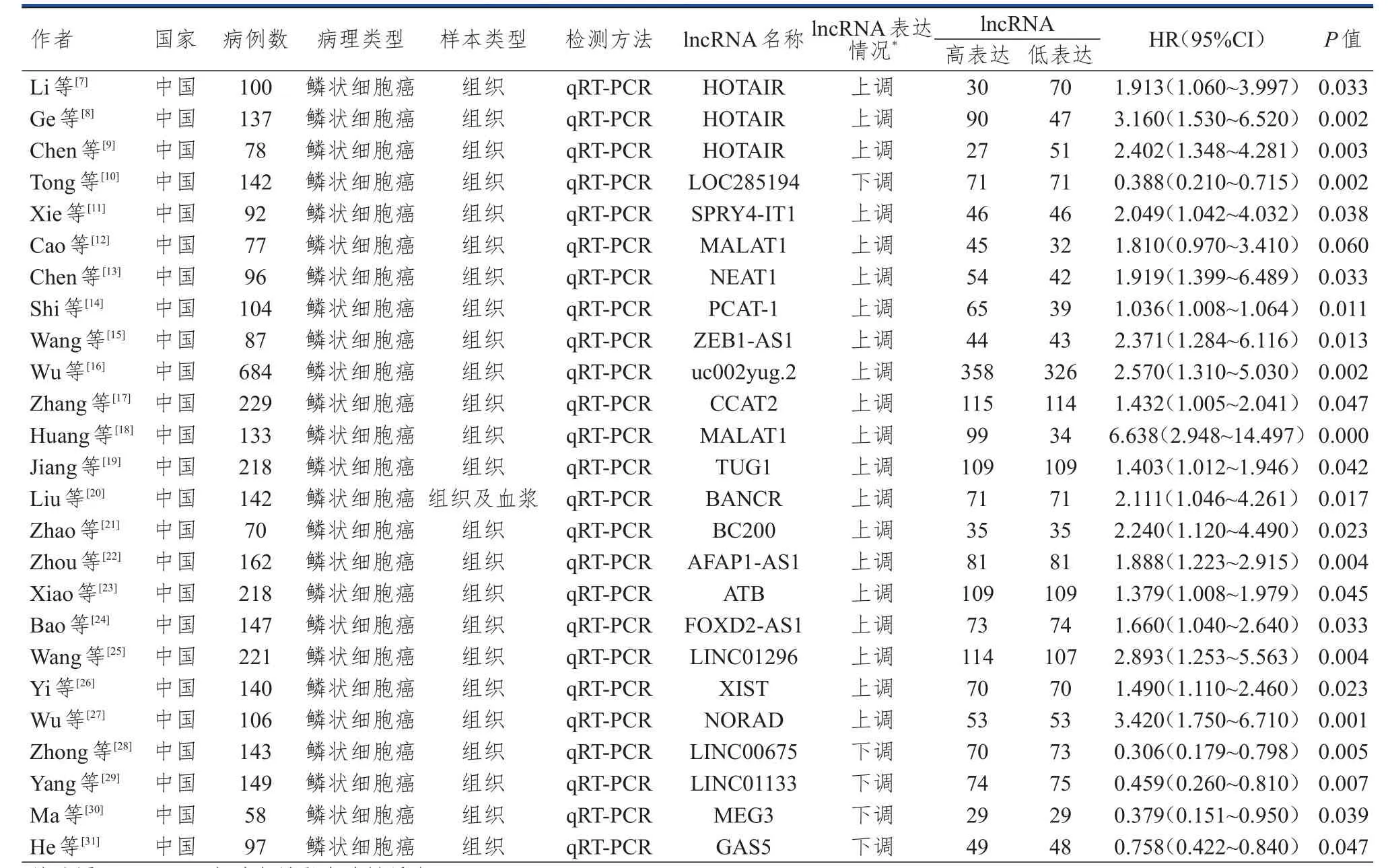
表1 食管癌患者lncRNA表达情况及预后的回顾性研究
3 讨论
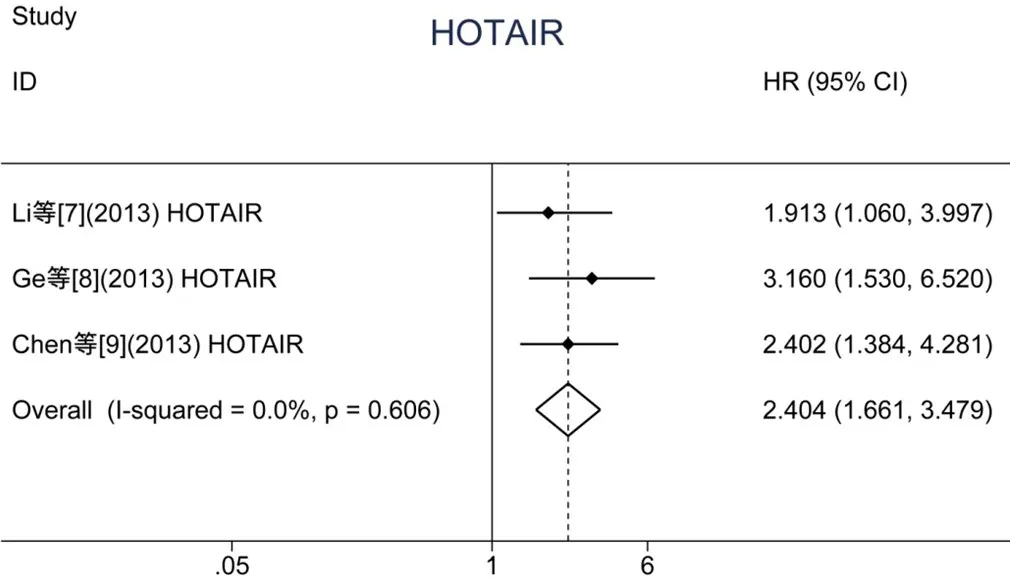
图2 HOTAIR表达与食管癌患者总生存期的森林图

图3 MALAT 1表达与食管癌患者总生存期的森林图
近年来,越来越多的研究证实了lncRNA参与肿瘤细胞的增殖、凋亡、侵袭、转移等过程,并在肿瘤的发生发展过程中扮演重要角色[32]。先前的研究已经报道了一些lncRNA可能与食管癌的预后具有相关性。但由于样本量小等局限性,这些研究的结论可能存在争议。因此,本研究采用系统评价和荟萃分析的方法,评估lncRNA表达情况与食管癌患者预后的相关性。通过检索和筛选共纳入25篇原始文献,包含3830例患者。通过系统评价发现,食管癌患者中高表达的HOTAIR、SPRY4-IT1、MALAT1、NEAT1、PCAT-1、ZEB1-AS1、uc002yug.2、 CCAT2、 TUG1、 BANCR、BC200、AFAP1-AS1、ATB、FOXD2-AS1、LINC01296、XIST和NORAD预示着较差的生存期,低表达的LOC285194、LINC00675、LINC01133、MEG3和GAS5预示着较差的生存期。
虽然经系统评价发现了多个与食管癌预后相关的 lncRNA,但仅有两个 lncRNA(HOTAIR 和MALAT1)及其生存数据可以用于荟萃分析。荟萃分析结果显示,HOTAIR高表达与食管癌患者较短的生存期有关(HR=2.404,95%CI:1.661~3.479,P<0.01)。值得注意的是,尽管Huang等[18]研究显示MALAT1的表达与食管癌患者预后有关(HR=6.638,95%CI:2.948~14.497,P<0.01),但荟萃分析结果表明MALAT1的表达水平与食管癌患者的预后无明显相关性(HR=3.384,95%CI:0.948~12.082,P=0.060)。
HOTAIR可能作为一种促癌基因在多种肿瘤的发生发展中发挥作用[33]。研究表明,在多种肿瘤中高表达的HOTAIR预示着较差的生存期(HR=2.00,95%CI:1.77~2.27,P<0.01)[34],与本研究结果一致。此外,HOTAIR在细胞水平和分子水平上的功能可能解释了HOTAIR与食管癌预后的相关性。研究表明,HOTAIR可以促进食管癌细胞的侵袭和转移,并通过启动子甲基化下调WNT抑制因子 1(WNT inhibitory factor 1,WIF1)的表达,从而活化 WNT/β-联蛋白(β-catenin)信号通路[8]。另一项研究发现,敲除小鼠模型的HOTAIR基因可以抑制食管癌细胞的增殖[35]。
尽管本研究制订了严格的检索策略及纳入和排除标准,但仍存在一些局限性:①由于缺乏相关的研究数据,仅有5篇原始文献中的生存数据可以进行荟萃分析,这可能降低了研究结果的统计学效力;②纳入文献中lncRNA的表达水平的界定存在差异,这可能增加了lncRNA的检测难度;③所有的入组病例均为亚洲人群,且食管癌病理类型均为鳞状细胞癌。但目前亚洲,尤其是中国为世界食管癌的高发区域,且绝大多数为食管鳞状细胞癌,这一不足似乎无法避免。
综上所述,系统评价和荟萃分析表明,多种lncRNA与食管癌患者的预后具有相关性,因此lncRNA可能成为食管癌患者潜在的预后生物标志物,这一结论也需要更多的数据来证明。
