MALDI-TOF MS快速鉴定耐万古霉素屎肠球菌及其流行病学分型性能分析
2020-06-16吴思颖王晓庆刘山泉肖玉玲
吴思颖, 王晓庆, 刘山泉, 肖玉玲, 何 超, 马 莹, 谢 轶, 康 梅
(四川大学华西医院实验医学科,四川 成都 610041)
近年来,肠球菌的耐药率逐渐上升,尤其是耐万古霉素肠球菌(vancomycin-resistantEnterococcus,VRE)的出现,给临床治疗带来很大负担。在临床感染和定植的VRE中,以耐万古霉素屎肠球菌(vancomycin-resistantEnterococcus faecium,VREfm)最为常见[1-2]。目前,VRE的检测主要依靠体外药物敏感性试验,但该方法耗时长,缺乏及时性。分子生物学方法检测耐药基因成本高且操作复杂。建立VRE的快速检测方法,对指导临床治疗和控制细菌耐药性传播至关重要。基质辅助激光解吸电离飞行时间质谱(matrix-assisted 1aser desorption ionization-time of flight mass spectrometry,MALDI-TOF MS)是一种新型的软电离分析技术,不同的细菌经MALDI-TOF MS检测可以形成特异性的指纹图谱,将其与数据库中的指纹图谱进行比对,从而鉴定病原菌,目前已被用于临床实验室微生物的鉴定。除了用于鉴定病原菌,MALDI-TOF MS还被用来识别抗菌药物耐药性以及对菌株进行分型,以确定流行病学相关性。如MALDI-TOF MS用于产碳青霉烯酶细菌的检测和铜绿假单胞菌对黏菌素耐药机制的研究,以及vanB阳性屎肠球菌的流行病学分型[3-6]。本研究旨在评估MALDITOF MS快速鉴定vanA型耐万古霉素屎肠球菌,及对VREfm进行流行病学分型的能力。
1 材料和方法
1.1 研究对象
收集近2015年1月—2018年9月四川大学华西医院66株VREfm临床分离株及73株万古霉素敏感屎肠球菌(vancomycin-susceptibleEnterococcus faecium,VSEfm)临床分离株。
1.2 仪器与试剂
MALDI-TOF MS仪(德国Bruker公司),VITEK-2 Compact 全自动微生物分析系统(法国生物梅里埃公司),Veriti 96-Well PCR扩增仪(美国ABI公司);血琼脂平板、MH琼脂平板(郑州安图公司),万古霉素E-test(英国Oxoid公司),α-氰基-4-羟基肉桂酸(德国Bruker公司),无水乙醇(成都科龙公司),三氟乙酸(德国MERCK公司),乙腈(美国Tedia公司),甲酸(美国Anaqua Chemicals Supply公司)。
1.3 方法
1.3.1 菌株鉴定及体外药物敏感性试验 将待测菌株复苏于血琼脂平板,35 ℃孵育18~20 h。挑选血琼脂平板上的可疑菌落经VITEK-2 Compact全自动微生物分析系统进行鉴定和体外药物敏感性试验,对VITEK-2 Compact 鉴定为VREfm的菌株,经万古霉素E-test 试验确证。
1.3.2 耐药基因检测 参照文献[7]扩增万古霉素耐药基因vanA、vanB、vanC1/vanC2。
1.3.3 MALDI-TOF MS鉴定 MALDI-TOF MS系统采用线性正性模式,采集相对分子质量为2~2 000的图谱。采用甲酸提取法,即取待测菌株,加入300 μL水和900 μL乙醇,混匀(此步骤用于灭活细菌)后加入30 μL 70%甲酸溶液,混匀后再加入30 μL乙腈混匀,离心,取1 μL上清在靶板上点样,晾干后添加1 μL α-氰基-4-羟基肉桂酸,再晾干,应用FlexControl和MALDI Biotyper 3.1软件对139株菌株进行鉴定。具体操作步骤按照仪器说明书进行。
1.3.4 建立模型 保持基本参数不变,应用ClinProTools 3.0软件随机比较45株VREfm和50株VSEfm,分析VREfm和VSEfm的差异蛋白质谱峰。分别选用遗传算法、监督神经网络算法和快速分类算法建立相应的模型。
1.3.5 外部验证 以剩下的21株VREfm和23株VSEfm为实验对象,利用遗传算法模型对其进行分类,软件可自动判断屎肠球菌对万古霉素的敏感性,计算MALDI-TOF MS检测VREfm的敏感性、特异性、阴性预测值和阳性预测值。
1.3.6 多位点序列分型(multilocus sequence typing,MLST) 从66株VREfm中随机挑选21株进行MLST分析。扩增屎肠球菌的7个管家基因(atpA、ddl、gdh、purK、gyd、pstS和adk),扩增方法参见http://efaecium.mlst.net/.。将测得的序列在线提交至E. faeciumMLST website(http://efaecium.mlst.net/),得到21株VREfm的序列分型(sequence type,ST)型别。1.3.7 层次聚类分析 采用ClinProTools 3.0软件对21株VREfm进行层次聚类分析。
2 结果
2.1 屎肠球菌药物敏感性试验结果
21株万古霉素耐药屎肠球菌中,有17株对万古霉素的最小抑菌浓度(minimum inhibitory concentration,MIC)>256,有4株同时对利奈唑胺耐药(MIC≥8);21株均对替加环素敏感。73株万古霉素敏感屎肠球菌中,有65株对万古霉素的MIC≤0.5,有55株同时对环丙沙星、莫西杀星、左氧氟沙星、克林霉素和红霉素耐药;73株均对利奈唑胺和替加环素敏感。
2.2 万古霉素耐药基因携带情况
66株VREfm均含有vanA基因,未发现vanB和vanC1/vanC2基因。
2.3 MALDI-TOF MS鉴定结果
139株临床分离株MALDI-TOF MS鉴定结果均为屎肠球菌,66株VREfm的鉴定分值为2.321~2.521,73株VSEfm的鉴定分值为2.240~2.602。质谱鉴定结果分值均>2,表示可信度非常高,均能鉴定到种的水平。
2.4 ClinProTools 3.0软件分析结果
2.4.1 质谱图统计分析 采用ClinProTools 3.0软件进行质谱图统计之后,将VREfm和VSEfm分成了比较明显的2个区域,大多数菌株都落在相应的VREfm或VSEfm分组中,见图1。质谱图统计分析显示,峰97、20、117、135和85是区分VREfm和VSEfm比较重要的差异质谱峰,峰值统计数据见表1。5个差异质谱峰的受试者工作特征(receiver operating characteristic,ROC)曲线的曲线下面积(area under curve,AUC)分别为0.951 972、0.925 177、0.910 01、0.883 215和0.898 888,表明这些峰在区分VREfm和VSEfm时具有较好的敏感性和特异性,见图2。
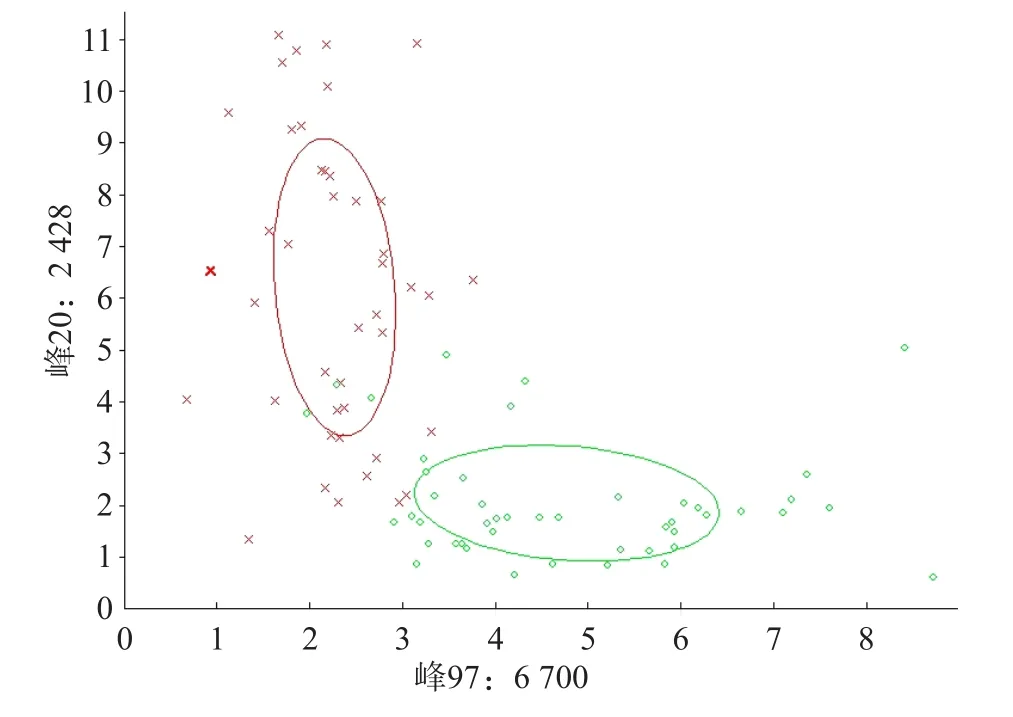
图1 VREfm和VSEfm的二维分布图
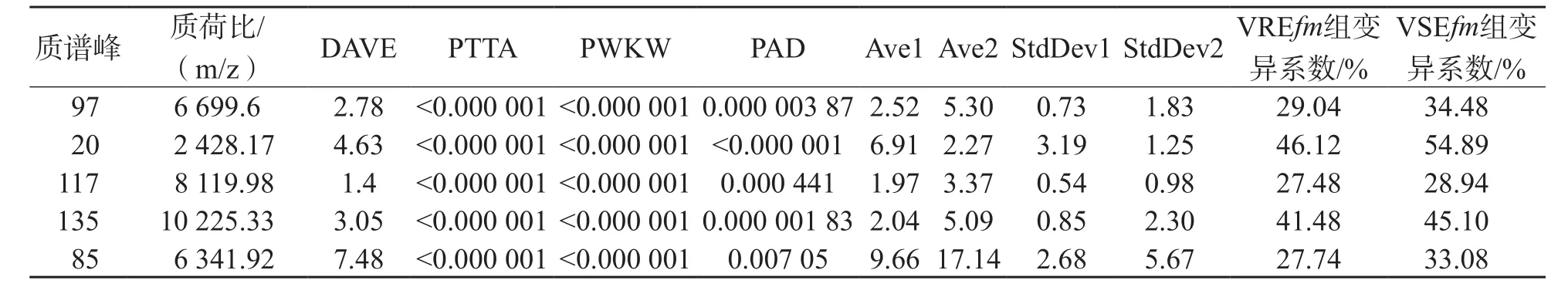
表1 质谱峰的峰值统计数据
2.4.2 3种算法建立模型结果 使用3种算法建立模型,遗传算法和监督神经网络算法具有相似的交叉验证能力(有效敏感性),分别为91.40%和90.89%,识别能力(等同于特异性)均为100%,快速分类算法的交叉验证能力和识别能力最低,分别为82.28%和84.56%。尽管差异不明显,遗传算法模型仍是区分VREfm和VSEfm最可靠的模型。
2.4.3 外部验证结果 采用遗传算法建立的模型对21株VREfm和23株VSEfm进行外部验证。遗传算法的敏感性和特异性分别为95.2%(20/21)和95.7%(22/23)。阳性预测值和阴性预测值分别为95.2%(20/21)和95.7%(22/23)。
2.4.4 MLST和层次聚类分析结果 21株VREfm中有2株未查询到ST型别,其余19株共有6种ST型别,包括ST78、 ST389、ST192、ST64、ST414和ST17。其中以ST78最常见,共12株,ST389和ST192各2株,ST64、ST414和ST17各1株。基于18的任意相似性截断值,将21株VREfm分为4个集群。集群1包含2种ST型别,分别为ST78和ST17;集群2只有1种ST型别,即ST414;集群3有2种ST型别,分别为ST78和ST389;集群4菌株数量最多(16株),其中大部分为ST78(10株),其他分别为2株ST192、1株ST64和1株ST389,2株未查询到ST型别的菌株也被分到这个集群。见图3。
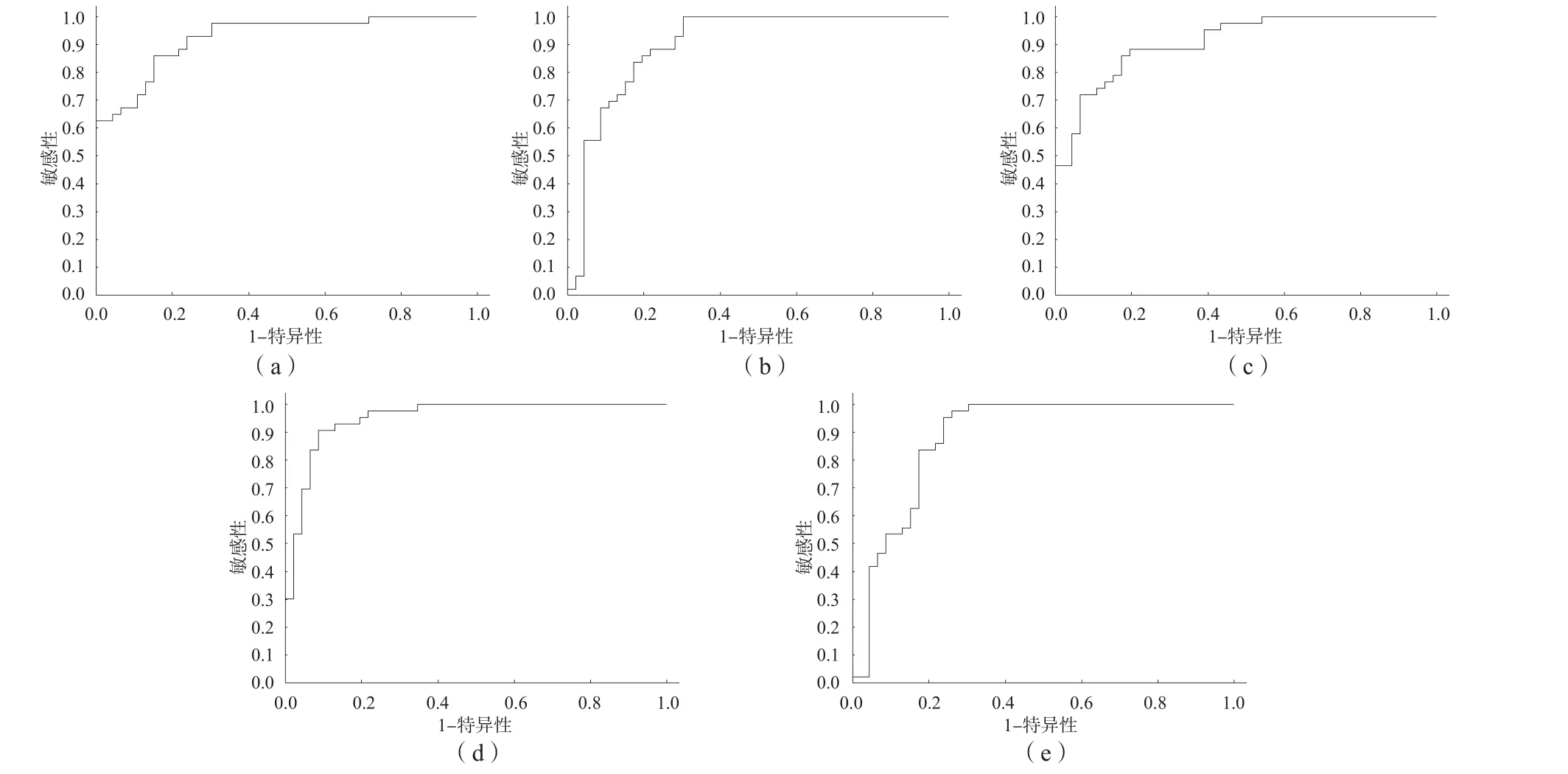
图2 质谱峰ROC曲线
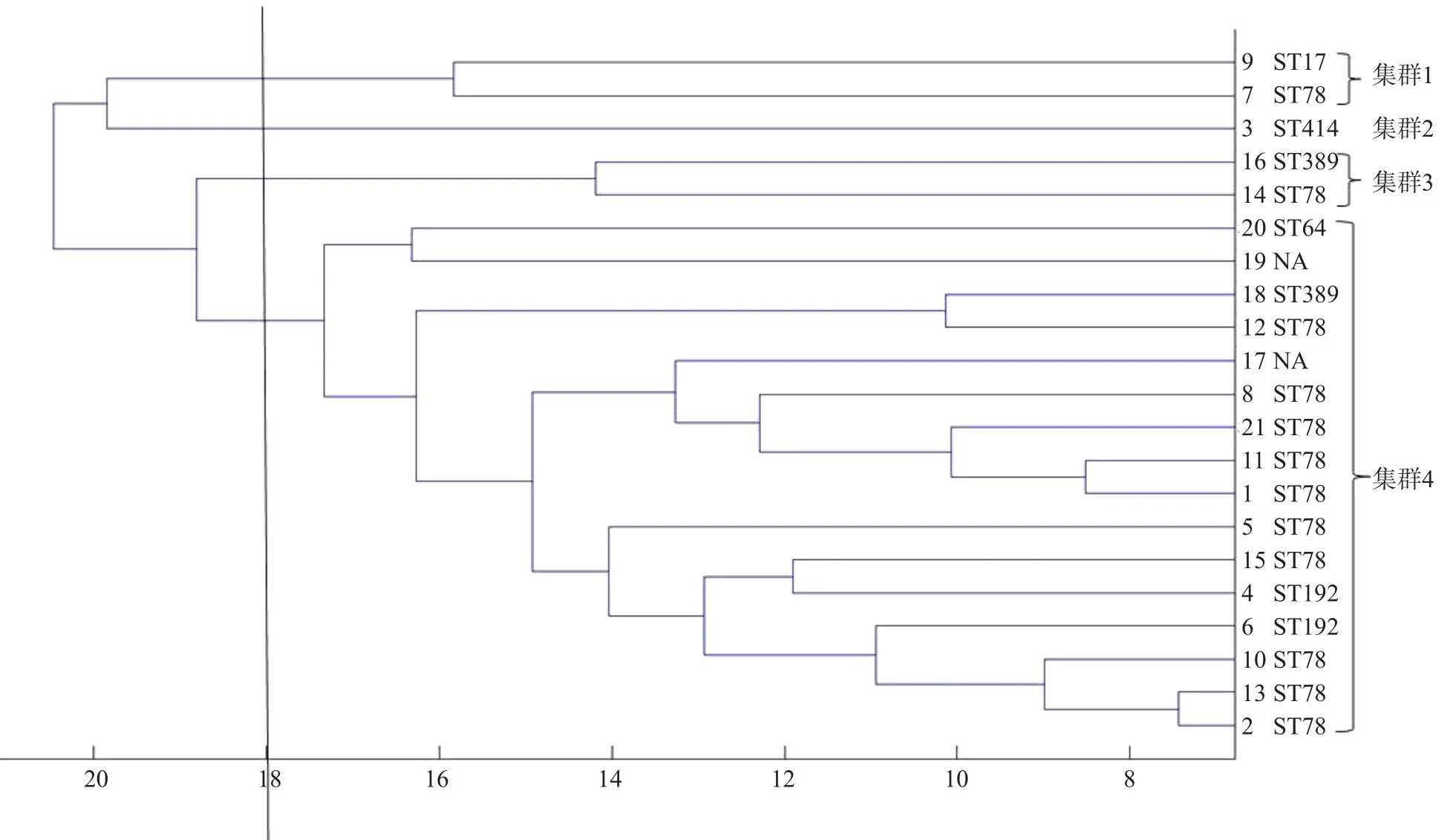
图3 21株VREfm层次聚类分析
3 讨论
目前常用的VRE筛选方法包括chromID VRE显色培养基(法国生物梅里埃公司)、万古霉素药物敏感性试验(VITEK-2、E-test法)以及PCR扩增万古霉素耐药基因。VRE显色培养基主要用于肛拭子样本筛选VRE携带者。药物敏感性试验通常在收到样本后第3或第4天报告阳性结果。分子生物学方法价格昂贵,暂未在临床常规使用。MALDI-TOF MS因具有成本低、高效和快速的特点而被广泛用于常规临床微生物实验室病原菌的鉴定。本研究应用MALDITOF MS快速筛查vanA型VREfm,发现VREfm和VSRfm分成了比较明显的2个区域,区分VREfm和VSEfm比较重要的5个差异质谱峰的P值均<0.000 001,且这5个峰的AUC均在0.9左右,进一步说明这5个峰在区分VREfm和VSEfm时具有较高的准确性。使用3种算法建立模型对VREfm和VSEfm进行分析时发现,遗传算法是区分VREfm和VSEfm最可靠的模型。使用遗传算法建立的模型进行外部验证时,敏感性和特异性均为90%以上。吴薇等[8]对150株屎肠球菌的研究发现,使用遗传算法建立模型进行性能评估,MALDI-TOF MS筛查vanA型VREfm的敏感性和特异性分别为80%和90%,与本研究结果一致。有研究结果显示,MALDI-TOF MS能够快速、准确地鉴定vanB阳性VREfm,在纳入常规实验室工作流程后,前瞻性验证结果显示出较高的敏感性(96.7%)和特异性(98.1%)[3]。
培养基种类和培养条件能影响生物标记分子的出现,从而对质谱峰产生影响[9]。因此,本研究严格控制实验条件,包括培养基、培养时间和温度。采用直接涂布法直接挑取单个菌落涂抹到靶板上,自然晾干后在单菌落涂层上加入1 μL α-氰基-4-羟基肉桂酸,虽然简单快速,能够用于大多数细菌的常规鉴定,但发现进行MALDI-TOF MS分析时,使用直接涂布法对革兰阳性菌细胞壁肽聚糖的物理破坏并不能充分提取蛋白质,且会因代谢物、色素和/或琼脂干扰结晶过程而降低质谱的分辨率[10-11]。甲酸提取法包括裂解细菌细胞,将蛋白质释放到提取物中,然后将其应用于MALDI-TOF MS分析,克服了直接涂布法的缺点。陆文香等[12]发现,蛋白提取步骤获取质谱图进行外部验证实验时,MALDI-TOF MS对VREfm和VSEfm菌株的鉴定正确率相对较高。
MALDI-TOF MS可应用于碳青霉烯类耐药肺炎克雷伯菌以及引起医院暴发流行的弗劳地枸橼酸杆菌和黏质沙雷菌的分型,并且与MLST和全基因组测序(whole genome sequencing,WGS)分型结果具有较好的一致性[13-14]。MALDI-TOF MS用于屎肠球菌菌株分型的研究结果各不相同。在GRIFFIN等[3]的研究中,当使用2.5的相似性截断值时,MALDI-TOF MS与脉冲场凝胶电泳的结果相似。而LASCH等[15]将MALDI-TOF MS与MLST分型结果进行比较时,却无法推断出可靠的相关性。SCHLEBUSCH等[16]应用MALDI-TOF MS对引起医院暴发流行的VREfm进行分析,并与WGS进行了比较,MALDI-TOF MS分析显示VREfm暴发分离株的蛋白质组谱不完全相似,存在不同的集群,不是由单个菌株引起的暴发,使疫情的早期调查和管理得到了帮助,但如果需要更清楚地确定分离株之间的进化关系,以确认和进一步调查疫情,则可以使用详细基因组分析,如WGS。造成以上研究结果差异的原因可能是用于分析数据的软件和模型不同。另外,MALDI-TOF MS易受技术和生物变异的影响,因为结果是基于峰值强度,而峰值强度又与细菌基因表达和调控有内在联系。
本研究层次聚类分析结果显示,绝大多数ST78的菌株被归于同一集群,而和ST78归于同一集群的还有ST192、ST389和ST64,2株未查询到ST型别的菌株也被分到这个集群。ST78与ST192、ST64,ST17以及未查到的ST型别均只有1个等位基因的差异(single-locus variant,SLV),因此MALDI-TOF MS对于高度相关的ST型别,例如SLV,几乎不能区分。FREITAS等[17]研究发现,应用MALDI-TOF MS可区分屎肠球菌高风险克隆株(ST17、ST18和ST78等)和来自社区的克隆株(ST5、ST147和ST185)。本研究中的屎肠球菌均分离自医院,均属于CC17克隆复合体,即高风险克隆,后续需收集更多的菌株,特别是来自社区的分离株加以验证。
