基于SRAP标记的紫溪山华山松种子园无性系遗传多样性分析
2020-05-26刘成徐剑罗正平董云祥董章宏瞿绍宏李显煌辛培尧
刘成 徐剑 罗正平 董云祥 董章宏 瞿绍宏 李显煌 辛培尧
摘 要: 为明确云南楚雄市紫溪山华山松种子园内不同种源无性系间的遗传背景,该研究收集了园内6个种源的60个华山松无性系单株针叶,采用改良CTAB法提取其总DNA,并利用SRAP分子标记对其进行遗传多样性分析。结果表明:(1)从100对引物组合中共筛选出15对具有多态性的SRAP引物,经SRAP-PCR扩增后,共获得出194个位点,多态位点百分率(PPB)为85.05 %,Nei′s 基因多样性指数(H)为0.233 7,Shannons信息指数(I)为0.341 9,种源间的遗传分化系数(GST)为 0. 355 5。(2)华山松6个种源遗传多样性较高,且遗传变异主要存在于华山松种源内,种源地会泽(HZ)与巍山(WS)种源的遗传距离最近(D=0.050 1),会泽(HZ)与宜良(YL)种源的遗传距离最远(D=0.361 8)。(3)聚类分析显示将6个华山松种源一共聚为3类:会泽(HZ)和巍山(WS)种源聚为一类;楚雄(CX)、南华(NH)和宜良(YL)種源聚为一类;腾冲(TC)种源单独为一类。综合上述结果显示,紫溪山华山松种子园内无性系间的遗传分化处于较高水平,为华山松杂交育种时亲本的选配及种质资源的评价提供了分子水平的理论依据。
关键词: 华山松, 种子园, 无性系, SRAP, 遗传多样性
中图分类号: Q943 文献标识码: A
文章编号: 1000-3142(2020)04-0462-09
Abstract: In order to clear genetic background of different provenances Pinus armandii colnes from seed orchard in Zixi Mountain of Chuxiong City, Yunnan Province, the needles of 60 clones were taken from six provenances which was used to extract total DNA by modified CTAB method, and the genetic diversity was analyzed by SRAP molecular markers. The results were as follows: (1) Fifteen pairs of polymorphic SRAP primers were screened from 100 pairs of primer combinations. After SRAP-PCR amplification, a total of 194 loci were obtained, and the percentage of polymorphic loci PPB=85.05%, Nei′s gene diversity index H=0.233 7, Shannons information index I=0.341 9, genetic differentiation coefficient between population GST=0.355 5. (2) The genetic diversity of 6 provenances of P. armandii was high, and the genetic variation was mainly existed within provenances of P. armandii. The genetic distance between the Huize (HZ) provenance and the Weishan (WS) provenance was the closest (D=0.050 1), that the Huize (HZ) provenance and Yiliang (YL) provenance was the farthest (D =0.361 8). (3) The cluster analysis showed that six P. armandii provenances there were classified into three categories: Huize (HZ) and Weishan (WS) provenances were in one category; Chuxiong (CX), Nanhua (NH) and Yiliang (YL) provenances were in the second category; Tengchong (TC) provenance was a separate category. The results indicate that genetic differentiation in P. armandii clones from seed orchard in Zixi Mountain of Chuxiong City has a high level, and it can provide theoretical information and practical guidance for parent selections during the cross breeding and evaluation of germplasm resources of P. armandii as well.
Key words: Pinus armandii, seed orchard, clone, SRAP, genetic diversity
华山松(Pinus armandii)是松科(Pinaceae)松属(Pinus)单维管束亚属的植物(陈有民,1990; 袁红宁和陈文辉,2010),是我国特有的五针松高大乔木,同时也是高海拔地区重要的造林树种之一(赵杨等,2003;辛培尧等,2010)。20世纪70年代起,云南和贵州等地相继对华山松进行遗传改良,各自营建了数个优良无性系初级种子园,之后对其进行了遗传测定、优良家系选择及辅助授粉等工作(王秀荣等,2007;翟思万等,2007)。紫溪山华山松无性系种子园位于楚雄市紫溪山,为国家级林木良种基地。由于缺乏对园内种质的不断改良,该种子园仍然处于建园初期低级阶段,继而出现病虫害严重及生产力低下的问题,因此,急需对园内种质进行改良和创新。已有实践证实,杂交育种是迄今为止对植物不良性状进行遗传改良的最为有效的方法之一。多年来,华山松的杂交育种工作较为滞后,杂交亲本的选配多以形态及种源地为主要参考依据,不能明确所收集的种质资源间遗传多样性的丰度及亲缘关系的远近,从而影响改良的程度及效果。因此,在种子园内进行华山松杂交育种时,需有针对性地明确园内种质的遗传多样性程度及各种质间的亲缘关系显得尤为重要。随着分子生物学研究的不断深入及相关领域技术水平的进步,华山松遗传多样性的检测方法不断得到完善。例如:赵杨等(2012)利用ISSR标记对贵州平坝华山松无性系种子园内的85个无性系进行遗传多样性分析后发现,其遗传变异主要在种源内;祝娟(2016)利用SSR分子标记对华山松52个自然分布群体进行遗传多样性分析,结果表明遗传变异主要存在于个体内,且其遗传多样性水平较高;朱晓丹(2006)利用RAPD分子标记对紫溪山华山松种子园内的部分无性系进行了遗传多样性研究,结果表明各种源之间具有较大的遗传差异,且遗传多样性较丰富。但由于RAPD(Random Amplified Polymorphic DNA)标记相对较早,且与新型分子标记比较,存在一定的缺陷(阮桢媛等,2016),其研究结果可能存在一定的局限性。上述相关研究为华山松的遗传改良及种质资源的分子评价提供了重要的理论基础。
在众多的分子标记中,SRAP(Sequence-related Amplified Polymorphism)凭借其多态性高、操作简单、易于分离测序,且多数标记在相关实验样本基因组中相对均匀分布的特点(Li et al., 2001;Tilman et al., 2006),目前在较多的林木遗传多样性分析中得到广泛应用(张冬东,2008;向晖等,2016;陈慧等,2017;孟慧等,2018;张安世等,2018;姜武等,2019)。赵杨等(2012)对贵州平坝华山松种子园无性系进行了SRAP-PCR反应体系的优化,但未对其遗传多样性进行具体分析。目前,对华山松遗传多样性进行SRAP分析的相关研究,国内外鲜见报道。
针对云南省楚雄市紫溪山华山松种子园的生产现状,实验选取6个种源共60个华山松优良无性系,开展了以杂交育种及种质资源评价为最终目的的华山松SRAP遗传标记分析,研究结果可为该种子园内华山松种质资源的评价以及杂交育种时亲本的选配提供分子水平的理论与实践指导。
1 材料与方法
1.1 材料来源
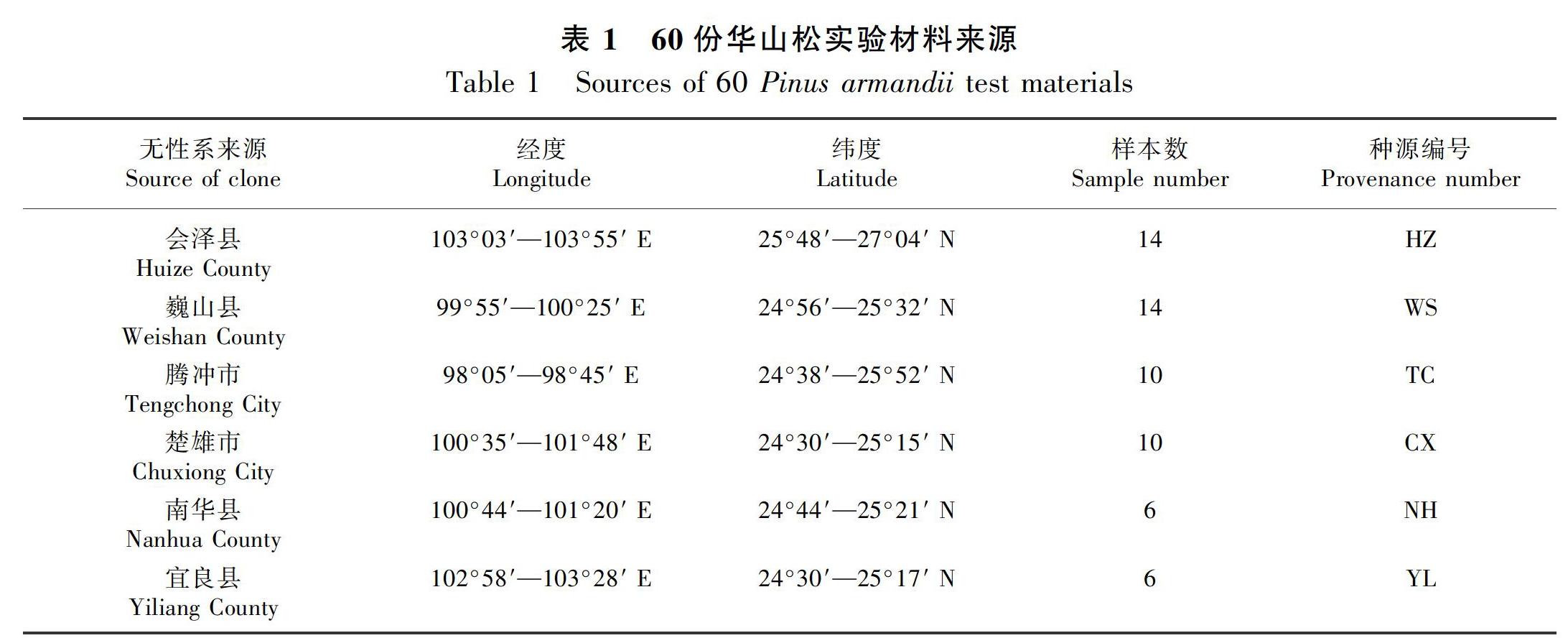
材料采集于云南省楚雄市紫溪山华山松种子园内60个无病虫害的不同无性系新鲜幼嫩针叶,分别按无性系单株编号后装入自封袋,带回实验室后置于 -80 ℃冰箱,备用。所有材料均来自云南省内的6个种源(表1)。
1.2 总DNA提取
取4~5根松针,采用改良的CTAB法,提取华山松基因组DNA(谢冬梅等,2014)。加入50 μL TE buffer稀释所提取的DNA至50 ng·μL-1,然后用1%的琼脂糖凝胶电泳进行检测,并保存于-20 ℃冰箱中。
1.3 SRAP-PCR擴增
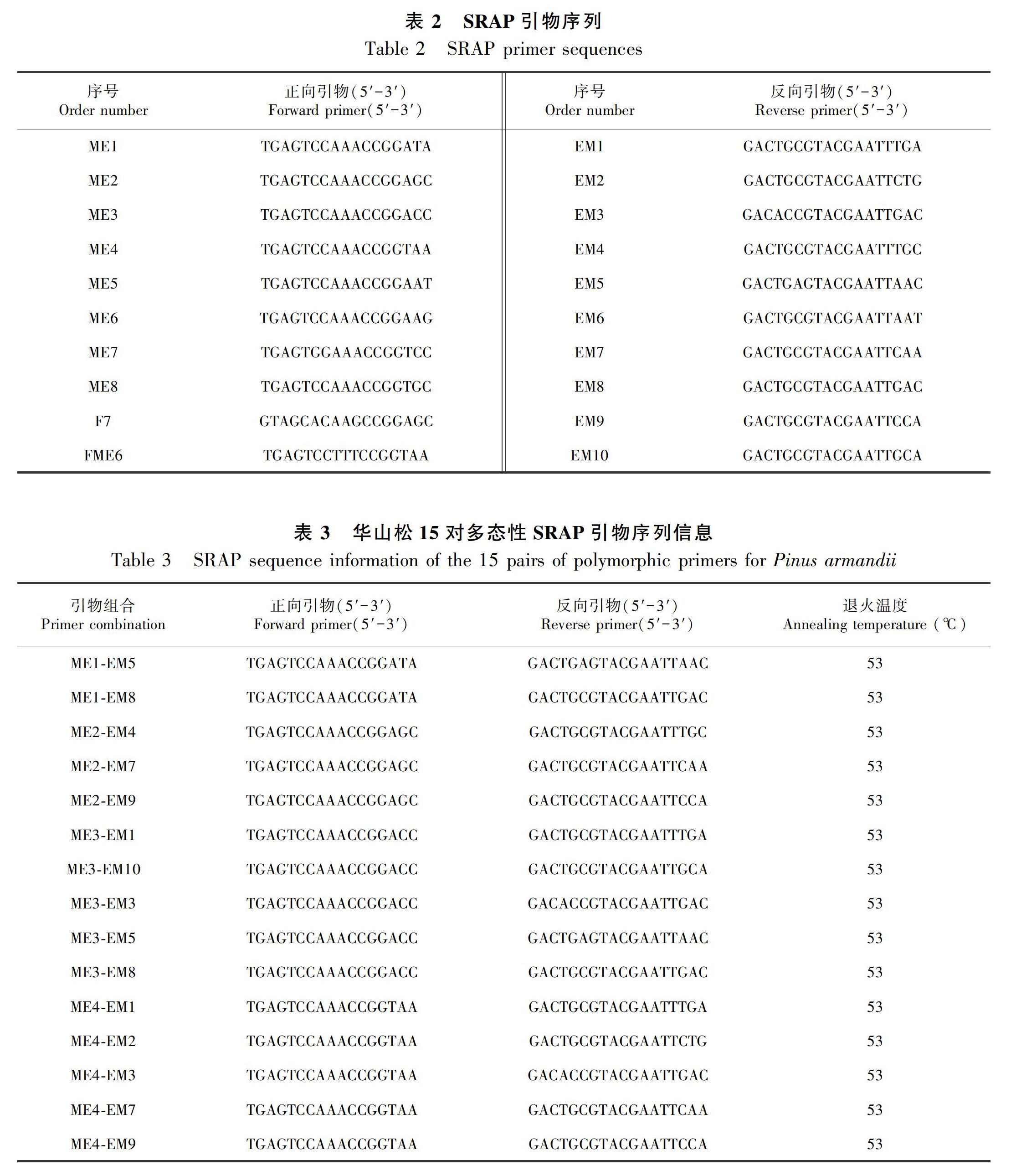
实验扩增所用引物序列来自公开发表的相关报道(李正鹏等,2011;蔡元保等,2014)。选择正向和反向引物各10条,随机组合后获得100对引物(表2)。经初筛和复筛后成功筛选出15对多态性较好且条带清晰的SRAP引物(表3)。华山松SRAP-PCR 25 μL反应体系:DNA 模板1 μL、Green Taq Mix12.5 μL、ddH2O 9.5 μL、正向和反向引物各1 μL。其PCR扩增反应程序:94 ℃预变性5 min;94 ℃变性1 min,37 ℃退火1 min,72 ℃延伸1 min,5个循环;94 ℃变性1 min,53 ℃退火1 min,72 ℃延伸1 min,30个循环;72 ℃延伸10 min;4 ℃保存。
1.4 聚丙烯酰胺凝胶电泳
吸取6 μL PCR扩增产物和2 μL 6×Loading buffer混合后,加入到8%聚丙烯酰氨凝胶孔中进行电泳检测,电压设为230 V,电泳时间为150 min。电泳结束后进行凝胶的固定、染色和显影,拍照保存检测结果。
1.5 数据处理
将扩增得到清晰可辨的多态性条带, 作为参考带型,根据实验得到的聚丙烯凝胶电泳图,同一位点清晰的条带,记“1”;无带或者弱带记“0”,模糊不清的不予计数,从而构成(0,1)矩阵,调整格式以符合软件分析要求。
利用Excel表对60株华山松进行多态位点数、多态性百分比统计计算;通过POPGENE version 1. 32分析软件进行遗传多样性分析[观测等位基因(Na)、有效等位基因数(Ne)、基因多样度(H)等参数];利用NTSYS 2.10软件对6个种源的华山松进行聚类,形成聚类图。
2 结果与分析
2.1 华山松基因组DNA提取结果
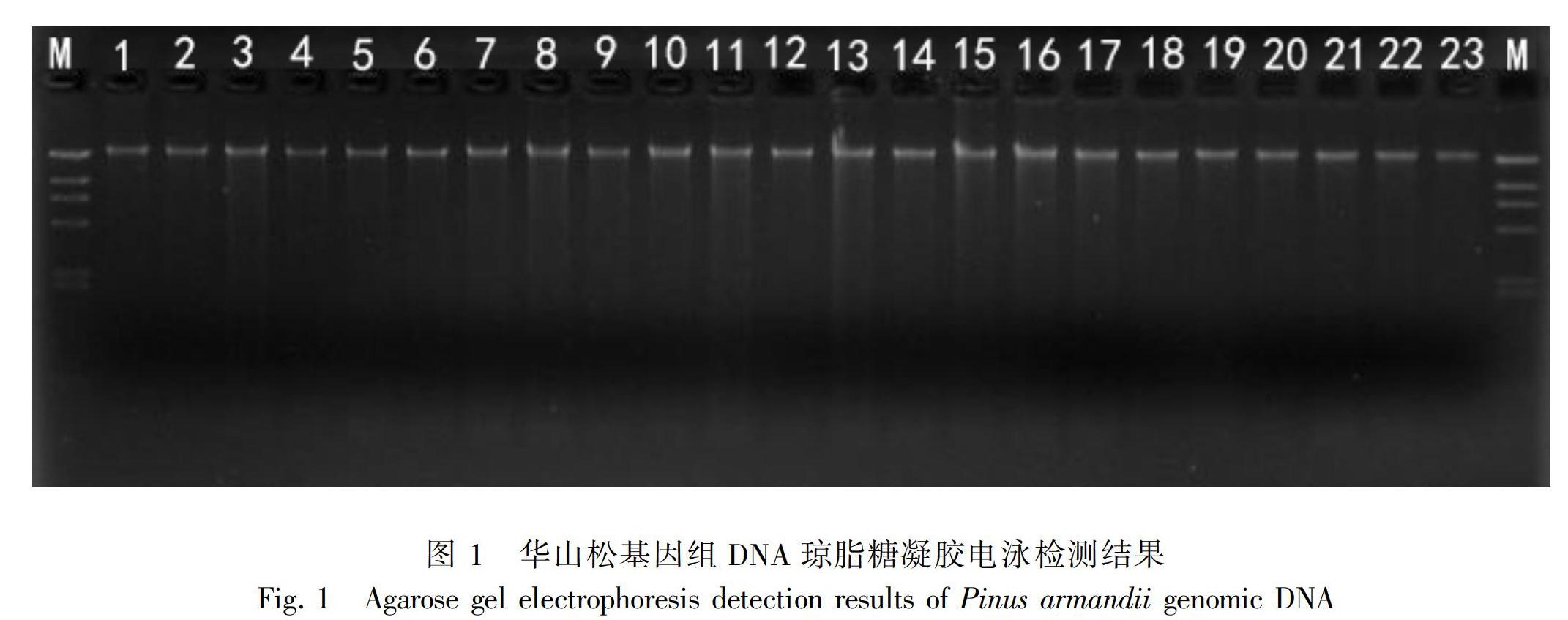
将华山松基因组DNA用1%的琼脂糖凝胶检测,部分结果如图1所示,DNA条带清晰且无拖带和弥散现象,可满足后续实验的要求。
2.2 SRAP-PCR扩增结果
用15对具有多态性的引物对6个种源共60株不同华山松无性系进行PCR扩增反应,共扩增出165个多态性位点,其结果列于表4。
从表4可以看出,在各种源间,华山松多态位点百分率(PPB)为 85.05%;在各种源内,各个种源的PPB为41.24%~81.96%,平均值为59.37%。PPB最低的是宜良(YL)种源,为41.24%;PPB最高的是腾冲(TC)种源,为81.96%。把华山松6个种源根据PPB大小进行排序:腾冲(TC)>会泽(HZ)>巍山(WS)>楚雄(CX)>南华(NH)>宜良(YL)。
2.3 遗传多样性分析
华山松6个不同种源的Nei′ s基因多样性(H)范围为0.187 3~0.323 4,楚雄(CX)的最低,为0.187 3,腾冲(TC)的最高,为0.323 4。Shannons信息指数(I)范围为0.277 0~0.471 6,楚雄(CX)的最低,为0.277 0,腾冲(TC)的最高,为0.471 6。等位基因数(Na)最高的是腾冲(TC),为1.819 6,最低的是宜良(YL),为1.142 4,有效等位基因数(Ne)最高的是腾冲(TC),为1.580 9,最低的是宜良(YL),为1.269 1。6个种源的H和I均表明,各种源的遗传变异从大到小依次为腾冲(TC)>会泽(HZ)>巍山(WS)>南华(NH)>宜良(YL)>楚雄(CX)(表5)。
2.4 遗传分化分析
由Nei′s指数估算可知,6个华山松种源的总基因多样度(Ht)为0.342 0,种源内基因多样性(Hs)为0.220 4。遗传分化系数(GST)为0.355 5,说明在华山松种源间有35.55%的遗传变异, 而在华山松种源内则有64.45%的遗传分化,体现了华山松种源内的遗传分化远大于华山松种源间的遗传分化。华山松各种源间的基因流(Nm)为0.906 5<1,表明华山松各种源间的基因交流较少。
2.5 遗传相似度与遗传距离分析
为了进一步分析华山松种源间的遗传关系,对华山松种源的遗传相似度和遗传距离进行分析,其结果如表6所示。由表6可知,华山松6个种源的遗传相似度在0.696 4~0.951 1之间,平均值为0.814 1;遗传距离在0.050 1~0.361 8之间,平均值为0. 212 3。其中,遗传距离最大的是宜良(YL)和会泽(HZ)(D=0.361 8),而它们之间的遗传相似度相对较小(Gs=0.696 4),说明宜良(YL)和会泽(HZ)之间的亲缘关系最远;会泽(HZ)与巍山(WS)之间的遗传距离最小(D=0.050 1),它们之间的遗传相似度最大(Gs =0.951 1),这表明会泽(HZ)与巍山(WS)的遗传差异较小,亲缘关系最近。
2.6 群体间遗传关系聚类分析
根据6个华山松种源遗传相似度,对华山松种源进行聚类分析,其结果如图2所示。以0.87为阈值,华山松6个种源被分为三个类群。其中,会泽(HZ)和巍山(WS)种源分为一类;楚雄(CX)、南华(NH)、宜良(YL)种源被聚为一类;腾冲(TC)种源单独聚为一类。
3 讨论与结论
遗传分化系数(GST)是衡量种群基因分化程度的重要指标,能较好地反映群体的遗传分化水平。按照GST的大小可分为4种情况:GST在0~0.05之间时,种群的遗传分化水平基本不存在;GST在0.05~0.15之间时,种群遗传分化水平为中等水平;GST在0.15~0.25之间时,种群遗传分化水平较高;GST大于0.25时,种群遗传分化水平相当高(孙文婷等,2016;蒋谦才等,2018)。本实验结果表明,华山松6个种源的总基因多样度(Ht)为0.342 0,种源内基因多样性(Hs)为0.220 4,GST为0.355 5,其遗传变异达35.55%,在华山松种源内有64.45%的遗传分化,体现了华山松种源内的遗传分化远大于华山松种源间的遗传分化,说明这6个种源的遗传分化处于较高的水平。这和Wheeler & Jech(1992)研究发现种子园不会因为群体数量有限可能导致遗传多样性较天然种群低相一致。此外,朱晓丹(2006)曾对楚雄华山松种子园4个种源不同无性系进行RAPD遗传多样性分析,其PPB为71.42%,Ne为1.364 9,H为0.140 4,I为0.219 3,说明这4个种源64个个体间的遗传差异较大,该种子园遗传多样性较丰富。本实验利用与朱晓丹(2006)所研究的同一种子园内材料进行SRAP遗传多样性分析,得到多态位点百分率(PPB)为85.05%,Ne为1.386 6,H为0.233 7,I为0.341 9,各项参数均有显著提高,且主要遗传变异也存在于种源内。由于RAPD标记本身具有重复性低,扩增产物不稳定,特异性不强等缺点,该实验结果可能有一定的局限性,而SRAP标记,多态性强、标记分布均匀且比较稳定(周延清,2008)。因此,相比RAPD标记可以获得更加丰富且可靠的遗传变异信息,同时,种子园内各单株间不同程度的遗传变异,也有利于在园内更加方便地就地进行华山松的遗传改良工作。
在生物自然进化中,基因流(gene flow)有助于提高物种种源的遗传多样性水平,防止种群分化(Ellstrand et al., 1993)。Widen & Svensson(1992)统计了124种植物的基因流(Nm)认为,遗传漂变对种群分化的影响较大。而Wright(1950)认为当基因流Nm<1时,遗传漂变是导致种群之间存在明显遗传分化的主要原因。有学者还指出,如果植物种群的基因流(Nm>0.5)从原来的较高水平,到后来呈现较大下降趋势,则猜想原因可能是植物生存的环境斑块化、传粉的减少、自然选择作用的加强等降低了基因流(杨章旗等,2014)。本研究结果表明,紫溪山华山松种子园内无性系Nm= 0.906 5<1,说明供试华山松6个种群间基因交流水平相对较低,华山松的遗传变异主要存在于种源内,即种源内的不同优良单株之间的遗传差异较大。那么,在杂交育种时,可在种源内进行亲本的选配,这样不仅有利用于提高其杂交亲合性,同时还可丰富杂种后代的基因型,从而更有利于人工选择。
在进行植物遗传多样性分析时,遗传距离是一个用来反映物种和种源间遗传变异水平及遗传分化程度的重要指标,可为杂种优势提供基本遗传参数(睢鑫洲等,2017)。利用SRAP分子标记技术对6个华山松种源共60个单株样本进行聚类分析发现:在阈值为0.95时,可以分成五类,其中,会泽(HZ)和巍山(WS)种源最先聚为一类,楚雄(CX)、南华(NH)、腾冲(TC)、宜良(YL)4个种源各自被聚为一类,而在阈值为0.87时,6个种源被聚为三类,其中腾冲(TC)单独聚为一类;会泽(HZ)和巍山(WS)种源分为一类;南华(NH)、宜良(YL)和楚雄(CX)三个种源被聚为一类,说明会泽(HZ)和巍山(WS)之间的亲缘关系较近。由于种源间的亲缘关系与其分布的地理种源存在相关性。一般认为,地理位置相近的种源其遗传基础也较为接近。实验结果表明,曲靖会泽(HZ)种源与大理巍山(WS)种源就被聚为一类,显然这种聚类与物种地域性的分布不相符。鄢家俊等(2009)研究老芒麦遗传多样性时也被发现这一现象。而对裸果木利用SCoT标记进行遗传多样性分析时发现,其种群地理距离与遗传距离也不存在显著相关性(狄林楠,2018)。研究认为,植物群体间当前的遗传构成不仅与目前的分布地理距离有关,还可能和该植物的进化历史和各种群受人为干扰程度等各种因素有关(刘柳,2012;宋春凤等,2014;狄林楠,2018)。因此,在进行植物杂交育种中,以目前两亲本地理位置的远近为选配亲本的主要標准,显然存在较大的局限性。在今后的育种工作中,应以DNA水平分析的结果为主要依据,结合形态,地理位置等综合评判,最后确定较为理想的亲本组合。通过杂交,从其子代中进行选择,有望获得更多的具有优异性状(如抗病虫害能力的提高、速生性等)的优良个体,再通过无性扩繁的手段对其优良性状加以固定,最终获得良种,继而在生产上大面积推广。
参考文献:
CAI YB, YANG XY, CHEN HJ, et al., 2014. Analysis of genetic diversity of Papaya germplasm by SRAP combined with SCoT marker [J]. J Plant Gene Resour, 15(2): 292-298. [蔡元保, 杨祥燕, 陈豪军, 等, 2014. SRAP结合SCoT标记分析番木瓜种质的遗传多样性 [J]. 植物遗传资源学报, 15(2): 292-298.]
CHEN H, QIE HL, HUANG YH, et al., 2017. Genetic diversity analysis of 66 Chinese Bayberry germplasm resources with SRAP [J]. S Chin Fruits, 46(4): 64-67. [陈慧, 郄红丽, 黄颖宏, 等, 2017. 66份杨梅种质SRAP标记遗传多样性分析 [J]. 中国南方果树, 46(4): 64-67.]
CHEN YM, 1990. The landscape dendrology [M]. Beijing: China Forestry Publishing House. [陈有民, 1990. 园林树木学. [M]. 北京: 中国林业出版社.]
DI LN, 2018. Genetic diversity of Gymnocarpos przewalskii based on SCoT markers [J]. Bull Bot Res, 38(5): 725-732. [狄林楠, 2018. 基于SCoT分子标记的裸果木遗传多样性分析 [J]. 植物研究, 38(5):725-732.]
ELLSTRAND NC, ELAM DR, 1993. Population genetic consequences of small population size: implications for plant conservation [J]. Ann Rev Ecol Sys, 24(1): 217-242.
JIANG QC, TAN ZJ, LIAO HB, et al., 2018. Genetic diversity of Aquilaria sinensis in Wuguishan, Zhongshan City, Guangdong Province [J]. Guihaia, 38(6): 804-811. [蒋谦才, 谭宗健, 廖浩斌, 等, 2018. 广东省中山市五桂山土沉香遗传多样性 [J]. 广西植物, 38(6): 804-811.]
JIANG W, WU ZG, TAO ZM, et al., 2019. Genetic diversity analysis of Gardenia jasminoides based on ISSR and SRAP molecular markers [J]. Chin Trad Herb Drugs, 50(2): 510-516. [姜武, 吴志刚, 陶正明, 等, 2019. 基于ISSR和SRAP标记的栀子种质遗传多样性研究 [J]. 中草药, 50(2): 510-516.]
LI G, QUIROS CF, 2001. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica [J]. Theoretical & Appl Gene, 103(2-3): 455-461.
LI ZP, LI TC, FAN HH, et al., 2011. Genetic diversity analysis of Papaya resources by SRAP and SCoT combination [J]. Acta Laser Biol Sin, 20(2): 236-244. [李正鵬, 李廷春, 樊洪泓, 等, 2011. 棉花遗传多样性SCoT和SRAP标记的研究及比较分析 [J]. 激光生物学报, 20(2): 236-244.]
LIU L, 2012. Population evolution history and speciation study of Pinus armandii and its three affinities [D]. Beijing: Graduate University of Chinese Academy of Sciences. [刘柳, 2012. 华山松(Pinus armandii)及其近缘种的群体进化历史与物种形成研究 [D]. 北京:中国科学院研究生院.]
MENG H, CHEN B, YANG Y, 2018. Analysis of genetic diversity of Dalbergia odorifera growing in Hainan by SRAP [J]. Chin J Trop Agric, 38(12): 37-42. [孟慧, 陈波, 杨云, 2018. SRAP技术分析海南降香檀遗传多样性 [J]. 热带农业科学, 38(12): 37-42.]
RUAN ZY, WANG BY, OUYANG ZQ, et al., 2016. Characterization of microsatellites in genome of Pinus squamata, a critically endangered species in the world [J]. Bull Bot Res, 36(5):775-781. [阮桢媛, 王兵益, 欧阳志勤, 等, 2016. 极度濒危植物巧家五针松基因组微卫星特征分析 [J]. 植物研究, 36(5):775-781.]
SONG CF, LIU QX, ZHOU YF, et al., 2014. Genetic diversity analysis of Glehnia littoralis (Apiaceae) revealed by SRAP [J]. Guihaia, 34(1): 15-18. [宋春凤, 刘启新, 周义峰, 等, 2014. 珊瑚菜居群遗传多样性的SRAP分析 [J]. 广西植物, 34(1):15-18.]
SUI XZ, ZHENG Y, QIU YP, et al., 2017. Genetic diversity of 4 Betula alnoides [J]. J SW For Univ (Nat Sci Ed), 37(3): 21-25. [睢鑫洲, 郑毅, 仇玉萍, 等, 2017. 4个西南桦品系的遗传多样性分析 [J]. 西南林业大学学报(自然科学版), 37(3): 21-25.]
SUN WT, YU DD, DONG ML, et al., 2016. Genetic diversity of control-pollinated progenies in seed orchard of Larix gmelinii var. principis-rupprechtii Mayr. [J]. Acta Bot Boreal-Occident Sin, 36(8): 1662-1670. [孙文婷, 于大德, 董明亮, 等, 2016. 华北落叶松种子园控制授粉子代遗传多样性分析 [J]. 西北植物学报, 36(8): 1662-1670.]
TILMAN D, REICH PB, KNOPS JM, 2006. Biodiversity and ecosystem stability in a decade-long grassland experiment [J]. Nature, 441(4): 629-632.
WANG XR, ZHAO Y, LI TS, 2007. Preliminary report on artificial supplementary pollination experiment in clonal seed orchard of Pinus armandii [J]. J SW For Univ, (3): 25-28. [王秀荣, 赵杨, 李桐森, 2007. 华山松无性系种子园辅助授粉试验初报 [J]. 西南林学院学报, (3): 25-28.]
WHEELER NC, JECH KS, 1992. The use of electrophoretic markers in seed orchard research [J]. New For, 6(1): 311-328.
WIDEN B, SVENSSON L, 1992. Conservation of genetic variation in plants: The importance of population size and gene flow [M]//HANSSON L(ed.). Ecological principles of nature conservation:Application in temperate and boreal environments [M]. New York: Elsevier Science Publishers Ltd: 113-161.
WRIGHT S, 1950. Genetical structure of populations [J]. Nature, 166(4215): 247-249.
XIANG H, YUAN DY, FAN XM, et al., 2016. The analysis of genetic diversity of Castanea henryi (Skan) Rehder & E.H. Wilson by using SRAP [J]. J Plant Gene Resour, 17(6): 1072-1081. [向晖, 袁德义, 范晓明, 等, 2016. 锥栗种质资源遗传多样性的SRAP分析 [J]. 植物遗传资源学报, 17(6): 1072-1081.]
XIE DM, CHEN GM, HE D, et al., 2014. Comparative study on the different organization of genomic DNA extraction in Pinus armandii Franch [J]. J Hubei Inst Natl(Nat Sci Ed), 32(3): 255-261. [謝冬梅, 陈国明, 何德, 等, 2014. 华山松不同部位DNA提取方法比较研究 [J]. 湖北民族学院学报(自然科学版), 32(3): 255-261.]
XIN PY, ZHOU J, DUAN AA, et al., 2010. Research advance of genetic improvement on Pinus armandii in China [J]. N Hortic, (19): 210-214. [辛培尧, 周军, 段安安, 等, 2010. 我国华山松遗传改良研究进展 [J]. 北方园艺, (19): 210-214.]
YAN JJ, BAI SH, ZHANG XQ, et al., 2010. Genetic diversity of wild Elymus sibiricus germplasm from the Qinghai-Tibetan Plateau in China detected by SRAP markers [J]. Acta Pratac Sin, 19(1):173-183. [鄢家俊, 白史且, 张新全, 等, 2010. 青藏高原老芒麦种质基于SRAP标记的遗传多样性研究 [J]. 草业学报, 19(1):173-183.]
YANG ZQ, FENG YH, WU DS, 2014. Analysis of genetic diversity of Pinus yunnanensis var. tenuifolia nature populations by SSR marker [J]. Guihaia, 34(1):10-14. [杨章旗, 冯源恒, 吴东山, 2014. 细叶云南松天然种源林遗传多样性的SSR分析 [J]. 广西植物, 34(1):10-14.]
YUAN HN, CHEN WH, 2010. Development and utilization status and protection measures of Pinus armandii [J]. For Shaanxi, (4): 39. [袁红宁, 陈文辉, 2010. 华山松的开发利用现状及保护措施 [J]. 陕西林业, (4): 39.]
ZHAI SW, CHEN QG, DAI Y, et al., 2007. Heritability analysis and superior family selection for Half-Sib test of Pinus armandii [J]. Seed, 26(12): 5-8. [翟思万, 陈启贵, 代毅, 等, 2007. 华山松半同胞子代测定遗传力分析及优良家系选择 [J]. 种子, 26(12): 5-8.]
ZHANG AS, SI QL, QI XJ, et al., 2018. Genetic diversity and fingerprints of Actinidia germplasm resource based on SRAP markers [J]. Jiangsu J Agric Sci, 34(1): 138-144. [张安世, 司清亮, 齐秀娟, 等, 2018. 猕猴桃种质资源的SRAP遗传多样性分析及指纹图谱构建 [J]. 江蘇农业学报, 34(1): 138-144.]
ZHANG DD, 2008. Study on genetic structure in different populations of Pinus koraiensis by SRAP [D]. Harbin: Northeast Forestry University. [张冬东, 2008. SRAP对不同地理种源红松遗传结构的研究 [D]. 哈尔滨: 东北林业大学.]
ZHAO Y, DAI Y, LI YP, 2012. Genetic diversity for clonal seed orchard of Pinus armandii [J]. J NE For Univ, 40(10): 4-6. [赵杨, 代毅, 李玉璞, 2012. 华山松无性系种子园遗传多样性分析 [J]. 东北林业大学学报, 40(10): 4-6.]
ZHAO Y, LI TS, DUAN AA, 2003. A research advance in genetic improvement and development strategy of Pinus armandii in China [J]. J SW For Univ, 23(1): 91-95. [赵杨, 李桐森, 段安安, 2003. 国内华山松遗传改良的研究进展及发展策略 [J]. 西南林学院学报, 23(1): 91-95.]
ZHAO Y, LI YP, DAI Y, 2012. Optimization of SRAP-PCR system for Pinus armandii [J]. J NW For Univ, 27(5): 87-90. [赵杨, 李玉璞, 代毅, 2012. 华山松SRAP-PCR反应体系的优化 [J]. 西北林学院学报, 27(5):87-90.]
ZHOU YQ, 2008. Biological genetic marking and application [M]. Beijing: Chemical Industry Publishing House: 135-137. [周延清, 2008. 生物遗传标记与应用 [M]. 北京:化学工业出版社: 135-137.]
ZHU J, 2016. Population genetics and speciation study of Pinus armandii and its three affinities [D]. Xian: Northwestern University. [祝娟, 2016. 华山松及其近缘种的群体遗传学和物种形成研究 [D]. 西安:西北大学.]
ZHU XD, 2006. Study on the genetic diversity and seed production in clone seed orchard of Pinus armandii Franch. [D]. Kunming: Southwest Forestry College. [朱晓丹, 2006. 华山松无性系种子园遗传多样性及结实研究 [D]. 昆明:西南林学院.]
(责任编辑 何永艳)
