细胞终末分化过程中三维基因组结构与功能调控的分子机制
2020-03-04杨科薛征吕湘
杨科,薛征,吕湘
综述
细胞终末分化过程中三维基因组结构与功能调控的分子机制
杨科,薛征,吕湘
中国医学科学院基础医学研究所病理生理学系,医学分子生物学国家重点实验室,北京 100730
真核细胞中的染色质DNA高度折叠形成复杂的三维结构,其空间组织方式对精准调控基因的表达和细胞发挥正常功能都起着重要的作用。细胞终末分化成熟过程中形态及基因表达谱常发生显著改变,同时伴随着明显的基因组三维结构变化。本文在简单介绍三维基因组多层次组织结构(染色质领域、A/B区室、拓扑相关结构域和成环构象等)基础上,重点综述了细胞终末分化过程中三维基因组结构变化与功能调控方面的研究进展,并探讨了当前三维基因组研究在解析细胞分化成熟过程时存在的问题和前景。
三维基因组;A/B区室;拓扑相关结构域;成环构象;细胞终末分化
自遗传密码被破解以来,尤其是多个种属基因组测序完成后,基因组的一维序列信息得到了广泛研究和阐释。然而基因组在细胞核内并非以简单的一维线性排布,而是以复杂的三维结构存在,基因组的三维结构对基因的表达调控和细胞正常功能的发挥有着重要影响[1]。染色体构象捕获技术(chromosome conformation capture, 3C)[2]及由其衍生而来的“C”系列技术[3~8]的发展,使研究者们得以在DNA分子水平研究染色质之间的相互作用。3C技术主要是通过对交联固定后的核内基因组DNA进行限制性酶切连接,使一维序列上远离而空间上邻近的酶切片段有机会连接形成嵌合片段,再通过PCR检测定量连接产物,以校正后的定量结果来反映染色质片段间相互作用的强度。该技术建立后迅速从检测两个特定基因座位点间相互作用的3C方法发展到检测单个位点与整个基因组相互作用的(circular chromosome conformation capture/chromosome conformation capture-on-chip) 4C[3,4]、检测多对基因座位点间相互作用的(chromosome conformation capture carbon copy) 5C[6]、以及适用于全基因组染色质相互作用的(high-throughput chromosome conformation capture) Hi-C[5]等。Hi-C技术在酶切片段末端引入生物素标记后再进行连接反应,末端消化去除未连接端的生物素,实现对新形成嵌合片段的特异富集,测序获得全基因组范围的染色质相互作用信息。对Hi-C数据的深入解析使人们可以从染色体领域(chromosome territory, CT)、染色体间相互作用、A/B区室(compartment)、拓扑相关结构域(topologically associated domain, TAD)以及成环构象(looping)等不同层次揭示染色质的三维结构情况(图1A)。
细胞分化和发育过程中,随着细胞功能的改变,染色质三维结构也会发生相应变化。有关早期胚胎发育和细胞定向分化的三维基因组研究近期已有Zhang等[9]综述文章给予了详细介绍。细胞终末分化成熟是干祖细胞发生特化、转变成为执行特定功能细胞的生理变化过程,该过程对应着基因表达谱的显著转变,并常伴随细胞核形态和大小的变化,进一步提示染色质三维结构可能发生了剧烈的转变。本文概述了染色质三维结构不同层次上的研究进展,重点讨论了细胞终末分化成熟过程中染色质三维结构功能的动态调节。
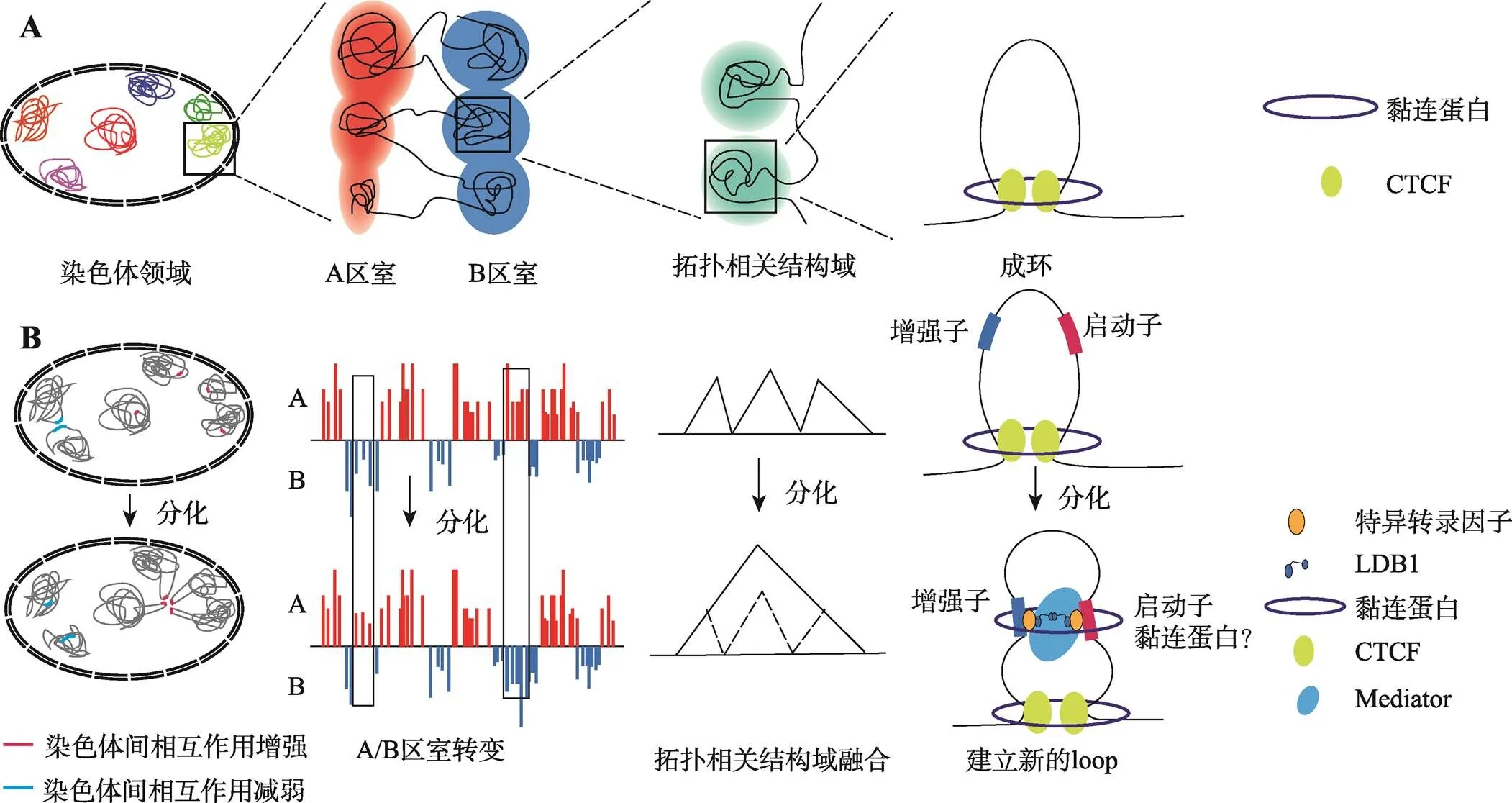
图1 三维基因组的多层次组织结构及其在细胞分化前后的变化
A:三维基因组的多层次组织结构;B:细胞分化过程中的三维基因组结构变化。
1 三维基因组的多层次组织结构
1.1 染色体领域
真核生物主要以染色体的形式承载遗传信息,染色体结构的存在为基因组以一定规律聚集或离散分布于细胞核中提供了基础和限制。在间期细胞核内,每条染色体占据一个相对独立但稳定的细胞核亚区域,称为染色体领域[10]。可以利用荧光原位杂交技术(fluorescenthybridization, FISH),通过源于特定染色体全序列的探针来获得染色体领域的直观图像[11]。Hi-C分析也发现同一染色体内即使相距很远的位点间相互作用频率也明显高于染色体间的相互作用,提示染色体内的DNA呈聚集分布,从分子水平上支持了CT模型[5]。研究表明,CT在细胞核内所处的位置并非随机,长染色体倾向与长染色体相互接近,而短染色体之间也更为靠近[12]。在同一CT中富含基因或活跃转录的DNA片段更高频率定位于CT的外围, 甚至突出到CT之外[13,14],这样更有利于接近位于CT之间(interchromatin compartment, IC)的转录机器,并和邻近染色体上转录活性类似的基因共转录[15]。在发育过程中,基因的激活常伴随其在CT中的定位由内部转移到外围[16]。CT具有组织和细胞类型特异性[17],细胞分裂间期核内的各个CT位置相对稳定,但最近有研究发现有丝分裂前后以及两个子细胞之间CT的位置会发生一定程度的变化[18],这可能与细胞之间的个体差异有关。
1.2 染色质核内定位与A/B区室
基因密度和转录活性不仅与其在CT中的定位有关,还与染色质在细胞核内的定位密切相关。基因密度低及不活跃转录的染色质倾向于定位在核周,其中与核纤层相互作用的区域称为核纤层蛋白相关结构域(lamin associated domains, LADs),富含H3K27me3和H3K9me2等非活性组蛋白修饰[19]。基因密度高或活跃表达的染色质则倾向定位于细胞核内部[20]。基因失去与核纤层的相互作用转而定位于核内部后,可以重新激活其表达,反之亦然[21]。在红系细胞中,复合物介导β-珠蛋白与其增强子基因座控制区(locus control region, LCR)间的相互作用,促进β-珠蛋白基因表达并定位于细胞核内部,敲低可抑制β-珠蛋白基因表达,其基因也从细胞核中部重新定位于核周[22]。此外,细胞核内邻近的活跃转录基因可以共定位并共享转录机器,形成转录工厂结构[23]。动态表达的基因在转录激活或抑制时移入或远离转录工厂,相比从头招募和组装转录复合物,将基因移动到预先装配好的转录工厂可能是更经济及常见的基因转录激活方式[23]。一个细胞核中通常存在多个不同类型的转录工厂[24],例如红系α-珠蛋白基因簇内的珠蛋白基因与其上游持家基因在空间上靠近并共同转录,是最早被鉴定的转录工厂之一[25]。类似的结构还有由不同染色体上的多个rRNA基因共定位,在核仁处形成的RNA聚合酶Ⅰ转录工厂[26]。转录工厂结构使细胞更有效的组织和调节自身基因的转录,协调细胞功能。
Hi-C分析提供了另一种核内染色质分区定位的方法。其结果提示具有转录活性的片段之间有着更高的相互作用频率,包括松散的染色质、具有活性组蛋白修饰、高基因密度和高表达水平的DNA片段等。同样基因组的非活性区段更倾向于和非活性区段相互作用[5]。通过主成分分析(principal component analysis, PCA)将这种Hi-C揭示的相互作用倾向性量化,可将基因组区分为相对分隔的A/B 两个区室。其中A区室基因密度较高,更富集DNaseⅠ高敏位点和H3K4me3等表观遗传修饰标志,与活性常染色质相对应,而B区室多处于基因荒漠区或为异染色质。染色质间相互作用更多发生在同一个区室内的位点之间。最近有研究通过分子模拟提出A/B区室是经由染色质片段相分离过程形成的:当一段染色质结合在核纤层或其它核内小体上时,其他与之相同状态的染色质也会随之结合进而引发相分离,形成A/B区室[27]。
1.3 拓扑相关结构域
提高Hi-C分析的测序深度和分辨率可以发现每条染色体均由多个结构单元组成,称为拓扑相关结构域(TAD)[28,29]。早期在果蝇胚胎中的Hi-C分析即发现TAD结构,表现为多个和染色质表观修饰标志高度相关的结构域,其内部染色质相互作用明显高于相邻结构域之间的互作等特点[30]。哺乳动物90%的基因组被约2000个TADs覆盖,其长度范围从数百kb到数Mb不等[28]。同一TAD内的基因往往处于相似的活性或者非活性状态。基因组活性与非活性区段的转变频繁发生在TAD边界附近,提示TAD间的边界序列具有隔离功能[31]。在不同细胞类型中,TAD活性状态常作为一个整体发生转变,呈协同表达模式。支持TAD作为相对独立的调控单元协调邻近基因的表达。分裂期细胞中未能检测到TAD结构,推测TAD主要在细胞分裂间期起作用,参与协调转录等过程[32]。
TAD位置及其边界序列在不同细胞类型间以及人和小鼠间均高度保守,进一步支持TAD边界及其隔离功能在哺乳动物细胞中具有重要意义。边界的存在使染色质片段间的相互作用限制在TAD内,从而将增强子等远距离调节元件功能局限于特定的靶基因[33]。TAD边界序列富含CTCF结合位点、转录起始位点和SINE元件[34]等序列。已知的TAD边界结合蛋白主要包括CTCF和黏连蛋白(cohesin)[35],CTCF可以起转录隔离的作用[36],阻断启动子和增强子的相互作用或表观遗传修饰标志向外延伸。黏连蛋白复合物形成环状结构,和CTCF共同参与TAD的形成。扰动CTCF/cohesin或其结合位点可增加邻近TAD间的相互作用,同时TAD内相互作用强度相对减弱,但并不完全破坏TAD边界,表明CTCF和cohesin在TAD的形成和维持中起一定作用[35,37~39],但不足以创建或消除TAD结构。
目前,TAD的鉴定主要建立在细胞群体Hi-C分析基础之上。单细胞Hi-C分析提示,TAD在单个细胞中可能不是固定的结构,而是群体细胞的染色质互作倾向性在总体上的反映[18,40]。最近,Bintu等[41]和Szabo等[42]基于高分辨成像的研究显示,即便在单细胞水平,类似TAD的结构也是存在的,这种差异也许与经典的分析流程对过于分散的单细胞Hi-C数据不太适用有关。
1.4 成环构象
细胞核内一维距离较远的DNA元件可以在空间上相互靠近,元件间的DNA片段环出,形成成环构象。Hi-C分析发现同一TAD中常存在多个染色质环,参与基因表达调节,其中尤以增强子和启动子间的成环构象在基因表达调控中的作用最为明确。β-珠蛋白基因簇是研究时空特异基因表达调控的理想模型[43]。其结构基因上游数十kb外的LCR作为超级增强子可显著增强簇内各类β-珠蛋白基因的表达。早前研究对远距离的LCR如何增强β-珠蛋白基因表达提出了成环模型(looping model),蛋白质联接介导模型(linking model)等多种假说[44]。3C技术从分子水平上证实了LCR和β-珠蛋白启动子在空间上靠近,支持增强子作用的成环模型[45]。启动子和增强子的相互作用常具有组织和发育阶段特异性,而非简单的一对一方式[46~49]。以LCR为例,在胚胎、胎儿和成年期分别与ε-、γ-和β-珠蛋白基因启动子空间靠近并激活其表达。反之,一个启动子也可与多个增强子互作。这种复杂的成环构象模式为基因选择性表达和精细调节提供了基础。
TAD内部常有多个CTCF结合位点,可招募CTCF/cohesin等分子介导其相互作用,为基因组 成环构象的形成和维持提供基础[50]。敲除CTCF或cohesin抑制这些位点间成环构象的形成[51]。在CTCF参与的成环模型中,环状的cohesin复合物结合在染色体上,形成染色质小环,cohesin顺着小环两端向外延伸移动,挤压导致染色质环增大,直至遭遇两个汇聚的(即方向朝环内的)CTCF结合位点,并和CTCF共同稳定该环状结构[52]。转录事件本身及cohesin释放因子共同参与cohesin在CTCF位点的募集[53]。在此基础上,转录因子招募互作蛋白或RNA聚合酶类形成复杂的转录复合物,介导增强子和启动子的相互作用及增强子转录调控功能,并通常将增强子的作用限制在CTCF/cohesin介导的成环构象及TAD边界以内。
核内染色质组织方式与基因转录活性密切相关,但其因果关系值得探讨。用DRB处理小鼠胎肝细胞抑制转录延伸对远距离基因共享转录工厂以及β-珠蛋白基因簇增强子与启动子的长距离相互作用均没有影响[54,55]。对转录起始的抑制因处理方法不同而效果有所差异,通过热激反应抑制转录起始显著影响转录工厂的形成并部分抑制β-珠蛋白启动子与LCR核心增强子HS2/3间的相互作用[55],而α-鹅膏蕈碱处理对β-珠蛋白启动子与LCR的相互作用没有影响[54]。这种差异可能和α-鹅膏蕈碱与热激具体抑制转录起始的方式不同有关。但两种转录起始抑制在总体上均对β-珠蛋白启动子与LCR相互作用形成的活性染色质结构域影响不大,表明染色质之间的相互作用并不是仅仅是基因转录的伴随现象。之后Deng等[56]在敲除的G1E细胞系中表达可识别基序的锌指结构与融合蛋白,人为把LCR和β-珠蛋白基因启动子联系在一起,促进了RNA聚合酶Pol II在β-珠蛋白基因启动子的募集,支持染色质成环构象可促进基因活跃转录。最近Ray等[57]的工作也发现热激不影响细胞的A/B分区和TAD结构,此时激活的基因多数在非热激状态已经建立增强子与启动子间相互作用,仅少部分与增强子启动子间的相互作用增强有关。综上提示,染色质三维结构是独立于转录事件的基因组特征,但又与基因转录密切相关并相互影响。
2 细胞终末分化过程中三维基因组结构变化
干祖细胞经由终末分化过程最终成为执行特定生理功能的成熟细胞,其细胞类型多样,对应复杂独特的细胞功能,并经常伴随细胞及细胞核形态大小上的不同变化,提示各种不同类型的终末分化过程中可能具有不同特色的三维基因组组织方式。对其三维基因组结构的详细解析将有助于了解终末分化阶段发挥重要功能基因(简称终末功能基因)的表达维持和相关疾病发生的分子机制。另外,细胞进入终末分化后许多终末功能基因的表达即已开启,细分的终末成熟各阶段细胞在三维基因组结构上是否仍存在显著变化,还是在原构架基础上微调值得关注。近年来,研究人员对T-细胞[58,59]、B-细胞[60,61]、巨噬细胞[62]、(骨骼/平滑)肌细胞[63]、神经细胞[64,65]、表皮角质细胞[66]和胰岛β-细胞[67]等多种细胞终末分化成熟过程中的染色质三维结构进行研究,在不同层次上展示了基因组三维结构伴随终末分化中重要基因表达呈现的各自特异的变化模式(图1B)。
2.1 细胞终末分化过程中的A/B区室翻转以及染色体间相互作用改变
基因组的A/B分区与染色质活性和抑制状态相对应,具有明显的组织特异性。细胞分化成熟过程往往伴随部分染色质区段的A/B分区转变。T细胞分化成熟过程经历造血干祖细胞(hematopoietic stem and progenitor cell, HSPC)、多潜能祖细胞(multipotent progenitor, MPP)、共同淋系祖细胞(common lymphoid progenitor, CLP)、早期T祖细胞(early T precursor, ETP)、CD4/CD8双阴T细胞2 (CD4 and CD8 double-negative 2, DN2)、DN3、DN4和CD4/CD8双阳T细胞(CD4 and CD8 double-positive, DP)等多个阶段。这个过程的基因组A/B分区持续发生转变,其中57.6%由B区室转变为A区室,37.4%由A区室转变为B区室,仅4.9%的区域在A/B分区上的存在反复。A/B区室的转变主要集中在DN2-DN3和DN4-DP阶段,提示这两个时间段可能是T细胞终末分化的关键。这与前期研究提出的DN2-DN3转变与T细胞命运决定相关,而DN4-DP转变是T细胞β选择关键步骤的观点相一致[68]。作为典型代表,T细胞分化和存活的关键转录因子基因由造血干细胞中的B区室向CD4/CD8双阳性细胞中活性的A区室转变,同时基因定位由核周转向核内部。相反造血干细胞自我更新的关键因子基因则由活跃的A区室转变为抑制状态的B区室,基因定位向核周转移。该过程中基因的沉默和开启均早于其A/B分区的翻转,提示区室转变并非基因表达变化的上游因素[59]。
除染色质A/B分区翻转外,同一区室内染色质相互作用强度和染色体间相互作用也伴随终末分化过程发生改变。小鼠皮质神经元终末分化时A/B区室均表现为大小渐增,其中A区室内部相互作用变弱、而B区室内部相互作用加强[65],与此前报道的神经细胞分化过程中异染色质聚集一致[69]。幼稚T细胞成熟分化为T辅Ⅰ、T辅Ⅱ细胞的过程中,11号染色体上调节元件和10号染色体γ干扰素基因启动子间的相互作用在分化后被染色体内相互作用所代替。在T辅Ⅰ细胞中,γ干扰素与其上游调控元件互作,产生更多的γ干扰素;在T辅Ⅱ细胞中,11号染色体上的调控元件与邻近的白介素基因相互作用并激活其表达[70,71]。
2.2 细胞终末分化过程中的TAD变化
研究表明TAD结构在不同细胞类型和物种间相对保守,但细胞分化过程中TAD内的局部染色质相互作用强度可发生变化。T细胞分化成熟过程中,TAD内部相互作用的强弱变化也主要发生在DN2- DN3和DN4-DP两个阶段,其变化与DNaseⅠ高敏位点信号及基因表达谱改变相吻合,但在时间上先后有别。沉默基因的表达下调先于相应TAD内染色质互作信号的减弱;相反激活基因TAD内染色质互作变强早于其基因表达上调[59],支持染色质互作只是基因转录的前提条件,转录激活还需要更多因素的参与。这种时序关系在转录抑制剂处理或热激引起转录改变时观察到的现象基本一致[54,55,57]。
虽然TAD结构相对稳定保守,在一些细胞分化过程中也观察到相邻TAD融合和TAD分裂的情况。幼稚B细胞(naïve B cells)向GC B细胞分化过程中TAD数目(>1500个)和平均大小(1.35 Mb)均无明显变化,但观察到其中171个TAD结构域与邻近TAD间融合形成更大的新型3D基因“城市”(city)结构。对三号染色体上一段6 Mb长的区域具体分析显示,该区段在幼稚B细胞中分为4个TAD结构域,其中多个基因在细胞分化为GC B细胞时跨越TAD边界建成新的染色质间相互作用,从而整段染色质融合形成一个大型TAD结构。跨界参与染色质相互作用的基因多在GC B细胞中特异表达上调并在其细胞特化过程中发挥重要作用。总体而言,相邻TAD的融合促进了基因的协同表达[60]。小鼠皮质神经元终末分化时也观察到TAD数目减少和平均大小增加,但同时一类不含CTCF的TAD边界明显增多。这类TAD边界附近富集终末阶段活跃表达基因的启动子,支持基因转录参与TAD边界形成,然而仅转录激活并不足以导致新TAD的出现[65]。
2.3 细胞终末分化过程的染色质成环情况
与TAD结构域的相对稳定保守相反的是,TAD内部染色质成环构象具有高度的细胞类型特异性,与不同细胞独特的基因表达谱形成密切相关。细胞终末分化过程中成环构象的研究有助于更深刻的理解细胞分化成熟的调控网络,鉴定新的调控元件和调节模式,以及发现终末分化过程中的关键基因。全基因组水平的成环构象分析对Hi-C数据量要求较高,ChIA-PET[7], capture Hi-C[8,72]和原位Hi-C[73]等方法的出现和优化以及二代测序效能的提高使在全基因组范围研究染色质成环构象成为可能。
在PMA诱导的THP-1体外巨噬细胞分化模型中,通过差异loop分析发现多个分化后新形成的染色质环(获得型loop),其出现与基因表达上调显著相关。另外,对大部分未随分化改变的染色质环而言,启动子所关联的远端锚定位点上H3K27ac信号增强(激活型loop)也与基因表达上调显著相关。基因本体论(Gene Ontology, GO)分析显示获得型loop和激活型loop所对应的靶基因均富集在巨噬细胞功能相关通路上,包含、和等参与巨噬细胞发育和功能的重要基因[62]。在人表皮角质细胞终末分化模型中观察到类似现象,基因表达上调与两种不同的增强子启动子成环构象有关:其中一类是在终末分化过程中新形成的染色质环,即获得型loop,同时伴随H3K27ac修饰水平增加;另一类环则在祖细胞阶段已经形成,并在终末分化过程中稳定存在(稳定型loop),与巨噬细胞分化有所不同的是,表皮角质细胞中这类染色质环的增强子呈组成型H3K27ac修饰[66]。由此提出成环构象参与分化过程中基因表达调节的两种模型:在远端调控元件和基因启动子间建立新的染色质相互作用,或者通过已有成环构象的重塑对靶基因进行调控。值得注意的是,前述巨噬细胞分化时有2070个基因上调,而位于获得型loop或激活型loop的启动子只有527个,且其中只有156个在分化过程中显著上调,提示成环构象只是参与基因转录调控的诸多要素之一[62]。
巨噬细胞分化成熟过程中,存在由多个增强子与同一启动子共同形成的、含有多个染色质环的3D基因组“社区”或“中心”(hubs)。类似的活性染色质中心在红系终末分化的关键基因α-珠蛋白和β-珠蛋白基因簇上也观察到过[74]。此外,在B细胞分化为GC B细胞的过程中还观察到启动子间成环构象以及启动子与自身基因3¢-端的相互作用显著增多,伴随活跃染色质标志的富集和基因表达增加,其中富含GC B细胞特异表达基因[60]。3D基因组社区的组织方式有利于增加细胞核内局部增强子及其募集转录因子的浓度,共同调节分化过程中关键基因的激活。而一些关键调控元件又可同时调控与分化过程相关的多个基因,扮演染色质互作网络的核心元件。目前关于分化细胞中复杂成环构象形成的证据基本都来自对细胞群体的Hi-C分析,组成复杂成环构象的各种染色质相互作用是否同时存在于一个细胞当中尚有待通过新的实验或计算方法验证。
3 结构性因子与组织特异转录因子协同调控细胞分化成熟的染色质成环变换
CTCF是目前最广为人知的染色质成环介导因子。哺乳动物基因组上已发现的CTCF结合位点数多达5~6万。早期研究中发现的各种CTCF功能,包括转录激活、抑制、隔离和增强子阻断等,在很大程度上可以归结为CTCF作为一种结构性因子介导成环构象的形成。CTCF的具体功能取决于特定的辅因子种类和其所介导靠近的DNA序列性质[75]。一方面CTCF可与cohesin和等因子一起参与TAD边界的组建,发挥异染色质隔离和增强子阻断等功能;另一方面,它又可与cohesin、mediator和等因子共同介导启动子与增强子的相互作用,调控诸如神经细胞分化发育的基因启动子选择和胚胎干细胞到内胚层的分化等生物学过程[76]。细胞分化前后CTCF靶序列DNA甲基化水平,包括其经TET酶催化的各种氧化产物、CTCF自身Sumo-化修饰和多聚ADP化修饰、以及辅因子状态等均参与调节CTCF介导的成环构象形成[76,77]。近年研究显示非编码RNA在CTCF介导的染色质相互作用中也发挥重要作用[78]。最新研究对位于CTCF第11号锌指C-端的一段长38个氨基酸的RNA结合结构域进行敲除,发现胚胎干细胞中约一半的染色质环受到了明显影响[79]。同期文章还研究了CTCF第1及第10号锌指的RNA结合能力,其敲除亦可严重影响CTCF在染色质上的结合、成环构象形成和相应基因的表达[80]。CTCF还可以结合到基因5'-UTR序列和内含子上,调节RNA聚合酶Ⅱ的暂停和RNA加工,参与小鼠红系分化过程中基因的成环构象和转录延伸控制,并调节人淋巴细胞分化时CD45基因第5外显子的选择性剪接[81,82]。
是另一个在多种组织中普遍存在并介导增强子和启动子染色质相互作用的因子。可以结合在活性的增强子和启动子上,通过二聚化介导增强子和启动子间的成环相互作用,结合位点或者自身的敲除均会破坏增强子和启动子之间的成环构象并降低基因表达[83]。参与染色质构象组织的结构性因子还包括核基质结合蛋白、和果蝇的、因子等,在红系珠蛋白基因簇、基因成环构象和介导逆转座子拷贝间互作中发挥作用。近年还发现锌指蛋白可作为CTCF的重要辅因子广泛参与基因组成环构象的调节[84,85]。
组织特异转录因子在细胞分化特异的染色质高级构象形成当中发挥重要的作用。在单核细胞向巨噬细胞分化模型中,观察到巨噬细胞中Tn5转座酶敏感位点、新增和激活型成环构象锚定位点均富集AP-1家族转录因子基序,其富集程度甚至高于通常情况下最多见的CTCF基序。AP-1在远距离元件上的结合与染色质环另一端基因的表达激活正相关。提示AP-1及其辅因子介导了巨噬细胞特异成环构象形成并调节巨噬细胞分化[62]。细胞常利用多种因子分别参与不同类型染色质环的组织,协同调控细胞分化过程的基因表达。前述人表皮角质细胞终末分化模型中,发现仅稳定型loop上有cohesin的结合,组织特异的EHF因子也参与稳定型loop的形成并激活其基因表达;终末分化过程中新形成的染色质环上则缺乏cohesin结合,但高度富集C/EBP和KLF家族蛋白的结合基序,转录因子及其互作蛋白在这类获得型loop的组建和基因表达激活中发挥关键作用[66]。
越来越多的研究[62,64,67]发现LIM结构域结合蛋白1 (LIM domain binding 1, LDB1)在细胞分化成熟过程的染色质高级构象动态组织中发挥关键作用。与组织特异的转录因子共同介导不同类型细胞中特异染色质成环构象形成,促进分化特异基因表达:胰岛β-细胞中与、、和等转录因子共同介导了多种终末功能基因启动子与增强子的相互作用,促进人与小鼠胰岛β-细胞终末分化状态的维持。的敲除严重影响成熟β-细胞的胰岛素分泌和血糖稳态[67]。在红系终末分化过程中,//和核心因子分别结合LCR和β-珠蛋白启动子,并各自募集因子,通过自身二聚化促使LCR和珠蛋白启动子成环,增加β-珠蛋白基因表达[56,86]。在诱导胚胎干细胞向肌细胞分化的模型中,以关键转录因子结合位点为中心建立染色质成环构象。质谱分析发现可与和cohesin复合物核心成分相互作用,可独立于募集到部分结合的增强子元件并介导成环构象形成。敲低表达可导致结合位点H3K4me1表观修饰的降低和成环构象破坏,进而抑制驱动的基因表达和肌细胞分化过程[63]。介导的成环构象在小鼠嗅觉受体的选择型表达中也发挥重要作用。小鼠基因组中共有1000多个嗅觉受体基因,分布在18条不同染色体上[87]。每个成熟的嗅感觉神经元只表达一个嗅觉受体基因[88]。在水平基底细胞(horizontal basal cells)分化最终产生嗅感觉神经元的过程中,染色质高级构象分析显示,随机的某个嗅觉受体基因与63个处于不同染色体上的增强子相互作用,反式激活该嗅觉受体基因的表达。蛋白结合在这些称为“希腊小岛”(Greek islands)的嗅觉受体基因增强子上,通过其LIM结构域和辅因子结合,稳定“希腊小岛”间的反式染色质相互作用并促进关联嗅觉受体基因的表达[64]。另外,还与多种神经细胞以及心肌、乳腺和垂体的分化发育有关。介导的成环构象及组织特异转录因子是否及如何参与调控在这些组织细胞分化成熟的过程尚有待进一步探究。
4 结语与展望
目前大部分细胞分化的染色质高级构象研究是细胞群体上平均信号水平的反映,考虑到细胞的异质性,尤其细胞分化作为一个动态过程异质性更为明显,在单细胞水平研究细胞分化的三维基因组结构变化是未来的发展方向之一[8,18,40,89]。Hi-C策略需要通过酶切连接的方法检测位点之间的相互作用,每个酶切片段只有两个末端可与空间邻近的片段连接,从而限制了单细胞Hi-C中复杂成环构象,如转录工厂或者活性染色质中心等结构的鉴定。开发和优化新的单细胞染色质构象检测技术和分析方法,利用诸如串联体连接分析法(concatemer ligation assay, COLA)[90]、不依赖邻位连接的SPRITE[91]和阮一骏教授团队新发展的ChIA-Drop技术[92]等,在单细胞全基因组水平实现对多个成环构象共定位情况的同时检测;结合高分辨显微成像和细胞原位捕获等技术研究介导染色质构象形成的复合物组成等均有助于极大地促进人们对细胞分化过程中三维基因组组织及其调控功能的认识。
细胞分化成熟过程伴随终末功能基因表达的显著增加以及干性基因和其他组织细胞特异基因的表达抑制等转录组显著变化。细胞基因组三维组织方式的改变为其转录组变化提供了结构基础。从基因的染色质定位、染色体间的相互作用、A/B区室、TAD结构和成环构象等不同层次详细考察细胞终末分化过程的染色质高级构象动态变化不但能促进人们从理论上深入了解三维基因组多层次结构的建立过程和机制,加深对染色质高级构象结构和功能的认识;也有助于人们对细胞分化成熟过程的理解,发现分化成熟过程中的关键时期、新的关键基因或者调控模式,为体外培养细胞的高效终末分化和相关疾病的诊疗提供基础。
致谢
衷心感谢军事医学科学院生物工程研究所赵志虎研究员对本文提供的宝贵建议。
[1] Li GL, Ruan YJ, Gu RS, Du SM. Emergence of 3D genomics., 2014, 59(13): 1165–1172.李国亮, 阮一骏, 谷瑞升, 杜生明. 起航三维基因组学研究. 科学通报, 2014, 59(13): 1165–1172.
[2] Dekker J, Rippe K, Dekker M, Kleckner N. Capturing chromosome conformation., 2002, 295(5558): 1306–1311.
[3] Zhao Z, Tavoosidana G, Sjölinder M, Göndör A, Mariano P, Wang S, Kanduri C, Lezcano M, Sandhu KS, Singh U, Pant V, Tiwari V, Kurukuti S, Ohlsson R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra– and interchromosomal interactions., 2006, 38(11): 1341– 1347.
[4] Simonis M, Klous P, Splinter E, Moshkin Y, Willemsen R, de Wit E, van Steensel B, de Laat W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C)., 2006, 38(11): 1348–1354.
[5] Lieberman-Aiden E, van Berkum NL, Williams L, Imakaev M, Ragoczy T, Telling A, Amit I, Lajoie BR, Sabo PJ, Dorschner MO, Sandstrom R, Bernstein B, Bender MA, Groudine M, Gnirke A, Stamatoyannopoulos J, Mirny LA, Lander ES, Dekker J. Comprehensive mapping of long- range interactions reveals folding principles of the human genome., 2009, 326(5950): 289–293.
[6] Dostie J, Richmond TA, Arnaout RA, Selzer RR, Lee WL, Honan TA, Rubio ED, Krumm A, Lamb J, Nusbaum C, Green RD, Dekker J. Chromosome Conformation capture carbon copy (5C): a massively parallel solution for mapping interactions between genomic elements., 2006, 16(10): 1299–1309.
[7] Fullwood MJ, Liu MH, Pan YF, Liu J, Xu H, Mohamed YB, Orlov YL, Velkov S, Ho A, Mei PH, Chew EG, Huang PY, Welboren WJ, Han Y, Ooi HS, Ariyaratne PN, Vega VB, Luo Y, Tan PY, Choy PY, Wansa KD, Zhao B, Lim KS, Leow SC, Yow JS, Joseph R, Li H, Desai KV, Thomsen JS, Lee YK, Karuturi RK, Herve T, Bourque G, Stunnenberg HG, Ruan X, Cacheux-Rataboul V, Sung WK, Liu ET, Wei CL, Cheung E, Ruan Y. An oestrogen-receptor-alpha-bound human chromatin interactome., 2009, 462(7269): 58–64.
[8] Hughes JR, Roberts N, McGowan S, Hay D, Giannoulatou E, Lynch M, De Gobbi M, Taylor S, Gibbons R, Higgs DR. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment., 2014, 46(2): 205–212.
[9] Zheng H, Xie W. The role of 3D genome organization in development and cell differentiation., 2019, 20(9): 535–550.
[10] Cremer T, Cremer M. Chromosome territories., 2010, 2(3): a003889.
[11] Macville M, Veldman T, Padilla-Nash H, Wangsa D, O'Brien P, Schröck E, Ried T. Spectral karyotyping, a 24-colour FISH technique for the identification of chromosomal rearrangements., 1997, 108(4–5): 299–305.
[12] Zhang Y, McCord RP, Ho YJ, Lajoie BR, Hildebrand DG, Simon AC, Becker MS, Alt FW, Dekker J. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations., 2012, 148(5): 908–921.
[13] Boyle S, Rodesch MJ, Halvensleben HA, Jeddeloh JA, Bickmore WA. Fluorescence in situ hybridization with high-complexity repeat-free oligonucleotide probes generated by massively parallel synthesis., 2011, 19(7): 901–909.
[14] Heard E, Bickmore W. The ins and outs of gene regulation and chromosome territory organisation., 2007, 19(3): 311–316.
[15] Schoenfelder S, Sexton T, Chakalova L, Cope NF, Horton A, Andrews S, Kurukuti S, Mitchell JA, Umlauf D, Dimitrova DS, Eskiw CH, Luo YY, Wei CL, Ruan YJ, Bieker JJ, Fraser P. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells., 2010, 42(1): 53–61.
[16] Morey C, Da Silva NR, Perry P, Bickmore WA. Nuclear reorganisation and chromatin decondensation are conserved, but distinct, mechanisms linked to Hox gene activation., 2007, 134(5): 909–919.
[17] Bolzer A, Kreth G, Solovei I, Koehler D, Saracoglu K, Fauth C, Müller S, Eils R, Cremer C, Speicher MR, Cremer T. Three-dimensional maps of all chromosomes in human male fibroblast nuclei and prometaphase rosettes., 2005, 3(5): e157.
[18] Nagano T, Lubling Y, Stevens TJ, Schoenfelder S, Yaffe E, Dean W, Laue ED, Tanay A, Fraser P. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure., 2013, 502(7469): 59–64.
[19] Akhtar W, de Jong J, Pindyurin AV, Pagie L, Meuleman W, de Ridder J, Berns A, Wessels LF, van Lohuizen M, van Steensel B. Chromatin position effects assayed by thousands of reporters integrated in parallel., 2013, 154(4): 914–927.
[20] Tanabe H, Müller S, Neusser M, von Hase J, Calcagno E, Cremer M, Solovei I, Cremer C, Cremer T. Evolutionary conservation of chromosome territory arrangements in cell nuclei from higher primates., 2002, 99(7): 4424–4429.
[21] Peric-Hupkes D, Meuleman W, Pagie L, Bruggeman SW, Solovei I, Brugman W, Gräf S, Flicek P, Kerkhoven RM, van Lohuizen M, Reinders M, Wessels L, van Steensel B. Molecular maps of the reorganization of genome-nuclear lamina interactions during differentiation., 2010, 38(4): 603–613.
[22] Song SH, Kim A, Ragoczy T, Bender MA, Groudine M, Dean A. Multiple functions of Ldb1 required for beta-globin activation during erythroid differentiation., 2010, 116(13): 2356–2364.
[23] Osborne CS, Chakalova L, Brown KE, Carter D, Horton A, Debrand E, Goyenechea B, Mitchell JA, Lopes S, Reik W, Fraser P. Active genes dynamically colocalize to shared sites of ongoing transcription., 2004, 36(10): 1065–1071.
[24] Papantonis A, Cook PR. Transcription factories: genome organization and gene regulation., 2013, 113(11): 8683–8705.
[25] Zhou GL, Xin L, Song W, Di LJ, Liu G, Wu XS, Liu DP, Liang CC. Active chromatin hub of the mouse alpha- globin locus forms in a transcription factory of clustered housekeeping genes., 2006, 26(13): 5096– 5105.
[26] Faro-Trindade I, Cook PR. Transcription factories: structures conserved during differentiation and evolution., 2006, 34(Pt 6): 1133–1137.
[27] Nuebler J, Fudenberg G, Imakaev M, Abdennur N, Mirny LA. Chromatin organization by an interplay of loop extrusion and compartmental segregation., 2018, 115(29): E6697–E6706.
[28] Dixon JR, Selvaraj S, Yue F, Kim A, Li Y, Shen Y, Hu M, Liu JS, Ren B. Topological domains in mammalian genomes identified by analysis of chromatin interactions., 2012, 485(7398): 376–380.
[29] Nora EP, Lajoie BR, Schulz EG, Giorgetti L, Okamoto I, Servant N, Piolot T, van Berkum NL, Meisig J, Sedat J, Gribnau J, Barillot E, Blüthgen N, Dekker J, Heard E. Spatial partitioning of the regulatory landscape of the X-inactivation centre., 2012, 485(7398): 381–385.
[30] Sexton T, Yaffe E, Kenigsberg E, Bantignies F, Leblanc B, Hoichman M, Parrinello H, Tanay A, Cavalli G. Three- dimensional folding and functional organization principles of the drosophila genome., 2012, 148(3): 458–472.
[31] Sofueva S, Yaffe E, Chan WC, Georgopoulou D, Vietri Rudan M, Mira-Bontenbal H, Pollard SM, Schroth GP, Tanay A, Hadjur S. Cohesin-mediated interactions organize chromosomal domain architecture., 2013, 32(24): 3119–3129.
[32] Naumova N, Imakaev M, Fudenberg G, Zhan Y, Lajoie BR, Mirny LA, Dekker J. Organization of the mitotic chromosome., 2013, 342(6161): 948–953.
[33] Smallwood A, Ren B. Genome organization and long- range regulation of gene expression by enhancers., 2013, 25(3): 387–394.
[34] Hou C, Li L, Qin ZS, Corces VG. Gene density, transcription, and insulators contribute to the partition of the drosophila genome into physical domains., 2012, 48(3): 471–484.
[35] Zuin J, Dixon JR, van der Reijden MI, Ye Z, Kolovos P, Brouwer RW, van de Corput MP, van de Werken HJ, Knoch TA, van IJcken WF, Grosveld FG, Ren B, Wendt KS. Cohesin and CTCF differentially affect chromatin architecture and gene expression in human cells., 2014, 111(3): 996–1001.
[36] Phillips JE, Corces VG. CTCF: master weaver of the genome., 2009, 137(7): 1194–1211.
[37] Rao SSP, Huang SC, Glenn St Hilaire B, Engreitz JM, Perez EM, Kieffer-Kwon KR, Sanborn AL, Johnstone SE, Bascom GD, Bochkov ID, Huang X, Shamim MS, Shin J, Turner D, Ye Z, Omer AD, Robinson JT, Schlick T, Bernstein BE, Casellas R, Lander ES, Aiden EL. Cohesin loss eliminates all loop domains., 2017, 171(2): 305–320 e324.
[38] Nora EP, Goloborodko A, Valton AL, Gibcus JH, Uebersohn A, Abdennur N, Dekker J, Mirny LA, Bruneau BG. Targeted degradation of CTCF decouples local insulation of chromosome domains from genomic compartmentalization., 2017, 169(5): 930–944.e922.
[39] Schwarzer W, Abdennur N, Goloborodko A, Pekowska A, Fudenberg G, Loe-Mie Y, Fonseca NA, Huber W, Haering CH, Mirny L, Spitz F. Two independent modes of chromatin organization revealed by cohesin removal., 2017, 551(7678): 51–56.
[40] Stevens TJ, Lando D, Basu S, Atkinson LP, Cao Y, Lee SF, Leeb M, Wohlfahrt KJ, Boucher W, O'Shaughnessy- Kirwan A, Cramard J, Faure AJ, Ralser M, Blanco E, Morey L, Sansó M, Palayret MGS, Lehner B, Di Croce L, Wutz A, Hendrich B, Klenerman D, Laue ED. 3D structures of individual mammalian genomes studied by single-cell Hi-C., 2017, 544(7648): 59–64.
[41] Bintu B, Mateo LJ, Su JH, Sinnott-Armstrong NA, Parker M, Kinrot S, Yamaya K, Boettiger AN, Zhuang XW. Super-resolution chromatin tracing reveals domains and cooperative interactions in single cells., 2018, 362(6413): eaau1783.
[42] Szabo Q, Jost D, Chang JM, Cattoni DI, Papadopoulos GL, Bonev B, Sexton T, Gurgo J, Jacquier C, Nollmann M, Bantignies F, Cavalli G. TADs are 3D structural units of higher-order chromosome organization in Drosophila., 2018, 4(2): eaar8082.
[43] Noordermeer D, de Laat W. Joining the loops: beta-globin gene regulation., 2008, 60(12): 824–833.
[44] Bulger M, Groudine M. Looping versus linking: toward a model for long-distance gene activation., 1999, 13(19): 2465–2477.
[45] Carter D, Chakalova L, Osborne CS, Dai YF, Fraser P. Long-range chromatin regulatory interactions., 2002, 32(4): 623–626.
[46] Li G, Ruan X, Auerbach RK, Sandhu KS, Zheng M, Wang P, Poh HM, Goh Y, Lim J, Zhang J, Sim HS, Peh SQ, Mulawadi FH, Ong CT, Orlov YL, Hong S, Zhang Z, Landt S, Raha D, Euskirchen G, Wei CL, Ge W, Wang H, Davis C, Fisher-Aylor KI, Mortazavi A, Gerstein M, Gingeras T, Wold B, Sun Y, Fullwood MJ, Cheung E, Liu E, Sung WK, Snyder M, Ruan Y. Extensive promoter- centered chromatin interactions provide a topological basis for transcription regulation., 2012, 148(1–2): 84–98.
[47] Sanyal A, Lajoie BR, Jain G, Dekker J. The long-range interaction landscape of gene promoters., 2012, 489(7414): 109–113.
[48] Kieffer-Kwon KR, Tang Z, Mathe E, Qian J, Sung MH, Li G, Resch W, Baek S, Pruett N, Grøntved L, Vian L, Nelson S, Zare H, Hakim O, Reyon D, Yamane A, Nakahashi H, Kovalchuk AL, Zou J, Joung JK, Sartorelli V, Wei CL, Ruan X, Hager GL, Ruan Y, Casellas R. Interactome maps of mouse gene regulatory domains reveal basic principles of transcriptional regulation., 2013, 155(7): 1507– 1520.
[49] Jin F, Li Y, Dixon JR, Selvaraj S, Ye Z, Lee AY, Yen CA, Schmitt AD, Espinoza CA, Ren B. A high-resolution map of the three-dimensional chromatin interactome in human cells., 2013, 503(7475): 290–294.
[50] Yan J, Enge M, Whitington T, Dave K, Liu J, Sur I, Schmierer B, Jolma A, Kivioja T, Taipale M, Taipale J. Transcription factor binding in human cells occurs in dense clusters formed around cohesin anchor sites., 2013, 154(4): 801–813.
[51] Chien R, Zeng W, Kawauchi S, Bender MA, Santos R, Gregson HC, Schmiesing JA, Newkirk DA, Kong X, Ball AR, Jr., Calof AL, Lander AD, Groudine MT, Yokomori K. Cohesin mediates chromatin interactions that regulate mammalian β-globin expression., 2011, 286(20): 17870–17878.
[52] Sanborn AL, Rao SS, Huang SC, Durand NC, Huntley MH, Jewett AI, Bochkov ID, Chinnappan D, Cutkosky A, Li J, Geeting KP, Gnirke A, Melnikov A, McKenna D, Stamenova EK, Lander ES, Aiden EL. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes., 2015, 112(47): E6456–E6465.
[53] Busslinger GA, Stocsits RR, van der Lelij P, Axelsson E, Tedeschi A, Galjart N, Peters JM. Cohesin is positioned in mammalian genomes by transcription, CTCF and Wapl., 2017, 544(7651): 503–507.
[54] Palstra RJ, Simonis M, Klous P, Brasset E, Eijkelkamp B, de Laat W. Maintenance of long-range DNA interactions after inhibition of ongoing RNA polymerase II transcription., 2008, 3(2): e1661.
[55] Mitchell JA, Fraser P. Transcription factories are nuclear subcompartments that remain in the absence of transcription., 2008, 22(1): 20–25.
[56] Deng W, Lee J, Wang H, Miller J, Reik A, Gregory PD, Dean A, Blobel GA. Controlling long-range genomic interactions at a native locus by targeted tethering of a looping factor., 2012, 149(6): 1233–1244.
[57] Ray J, Munn PR, Vihervaara A, Lewis JJ, Ozer A, Danko CG, Lis JT. Chromatin conformation remains stable upon extensive transcriptional changes driven by heat shock., 2019, 116(39): 19431–19439.
[58] Isoda T, Moore AJ, He Z, Chandra V, Aida M, Denholtz M, Piet van Hamburg J, Fisch KM, Chang AN, Fahl SP, Wiest DL, Murre C. Non-coding transcription instructs chromatin folding and compartmentalization to dictate enhancer-promoter communication and T cell fate., 2017, 171(1): 103–119 e118.
[59] Hu GQ, Cui KR, Fang DF, Hirose S, Wang X, Wangsa D, Jin WF, Ried T, Liu PT, Zhu JF, Rothenberg EV, Zhao KJ. Transformation of accessible chromatin and 3D nucleome underlies lineage commitment of early T cells., 2018, 48(2): 227–242.e228.
[60] Bunting KL, Soong TD, Singh R, Jiang Y, Béguelin W, Poloway DW, Swed BL, Hatzi K, Reisacher W, Teater M, Elemento O, Melnick AM. Multi-tiered reorganization of the genome during b cell affinity maturation anchored by a germinal center-specific locus control region., 2016, 45(3): 497–512.
[61] Kieffer-Kwon KR, Nimura K, Rao SSP, Xu JL, Jung S, Pekowska A, Dose M, Stevens E, Mathe E, Dong P, Huang SC, Ricci MA, Baranello L, Zheng Y, Tomassoni Ardori F, Resch W, Stavreva D, Nelson S, McAndrew M, Casellas A, Finn E, Gregory C, St Hilaire BG, Johnson SM, Dubois W, Cosma MP, Batchelor E, Levens D, Phair RD, Misteli T, Tessarollo L, Hager G, Lakadamyali M, Liu Z, Floer M, Shroff H, Aiden EL, Casellas R. Myc regulates chromatin decompaction and nuclear architecture during B cell activation., 2017, 67(4): 566–578 e510.
[62] Phanstiel DH, Van Bortle K, Spacek D, Hess GT, Shamim MS, Machol I, Love MI, Aiden EL, Bassik MC, Snyder MP. Static and Dynamic DNA loops form AP-1-Bound activation hubs during macrophage development., 2017, 67(6): 1037–1048 e1036.
[63] Magli A, Baik J, Pota P, Cordero CO, Kwak IY, Garry DJ, Love PE, Dynlacht BD, Perlingeiro RCR. Pax3 cooperates with Ldb1 to direct local chromosome architecture during myogenic lineage specification., 2019, 10(1): 2316.
[64] Monahan K, Horta A, Lomvardas S. LHX2- and LDB1-mediated trans interactions regulate olfactory receptor choice., 2019, 565(7740): 448–453.
[65] Bonev B, Mendelson Cohen N, Szabo Q, Fritsch L, Papadopoulos GL, Lubling Y, Xu X, Lv X, Hugnot JP, Tanay A, Cavalli G. Multiscale 3D genome rewiring during mouse neural development., 2017, 171(3): 557–572.e524.
[66] Rubin AJ, Barajas BC, Furlan-Magaril M, Lopez-Pajares V, Mumbach MR, Howard I, Kim DS, Boxer LD, Cairns J, Spivakov M, Wingett SW, Shi M, Zhao Z, Greenleaf WJ, Kundaje A, Snyder M, Chang HY, Fraser P, Khavari PA. Lineage-specific dynamic and pre-established enhancer- promoter contacts cooperate in terminal differentiation., 2017, 49(10): 1522–1528.
[67] Ediger BN, Lim HW, Juliana C, Groff DN, Williams LT, Dominguez G, Liu JH, Taylor BL, Walp ER, Kameswaran V, Yang J, Liu C, Hunter CS, Kaestner KH, Naji A, Li C, Sander M, Stein R, Sussel L, Won KJ, May CL, Stoffers DA. LIM domain-binding 1 maintains the terminally differentiated state of pancreatic β cells., 2017, 127(1): 215–229.
[68] Carpenter AC, Bosselut R. Decision checkpoints in the thymus., 2010, 11(8): 666–673.
[69] Solovei I, Wang AS, Thanisch K, Schmidt CS, Krebs S, Zwerger M, Cohen TV, Devys D, Foisner R, Peichl L, Herrmann H, Blum H, Engelkamp D, Stewart CL, Leonhardt H, Joffe B. LBR and lamin A/C sequentially tether peripheral heterochromatin and inversely regulate differentiation., 2013, 152(3): 584–598.
[70] Spilianakis CG, Flavell RA. Long-range intrachromosomal interactions in the T helper type 2 cytokine locus., 2004, 5(10): 1017–1027.
[71] Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA. Interchromosomal associations between alternatively expressed loci., 2005, 435(7042): 637–645.
[72] Mifsud B, Tavares-Cadete F, Young AN, Sugar R, Schoenfelder S, Ferreira L, Wingett SW, Andrews S, Grey W, Ewels PA, Herman B, Happe S, Higgs A, LeProust E, Follows GA, Fraser P, Luscombe NM, Osborne CS. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C., 2015, 47(6): 598–606.
[73] Rao SS, Huntley MH, Durand NC, Stamenova EK, Bochkov ID, Robinson JT, Sanborn AL, Machol I, Omer AD, Lander ES, Aiden EL. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping., 2014, 159(7): 1665–1680.
[74] Palstra RJ, Tolhuis B, Splinter E, Nijmeijer R, Grosveld F, de Laat W. The beta-globin nuclear compartment in development and erythroid differentiation., 2003, 35(2): 190–194.
[75] Zhai YN, Xu Q, Guo Y, Wu Q. Characterization of a cluster of CTCF-binding sites in a protocadherin regulatory region., 2016, 38(4): 323–336. 翟亚男, 许泉, 郭亚, 吴强. 原钙粘蛋白基因簇调控区域中成簇的CTCF结合位点分析. 遗传, 2016, 38(4): 323–336.
[76] Ong CT, Corces VG. CTCF: an architectural protein bridging genome topology and function., 2014, 15(4): 234–246.
[77] Ghirlando R, Felsenfeld G. CTCF: making the right connections., 2016, 30(8): 881–891.
[78] Sun S, Del Rosario BC, Szanto A, Ogawa Y, Jeon Y, Lee JT. Jpx RNA activates xist by evicting CTCF., 2013, 153(7): 1537–1551.
[79] Hansen AS, Hsieh THS, Cattoglio C, Pustova I, Saldaña- Meyer R, Reinberg D, Darzacq X, Tjian R. Distinct classes of chromatin loops revealed by deletion of an RNA-Binding region in CTCF., 2019, 76(3): 395–411.e313.
[80] Saldaña-Meyer R, Rodriguez-Hernaez J, Escobar T, Nishana M, Jácome-López K, Nora EP, Bruneau BG, Tsirigos A, Furlan-Magaril M, Skok J, Reinberg D. RNA interactions are essential for CTCF-Mediated genome organization., 2019, 76(3): 412–422.e415.
[81] Stadhouders R, Thongjuea S, Andrieu-Soler C, Palstra RJ, Bryne JC, van den Heuvel A, Stevens M, de Boer E, Kockx C, van der Sloot A, van den Hout M, van Ijcken W, Eick D, Lenhard B, Grosveld F, Soler E. Dynamic long- range chromatin interactions control Myb proto-oncogene transcription during erythroid development., 2012, 31(4): 986–999.
[82] Shukla S, Kavak E, Gregory M, Imashimizu M, Shutinoski B, Kashlev M, Oberdoerffer P, Sandberg R, Oberdoerffer S. CTCF-promoted RNA polymerase II pausing links DNA methylation to splicing., 2011, 479(7371): 74–79.
[83] Weintraub AS, Li CH, Zamudio AV, Sigova AA, Hannett NM, Day DS, Abraham BJ, Cohen MA, Nabet B, Buckley DL, Guo YE, Hnisz D, Jaenisch R, Bradner JE, Gray NS, Young RA. YY1 is a Structural Regulator of enhancer- promoter loops., 2017, 171(7): 1573–1588.e1528.
[84] Heidari N, Phanstiel DH, He C, Grubert F, Jahanbani F, Kasowski M, Zhang MQ, Snyder MP. Genome-wide map of regulatory interactions in the human genome., 2014, 24(12): 1905–1917.
[85] Ye BY, Shen WL, Wang D, Li P, Zhang Z, Shi ML, Zhang Y, Zhang FX, Zhao ZH. ZNF143 is involved in CTCF- mediated chromatin interactions by cooperation with cohesin and other partners., 2016, 50(3): 496–503.
[86] Deng W, Rupon JW, Krivega I, Breda L, Motta I, Jahn KS, Reik A, Gregory PD, Rivella S, Dean A, Blobel GA. Reactivation of developmentally silenced globin genes by forced chromatin looping., 2014, 158(4): 849–860.
[87] Magklara A, Yen A, Colquitt BM, Clowney EJ, Allen W, Markenscoff-Papadimitriou E, Evans ZA, Kheradpour P, Mountoufaris G, Carey C, Barnea G, Kellis M, Lomvardas S. An epigenetic signature for monoallelic olfactory receptor expression., 2011, 145(4): 555–570.
[88] Monahan K, Lomvardas S. Monoallelic expression of olfactory receptors., 2015, 31: 721–740.
[89] Flyamer IM, Gassler J, Imakaev M, Brandão HB, Ulianov SV, Abdennur N, Razin SV, Mirny LA, Tachibana- Konwalski K. Single-nucleus Hi-C reveals unique chromatin reorganization at oocyte-to-zygote transition., 2017, 544(7648): 110–114.
[90] Darrow EM, Huntley MH, Dudchenko O, Stamenova EK, Durand NC, Sun Z, Huang SC, Sanborn AL, Machol I, Shamim M, Seberg AP, Lander ES, Chadwick BP, Aiden EL. Deletion of DXZ4 on the human inactive X chromosome alters higher-order genome architecture., 2016, 113(31): E4504–E4512.
[91] Quinodoz SA, Ollikainen N, Tabak B, Palla A, Schmidt JM, Detmar E, Lai MM, Shishkin AA, Bhat P, Takei Y, Trinh V, Aznauryan E, Russell P, Cheng C, Jovanovic M, Chow A, Cai L, McDonel P, Garber M, Guttman M. Higher-order inter-chromosomal hubs shape 3D Genome organization in the nucleus., 2018, 174(3): 744– 757.e724.
[92] Zheng M, Tian SZ, Capurso D, Kim M, Maurya R, Lee B, Piecuch E, Gong L, Zhu JJ, Li Z, Wong CH, Ngan CY, Wang P, Ruan X, Wei CL, Ruan Y. Multiplex chromatin interactions with single-molecule precision., 2019, 566(7745): 558–562.
Molecular mechanism of the 3D genome structure and function regulation during cell terminal differentiation
Ke Yang, Zheng Xue, Xiang Lv
The eukaryotic chromatin is folded into highly complex three-dimensional (3D) structures, which plays an important role in the precise regulation of gene expression and normal physiological function. During differentiation and terminal maturation, cells usually undergo dramatic morphology and gene expression changes, accompanied by significant changes in the 3D structure of the genome. In this review, we provide a comprehensive view of the spatial hierarchical organization of the genome, including chromosome territories, A/B compartment, topologically associating domains (TADs) and looping, focusing on recent progresses in the dynamic 3D genomic structural changes and functional regulation during cell differentiation and terminal maturation. In the end, we summarize the unsolved issues as well as prospects of the 3D genome research in cell differentiation and maturation.
3D genome; A/B compartment; topologically associating domain (TAD); looping; cell terminal differentiation
2019-09-05;
2019-12-13
国家重点研发计划干细胞及转化研究专项(编号:2016YFA0100603),中国医学科学院医学与健康科技创新工程协同创新团队项目(编号:2016-I2M-3-002)和国家自然科学基金面上项目(编号:81670108) 资助[Supported by the National Key Research and Development Program for Stem Cells and Transformation Research (No. 2016YFA0100603), Chinese Academy of Medical Sciences Medical and Health Science and Technology Innovation Engineering Collaborative Innovation Team Project (No. 2016-I2M-3-002) and General Program of the National Natural Science Foundation of China (No. 81670108)]
杨科,博士研究生,研究方向:造血调控与三维基因组。E-mail: yangk_92@163.com
吕湘,博士,教授,研究方向:造血调控与三维基因组。E-mail: lvxiang@pumc.edu.cn
10.16288/j.yczz.19-270
2019/12/26 9:41:30
URI: http://kns.cnki.net/kcms/detail/11.1913.r.20191224.1742.004.html
(责任编委: 方向东)
