CRISPR/Cas9基因编辑在三维基因组研究中的应用
2020-03-04刘沛峰吴强
刘沛峰,吴强
综述
CRISPR/Cas9基因编辑在三维基因组研究中的应用
刘沛峰,吴强
上海交通大学系统生物医学研究院比较生物医学研究中心,系统生物医学教育部重点实验室,上海 200240
CRISPR/Cas9系统在基因编辑方面具有巨大优势,能够低成本、可编程、方便快捷地用于动物、植物以及微生物的基因组靶向编辑和功能改造。三维基因组学是近年来兴起的一门研究染色质高级结构动态调控及基因组生物学功能的交叉学科。在三维基因组研究中,通常采用对DNA片段进行基因编辑以模拟基因组结构性变异,标记特定DNA片段,进而研究调控元件对于基因调控、细胞分化、组织发生、器官形成、个体发育的影响,最终阐明三维基因组的组装调控机制和生物学功能。因此,CRISPR及其衍生技术为研究三维基因组提供了极好的遗传学工具。本文主要综述了CRISPR片段编辑及其衍生技术在三维基因组调控与功能研究中的应用,以期为后续研究工作提供理论参考以及新的研究思路。
CRISPR/Cas9系统;三维基因组;DNA片段编辑;染色质重排;Cas9核酸酶内切机制
成簇规律间隔短回文重复序列及其相关蛋白(clustered regularly interspaced short palindromic repeats/CRISPR-associated protein, CRISPR/Cas)是古菌和细菌中一种用于抵抗噬菌体、病毒DNA等外来遗传因子的原核生物获得性免疫系统。该系统可以将外源入侵DNA的小片段整合到宿主基因组的CRISPR重复序列中靠近前导序列(leader sequence)的位置,形成CRISPR重复-间隔基因座。这种为外源DNA侵染所做的遗传记录使宿主产生遗传记忆并在下次面对同样的入侵者时可以进行防御[1]。当相同的外源DNA再次入侵时,CRISPR重复-间隔基因座会转录并加工为crRNA (CRISPR RNA),并引导DNA核酸内切酶Cas家族蛋白对入侵的核苷酸序列进行靶向切割。Cas9蛋白是Cas蛋白家族中的一员,Cas9蛋白参与组成的CRISPR/Cas9系统是CRISPR/Cas系统的子系统之一。自然界的CRISPR/ Cas9系统经过原始创新性改造可以在一个单链引导RNA (single guide RNA, sgRNA)介导下编辑基因组特定位点(图1)[2~4]。因其灵活、高效、方便、操作性强等特点,该基因组编辑系统很快被广泛应用于生物医药及人类健康等领域。近年来,随着进一步的研究发展,更多CRISPR/Cas9系统的应用方法被开发出来,在不同的研究领域发挥重要作用,三维基因组即是受益于CRISPR/Cas9系统的研究领域之一。
三维基因组是近年来兴起的研究生物体遗传物质空间结构及其动态调控和信息传递规律的学科,主要研究染色体内部的空间盘曲折叠或染色体间的立体相互关系对各种生命过程的影响及其调控机制。在真核细胞间期,不同染色体被规则排列在特定的“领地”内[5,6],而在一条染色体内部,不同染色质区域的开放状态、盘曲折叠的方式也不尽相同[7,8],形成不同的拓扑结构域[9,10]。以往人们只注重研究染色体上DNA的线性序列及其与蛋白质的结合和调控机制,因为线性序列承载了遗传信息的传递功能。但后来科学研究发现,基因组DNA一维线性序列中包含有三维基因组的架构信息,并且细胞核内复杂的三维基因组结构可以控制调控元件与靶基因之间的特异性相互作用,从而影响基因的时空表达,从立体层面调控着生命过程[11]。此外,许多疾病的发生发展已经被证明与三维基因组密切相关[12,13]。随着3C (chromosome conformation capture)、4C (circular chromosome conformation capture)、Hi-C (high- resolution chromosome conformation capture)和ChIA- PET (chromatin interaction analysis by paired-end tag sequencing)等各种构象捕获技术[14~18]以及高分辨率显微成像技术的发展,人们对染色体空间组织原则和三维架构规律的认识越来越深入。CRISPR/Cas9系统及其衍生技术更是极大地促进了三维基因组的研究。本文主要综述了CRISPR/Cas9基因编辑技术在三维基因组研究中的应用。
1 CRISPR/Cas9基因编辑机制
1.1 基因组单位点编辑
20世纪80年代开发的基因打靶技术实现了基因组中任何位点的编辑或改造,结合干细胞技术能够产生基因组靶向修饰的小鼠,有力推动了小鼠反向遗传学的发展。基因打靶技术通过直接同源重组的方法将外源DNA序列导入目标基因组,实现目标位点DNA序列的突变,删除或插入[19~21](图1A)。随后开发的锌指核酸酶(zinc finger nuclease, ZFN)和类转录激活因子效应物核酸酶(transcription activator- like effector nuclease, TALEN)分别将可以特异性识别DNA序列的锌指蛋白和类转录激活因子效应物与核酸内切酶Ⅰ结合,造成基因组特定位点的双链断裂(double-strand break, DSB),接着利用细胞内的DNA修复系统,完成对基因组特定位点的改造。这两种技术的出现进一步促进了基因组的靶向切割和编辑领域的发展[22~26](图1,B和C)。2012年兴起的CRISPR/Cas9技术使得人们可以非常容易地靶向编辑几乎任何物种的基因组[2~4,27]。具体地讲,Cas9核酸内切酶在单个sgRNA的引导下能够切割基因组符合PAM (protospacer adjacent motif)规则的任意位点,引起DNA双链断裂损伤。在细胞内DNA损伤修复系统的作用下,单位点切割(单切)断裂处虽然被修复,但却可能会被引入突变,最终达到编辑基因组的效果(图1D)。
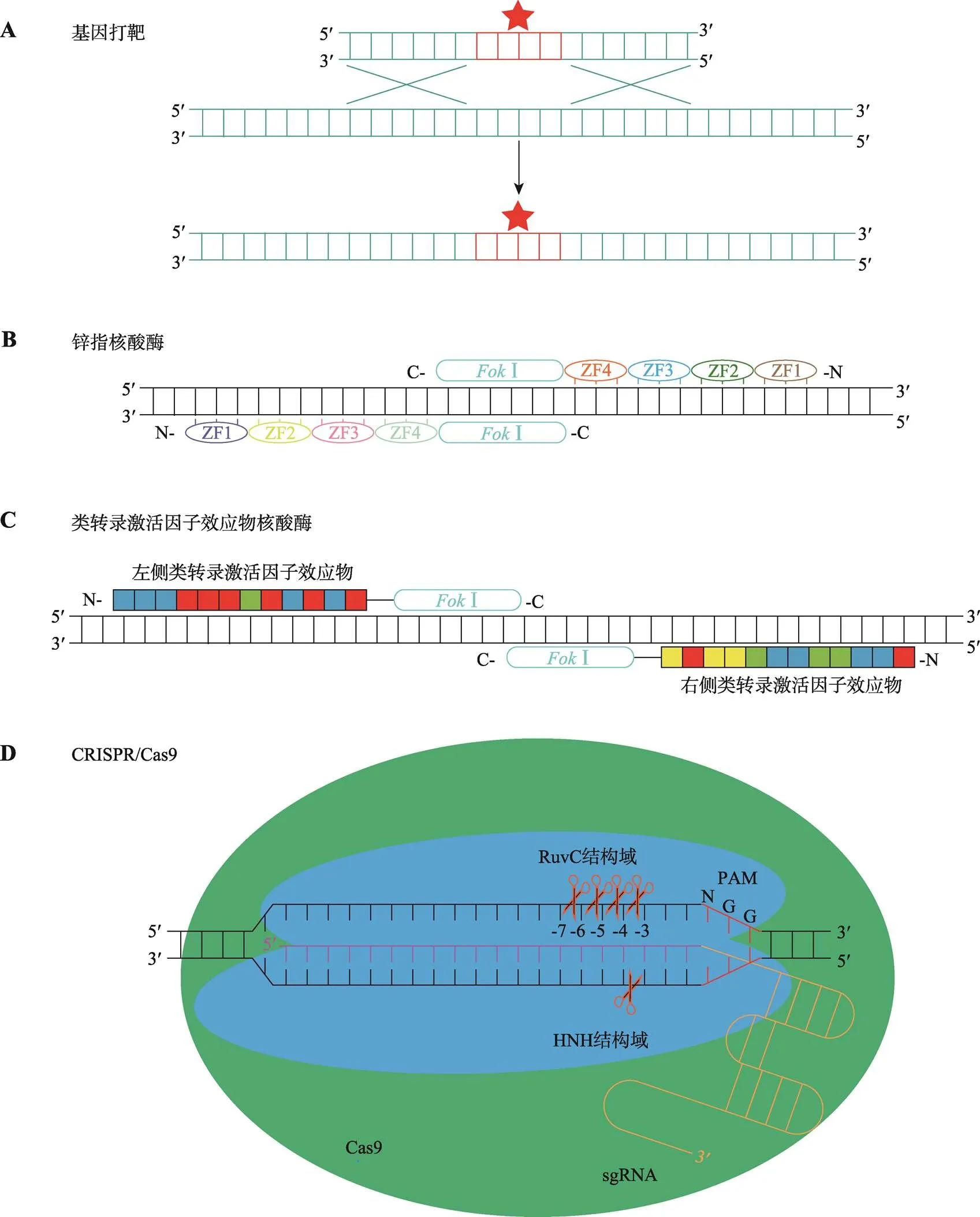
图1 基因组编辑技术
A:基因打靶技术原理。基因打靶技术利用同源重组的方法将目的片段导入基因组。B:锌指核酸酶基因组编辑技术原理。将特异性识别DNA序列的锌指结构串联,与Ⅰ核酸内切酶融合,实现基因组靶向切割。再利用DNA修复系统实现基因组编辑。C:类转录激活因子效应物核酸酶基因组编辑技术原理。将特异性识别DNA序列的类转录激活因子效应物串联,与Ⅰ核酸内切酶融合,实现基因组靶向切割,再利用DNA修复系统实现基因组编辑。D:CRISPR/Cas9基因组编辑技术原理。Cas9蛋白可以被sgRNA引导至基因组特定位点,Cas9不具有核酸外切酶活性,仅仅具有核酸内切酶活性。Cas9的HNH和RuvC结构域分别催化互补链和非互补链的核酸内切反应造成DNA的双链断裂。HNH结构域造成的断裂位点位于PAM序列上游第3与第4个核苷酸之间,而RuvC结构域在PAM序列上游可造成多达4种不同的DNA断裂。
细胞的DNA修复系统主要包括同源重组(homologous recombination, HR)、非同源末端连接(nonhomologous end joining, NHEJ)和微同源末端连接(microhomology-mediated end joining, MMEJ)等。在没有模板的情况下,细胞主要选择NHEJ和MMEJ修复通路,产生随机突变进行基因组编辑。在提供模板的情况下,细胞利用HR修复通路产生遵照模板的精准编辑[27,28]。
1.2 CRISPR/Cas9双位点DNA片段编辑
单个sgRNA介导的单位点编辑很难造成基因组结构性变异(structural variation, SV),而一对sgRNA介导的双位点切割(CRISPR/Cas9双切系统)能够引起各种各样的染色质重排(chromosomal rearrangement),模拟基因组结构性变异。在一对sgRNA的引导下,Cas9核酸内切酶能够同时产生两个切口形成4个DNA双链断裂末端(Ⅰ~Ⅳ) (图2A)。如果断裂末端Ⅰ和断裂末端Ⅳ连接,则会造成两接口间的DNA片段删除(图2A)。由于DNA双链的Watson链和Crick链反向互补且都具有方向性,中间的DNA片段反转后仍然能够连接到基因组原位,也就是说断裂末端Ⅰ和Ⅲ以及断裂末端Ⅱ和Ⅳ相互连接,最终会形成DNA片段反转(图2A)。如果断裂末端Ⅲ和断裂末端Ⅱ连接,则会造成断裂末端Ⅰ和断裂末端Ⅳ中间的DNA片段重复或成环(图2A)。所以,CRISPR/Cas9双切系统能够形成DNA片段删除、反转、成环或重复,即所谓的DNA片段编辑[28~30]。由于三维基因组中存在大量的染色质重排,包括DNA片段删除、反转、重复、易位等,而这些染色质重排现象都可以利用DNA大片段编辑技术进行模拟,所以三维基因组结构和功能的研究为CRISPR/Cas9系统的DNA片段编辑技术提供了广阔的应用空间。同时,一对sgRNA编辑基因组的方法在应用于二倍体或多倍体生物中的高度同源序列时也表现出了足够的效率,这进一步扩展了CRISPR/ Cas9双切系统的应用前景[31]。
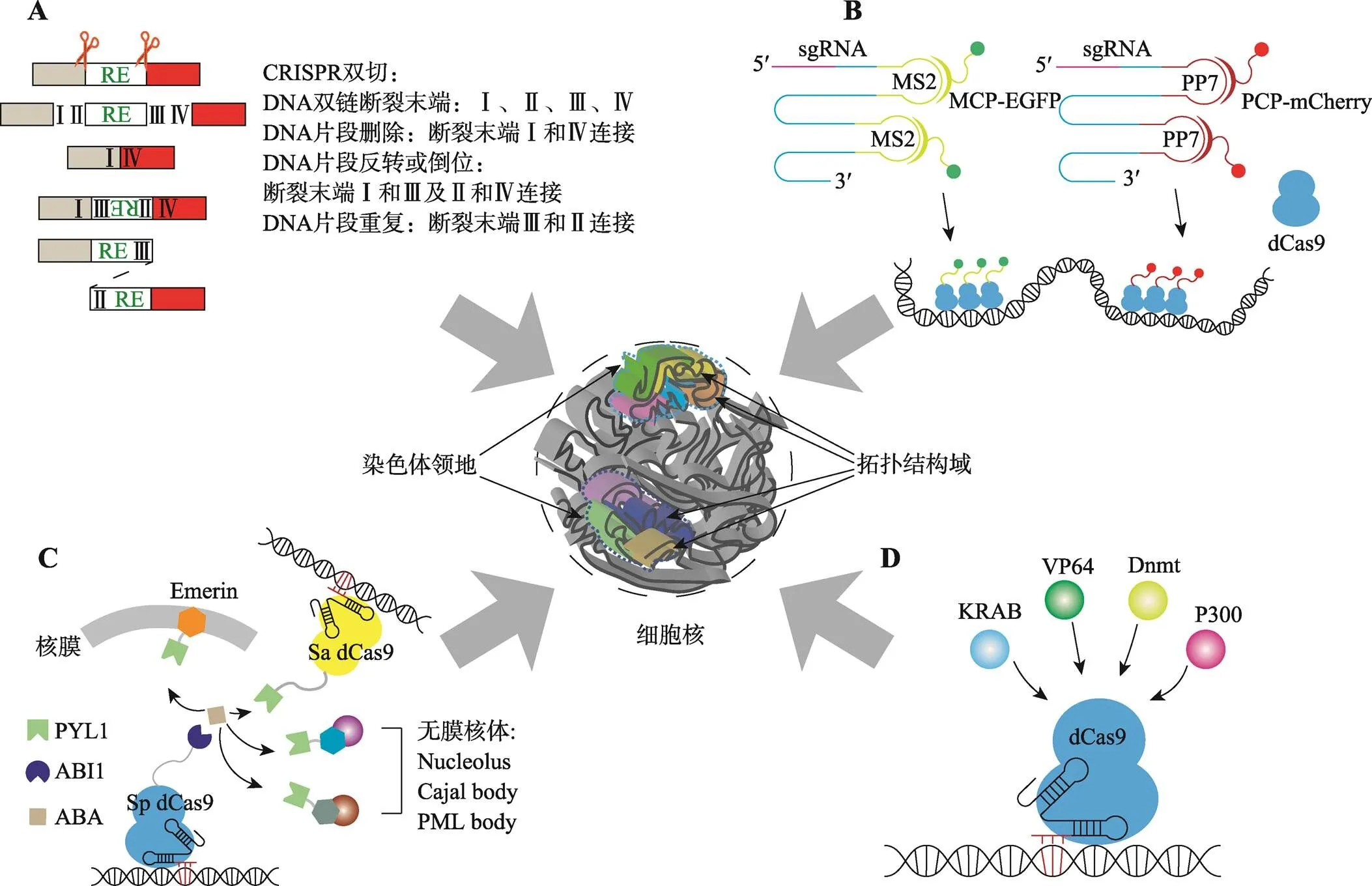
图2 CRISPR在三维基因组研究中的技术方法
A:CRISPR介导的DNA片段编辑。通过一对sgRNA介导Cas9在基因组中进行靶向双位点切割,可以造成基因组片段删除、反转、重复或成环等现象,为三维基因组研究提供模型。B:CRISPR介导的基因组位点可视化。通过在sgRNA上加入MS2与PP7等茎环结构,招募融合了荧光蛋白的MCP、PCP等蛋白,将荧光信号特异性地标记在特定的基因组位点。C:CRISPR介导的基因组位点重定位。利用植物激素ABA介导的PYL1与ABI1的相互作用,将PYL1和ABI1分别结合在不同系统的dCas9或其他核体特有的蛋白上,可以将特定的基因组位点定位到新的目标位点。D:以CRISPR为平台的靶向效应系统。将激活或抑制转录以及表观遗传修饰相关蛋白结合在dCas9上,可以特异性地使基因组特定位点的转录水平以及表观遗传学修饰改变。
1.3 精准且可预测的基因编辑
通常只有同源重组通路介导的DNA修复被认为是精准的,所以CRISPR/Cas9基因编辑只有在提供模板的情况下通过HR通路实现精准编辑。但近年来的研究发现,经典的或传统的非同源末端连接修复通路(classic or canonic nonhomologous end joining, cNHEJ)也是细胞的精准修复通路,而选择性非同源末端连接修复通路(alternative nonhomologous end joining, aNHEJ或alt-NHEJ)和微同源末端连 接修复通路才是易错的(error-prone)[32]。所以,提高cNHEJ修复通路效率或抑制aNHEJ和MMEJ修复通路的效率,例如敲低CtIP或FANCD2等在aNHEJ修复通路中发挥作用的蛋白,可以在没有外源模板的情况下实现精准基因编辑[33]。与HR类似,MMEJ在修复过程中也利用了同源模板,不过其利用的同源序列较短,一般为5~25 bp左右。并且MMEJ使用的同源模板不是来自于外源导入而是位于DSB附近[34]。通过该通路,在不提供模板的情况下,精确的基因组编辑(如反转和删除)也可以被高质量地完成。
Cas9切割DNA的功能是由HNH结构域以及RuvC结构域共同实现的。Cas9切割DNA时,HNH结构域切割sgRNA互补链,而RuvC结构域切割sgRNA非互补链。两个结构域制造的切点分别位于两条链上的PAM序列上游第3与第4个核苷酸之间,产生平头的双链DNA断裂末端。最近的研究发现,Cas9切割基因组时,RuvC结构域也有一定概率在PAM序列上游第4和第5个核苷酸间造成DNA断裂,从而在切割产物的5¢端位置产生一个碱基突出的粘性末端。这些突出的粘性末端在细胞内源DNA聚合酶作用下能够被补平,并在DNA连接酶的作用下被精准连接。这就为CRISPR/Cas9基因编辑后可预测的单碱基插入提供了一条可能途径[32,33,35,36]。进一步利用DNA片段编辑方法研究发现,SpCas9核酸内切酶的RuvC结构域在PAM位点上游第5与第6以及第6与第7个核苷酸间均有一定几率造成DNA断裂,使切割产物产生两个或3个碱基突出的粘性末端,从而实现两个和3个碱基插入的可预测CRISPR/Cas9基因组编辑[37](图1D)。
因为单位点Cas9切割后产生的粘性末端很容易被完全修复并被再次切割,而一对sgRNA介导的双位点切割后产生的粘性末端在染色体重排后几乎不可能配对,所以在CRISPR/Cas9双切系统中,Cas9切割后产生的突出末端经过补平和连接后更容易被观察到[33,37]。也就是说,两个和3个碱基插入的效率在双切的情况下远远高于单切的情况[37],这就解释了为什么单切时几乎看不到一个以上碱基的可预测插入[35,36]。
2 CRISPR研究三维基因组的技术方法
CRISPR/Cas系统在三维基因组中的应用主要依靠的是其序列特异性结合的特性。利用该特性,改造或未被改造的多种Cas9蛋白可以被招募到基因组特定位点,进行切割或实现其他功能。
2.1 CRISPR介导的DNA片段编辑
通过一对sgRNA介导的CRISPR/Cas9双位点切割,研究人员可以制作基因组调控元件插入、删除、反转的细胞或动植物模型,并利用这些模型研究三维基因组如何调控基因表达等生命过程,以及疾病发生发展的规律[29](图2A)。例如,染色质架构蛋白CTCF (CCCTC-binding factor)的结合位点反转、插入或敲除等都表明了CTCF对于细胞三维基因组架构维持和动态调控的重要性[11,38,39];CRISPR/Cas9双切系统介导的DNA大片段删除还能够筛选出基因组潜在的调控元件[40]。最近,利用DNA片段编辑技术发现了控制哺乳动物基因组复制事件时空特性的重要顺式元件[41]。删除与反转基因组部分片段会导致DNA复制时间的变化,说明这些片段包含着调控DNA复制的元件。单独删除与组合删除这些元件对DNA复制的影响不同,说明了这些元件在发挥作用时的相互依赖性。Hi-C结果也显示,这些元件的删除对于基因组区室划分以及拓扑结构域的强度都有影响。除此之外,多个靶向同一染色体不同位置的sgRNA介导的CRISPR/Cas9系统可以在老鼠或人类细胞中敲除一整条染色体,甚至将单倍体酵母的所有染色体融合在一起,创造出可以生存的只有一条染色体的酵母[42,43]。
2.2 CRISPR介导的基因组位点可视化
生命信息传递以及基因组功能调节与染色质的空间组织架构和动态变化关系密切,而基因组位点在三维空间的直接可视化能够帮助理解基因组的空间组织架构以及其与基因表达调控等生命活动的关系。以往人们利用荧光原位杂交技术(fluorescence in situ hybridization, FISH)观察基因组位点在细胞核中的位置。而如今,利用CRISPR/Cas系统结合基因组的序列特异性,失活的dCas9 (nuclease-deactivated Cas9, D10A和H841A)荧光蛋白嵌合体可以被sgRNA招募到基因组的特定位点,从而使活细胞的特定染色体片段的时空动态可视化。Chen等[44]利用该方法观察了端粒在活细胞中的动态变化,发现端粒在受损后,在细胞核内的移动显著增加。同时他们还观察了编码基因在有丝分裂中的细胞核内的位置变化[44]。
dCas9/sgRNA复合体的稳定性以及dCas9蛋白结合DNA的高亲和性使得该方法可以被方便高效广泛地应用于细胞基因组特定位点标记。在体外孵育组装的dCas9/sgRNA复合物也可以与细胞内的DNA杂交,并且这样在体外组装的系统可以在基因组不同的位点标记不同的颜色,从而同时检测多个位点的位置,而这是研究染色质相互作用的关键[45]。同时使用多种细菌的同源dCas9/sgRNA组合也可以构建多色CRISPR/dCas9可视化系统[46,47]。除此之外,另一种策略也可以解决多色标记问题:在sgRNA的支架上加入两个拷贝的MS2或PP7 RNA茎环结构(图2B)。MS2可以被MCP (MS2 coat protein)蛋白特异性识别,而PP7可以被PCP (PP7 coat protein)蛋白特异性识别。向细胞内转入MCP-EGFP和PCP- mCherry融合蛋白的表达载体后,不同基因组靶点就可以被EGFP和mCherry同时标记[48,49]。在此基础上,CRISPRainbow等更加高效的技术进一步促进了基因组可视化的发展[50]。此外,dCas9的N端与C端的核定位信号(nuclear localization sequences, NLS)插入,sgRNA支架上的U-A翻转、发夹结构延长等一些技术上的难题被攻克也使得可视化更加高效稳定[44,51]。染色体上重复的DNA元件可以被单独一种特异性的sgRNA结合CRISPR/dCas9系统稳定地显示,而非重复的DNA元件也可以被一系列串联覆盖目的片段的sgRNA协同CRISPR/dCas9系统标记出来。在sgRNA支架上加入更多的MS2等茎环结构可以增强单个位点的荧光强度,减少标记非重复区域时所需的sgRNA的数量[52]。提高sgRNA的表达水平并利用诸如强力霉素(doxycycline)介导的启动子等方法降低dCas9蛋白的表达可以降低信噪比。
2.3 CRISPR介导的基因组位点重定位
基因组位点在核内的位置与基因表达是否有联系?这是三维基因组学关注的关键问题之一。程序性的控制基因组空间架构对于研究细胞核内结构如何影响基因调控和细胞功能十分重要。参考LacI- LacO方法[53,54],依赖于CRISPR/dCas9系统的CRISPR- GO (CRISPR-genome organization)方法被建立起来。该方法利用植物激素脱落酸(abscisic acid, ABA)介导的PYL1 (pyrabactin resistance (PYR)-like)与ABI1 (ABA insensitive 1)的相互作用,将ABI1融合至dCas9蛋白上,而将PYL1融合至核膜蛋白Emerin上或其他核体如卡哈尔体(Cajal body)的标志物Coilin上。这样通过dCas9/sgRNA的靶向作用,就可以将基因组特定片段重定位至细胞核外周或与无膜核体共定位。该方法对于基因组重复和非重复区域都十分有效,并且在除去ABA后,重定位的效果是可逆的[55]。
类似的,Morgan等[56]利用金黄色葡萄球菌(,Sa)和酿脓性链球菌(, Sp)的dCas9,结合ABA信号通路相关组件,开发出了介导两个基因组位点共定位的工具CLOuD9 (chromatin loop reorganization using CRISPR-dCas9)。该工具可以可逆地介导染色质环的形成,改变特定区域基因的表达(图2C)。
2.4 以CRISPR为平台的靶向效应系统
CRISPR/Cas系统为蛋白靶向DNA片段提供了一个特异性良好的稳定平台。将dCas9与具有不同调节功能的效应因子结合,利用sgRNA可以将dCas9靶向传递到特定的基因组位点的特性,就可以实现特定位点的功能调控。研究人员在这一方面已经做了大量的尝试[57~62],其中以CRISPR interference (CRISPRi)最为典型[63]。在大肠杆菌和人类细胞中,CRISPRi通过sgRNA将dCas9靶向特定的蛋白编码区域,利用dCas9结合DNA的空间位阻,通过干扰RNA聚合酶介导的转录延伸抑制相关基因的表达。这种方法可逆且高效,可以同时针对多个基因进行干扰。在此基础上,在dCas9后加入转录抑制或激活因子(如KRAB或VP64),可以实现特定基因转录的抑制或激活[64]。除此之外,将激活或抑制效应因子与植物激素诱导系统和CRISPR/Cas系统相结合,可以实现可诱导可逆转的基因表达激活或抑制。两套植物激素诱导系统同时联用使得“AND”或“OR”这样的逻辑调控成为可能[65]。但是,dCas9融合蛋白在活体内的转导效率很低。另外,在体转导经常使用病毒载体,而编码dCas9/ gRNA和转录激活因子的序列长度超过了大部分常见病毒载体可以承受的范围,因此将靶向基因激活系统用于在体研究是一个难点。Liao等[62]开发了一个能够装载在一个病毒载体内、dgRNA (dead sgRNA, 识别序列为14或15 bp,可以抑制Cas9的切割活性)与MPH (MS2:P65:HSF1)转录激活复合物融合、具有高靶基因激活水平的系统,将其注射入表达Cas9的小鼠体内,可以有效地调节基因表达,改变基因位点的表观遗传学状态,从而改善小鼠模型的疾病表型。并且该系统因其使用的Cas9并未失去核酸酶活性,故可以同时介导不同基因的转录激活和基因敲除[62]。该系统不仅进一步推动了靶向基因激活系统的发展,同时也证明了CRISPR在疾病治疗应用中的前景。
将dCas9与胞嘧啶脱氨酶融合在一起,可以作为靶向的单碱基编辑器[61,66,67]。这一技术可以用于修正某些疾病中的单碱基突变,同样也可以用于验证转录因子结合基序(motif)中的关键碱基,阐明转录因子与DNA的结合方式。
通过dCas9与具有甲基化、去甲基化、乙酰化和去乙酰化等功能的蛋白质的融合,CRISPR/Cas系统亦可用于特定位点表观遗传的修饰[60,68~70]。这种方法不会改变全基因组的表观遗传修饰,仅仅改变基因组特定位点和组蛋白的表观遗传状态,靶向操纵常染色质与异染色质状态的转变。因此,这样的方法可以操控特定位点的表观遗传状态,提供以往难以得到的证据与结论,在三维基因组研究中尤为重要。最后,通过靶向调控特定基因的表达,该技术亦具有癌症治疗的潜能。例如,将dCas9的C端融合KRAB (the Krüppel-associated box transcriptional repression domain)等表观遗传抑制因子,靶向抑制肝癌中表达上调且促进肿瘤发展的颗粒蛋白(Granulin, GRN)基因的表达,可以抑制肿瘤细胞增殖,减少肿瘤球的形成[70]。此外,使用dCas9靶向激活肿瘤细胞中肿瘤相关抗原(tumor-associated antigens, TAAs)的表达,可以诱导机体对肿瘤细胞产生更强的免疫识别,从而利用免疫系统清除肿瘤细胞[71](图2D)。
3 CRISPR研究三维基因组架构的机制
3.1 研究三维基因组的调控元件
人类基因组约2%的编码区包含2万多个基因,但这2万多个基因的时空表达却受到存在于98%的人类基因组非编码区中多达几百万个调控元件的精细控制。常见的基因组调控元件有启动子(promoter)、增强子(enhancer)、绝缘子(insulator)、基因座控制区(LCR: locus control region)等,而增强子、绝缘子、基因座控制区等远程调控元件通常通过与启动子之间的特异性空间相互作用调控基因表达,所以它们在基因表达调控以及三维基因组架构调节中发挥着重要作用。如何研究它们的功能及其具体机制是一个重要的难题,而CRISPR/Cas9系统则为解决这个难题提供了机遇。
因为一个sgRNA介导的Cas9单位点切割,能够在编码区造成小的插入或删除,引起蛋白移码突变,从而破坏基因功能,所以利用一个sgRNA的CRISPR/Cas9单位点切割系统在研究基因组编码区的功能中发挥了重要作用。由于非编码区不存在移码突变,Cas9单切后通常不能完全破坏非编码区的功能,单位点切割的CRISPR/Cas9系统在非编码区研究中难以发挥作用。而利用两个sgRNA的CRISPR/Cas9双位点切割系统(CRISPR/Cas9双切系统)很容易把单个调控元件完全敲除,从而容易研究成千上万的三维基因组调控元件。另外CRISPR/ Cas9双切系统除了造成DNA片段敲除,还可以造成DNA片段的反转、重复、易位等染色体结构性变异,因此DNA片段编辑对于研究DNA元件在三维基因组调控与功能中作用机制非常重要。例如,秀丽线虫()中X染色体的计量补偿效应是性染色体特殊的空间构象造成的现象。此现象是由于计量补偿复合物(dosage compensation complex, DCC)被招募到(recruitment elements on X)位点引起的。使用CRISPR/Cas9技术敲除位点能够引起这种特殊的空间构象消失,同时DCC对X染色体基因表达的抑制作用也随之消失[72]。此外,将CRISPRi系统与单细胞RNA-seq联用,通过对假定的调控元件进行组合干扰,能够筛选出基因的调控元件[73]。
3.2 CTCF架构机制
利用CRISPR/Cas9双切系统对染色质架构蛋白CTCF结合的DNA片段进行删除或反转,结合染色质构象捕获技术,证明了CTCF对于三维基因组结构的重要性以及其DNA结合序列的方向性[11,74~76]。首先,一对方向收敛或者说正向-反向(convergent or forward-reverse)的CTCF的结合位点(CTCF-binding site, CBS)能够更容易地介导CTCF与cohesin一起控制染色质环形成。其次,一对方向发散或者说反向–正向(divergent or reverse-forward)的CBSs通常位于拓扑结构域(topologically associating domains, TADs)的边界。再次,串联排列方向一致的CBSs能够平衡启动子的选择[77]。CTCF/cohesin介导的染色质环保证了基因组的增强子与合适的启动子相互作用,隔绝错误的DNA调控元件之间的联系,维持正常的基因表达,而CTCF结合位点的删除或反转的转变会改变染色质环的结构从而影响基因表达、发育分化甚至导致畸形或癌症等疾病的发生[11,78,79]。
3.3 增强子的方向性
哺乳动物基因组内许多增强子元件的附近都含有CBS,而CBS的方向可以决定CTCF结合DNA的方向,从而影响CTCF介导的染色质环的形成。这使得一些含有CBS的增强子方向特异性地与合适的启动子相互作用,如原钙粘蛋白(protocadherin,)基因簇的增强子HS5-1[80,81](图3A)。
原钙粘蛋白基因座包含α、β、γ 3个基因簇,并且基因启动子与增强子区域含有大量的CBSs (图3A)。在原钙粘蛋白基因座中,除了αc2、β1、γc4和γc5的启动子外,每个启动子区都含有正向的CBS,而增强子通常含有反向的CBS。正向-反向的CBS可以介导CTCF与cohesin一起形成染色质环,控制启动子与增强子间的相互作用。原钙粘蛋白基因簇的表达主要由其下游的两个增强子HS7以及HS5-1调控。原钙粘蛋白基因簇位点的表达主要由其下游的超级增强子调控[82,83]。
原钙粘蛋白基因簇的增强子HS5-1含有两个反向的CBSs,通常情况下,这两个反向的CBSs会介导HS5-1与上游的含有正向CBS的原钙粘蛋白启动子形成染色质环,从而使得HS5-1激活原钙粘蛋白而不是下游的原钙粘蛋白基因簇表达。如果使用CRISPR/Cas9双切系统在小鼠与细胞内对HS5-1进行反转,可以观察到HS5-1与下游的基因簇相互作用显著增加,同时,与上游的基因簇启动子的相互作用减少(图3B)。原钙粘蛋白的转录也相应地发生了显著下降[11,84]。这些现象都说明HS5-1在染色体原位环境中是以方向依赖性的方式调控原钙粘蛋白基因簇的表达。
3.4 绝缘子作用机制
哺乳动物基因组中的绝缘子元件可以隔绝增强子与不合适的启动子间的相互作用,也可以分割常染色质与异染色质以及不同的TAD区域。那么其潜在的机制是怎样的呢?CRISPR/Cas9双切系统介导的DNA片段插入、删除与反转技术证明了CBS可以不依赖方向地实现绝缘子的功能[77]。
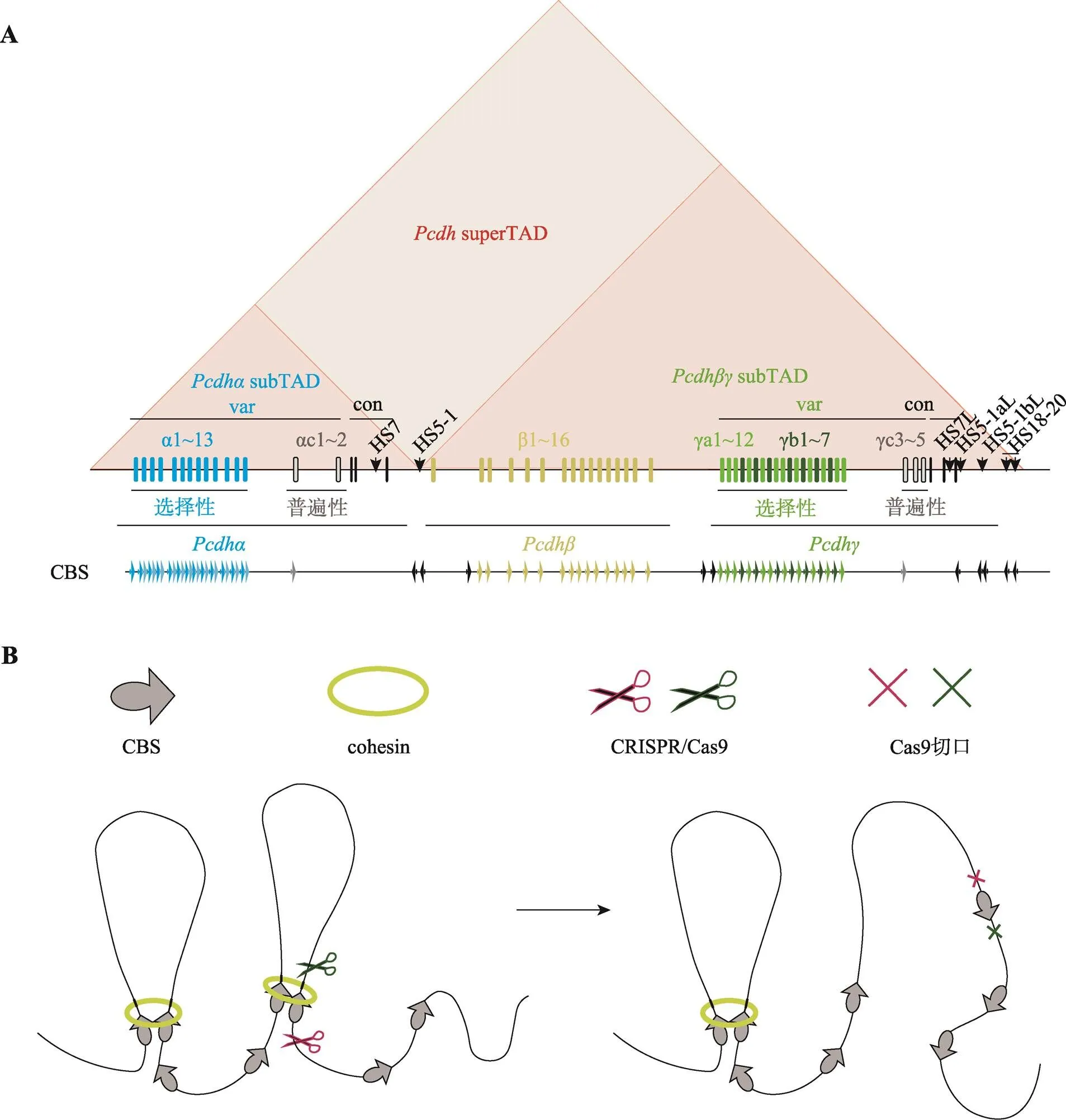
图3 人类原钙粘蛋白基因簇结构以及CRISPR介导的基因组编辑
A:人类原钙粘蛋白基因簇。人类原钙粘蛋白基因座包含α、β、γ 3个基因簇,并且在基因启动子与增强子区域包含大量的CTCF位点(CTCF-binding site, CBS)。通过方向性结合这些CBSs,CTCF可以介导染色质环形成,调控原钙粘蛋白基因表达。B:CRISPR系统介导的基因组元件反转改变染色质拓扑结构。基因组折叠的染色质环挤出模型(chromatin loop extrusion):沿染色质滑动的cohesin可以将染色质挤出形成染色质环,而cohesin的滑动会被一对方向收敛或者说正向–反向(convergent or forward-reverse)的CBS上的CTCF阻滞,这使得染色质环挤出的进程终止,两个CBS在三维空间上被拉近。利用CRISPR/Cas9系统反转CBS方向能够改变染色质环化方向,证明了CTCF位点的方向性在三维基因组折叠中的关键作用。
在原钙粘蛋白基因簇的启动子与增强子HS5-1之间插入单个或多个,正向或反向,成对的方向收敛或方向发散的CBSs后,使用定量高分辨率4C技术(quantitative high-resolution chromosome-conformation capture, QHR-4C)测定这些CBSs与基因组位点间的相互作用强弱。QHR-4C数据表明,这些CBSs都发挥了绝缘子的功能。这些插入的CBSs替代了与自己同向的CBSs的功能,和远处方向与自己相对的CBSs形成染色质环,从而破坏了原有的启动子–增强子相互作用,承担绝缘子的角色[77]。
删除原钙粘蛋白基因簇内原有的CBS会导致启动子或增强子跨过该CBS与更远处的DNA元件相互作用。这些结果说明原钙粘蛋白基因簇内原有的CBSs也作为绝缘子而存在着。
综合上面的现象可以发现,CBS其实是通过与其他方向相对的CBS形成染色质环从而隔离出一个个区室,防止不适当的DNA元件间相互作用,实现其绝缘子功能。
4 结语与展望
三维基因组研究对于人们深入理解发育、分化、基因表达调控等生命过程至关重要,而CRISPR/ Cas9系统则是研究三维基因组的重要工具。与其他技术相比,CRISPR/Cas9系统在精确性、可塑性、性价比和适用性等方面都有巨大的优势。但是如何利用这些优势并开发新的衍生技术则是未来努力的方向。
随着CRISPR/Cas9介导的基因组编辑越来越精准和可预测以及一对sgRNA介导DNA片段编辑技术的广泛使用[33],研究者可以花费更少的精力得到想要的突变类型,这对于快速发展的三维基因组研究具有巨大的推动作用。而开发编辑效果更为准确且脱靶效应更少的Cas9突变体以及更准确的CRISPR编辑方法,将会有助于CRISPR/Cas9系统更多更好地应用于三维基因组研究[85,86]。。
利用CRISPR/Cas9系统对染色质架构蛋白进行高通量的筛选,可以找到在基因组复杂网络中在三维架构方面发挥重要功能的因子。使用CRISPR文库干扰或敲除一系列潜在的染色质架构蛋白后,在经典的基因座如原钙粘蛋白[80,81]和珠蛋白()[11]等位点及其超级增强子区域中,利用染色质构象捕获等技术可以筛选出对染色质环或TADs等三维基因组重要组件的形成具有重要意义的蛋白。
对于原钙粘蛋白基因簇内增强子HS5-1的反转降低了HS5-1与上游原钙粘蛋白各启动子的相互作用,并且增加了HS5-1与下游原钙粘蛋白基因簇的相互作用(图3B)。这样的改变虽然显著降低了原钙粘蛋白的转录水平,却没有增强原钙粘蛋白的转录水平[11]。增强子和启动子之间通过远程相互作用形成的染色质环调控基因转录水平是一个研究热点和难点。
虽然有很多研究证明染色质的拓扑结构性质与TAD对于基因表达十分重要[38,78,79],但是最近的研究发现,将果蝇的染色体高度重排后,果蝇的基因表达受到的影响并不大[87]。虽然重排后的基因组内出现了反转、删除和重复等许多结构性变异,拓扑结构性质发生巨大的变化,染色质环和TAD等三维基因组高级结构被破坏,但是表达受到影响的基因只是一小部分。TAD的变化并不能很好的预测基因表达的变化[87]。这样的发现挑战了三维基因组学的以往认知。这一发现是否说明TADs对于全基因组基因表达的影响没有以前预测的那么重要?是否是果蝇染色体的特殊性导致了这样的结果?染色质环等三维基因组结构是否只在小范围的尺度影响着基因表达?这些猜测都有待进一步的研究验证。
总之,CRISPR/Cas9系统在靶向三维基因组的精确性、易用性等方面都具有巨大的优势。相信随着CRISPR/Cas9基因编辑及其衍生技术的进一步发展,这些新兴遗传学技术在三维基因组研究中会发挥越来越重要的作用。
[1] Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes., 2007, 315(5819): 1709–1712.
[2] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816–821.
[3] Gasiunas G, Barrangou R, Horvath P, Siksnys V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria., 2012, 109(39): E2579–E2586.
[4] Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system., 2013, 8(11): 2281–2308.
[5] Cremer T, Cremer M. Chromosome territories., 2010, 2(3): a003889.
[6] Tan LZ, Xing D, Chang CH, Li H, Xie XS. Three- dimensional genome structures of single diploid human cells., 2018, 361(6405): 924–928.
[7] de Laat W, Duboule D. Topology of mammalian developmental enhancers and their regulatory landscapes., 2013, 502(7472): 499–506.
[8] Gibcus JH, Dekker J. The hierarchy of the 3D genome., 2013, 49(5): 773–782.
[9] Dixon JR, Gorkin DU, Ren B. Chromatin domains: The unit of chromosome organization., 2016, 62(5): 668–680.
[10] Dekker J, Mirny L. The 3D genome as moderator of chromosomal communication., 2016, 164(6): 1110– 1121.
[11] Guo Y, Xu Q, Canzio D, Shou J, Li J, Gorkin DU, Jung I, Wu H, Zhai Y, Tang Y, Lu Y, Wu Y, Jia Z, Li W, Zhang MQ, Ren B, Krainer AR, Maniatis T, Wu Q. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function., 2015, 162(4): 900–910.
[12] Krijger PH, de Laat W. Regulation of disease-associated gene expression in the 3D genome., 2016, 17(12): 771–782.
[13] Norton HK, Phillips-Cremins JE. Crossed wires: 3D genome misfolding in human disease., 2017, 216(11): 3441–3452.
[14] Dekker J, Rippe K, Kleckner N. Capturing chromosome conformation., 2002, 295(5558): 1306–1311.
[15] Zhao Z, Tavoosidana G, Sjölinder M, Göndör A, Mariano P, Wang S, Kanduri C, Lezcano M, Sandhu KS, Singh U, Pant V, Tiwari V, Kurukuti S, Ohlsson R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions., 2006, 38(11): 1341– 1347.
[16] Simonis M, Klous P, Splinter E, Moshkin Y, Willemsen R, de Wit E, van Steensel B, de Laat W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C)., 2006, 38(11): 1348–1354.
[17] Lieberman-Aiden E, van Berkum NL, Williams L, Imakaev M, Ragoczy T, Telling A, Amit I, Lajoie BR, Sabo PJ, Dorschner MO, Sandstrom R, Bernstein B, Bender MA, Groudine M, Gnirke A, Stamatoyannopoulos J, Mirny LA, Lander ES, Dekker J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome., 2009, 326(5950): 289–293.
[18] Fullwood MJ, Liu MH, Pan YF, Liu J, Xu H, Mohamed YB, Orlov YL, Velkov S, Ho A, Mei PH, Chew EG, Huang PY, Welboren WJ, Han Y, Ooi HS, Ariyaratne PN, Vega VB, Luo Y, Tan PY, Choy PY, Wansa KD, Zhao B, Lim KS, Leow SC, Yow JS, Joseph R, Li H, Desai KV, Thomsen JS, Lee YK, Karuturi RK, Herve T, Bourque G, Stunnenberg HG, Ruan X, Cacheux-Rataboul V, Sung WK, Liu ET, Wei CL, Cheung E, Ruan Y. An oestrogen- receptor-alpha-bound human chromatin interactome., 2009, 462(7269): 58–64.
[19] Evans MJ, Kaufman MH. Establishment in culture of pluripotential cells from mouse embryos., 1981, 292(5819): 154–156.
[20] Smithies O, Gregg RG, Boggs SS, Koralewski MA, Kucherlapati RS. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination., 1985, 317(6034): 230–234.
[21] Thomas KR, Folger KR, Capecchi MR. High frequency targeting of genes to specific sites in the mammalian genome., 1986, 44(3): 419–428.
[22] Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes: Zinc finger fusions toⅠ cleavage domain., 1996, 93(3): 1156–1160.
[23] Bibikova M, Carroll D, Segal DJ, Trautman JK, Smith J, Kim YG, Chandrasegaran S. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases., 2001, 21(1): 289–297.
[24] Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors., 2009, 326(5959): 1501.
[25] Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, Kay S, Lahaye T, Nickstadt A, Bonas U. Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors., 2009, 326(5959): 1509–1512.
[26] Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove AJ, Voytas DF. Targeting DNA double-strand breaks with TAL effector nucleases., 2010, 186(2): 757–761.
[27] Li J, Zhang Y, Chen KL, Shan QW, Wang YP, Liang Z, Gao CX. CRISPR/Cas: A novel way of RNA-guided genome editing., 2013, 35(11): 1265– 1273.李君, 张毅, 陈坤玲, 单奇伟, 王延鹏, 梁振, 高彩霞. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术. 遗传, 2013, 35(11): 1265–1273.
[28] Huang H, Wu Q. CRISPR double cutting through the labyrinthine architecture of 3D genomes., 2016, 43(5): 273–288.
[29] Li JH, Shou J, Guo Y, Tang YX, Wu YH, Jia ZL, Zhai YA, Chen ZF, Xu Q, Wu Q. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9., 2015, 7(4): 284–298.
[30] Li JH, Shou J, Wu Q. DNA fragment editing of genomes by CRISPR/Cas9., 2015, 37(10): 992– 1002.李金环, 寿佳, 吴强. CRISPR/Cas9系统在基因组DNA片段编辑中的应用. 遗传, 2015, 37(10): 992–1002.
[31] Wang LY, Huang HY, Wu Q. The diversity of DNA fragment editing by CRISPR/Cas9 in highly homologous or repetitive sequences., 2017, 39(4): 313–325.汪乐洋, 黄海燕, 吴强. 利用CRISPR/Cas9对基因组中高度同源DNA片段编辑多样性的遗传学研究. 遗传, 2017, 39(4): 313–325.
[32] Long CZ. God does not play dice, and neither does CRISPR/Cas9., 2019, 6(3): 393–393.
[33] Shou J, Li J, Liu Y, Wu Q. Precise and predictable CRISPR chromosomal rearrangements reveal principles of Cas9-mediated nucleotide insertion., 2018, 71(4): 498–509.e4.
[34] McVey M, Lee SE. MMEJ repair of double-strand breaks (director's cut): Deleted sequences and alternative endings., 2008, 24(11): 529–538.
[35] Allen F, Crepaldi L, Alsinet C, Strong AJ, Kleshchevnikov V, De Angeli P, Páleníková P, Khodak A, Kiselev V, Kosicki M, Bassett AR, Harding H, Galanty Y, Muñoz- Martínez F, Metzakopian E, Jackson SP, Parts L. Predicting the mutations generated by repair of Cas9-induced double- strand breaks., 2018, 37(1): 64–72
[36] Shen MW, Arbab M, Hsu JY, Worstell D, Culbertson SJ, Krabbe O, Cassa CA, Liu DR, Gifford DK, Sherwood RI. Predictable and precise template-free CRISPR editing of pathogenic variants., 2018, 563(7733): 646–651.
[37] Shi X, Shou J, Mehryar MM, Li JW, Wang LY, Zhang M, Huang HY, Sun XF, Wu Q. Cas9 has no exonuclease activity resulting in staggered cleavage with overhangs and predictable di- and tri-nucleotide CRISPR insertions without template donor., 2019, 5: 53.
[38] Dowen JM, Fan ZP, Hnisz D, Ren G, Abraham BJ, Zhang LN, Weintraub AS, Schujiers J, Lee TI, Zhao K, Young RA. Control of cell identity genes occurs in insulated neighborhoods in mammalian chromosomes., 2014, 159(2): 374–387.
[39] Despang A, Schöpflin R, Franke M, Ali S, Jerković I, Paliou C, Chan WL, Timmermann B, Wittler L, Vingron M, Mundlos S, Ibrahim DM. Functional dissection of thelocus identifies nonessential and instructive roles of TAD architecture., 2019, 51(8): 1263–1271.
[40] Kragesteen BK, Spielmann M, Paliou C, Heinrich V, Schöpflin R, Esposito A, Annunziatella C, Bianco S, Chiariello AM, Jerković I, Harabula I, Guckelberger P, Pechstein M, Wittler L, Chan WL, Franke M, Lupiáñez DG, Kraft K, Timmermann B, Vingron M, Visel A, Nicodemi M, Mundlos S, Andrey G. Dynamic 3D chromatin architecture contributes to enhancer specificity and limb morphogenesis., 2018, 50(10): 1463–1473.
[41] Sima J, Chakraborty A, Dileep V, Michalski M, Klein KN, Holcomb NP, Turner JL, Paulsen MT, Rivera-Mulia JC, Trevilla-Garcia C, Bartlett DA, Zhao PA, Washburn BK, Nora EP, Kraft K, Mundlos S, Bruneau BG, Ljungman M, Fraser P, Ay F, Gilbert DM. Identifying cis elements for spatiotemporal control of mammalian DNA replication., 2019, 176(4): 816–830.e18.
[42] Zuo E, Huo X, Yao X, Hu X, Sun Y, Yin J, He B, Wang X, Shi L, Ping J, Wei Y, Ying W, Wei W, Liu W, Tang C, Li Y, Hu J, Yang H. CRISPR/Cas9-mediated targeted chromosome elimination., 2017, 18(1): 224.
[43] Shao Y, Lu N, Wu Z, Cai C, Wang S, Zhang LL, Zhou F, Xiao S, Liu L, Zeng X, Zheng H, Yang C, Zhao Z, Zhao G, Zhou JQ, Xue X, Qin Z. Creating a functional single- chromosome yeast., 2018, 560(7718): 331–335.
[44] Chen B, Gilbert LA, Cimini BA, Schnitzbauer J, Zhang W, Li GW, Park J, Blackburn EH, Weissman JS, Qi LS, Huang B. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system., 2013, 155(7): 1479–1491.
[45] Deng W, Shi X, Tjian R, Lionnet T, Singer RH. Casfish: CRISPR/Cas9-mediated in situ labeling of genomic loci in fixed cells., 2015, 112(38): 11870–11875.
[46] Ma H, Naseri A, Reyes-Gutierrez P, Wolfe SA, Zhang S, Pederson T. Multicolor CRISPR labeling of chromosomal loci in human cells., 2015, 112(10): 3002–3007.
[47] Chen B, Hu J, Almeida R, Liu H, Balakrishnan S, Covill-Cooke C, Lim WA, Huang B. Expanding the CRISPR imaging toolset withCas9 for simultaneous imaging of multiple genomic loci., 2016, 44(8): e75.
[48] Fu Y, Rocha PP, Luo VM, Raviram R, Deng Y, Mazzoni EO, Skok JA. CRISPR-dCas9 and sgRNA scaffolds enable dual-colour live imaging of satellite sequences and repeat- enriched individual loci., 2016, 7: 11707.
[49] Maass PG, Barutcu AR, Weiner CL, Rinn JL. Inter- chromosomal contact properties in live-cell imaging and in Hi-C., 2018, 69(6): 1039–1045.e3.
[50] Ma H, Tu LC, Naseri A, Huisman M, Zhang S, Grunwald D, Pederson T. Multiplexed labeling of genomic loci with dCas9 and engineered sgRNAs using CRISPRainbow., 2016, 34(5): 528–530.
[51] Shao S, Zhang W, Hu H, Xue B, Qin J, Sun C, Sun Y, Wei W, Sun Y. Long-term dual-color tracking of genomic loci by modified sgRNAs of the CRISPR/Cas9 system., 2016, 44(9): e86.
[52] Qin P, Parlak M, Kuscu C, Bandaria J, Mir M, Szlachta K, Singh R, Darzacq X, Yildiz A, Adli M. Live cell imaging of low- and non-repetitive chromosome loci using CRISPR-Cas9., 2017, 8: 14725.
[53] Finlan LE, Sproul D, Thomson I, Boyle S, Kerr E, Perry P, Ylstra B, Chubb JR, Bickmore WA. Recruitment to the nuclear periphery can alter expression of genes in human cells., 2008, 4(3): e1000039.
[54] Kumaran RI, Spector DL. A genetic locus targeted to the nuclear periphery in living cells maintains its transcriptional competence., 2008, 180(1): 51–65.
[55] Wang H, Xu X, Nguyen CM, Liu Y, Gao Y, Lin X, Daley T, Kipniss NH, La Russa M, Qi LS. CRISPR-mediated programmable 3D genome positioning and nuclear organization., 2018, 175(5): 1405–1417.e14.
[56] Morgan SL, Mariano NC, Bermudez A, Arruda NL, Wu FT, Luo YH, Shankar G, Jia L, Chen HL, Hu JF, Hoffman AR, Huang CC, Pitteri SJ, Wang KC. Manipulation of nuclear architecture through CRISPR-mediated chromosomal looping., 2017, 8: 15993.
[57] Cheng AW, Wang HY, Yang H, Shi YL, Katz Y, Theunissen TW, Rangarajan S, Shivalila CS, Dadon DB, Jaenisch R. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system., 2013, 23(10): 1163–1171.
[58] Black JB, Adler AF, Wang HG, D'Ippolito AM, Hutchinson HA, Reddy TE, Pitt GS, Leong KW, Gersbach CA. Targeted epigenetic remodeling of endogenous loci by CRISPR/ Cas9-based transcriptional activators directly converts fibroblasts to neuronal cells., 2016, 19(3): 406–414.
[59] Himeda CL, Jones TI, Jones PL. CRISPR/dCas9-mediated transcriptional inhibition ameliorates the epigenetic dysregulation at D4Z4 and repressesin FSH muscular dystrophy., 2016, 24(3): 527–535.
[60] Liu XS, Wu H, Ji X, Stelzer Y, Wu X, Czauderna S, Shu J, Dadon D, Young RA, Jaenisch R. Editing DNA methylation in the mammalian genome., 2016, 167(1): 233– 247.e17.
[61] Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage., 2016, 533(7603): 420–424.
[62] Liao HK, Hatanaka F, Araoka T, Reddy P, Wu MZ, Sui Y, Yamauchi T, Sakurai M, O'Keefe DD, Núñez-Delicado E, Guillen P, Campistol JM, Wu CJ, Lu LF, Esteban CR, Izpisua Belmonte JC.target gene activation via CRISPR/Cas9-mediated trans-epigenetic modulation., 2017, 171(7): 1495–1507.e15.
[63] Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA- guided platform for sequence-specific control of gene expression., 2013, 152(5): 1173–1183.
[64] Gilbert LA, Larson MH, Morsut L, Liu Z, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR- mediated modular RNA-guided regulation of transcription in eukaryotes., 2013, 154(2): 442–451.
[65] Gao Y, Xiong X, Wong S, Charles EJ, Lim WA, Qi LS. Complex transcriptional modulation with orthogonal and inducible dCas9 regulators., 2016, 13(12): 1043–1049.
[66] Zuo E, Sun YD, Wei W, Yuan TL, Ying WQ, Sun H, Yuan LY, Steinmetz LM, Li YX, Yang H. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos., 2019, 364(6437): 289–292.
[67] Jin S, Zong Y, Gao Q, Zhu ZX, Wang YP, Qin P, Liang CZ, Wang DW, Qiu JL, Zhang F, Gao CX. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice., 2019, 364(6437): 292–295.
[68] Hilton IB, D'Ippolito AM, Vockley CM, Thakore PI, Crawford GE, Reddy TE, Gersbach CA. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers., 2015, 33(5): 510–517.
[69] Kearns NA, Pham H, Tabak B, Genga RM, Silverstein NJ, Garber M, Maehr R. Functional annotation of native enhancers with a Cas9-histone demethylase fusion., 2015, 12(5): 401–403.
[70] Wang H, Guo R, Du Z, Bai L, Li L, Cui J, Li W, Hoffman AR, Hu JF. Epigenetic targeting ofin hepatoma cells by synthetic CRISPR dCas9 epi-suppressors., 2018, 11: 23–33.
[71] Wang G, Chow R D, Bai Z, Zhu L, Errami Y, Dai X, Dong M B, Ye L, Zhang X, Renauer P A, Park J J, Shen L, Ye H, Fuchs C S, Chen S. Multiplexed activation of endogenous genes by CRISPRa elicits potent antitumor immunity., 2019, 20: 1494–1505.
[72] Crane E, Bian Q, McCord RP, Lajoie BR, Wheeler BS, Ralston EJ, Uzawa S, Dekker J, Meyer BJ. Condensin- driven remodeling of x-chromosome topology during dosage compensation., 2015, 523(7559): 240–244.
[73] Gasperini M, Hill AJ, McFaline-Figueroa JL, Martin B, Kim S, Zhang MD, Jackson D, Leith A, Schreiber J, Noble WS, Trapnell C, Ahituv N, Shendure J. A genome-wide framework for mapping gene regulation via cellular genetic screens., 2019, 176(1–2): 377–390.e19.
[74] de Wit E, Vos ES, Holwerda SJ, Valdes-Quezada C, Verstegen MJ, Teunissen H, Splinter E, Wijchers PJ, Krijger PH, de Laat W. CTCF binding polarity determines chromatin looping., 2015, 60(4): 676–684.
[75] Sanborn AL, Rao SS, Huang SC, Durand NC, Huntley MH, Jewett AI, Bochkov ID, Chinnappan D, Cutkosky A, Li J, Geeting KP, Gnirke A, Melnikov A, McKenna D, Stamenova EK, Lander ES, Aiden EL. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes., 2015, 112(47): E6456–6465.
[76] Zheng XF, Huang HY, Wu Q. Chromatin architectural protein CTCF regulates gene expression of the UGT1 cluster., 2019, 41(6): 509–523.郑晓飞, 黄海燕, 吴强. 染色质架构蛋白CTCF调控UGT1基因簇的表达. 遗传, 2019, 41(6): 509–523.
[77] Wu Q, Guo Y, Lu YJ, Li JW, Wu YH, Jia ZL. Tandem directional CTCF sites balance protocadherin promoter usage., 2019, 10.1101/525543
[78] Lupiáñez D G, Kraft K, Heinrich V, Krawitz P, Brancati F, Klopocki E, Horn D, Kayserili H, Opitz JM, Laxova R, Santos-Simarro F, Gilbert-Dussardier B, Wittler L, Borschiwer M, Haas SA, Osterwalder M, Franke M, Timmermann B, Hecht J, Spielmann M, Visel A, Mundlos S. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions., 2015, 161(5): 1012–1025.
[79] Hnisz D, Weintraub AS, Day DS, Valton AL, Bak RO, Li CH, Goldmann J, Lajoie BR, Fan ZP, Sigova AA, Reddy J, Borges-Rivera D, Lee TI, Jaenisch R, Porteus MH, Dekker J, Young RA. Activation of proto-oncogenes by disruption of chromosome neighborhoods., 2016, 351(6280): 1454–1458.
[80] Guo Y, Monahan K, Wu H, Gertz J, Varley KE, Li W, Myers RM, Maniatis T, Wu Q. CTCF/cohesin-mediated DNA looping is required for protocadherin α promoter choice., 2012, 109(51): 21081– 21086.
[81] Zhai YN, Xu Q, Guo Y, Wu Q. Characterization of a cluster of CTCF-binding sites in a protocadherin regulatory region., 2016, 38(4): 323–336.翟亚男, 许泉, 郭亚, 吴强. 原钙粘蛋白基因簇调控区域中成簇的CTCF结合位点分析. 遗传, 2016, 38(4): 323–336.
[82] Wu Q, Maniatis T. A striking organization of a large family of human neural cadherin-like cell adhesion genes., 1999, 97(6): 779–790.
[83] Guo Y, Wu Q. Inversion of CTCF binding sites by DNA fragment editing alters genome topology and enhancer/ promoter functions., 2015, 37(10): 1073–1074.郭亚, 吴强. 采用DNA片段编辑技术反转CTCF结合位点改变基因组拓扑结构和增强子与启动子功能. 遗传, 2015, 37(10): 1073–1074.
[84] Lu YJ, Shou J, Jia ZL, Wu YH, Li JH, Guo Y, Wu Q. Genetic evidence for asymmetric blocking of higher-order chromatin structure by CTCF/cohesin., 2019, 1–7.
[85] Kleinstiver BP, Sousa AA, Walton RT, Tak YE, Hsu JY, Clement K, Welch MM, Horng JE, Malagon-Lopez J, Scarfò I, Maus MV, Pinello L, Aryee MJ, Joung JK. Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene, epigenetic and base editing., 2019, 37(3): 276–282.
[86] Liu L, Li X, Ma J, Li Z, You L, Wang J, Wang M, Zhang X, Wang Y. The molecular architecture for RNA-guided RNA cleavage by Cas13a., 2017, 170(4): 714–726.e10.
[87] Ghavi-Helm Y, Jankowski A, Meiers S, Viales RR, Korbel JO, Furlong EEM. Highly rearranged chromosomes reveal uncoupling between genome topology and gene expression., 2019, 51(8): 1272–1282.
Probing 3D genome by CRISPR/Cas9
Peifeng Liu, Qiang Wu
CRISPR/Cas9 system has significant advantages in gene editing strategy, offering cost-effective and efficient means to modify and edit the genomes of animals, plants, and microorganisms. Three-dimensional (3D) genome is an emerging and interdisciplinary field catapulted by combined technological breakthroughs of chromosome conformation capture with next-generation sequencing and live imaging with super-resolution microscopy. An important aspect of 3D genomics is to model structural variations and label specific genomic fragments to investigate the effects of manipulation of genomic elements on gene expression regulation, cell development and differentiation, and spatial location of chromosomal regions. Therefore, CRISPR/Cas9 system and its derivative technologies of DNA-fragment editing are excellent toolboxes for investigating dynamics and functions of the higher-order chromatin organization and three-dimensional genome structure. In this review, we describe the opportunities and challenges of CRISPR as well as its derivative technologies in 3D genome research, thereby providing some critical references and future research directions in the field.
CRISPR/Cas9 system; 3D genome; DNA fragment editing; chromosome rearrangement; Cas9 endonuclease cleavage mechanism
2019-08-22;
2019-11-18
国家自然科学基金项目(编号:31630039,91640118),国家重点研发计划课题项目(编号:2017YFA0504203,2018YFC1004504)和上海市科学技术委员会项目(编号:19JC1412500)资助[Supported by the National Natural Science Foundation of China (Nos. 31630039, 91640118), the National Key Research and Development Program of China (Nos. 2017YFA0504203, 2018YFC1004504), and Science and Technology Commission of Shanghai Municipality program (No. 19JC1412500)]
刘沛峰,博士研究生,专业方向:生物学。E-mail: lpfmail@foxmail.com
吴强,博士,教授,研究方向:分子遗传学。E-mail: qwu123@gmail.com
10.16288/j.yczz.19-246
2019/12/19 14:29:18
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20191218.1615.001.html
(责任编委: 张飞雄)
