基于matK序列将中药石斛鉴定到属
2020-02-18黄周英桑庆亮陈怀宇
黄周英 桑庆亮 陈怀宇

摘 要:以兰科的石斛属、石仙桃属、石豆兰属等14属matK全长CDS为研究对象,通过比较属内、属间序列两两p-距离,得到属内p-距离上限和属间p-距离下限,并以石斛属、石豆兰属和石仙桃属3个近缘属的matK部分CDS为待检序列进行实地鉴定.结果表明,基于matK可清楚地将石斛属、石仙桃属、石豆兰属序列鉴定到其对映的属,表明基于matK序列可将中药石斛鉴定到属.
关键词:中药石斛;matK;鉴定
中图分类号:S567.239 文献标识码:A 文章编号:1673-260X(2020)01-0052-04
石斛属在分类学上属于兰科树兰族石斛亚族,我国共有74种2变种[1].我国石斛属植物主要分布在秦岭——淮河以南各省区,其中以云南省石斛种类为最多[2].2015年版《中华人民共和国药典》[3]收载的中药石斛为金钗石斛、鼓槌石斛和流苏石斛的栽培品及其同属植物近似种新鲜或干燥茎,其干燥品具益胃生津,滋阴清热的功效,同时提及“及其同属植物近似种”,即表示原植物仍未明确之意.
实际上,各地栽培和开发的中药石斛多达近40种[4,5],但究竟哪种石斛属物种为中药石斛的正源尚无定论[6,7];中药石斛中常混有金石斛属、石仙桃属、石豆兰属某些物种[4],这给中药石斛在医学上的应用带来了很大的问题,因此,建立一套行之有效的鉴定方法是解决这些问题的重要一环.
matK基因是位于叶绿体trnK基因第1号内含子中的成熟酶基因,参与II类内含子的剪接,是已知进化速度最快的叶绿体编码基因,一般认为matK可以作为单独使用的植物条形码[8,9],且因此得到CBOL的Plant Working Group推荐作为植物的条形码[10].事实上,单独使用matK仍无法完全解决问题,尤其是对于复杂的类群需有针对性地将matK与rbcL组合才可达到目的[10],也就是说,仍没有一种可以通用的植物条形码序列.
研究表明,matK能识别90%的兰科植物[11],在将matK应用于石斛属鉴定的实践中,刘静等[12]、腾艳芬等[13]、Srikulnath等[14]、钟志敏[15]的研究均认为matK至少在不同程度上可以用于石斛属物种的分子鉴定,而黄海等[16]、Xu等[17]认为matK序列并不适于作为石斛属DNA条形码鉴定.为此,十分有必要对matK在石斛属,乃至兰科植物鉴定中的作用进行全面的评估,为建立完整的石斛属植物鉴定方法提供依据.
本文以叶绿体基因matK全长CDS为依据,比较分析了石斛属、石仙桃属、石豆兰属等14个属的属内、属间p-距离差异,并选取石斛属、石仙桃属、石豆兰属matK部分CDS进行鉴定,以依据matK序列将物种鉴定到属.
1 材料与方法
1.1 材料
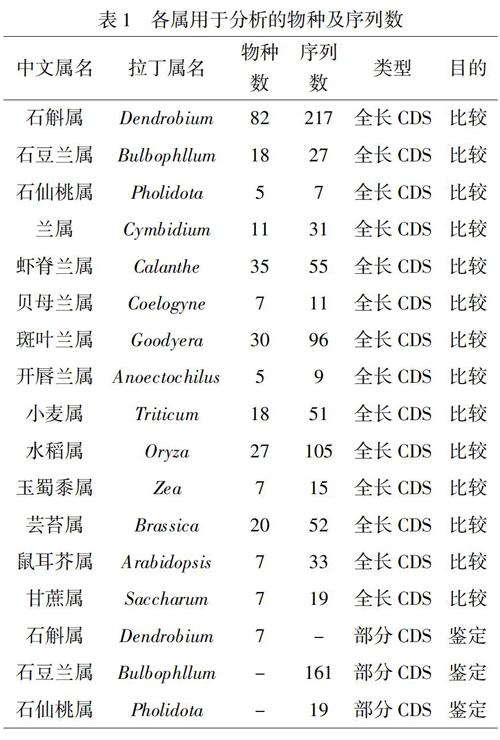
从GenBank数据库提取石斛属、石豆兰属、石仙桃属、贝母兰属、虾脊兰属、斑叶兰属、兰属、开唇兰属共8个兰科属,与水稻属、玉蜀黍属、小麦属、甘蔗属共个禾本科属以及芸苔属、鼠耳芥属共2个十字花科属的matK全长CDS及石仙桃属、开唇兰属、石豆兰属长度大于1100bp的matK序列(表1).
1.2 方法
1.2.1 序列获取
使用Biopython[18]的Entrez模块调用NCBI的Entrez检索系统检索并获取所需matK序列,对数据文件解析后提取其序列及相关信息.
1.2.2 序列比对、p-距离计算及绘图
采用muscle[19]进行多序列比对,采用megacc[20]计算p-距离(p-distance)[21],提取p-距离数据后使用python环境下的matplotlib模块和seaborn模块绘图.
1.2.3 统计检验
数据组间的差异采用Wilcoxon检验[22],在Python环境下使用scipy.stats模块实现.
2 结果与分析
2.1 属内、属间p-距离比较
2.1.1 各属属内p-距离比较
将供试14个属matK序列属内p-距离绘成箱形图(图1),结果表明,不同属matK序列属内p-距离变化范围差异较大,大部分小于0.04,个别属内差异较大,如开唇兰属(属内p-距离可达0.08).因此,属内p-距离上限需根据各属的具体情况而定,对于石斛属来说,该值可定为0.02,而对于开唇兰属来说,则可定为0.04,即当某未知序列与石斛属matK序列的p-距离小于0.02,即可认为该序列来自石斛属物种.
2.1.2 石斛属与各属间两两p-距离比较
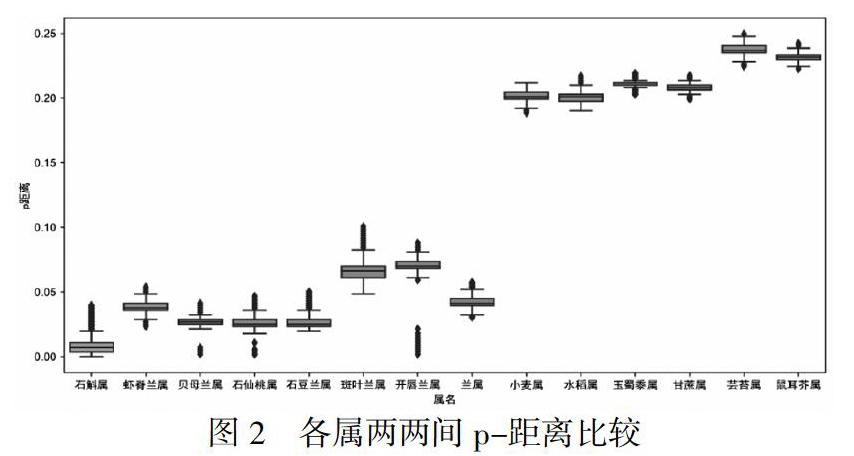
将石斛属matK序列分别与供试14个属的matK序列进行属间两两序列p-距离计算,结果绘成箱形图(图2),可以看出,按照与石斛属间p-距离由小到大排列为石斛属(属内)、虾脊兰属、贝母兰属、石仙桃属、石豆兰属、兰属、斑叶兰属、开唇兰属、小麦属、水稻属、玉蜀黍属、甘蔗属、芸苔属和鼠耳芥属.石斛属与同科其他属间p-距离数值均小于0.1,而与禾本科4个属之间的属间p-距离为0.2~0.25之间,表明亲缘关系越远,matK序列差异越大.
但石斛属matK序列与同科的虾脊兰属、贝母兰属、石仙桃属和石豆兰属差异较小,p-距离数值小于0.05且与石斛属内p-距离相比差异不显著(P>0.05),因此难以准确鉴别;开唇兰属、斑叶兰属及兰属与石斛属间的p-距离则与石斛属内p-距离相比有显著差异(P<0).兰科与禾本科之间0.1-0.2之间为科之间的gap区,说明matK序列可用于区分不同科.
2.1.3 近缘属两两之间p-距离的比较
为进一步弄清这些近缘属间是否可基于matK这一单一序列可区分,将石斛属与其他各属进行两两作图(图3),可以明显看出,石斛属与贝母兰属、石仙桃属、石豆兰属、虾脊兰属、开唇兰属、斑叶兰属、兰属及不同科的鼠耳芥屬均可以借助matK序列明确可分(P<0.01),属内与属间p-距离的交叠区很小.因此,可基于matK序列鉴定到属.
其中,A-H分别为石斛属与虾脊兰、贝母兰属、兰属、石仙桃属、石豆兰属、斑叶兰属、开唇兰属及鼠耳芥属两两之间属内、属间的p-距离比较;Wilcoxon检验结果标记在两属箱图上方,“*”代表显著差异(p<0.05),“**”代表极显著差异(p<0.01).
2.2 matK序列归属鉴定
以石斛属、石仙桃属、石豆兰属的matK序列序列作为待检序列,分别与上述14个属两两组合,比对并计算两两属间p-距离,依据以下标准判断等检序列的归属:
(1)在所分析属中值最小;(2)低于该属内上限.
2.2.1 石斛属matK部分CDS的鉴定结果
将获取的石斛属matK序列作为待检序列分别与各属matK全长CDS组合后比对并计算p-距离,石斛属待检序列与其余各属间的p-距离分布情况见图4,很明显兰科以外的属与待检序列的p-距离为0.2~0.3,远高于属内p-距离上限,同为兰科的属中,兰属、虾脊兰属、开唇兰属和斑叶兰属与待检序列的p-距离为0.1上下,与石斛属之间比较有显著差异(P<0.01).因此,待检序列不能鉴定为来自这10个属的物种.
余下的4个属均为兰科石斛属的近缘属,即石斛属、石仙桃属、石豆兰属、贝母兰属,与待检序列间p-距离无显著差异(P值均大于0.05),因此,通过Wilcoxon检验不能找出唯一的结果.但是,比较后发现,待检序列与石斛属间的p-距离最小,石仙桃属次之,然后是贝母兰属和石豆兰属,可据此将待检序列定为石斛属.
A-H分别为石斛属matK待检序列与石仙桃属、石斛属、石豆兰属、开唇兰属、兰属、贝母兰属、斑叶兰属及虾脊兰属两两属间p-距离比较结果.
2.2.2 石仙桃属鉴定结果
以石仙桃属matK部分CDS作为待检序列,分别与各属matK全长CDS两两组合,比对并计算p-距离并绘制箱形图,由图6可见,非兰科的属可很容易地排除在外,同属兰科的斑叶兰属和开唇兰属也有明显gap,石斛属、石豆兰属、兰属和虾脊兰属应也有显著差异(P<0.01),石仙桃属和贝母兰属则明显差异较小,且有明显交叠区(图7F),难以区分(P>0.05).
A-H分别为石仙桃属matK待检序列与石仙桃属、石斛属、石豆兰属、开唇兰属、兰属、贝母兰属、斑叶兰属及虾脊兰属两两属间p-距离比较结果.
2.2.3 石豆兰属鉴定结果
以石豆兰属matK部分CDS作为待检序列,分别与各属matK全长CDS两两组合,比对并计算p-距离并绘制箱形图,由图8可知,禾本科和十字花科的属与兰科各属间具很大的gap,同属兰科的斑叶兰属、虾脊兰属和开唇兰属与待检序列间的p-距离显著高于石豆兰属(P<0.05),石斛属、石豆兰属、石仙桃属及贝母兰属互有叠区(图9A、B、C),难以区分(P值均大于0.05).
A-H分别为石豆兰属matK待检序列与石仙桃属、石斛属、石豆兰属、开唇兰属、兰属、贝母兰属、斑叶兰属及虾脊兰属两两属间p-距离比较结果.
3 讨论
3.1 matK序列可将植物鉴定至属
本研究通过对来自兰科、禾本科、十字花科的14个属的比较分析,明确了属内p-距离上限为0.1,属间p-距离下限为0.3.以此gap为基础,建立将matK序列鉴定到属的方法,即将待检序列与目标属的所有matK全长CDS进行两两p-距离计算,从中选择p-距离小于0.1且排在各属最末尾(中位数最小)的属定为该matK序列的物种来源.
然后,以鉴定的属分别对其余各属进行组间Wilcoxon检验,以确定鉴定结果在95%或99%的置信区间内可信,若P>0.05,则可能有多种可能,即结果不唯一.
采用该方法,将石斛属、石仙桃属、石豆兰属物种的待检序列(石仙桃属、石豆兰属与之前建立方法所有序列有部分重叠,其余均不同,且待检对象均为matK序列序列).待检序列均可成功鉴定至所在属.
3.2 中药石斛鉴定到属的意义
据考证[6,23-25],中药石斛中常混有来处近缘属,如石豆兰属、石仙桃属、金石斛属等的物种,而且即使来自石斛属的植物,也有多达40种左右被认作中药石斛的原植物[4],且栽培中经常是多种混种混收[15],因此混用石斛属植物作为中药石斛成为目前的常态.相比于同属的近缘种,确定待检样品是否属于石斛属对中药石斛道地性的鉴别的第一步,因此,我们认为应首先将中药石斛鉴定到属.
为此,采用matK通过计算比较p-距离的方式,更为精细的鉴别,如种及种以下分类阶的鉴别,有赖于对中药石斛原植物的明确的结论,以及较为细致的序列分析和更为有效的分子序列片段的发掘.
3.3 影响分子鉴定的因素
首先,传统分类中仍然存在很多问题,对很多物种的归属仍存在争议,且中药石斛原植物不明确,这使得分子鉴定缺乏可靠的参照;其次,中药石斛干品难以获取理想的DNA模板,造成序列扩增困难;第三,序列数据库中的序列数量多少不一、质量参差不齐,造成分子鉴定难以获得理想的结果;最后,问题序列的存在有可能造成错误的结论.
参考文献:
〔1〕中国科学院中国植物志编辑委员会.中国植物志[M].北京:科学出版社,1999.
〔2〕白音.药用三角鉴定方法的系统研究[D].北京:北京中医药大学,2007.
〔3〕国家药典委员会.中华人民共和国药典:2015年版[M].北京:中国医药科技出版社,2015.
〔4〕李满飞,徐国钧,徐珞珊,等.商品石斛的调查与鉴定(ii)[J].中草药,1991,22(4):173-178.
〔5〕馬国祥,徐国钧,徐珞珊,等.商品三角的调查与鉴定(iii)[J].中草药,1995,26(7):370-373.
〔6〕金效华,黄璐琦.40[J].中国中药杂志,2015,40(13):247-251.
〔7〕顺庆生,徐一帆,魏刚,等.中药三斛正本清源之霍山石斛[J].广东药科大学学报,2019(1):1-4.
〔8〕HILU K. The matk gene: Sequence variation and application in plant systematics [J]. American Journal of Botany, 1997, 84 (6): 830-839.
〔9〕LAHAYE R, BANK M V D, BOGARIN D, et al. DNA barcoding the floras of biodiversity hotspots[J]. Proceeding of the National Academy of Sciences of the United States of America, 2008, 105 (8): 2923-2928.
〔10〕CBOL-PLANT-WORKING-GROUP. A DNA barcode for land plants[J]. Proceeding of the National Academy of Sciences of the United States of America, 2009, 106 (31): 12794-12797.
〔11〕KRESS W J, ERICKSON D L. A two-locus global DNA barcode for land plants: The coding rbcl gene complements the non-coding trnh-psba spacer region[J]. PLoS One, 2007, 2 (6): e508.
〔12〕刘静,何涛,淳泽.药用石斛的叶绿体matk基因序列分析及鉴别[J].药学学报,2009,44(9):1051-1055.
〔13〕腾艳芬,吴晓俊,徐红,等.石斛及其常见混淆品的matk基因序列比较[J].中国药科大学学报,2010,33(4):280-283.
〔14〕SRIKULNATH K, SAWASDICHAI S. Phylogenetic relationship of dendrobium species in thailand inferred from chloroplast matk gene and nuclear rdna its region[J]. The Horticulture Journal, 2015, 84 (3): 243-252.
〔15〕钟志敏.石斛dna条形码鉴定及系统分类研究[D].广州:广州中医药大学,2018.
〔16〕黄海,李劲松,符岸军,等.石斛属植物dna条形码序列的筛选[J].热带作物学报,2011,31(10):1769-1777.
〔17〕XU S, LI D, XIANG X, et al. Evaluation of DNA barcodes in dendrobium (orchidaceae) from mainland asia[J]. PLoS One, 2015, 10 (1): e0115168.
〔18〕COCK P J A, ANTAO T, CHANG J T, et al. Biopython: Freely available python tools for computational molecular biology and bioinformatics[J]. Bioinformatics, 2009, 25 (11): 1422-1423.
〔19〕EDGAR R C. Muscle: Multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32 (5): 1792-1797.
〔20〕KUMAR S, STECHER G, PETERSON D, et al. Mega-cc: Computing core of molecular evolutionary genetics analysis program for automated and iterative data analysis[J]. Bioinformatics, 2012, 28 (20): 2685-2686.
〔21〕NEI M, KUMAR S. Molecular evolution and phylogenetics[M]. New York: Oxford University Press, 2000.
〔22〕吳喜之,赵博娟.非参数统计[M].北京:中国统计出版社,2011.
〔23〕沙文兰,罗金裕.中药石斛鉴定研究——i.石斛原植物和药用调查[J].药学学报,1980,5(6):351-358,385-386.
〔24〕郑博仁.云南石斛属药材现状及其原植物[J].中国中药杂志,1990,1990(15):9-14.
〔25〕腾建北,万德光,王孝勋.石斛名实及功效的本草考证[J].中药材,2013,36(11):1876-1880.
