裂腹鱼亚科鱼类Hif-α基因的分子进化及低氧诱导表达
2019-09-03吴蓉蓉晁燕赵永丽陈祺昌郑志琴夏明哲祁得林
吴蓉蓉,晁燕,赵永丽,陈祺昌,郑志琴,,夏明哲,祁得林*
(1.青海大学省部共建三江源生态与高原农牧业国家重点实验室,西宁 810016;2. 青海大学生态环境工程学院,西宁 810016;3. 青海大学农牧学院动物科学系,西宁 810016)
裂腹鱼亚科(Schizothoracinae)鱼类属于鲤形目(Cypriniforme),鲤科(Cyprinidae),是青藏高原水生鱼类区系中主要类群之一,广泛分布于上至海拔5000~6000 m雪线附近和下至海抜1000 m左右的高原边缘的湖泊河流[1],包含11~12属共100多种[2],根据形态特征将裂腹鱼分为3个类群,即原始、特化和高度特化类群[3],研究表明三个类群代表了青藏高原持续隆起过程的特定阶段[4-5]。青藏高原具有高寒、低氧、强紫外线辐射等特征[6],高原水体溶解氧波动较大(4.0~7.4 mg/L)[5],这种极端的水体环境对土著鱼类的分布、生存适应及进化模式等产生了深刻的影响,同时赋予了裂腹鱼亚科鱼类抗缺氧、耐高寒的优良生物学特性。目前,对裂腹鱼类的研究多集中在形态分类及生物学特征等方面[7],而在低氧适应方面的研究仍有欠缺。
氧是机体进行新陈代谢和维持生存的必要因素[8],环境缺氧将严重影响鱼类的生存、生长和繁殖[9-10]。鱼类在长期进化过程中具有独特的适应机制,如通过低氧诱导因子(hypoxia inducible factors,HIFs)信号途径调节相关基因的转录进行低氧应答,增强低氧时的耐受性[11-12]。HIFs是一组由低氧调节的α亚基和氧不敏感的β亚基组成的异二聚体转录因子[13],β亚基又称芳香烃受体核转运蛋白。α亚基是HIF唯一的氧调节亚基,即是低氧诱导的,它决定HIF的活性。在脊椎动物中发现HIF-α基因至少存在3种亚型:Hif-1α、Hif-2α和Hif-3α,其皆具有N端保守的bHLH结构域,PAS和PAC结构域,氧敏感的ODD结构域以及C端负责转录激活的TAD结构域。其中HIF-3α仅有一个N-TAD,C-TAD缺失[14]。
2001年,由Soitamo等[15]从虹蹲(Oncorhynchusmykiss)中首先克隆得到Hif-1α,在低氧时Hif-1α表达量明显上升。Rojas等[16]从斑马鱼(Daniorerio)中克隆获得Hif-1α和Hif-2α基因序列,发现与其他脊椎动物Hif基因高度同源。Rytkönen等[17]研究发现硬骨鱼在早期发生的硬骨鱼类特异性全基因组复制中产生了6个Hif-α基因:1 A/B,2 A/B和3 A/B。目前多数硬骨鱼类有一个旁系同源基因发生丢失,但鲤科鱼类中两者都有保留[18]。为了深入研究青藏高原裂腹鱼亚科鱼类对高原低氧水体环境的适应机制,本研究选取裂腹鱼亚科鱼类不同等级13个代表物种,通过RT-PCR获得硬骨鱼类特异性Hif-α基因(Hif-1αΑ/Hif-1αB和Hif-2αΑ/Hif-2αB)序列,并开展分子进化分析。同时,选择广布种—花斑裸鲤,利用qRT-PCR方法检测了低氧胁迫下Hif-α基因在主要组织中的表达调控,为进一步探索Hif-α基因在高原鱼类低氧适应中的作用和调控机制奠定基础。
1 材料与方法
1.1 材料
1.1.1 实验动物
在青藏高原范围内采集13个裂腹鱼亚科鱼类代表物种,见表1。其中,澜沧裂腹鱼(Schizothoraxlantsangensis)、巨须裂腹鱼(Schizothoraxmacropogon)、光唇裂腹鱼(Schizothoraxlissolabiatus)、西藏裂腹鱼(Schizothoraxlabiatus)为原始类群;厚唇裸重唇鱼(Gymnodiptychuspachycheilus)、双须叶须鱼(Ptychobarbusdipogon)为特化类群;高原裸裂尻鱼(Schizopygopsisstoliczkae)、尖裸鲤(Oxygymnocyprisstewartii)、高原裸鲤(Gymnocypriswaddelli)、拉萨裸裂尻鱼(Schizopygopsisyounghusbandi)、花斑裸鲤(Gymnocypriseckloni)、黄河裸裂尻鱼(Schizopygopsispylzovi)、极边扁咽齿鱼(Platypharodonextremus)为高度特化类群。野外采样时,用100 mg/kg的戊巴比妥钠(C11H17N2NaO3)对鱼进行麻醉,分别现场采集13个物种的肌肉、脑和鳃保存于液氮中,用于HIF基因克隆。
花斑裸鲤30尾(体重约210 g)采集于青海省大通县湟水河支流—宝库河,活鱼运送并养于青海大学农牧实验室塑料桶中,经低氧处理后采集组织,检测各基因表达情况。
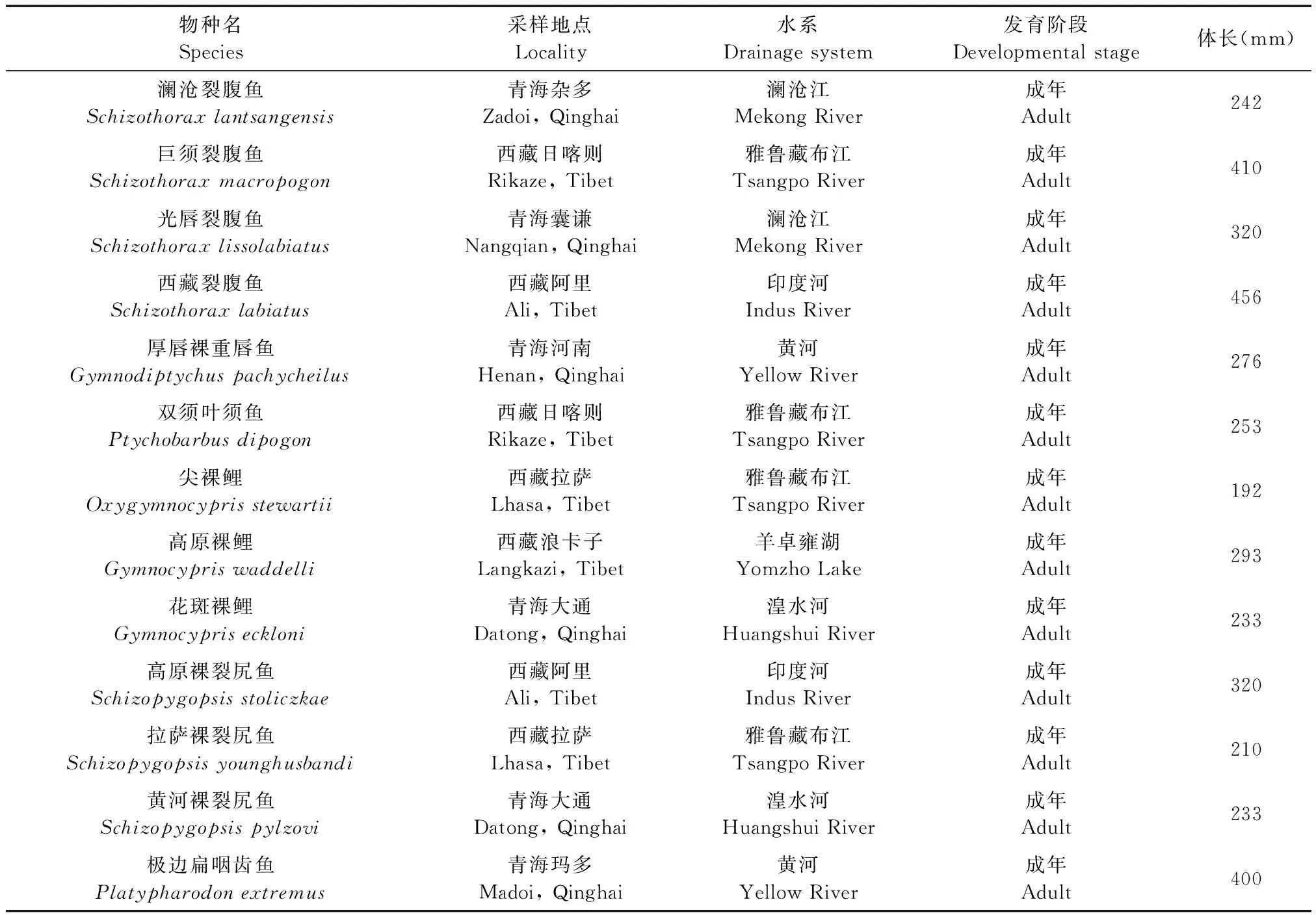
表1 裂腹鱼亚科鱼类采样信息Table 1 Sampling information of the schizothoracinae fishes
1.1.2 实验试剂
戊巴比妥钠(25 g高纯级,容创),RNA Simple Total RNA kit(天根),FastQuant RT Kit(天根),Premix Ex Taq® Version 2.0试剂盒(Takara),TIANgel Midi Purification Kit(天根),pMD19-T载体(天根),SuperReal PreMix Plus(SYBR Green)试剂盒(天根)。
1.1.3 实验仪器
溶解氧测定仪AZ8402(衡欣,台湾),氧泵(赛尔,浙江),高速冷冻离心机(Beckman Inc,美国),梯度PCR扩增仪(Bio-Rad,美国),Gel Doc XR+凝胶成像系统(Bio-Rad,美国),荧光定量PCR仪(Bio-Rad,美国),SX-500 高压灭菌锅(Tomy,日本)。
1.2 方法
1.2.1 总RNA提取及Hif-α基因cDNA序列扩增
分别提取13种裂腹鱼亚科鱼类肌肉、脑和鳃RNA,而后合成13个物种的cDNA第一链。以13个物种组织cDNA为模板,对Hif-1αΑ、Hif-1αB、Hif-2αΑ和Hif-2αB基因进行克隆,引物见表2。PCR扩增程序:94℃预变性3 min;94℃变性30 s,55℃退火30 s,72℃延伸1 min,循环36次;72℃续延伸5 min。PCR扩增产物经1.0%的琼脂糖凝胶电泳后,进行回收纯化,然后连接转化以进行测序。
1.2.2 序列分析
测序完成后,利用Lasergene 7.0软件[19]对测序结果分别进行分析;利用Blastn在线程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行确认并进行核酸比对;利用DNAMAN 6.0(http://www.lynnon.com)分别计算其核苷酸组成,碱基变异情况及氨基酸同源性;利用Smart(http://smart.embl-heidelberg.de/)和NCBI中的CDD数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行结构域分析。

表2 裂腹鱼Hif-α基因RT-PCR引物及qRT-PCR引物Table 2 Primers for RT-PCR and qRT-PCR of Hif-α gene in the schizothoracine fish
1.2.3 选择压力分析
在遗传学中,通过比较其非同义替换率(dN)和同义替换率(dS)之间的比率(ω),判断基因是否有选择压力作用。dN/dS>1,则为正向选择;dN/dS=1,则为中性选择;dN/dS<1,则为纯化选择。基于13个裂腹鱼亚科鱼类的物种进化树,使用Paml的codeml程序进行最大似然法分析,包括位点模型,支模型和支位点混合模型,具体方法见Qi等[20]文献。Bayes Empirical Bayes(BEB)用于计算模型中发现的正选择位点的可能性。
1.2.4 低氧处理
本研究中所涉及裂腹鱼亚科物种多分布在偏远地带,因此选择离实验室较近的裂腹鱼亚科代表种—花斑裸鲤开展低氧实验。活鱼运送至实验室后,暂养于塑料桶中的30尾花斑裸鲤,使水充分曝气将溶解氧(dissolved oxygen,DO)保持在(8.4 ± 0.1)mg/L,水温在10~13℃。预养3 d后,随机采集3尾花斑裸鲤白肌、红肌、心脏、脑、鳃组织作为对照组。24尾作为中度低氧组置于40 L塑料桶,1 h内将溶解氧浓度从(8.4 ± 0.1)mg/L降低至(3.0 ± 0.1)mg/L,并保持96 h,每隔12 h采集3尾各组织。取3尾作为重度低氧组置于另一40 L塑料桶,1 h内将溶解氧浓度降低至(0.3 ± 0.1)mg/L,并保持4 h,采集组织。低氧处理时,塑料桶顶部覆盖塑料膜,水体充氮除氧获得预期溶解氧浓度。维持低氧期间,利用溶解氧测定仪定期监控水体溶解氧浓度,氧含量若降低使用氧泵增氧至目的浓度。
1.2.5 qRT-PCR检测低氧诱导下Hif-α基因的表达
将花斑裸鲤各低氧条件处理下的白肌、红肌、心、脑和鳃组织分别提取总RNA,平衡各起始浓度合成cDNA第一链,然后进行荧光定量PCR分析。反应体系25 μL: 2× SuperReal PreMix Plus 12.5 μL、上下游引物(10 μmol/L)各1 μL、cDNA模板1 μL、ddH2O 9.5 μL。每个样品重复3次,结果采用2-ΔΔCT法分析[21]。qRT-PCR测定时β-actin基因表达量作为内标,引物见表2。由于所测荧光定量原始数据量过多,在本研究中不再以表格形式展示。
2 结果
2.1 13个裂腹鱼亚科鱼类Hif-α基因序列分析
13个物种的Hif-1αΑ、Hif-1αB、Hif-2αΑ和Hif-2αB基因编码区核苷酸序列分别为2196、2325、2544和2511 bp,分别编码731、774、847、836个氨基酸,见表3。13个物种间各HIF-α基因的同源性为93.6%~99.4%、92.7%~99.9%、92.6%~99.8%、93.5%~99.8%,各类群代表物种间同源性大于95%。同一物种的HIF-1αΑ与HIF-1αB的相似性为50%~53%,HIF-2αΑ与HIF-2αB的相似性为54%~58%。裂腹鱼亚科鱼类HIF-α具有bHLH结构域、PAS和PAC结构域、ODD结构域以及TAD结构域。在高度特化等级、特化等级和原始等级类群中分别选取高原裸鲤、厚唇裸重唇鱼和巨须裂腹鱼作为代表种,将三个物种的HIF-α蛋白序列进行多序列比对,结果见图1。HIF-α N端的bHLH-PAS结构域是高度保守的,但ODD区域保守性较低,特别是在氧依赖性脯氨酸羟基化位点附近。在13个物种的HIF-α序列中检测到两个保守的脯氨酸羟基化序列LxxLAP,但13个物种在HIF-1αΑ的NODD中检测到特异性缺失,并且特化及高度特化类群的HIF-1αB的CODD结构域中脯氨酸羟基化序列突变为PxxLAP。
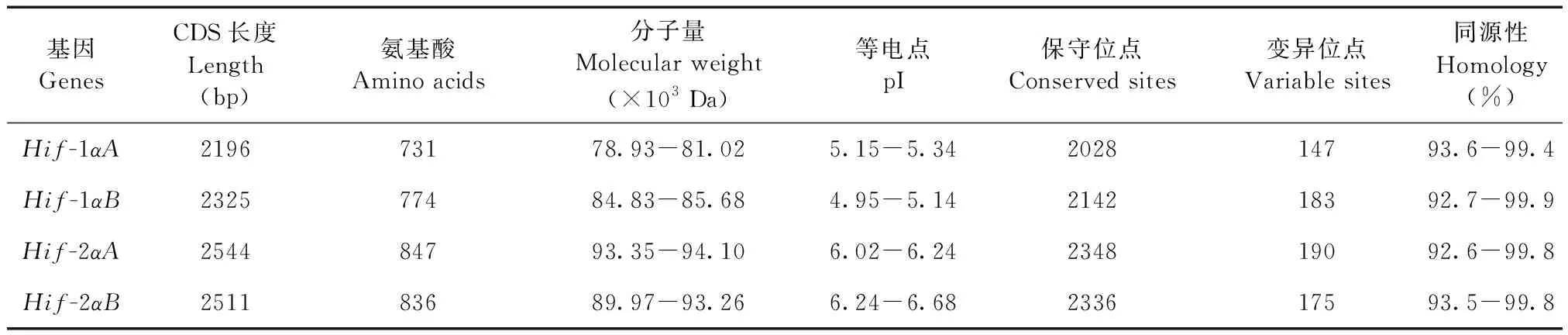
表3 基因编码区序列信息表Table 3 Sequence information of the gene coding region
2.2 HIF-α分子进化分析
为了探索HIF-1αΑ、HIF-1αB、HIF-2αΑ和HIF-2αB在裂腹鱼亚科鱼类不同类群中是否发生选择作用,本文以13个裂腹鱼亚科鱼类的物种进化树作为输入树(图2),运用PAML 4.6中CODEML程序的三个模型进行检测。位点模型中分析发现HIF-1αΑ、HIF-1αB、HIF-2αΑ和HIF-2αB存在正向选择作用(ω > 1),BEB分析显示HIF-1α和HIF-2α中皆存在后验概率大于0.95的潜在选择位点(表4)。支-位点混合模型中,以高度特化裂腹鱼亚科鱼类的7个代表物种作为前景支,其他物种作为背景支进行分子进化分析,结果显示HIF-1αΑ、HIF-1αB和HIF-2αB中均未检测到正向选择位点(ω=1),在HIF-2αA中检测到一个正向选择位点(516 N),但后验概率小于0.95。
为了进一步的检验HIF-1α和HIF-2α所有支系是否发生正向选择作用,本研究进行了支模型分析,结果表明HIF-1αΑ中拉萨裸裂尻鱼及其祖先支(支2、支14和支18,图2)、厚唇裸重唇鱼支(支9)和澜沧裂腹鱼及其祖先支(支12和支22)受到正向选择作用,HIF-1αB中极边扁咽齿鱼支(支6)受到选择作用,支15和支16也具有明显不同的进化速率,但具体的进化物种分支并未检测到。HIF-2αΑ中尖裸鲤支(支7)、双须叶须鱼支(支8)和西藏裂腹鱼支(支10)受到正向选择作用,且支14和支16也具有明显不同的进化速率。HIF-2αB中花斑裸鲤支(支5)受到选择作用,支16和支22也具有明显不同的进化速率,但具体的进化物种分支并未检测到。
2.3 低氧诱导下Hif-α基因的表达
在重度低氧处理4 h后,Hif-1αA基因在花斑裸鲤白肌组织中的表达量显著上升(P< 0.01),是常氧组的9.8倍;而在心脏中的表达量显著降低(P< 0.01);在脑、鳃和红肌组织中的表达量基本保持不变(P> 0.05)。Hif-1αB基因在白肌、红肌、心脏和脑组织中显著降低(P< 0.01);在鳃组织中表达量上调(P< 0.05)。Hif-2αA基因在心脏(P< 0.01)和红肌(P< 0.05)组织中表达量上升,且心脏组织中的表达量为常氧组的38倍;在脑组织中的表达量显著降低(P< 0.01)。Hif-2αB基因在白肌组织中的表达量上调(P< 0.01);在心脏和脑组织中表达量显著下调(P< 0.01)(图3)。

注:GWA:高原裸鲤;GPA:厚唇裸重唇鱼;SMA:巨须裂腹鱼。图1 HIF-α的蛋白序列比对Note: GWA: Gymnocypris waddelli; GPA: Gymnodiptychus pachycheilus; SMA: Schizothorax macropogon.Figure 1 Sequence alignment of HIF-α proteins

基因Genes参数估计Parameter estimates似然率值Likelihood values正向选择位点Positively selected sitesHif -1αAM0ω=0.561814753.99M1ap0=0.679, p1=0.3214683.99M2ap0=0.729, p1=0.154, p2=0.117, ω1=7.8994626.15M7p=0.005, q=0.005024695.43M8p0=0.89, p=0.005, q=0.021 (p1=0.11), ω1=8.4174626.2748I* 163N** 265G* 402C** 495L* 496 L*502S* 523P**571G* 572R** 589R**605G** 623P* 693I**Hif -1αBM0ω=0.434665.21M1ap0=0.561, p1=0.4394637.1M2ap0=0.9727, p1=0, p2=0.027, ω1=36.4334581.56M7p=0.005, q=0.007374637.64M8p0=0.999, p=0.005, q=0.007 (p1=0.00097), ω1=9994560.76532I* 644T*Hif -2αAM0ω=0.535213.57M1ap0=0.549, p1=0.451 5177.25M2ap0=0.558, p1=0.4408, p2=0.00098, ω1=9995097.15M7p=0.005, q=0.007475178.24M8p0=0.999, p=0.005, q=0.0075 (p1=0.00094), ω1=9995097.5039Q* 608P* 749S*Hif -2αBM0ω=0.4595084.73M1ap0=0.611, p1=0.3885032.04M2ap0=0.634, p1=0.356, p2=0.00965, ω1=86.5654960.77M7p=0.005, q=0.00735032.11M8p0=0.99, p=0.005, q=0.00754 (p1=0.00967), ω1=90.8674961.17427A* 435P**480E* 766C**
A
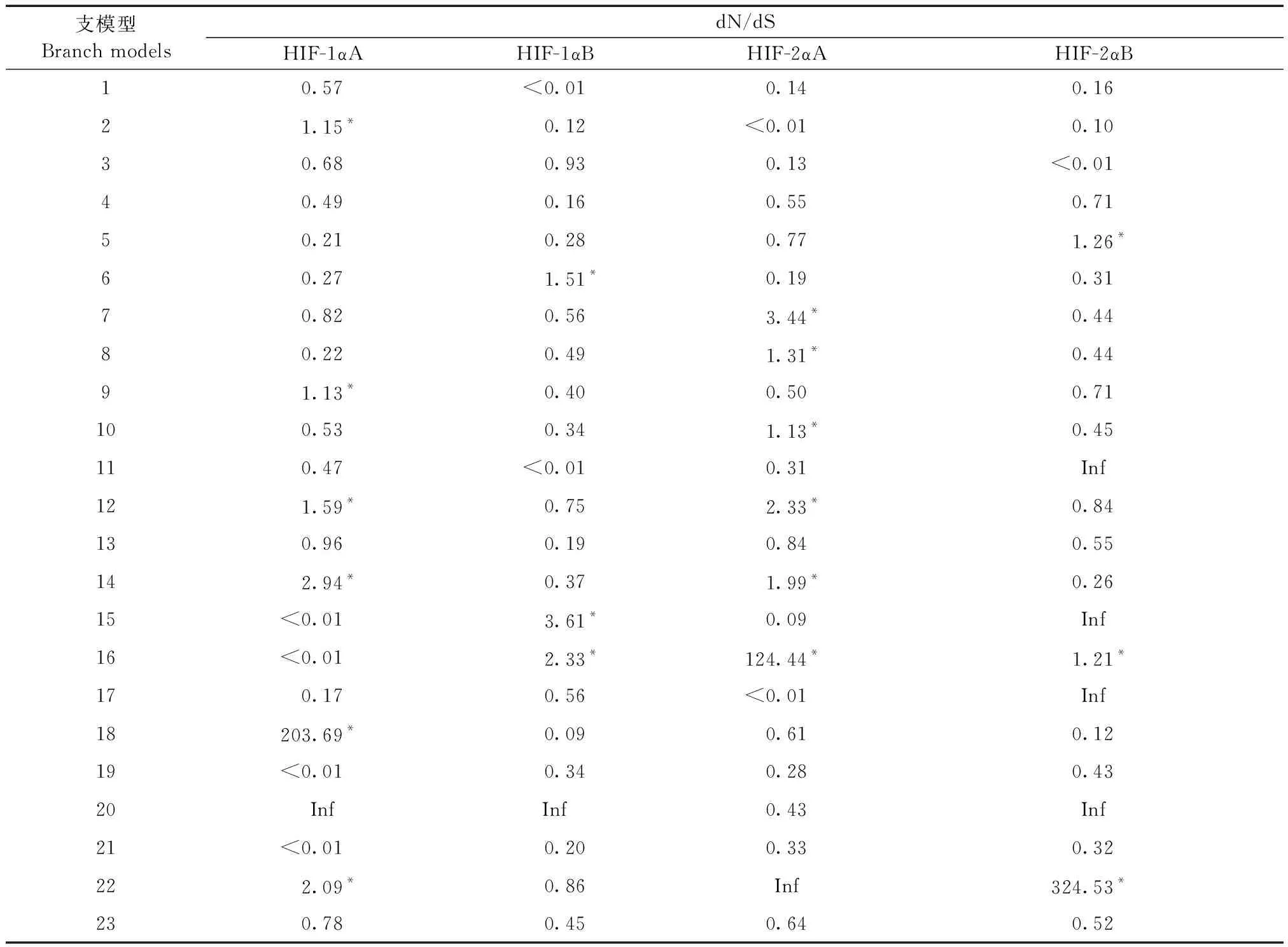
支模型Branch modelsdN/dSHIF-1αAHIF-1αBHIF-2αAHIF-2αB10.57<0.010.140.1621.15*0.12<0.010.1030.680.930.13<0.0140.490.160.550.7150.210.280.771.26*60.271.51*0.190.3170.820.563.44*0.4480.220.491.31*0.4491.13*0.400.500.71100.530.341.13*0.45110.47<0.010.31Inf121.59*0.752.33*0.84130.960.190.840.55142.94*0.371.99*0.2615<0.013.61*0.09Inf16<0.012.33*124.44*1.21*170.170.56<0.01Inf18203.69*0.090.610.1219<0.010.340.280.4320InfInf0.43Inf21<0.010.200.330.32222.09*0.86Inf324.53*230.780.450.640.52
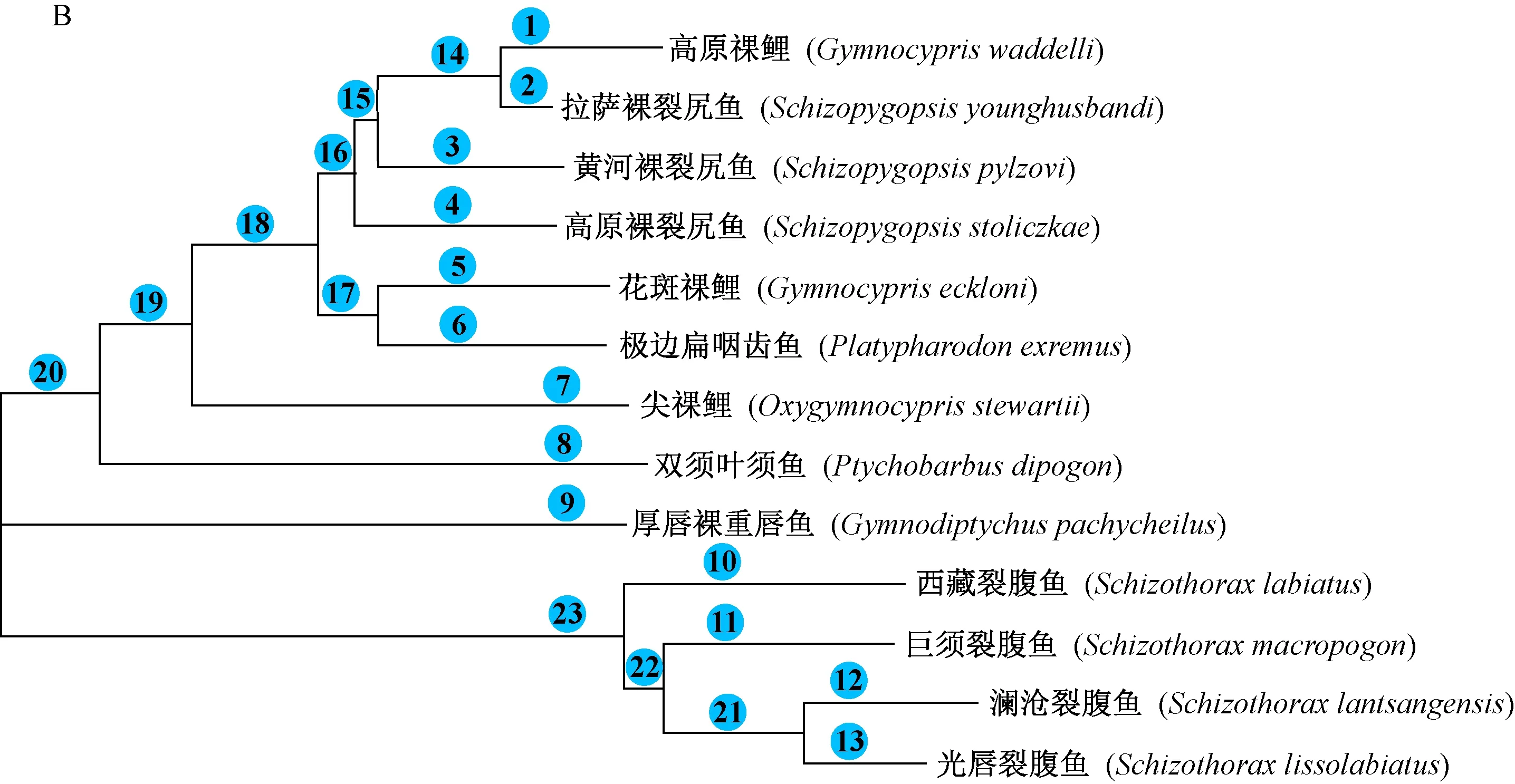
注:A为不同支系HIF-1αΑ、HIF-1αB、HIF-2αΑ和HIF-2αB的ω值,B中的分支编号是指A中系统发育树上的编号,*表示ω值大于1,Inf表示相应dN值为0,其中ω值表示非同义替换与同义替换比值,dN表示密码子的非同义替换。B是13个裂腹鱼亚科鱼类的系统发育树。图2 13个裂腹鱼亚科鱼类的系统发育树Note. A is a phylogenetic tree of 13 subfamilies of schizothoracine fishes. B indicates the ω values of HIF-1αΑ, HIF-1αB, HIF-2αΑ and HIF-2αB. The numbers in B refer to the numbers on the phylogenetic tree in A. The symbol* indicates that the ω value is greater than 1, and Inf indicates that the corresponding dN value is 0, where the ω value indicates the ratio of nonsynonymous to synonymous substitutions, and dN indicates nonsynonymous substitutions at codons.Figure 2 Phylogenetic tree of 13 subfamilies of schizothoracine fishes

注:单因子方差分析法检测各组织基因的表达量差异(n=3),与常氧组相比差异显著(**P< 0.01,*P< 0.05)。图3 重度低氧和常氧条件下花斑裸鲤主要组织Hif-α基因的表达量Note. Significant differences between tissues were analyzed by one-way ANOVA (n=3). Compared with the normoxia control group, the asterisks indicate significant difference (**P<0.01,*P<0.05).Figure 3 Expression of Hif-α gene in main tissues of Gymnocypris eckloni under severe hypoxia and normoxia

注:a:脑;b:心脏;c:鳃;d:白肌;e:红肌。单因子方差分析法检测各组组织的基因表达量差异(n=3),不同字母表示差异显著(P< 0.05)。图4 中度低氧和常氧条件下花斑裸鲤主要组织Hif-α基因的表达量Note. a: Brain. b: Heart. c: Gill. d: White muscle. e: Red muscle. Significant differences between tissues were analyzed by one-way ANOVA (n=3). Different letters indicate significant differences (P< 0.05).Figure 4 Expression of Hif-α gene in main tissues of Gymnocypris eckloni under moderate hypoxia and normoxia
中度低氧下,花斑裸鲤脑组织中Hif-1αA的表达量在48 h时达到最高,并且是0 h时的2.2倍,随后表达量急剧下调(P< 0.05);而Hif-1αB表达量从0 h开始逐渐上调,在48 h表达量最高,且72 h的表达量也明显高于0 h(P< 0.05);Hif-2αA表达量只在48 h和72 h时显著上调,在84 h时其表达量显著下调(P< 0.05);而Hif-2αB在脑组织中的表达量在12 h和36 h时显著高于0 h,且在48 h时表达量最高,随后其表达量急剧下调。心脏组织中Hif-1αA的表达量在72 h和96 h显著高于0 h(P< 0.05);在12 h时Hif-1αB和Hif-2αA的表达量最高,且Hif-2αA表达量在96 h时较0 h显著上调(P< 0.05)。鳃组织中,Hif-1αA的表达量除36 h外其他时间段均高于0 h的表达量(P< 0.05);Hif-1αB和Hif-2αB基因的表达量在36、84和96 h时基本保持不变(P> 0.05),其他时间段显著上调(P< 0.05);Hif-2αA的表达量在12 h和36 h表达量降低(P< 0.05)。白肌组织中,中度低氧胁迫时Hif-2αA的表达量远远高于其他三个基因,并在48 h时表达量最高;Hif-1αA和Hif-2αA基因在各个时间段的表达量与0 h相比均无明显变化(P> 0.05);Hif-1αB各个时间段的表达量均比0 h的表达量显著下调(P< 0.05)。红肌组织中,Hif-1αA和Hif-2αA基因在72 h表达量最高,而Hif-1αB和Hif-2αB表达量在24 h-96 h时间段较0 h均无明显变化(P> 0.05)(图4)。
3 讨论
鱼类具有同源基因Hif-αA和Hif-αB,并与人类HIF-α基因具有很高的相似性,同时鱼类中的Hif-α基因可能在氧依赖基因的表达中产生的作用[22]。裂腹鱼亚科鱼类HIF-αA和HIF-αB氨基酸序列长度与鲤科其他鱼类如鳙鱼(Hypophthalmichthysnobilis)、鲤鱼(Cyprinuscarpio)、鲫鱼(Carassiusauratus)及斑马鱼基本一致,并具有bHLH结构域、PAS和PAC结构域、ODD结构域以及TAD结构域。目前大多数硬骨鱼类丢失其中一个同源基因,鲤科鱼类二者皆有保存,以上表明HIF-αA和HIF-αB在鲤科鱼类长期进化过程中体现出较高的保守性,与其产生的生物学功能一致。本研究获得了13个裂腹鱼亚科代表物种Hif-1αΑ、Hif-1αB、Hif-2αΑ和Hif-2αB基因编码区完整序列,其N端的bHLH-PAS结构域是高度保守的,但ODD结构域保守性较低,特别是氧依赖性脯氨酸羟基化位点附近。HIF-α中检测到两个保守的脯氨酸羟基化序列LxxLAP,但在13个物种HIF-1αΑ的NODD中检测到特异性缺失,并且特化及高度特化的裂腹鱼的HIF-1αB的CODD结构域中脯氨酸羟基化序列突变为PxxLAP。HIF羟基化途径在后生动物中受氧依赖性脯氨酰羟化酶(prolyhydroxylases,PHD)家族的调控[23]。在哺乳动物中,CODD结构域中Pro-564是PHD与HIF-1α结合的重要位点[18]。缺氧条件下,PHD失活使HIF-α的羟基化受到抑制,从而与HIF-1β形成异源二聚体,结合DNA序列上的低氧反应元件以激活下游基因的表达进行耐氧适应性反应[24-25]。由此可以推断,发生在ODD区域中的缺失和特异突变可能是裂腹鱼亚科鱼类在低氧适应中进化表现。
目前,对于裂腹鱼亚科鱼类HIF-α分子进化研究报道甚少。管丽红等[18]在裂腹鱼亚科鱼类中首次克隆出Hif的旁系同源基因,并结合其他鲤科鱼HIF-α中序列仅发现HIF-1αB中存在正向选择作用,且在特化及高度特化的裂腹鱼中存在选择作用位点,Hif-1αB基因是裂腹鱼适应低氧环境中重要的调节因子,同时Hif-α基因的正向选择可能反映了裂腹鱼亚科鱼类在高海拔地区缺氧环境的适应性进化[26]。本研究进一步对裂腹鱼亚科不同进化等级物种间是否存在选择压力进行分析,结果表明HIF-1αΑ、HIF-1αB、HIF-2αΑ和HIF-2αB中皆存在潜在的受到正向选择作用的氨基酸位点,且后验概率大于0.95。同时,支模型表明HIF-α在特化各物种、高度特化各物种及其祖先支具有明显不同的进化速率,但具体的进化物种分支并未检测到。在支-位点混合模型中,以高度特化等级物种作为前景支,仅在HIF-2αA中存在正向选择作用。由此看来,HIF-α在13个物种间存在正向选择作用,但HIF-1αΑ、HIF-1αB和HIF-2αB仅检测到特化等级支及高度特化支中存在选择作用,具体物种分支有待进一步研究。
低氧诱导因子作为氧敏感的转录激活因子,在低氧条件下可被诱导表达参与反应调节。HIF-1α和HIF-2α两者具有不同的激活域从而形成了各自的特异性靶基因以及各自独特的功能[23]。本研究结果表明,重度缺氧条件下Hif-1αΑ和Hif-2αB基因在白肌组织中显著上升,且在中度缺氧时,Hif-2αB在白肌组织中的表达量远高于另外3个基因。Hif-1αB在重度缺氧条件下在鳃组织中表达量显著升高。Hif-2αA在重度缺氧条件下在心和红肌中的表达量显著上升,并在中度缺氧条件的12 h和96 h具有较高的表达量。在不同组织中Hif-αmRNA表达不同,可能是对氧气敏感度不同,缺氧表达的动力学取决于氧气水平并存在组织器官特异性。在对尼罗罗非鱼的研究中发现硬骨鱼Hif-1αΑ/B和Hif-2αA/B基因在缺氧条件下的转录调节中存在物种特异性,也还受缺氧的组织及缺氧状态的影响[27]。在重度缺氧条件下,花斑裸鲤的脑组织中Hif-1αB、Hif-2αΑ和Hif-2αB基因表达量显著下降,心脏组织中Hif-1αA、Hif-1αB和Hif-2αB基因表达量显著下降,可能在急性低氧环境中可以保护物种本身,在中度低氧的后期,心脏组织中的Hif-1αA和Hif-2αA基因表达量上调,可能是为了提高携氧能力从而弥补机体缺氧而做出的应激性应答。由此推断,裂腹鱼亚科鱼类在适应长期低氧和应激性低氧环境可能存在两种不同的机制。
