利用微卫星和线粒体D-loop区联合分析大尾寒羊的遗传多样性
2019-07-31刘泽民王岩超张淑二刘展生姜运良
刘泽民 王岩超 张淑二 刘展生 姜运良*
利用微卫星和线粒体D-loop区联合分析大尾寒羊的遗传多样性
刘泽民①王岩超①张淑二②刘展生②姜运良①*
(①山东农业大学动物科技学院 山东 泰安 271018 ②山东省畜牧总站 山东 济南)
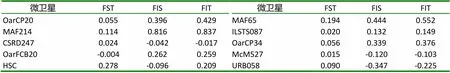
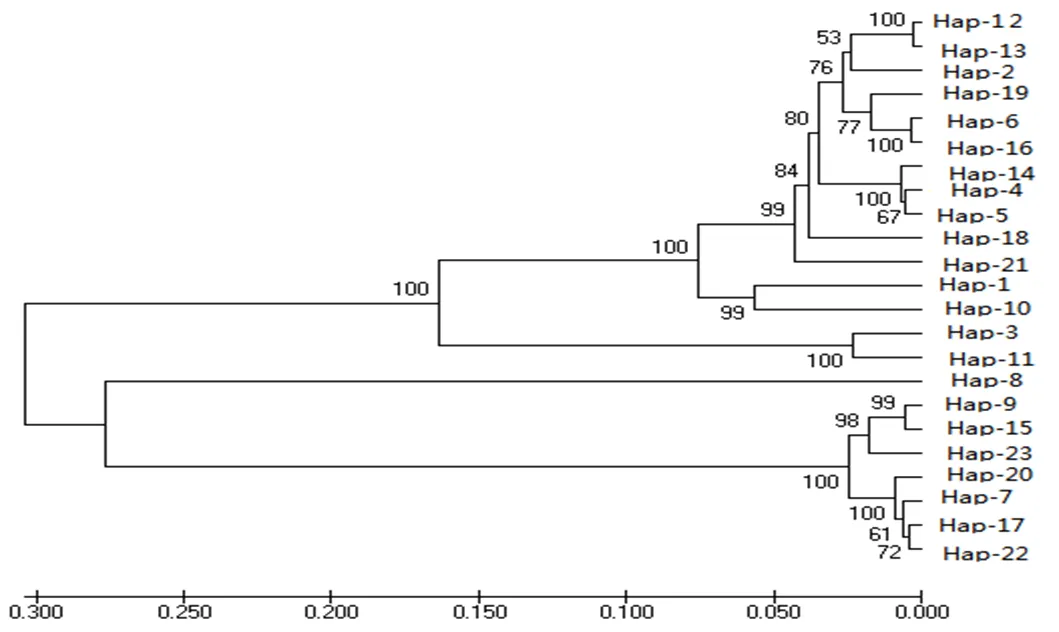
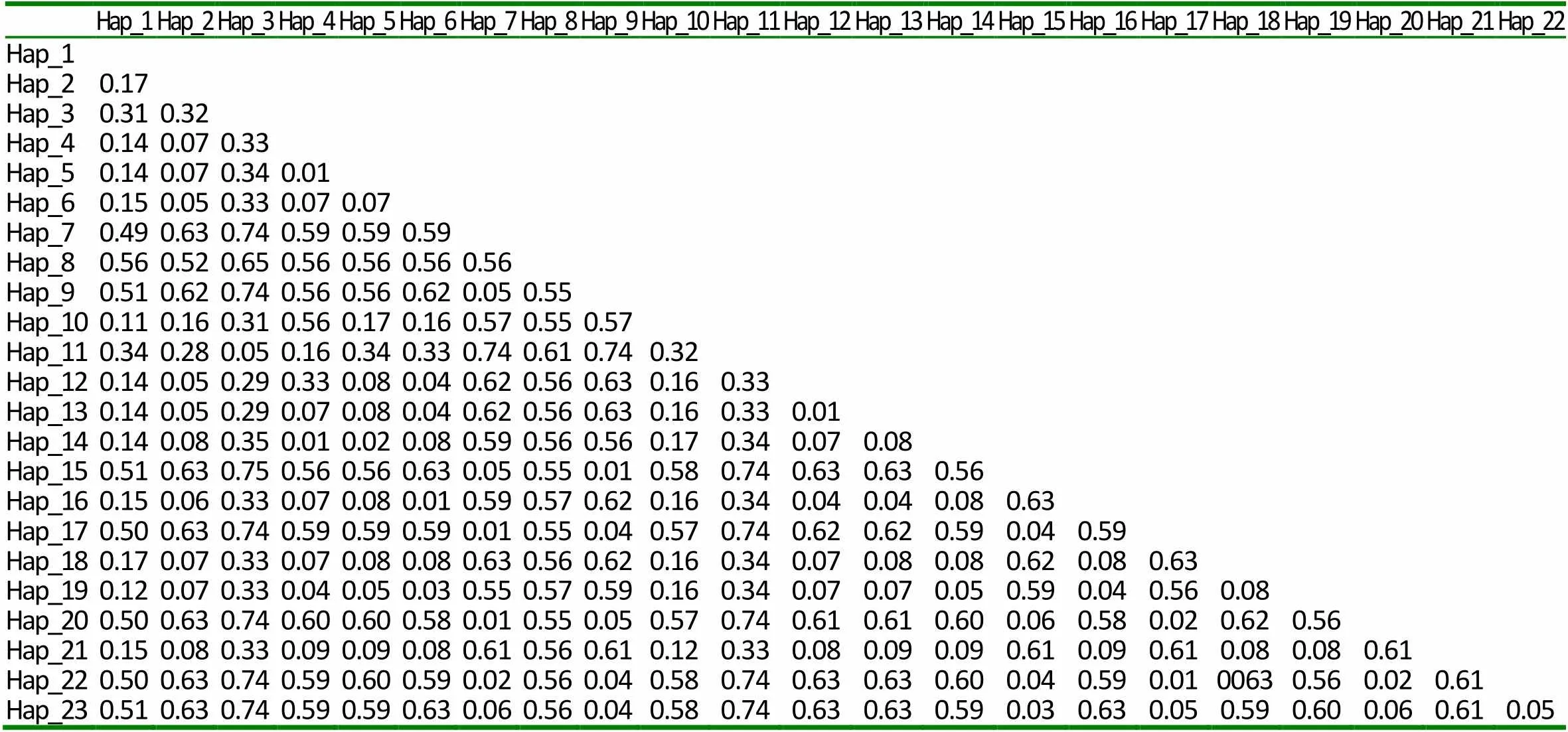
本研究对42只大尾寒羊的10个微卫星标记进行了遗传多样性分析,并对其中38只个体的线粒体DNA D-loop区的进行了测序和多态性分析。结果表明,10个微卫星位点共发现38个等位基因,平均观察杂合度为0.3631,平均PIC值为0.416,FST、FIS和FIT平均值分别为0.0842、0.1784和0.2466,且该群体的HO 大尾寒羊 微卫星 线粒体D-loop区 遗传多样性 大尾寒羊是我国华北地方品种羊,公母羊的尾都过飞节,长者可接地或拖及地面,尾尖向上翻卷,形成明显尾沟,属大脂尾羊[1]。作为我国优良绵羊品种,其遗传价值不容低估。由于保护开发投入不足,加之与其他品种的盲目杂交,导致大尾寒羊在部分地区濒临灭绝。妥善保存和利用大尾寒羊基因库,制订合理的保种方案,扩大其存栏数量已成为我省畜牧业生产中迫在眉捷的一项战略任务[2, 3]。 微卫星DNA是可变数目串状重复(VNTR)的一种[4]。微卫星具有多态信息含量丰富、杂合度高、共显性遗传、等位基因数目多、在畜群基因组DNA中分布广泛等优点[5, 6]。线粒体DNA (mitochondrial DNA, mtDNA)是存在于线粒体基质中的、独立于细胞核染色体外的基因组,与核基因相比,mtDNA 具有结构简单、以母性方式遗传、进化速度快和极少发生重组等特点[7-10]。其中D-loop区的进化速度最快,变异较大,适用于进行亚科内属、种间的系统学研究[11, 12]。 本研究分析了微卫星和线粒体D-loop区的多态性,旨在为大尾寒羊的种质资源保护提供理论依据。 42只大尾寒羊(包括27只母羊和15只公羊)于2016年3月31日采自山东省临清市大尾寒羊保种场,每只羊颈静脉采血15ml于自带肝素钠的采血管中,保存在-20℃冰箱中以备提取基因组。 1.2.1 微卫星DNA的多态性分析 选取的10个微卫星来源于GeneBank,由生工生物工程(上海)股份有限公司合成。PCR反应总体系为25μl,包括10×Ex Taq Buffer 2.5μl,d NTPs 2μl,上下游引物(1μM)各0.5μl,基因组 DNA(50~100ng)2μl,Ex Taq 0.25μl,灭菌ddH2O 17.25μl。PCR反应条件为95℃5min;94℃30s,50~60℃30s,72℃ 30s,38个循环;72℃延伸10min。1%的琼脂糖凝胶电泳检测。选择12%的非变性聚丙烯酰氨凝胶进行检测。预电泳结束后,取5μl PCR产物与3μl 6× Loading Buffer混匀,上样,160~180V电压,电泳12~16h,硝酸银染色,拍照。 1.2.2 线粒体DNA D-loop区的扩增与测序 根据NCBI数据库提交的绵羊线粒体基因组序列(KR868678)设计扩增大尾寒羊线粒体D-loop区部分序列的特异引物。上下游引物序列分别为F:CCACTATCAACACCCAA,R:CGAAGGGCGT TACTCACC。引物由生工生物工程(上海)股份有限公司合成。 1.2.3 分型分析与序列分析 利用Cervus3.0计算大尾寒羊群体10个微卫星位点的观察杂合度、多态信息含量(FIC)、哈代—温伯格平衡等,对大尾寒羊多样性进行评估。利用FSTAT3.9.2计算大尾寒羊总群体的近交系数(FIT)、不用亚群间的遗传分化程度(FST)、群内近交系数(FIS)等遗传多样性参数。测得的线粒体D-loop区序列用DNA MAN6.0软件进行整理对比,并进行人工校对。用DNAsp5.0软件统计单倍型及变异位点、计算单倍型多样性( Hd) 及核苷酸多样度(Pi)等,评价群体遗传多样性水平。采用MEGA5.0软件计算单倍型间的遗传距离,并构建UPGMA分子系统树。 表1 绵羊微卫星标记引物及退火温度 2.1.1 微卫星位点的PCR扩增及多态性检测 扩增结果显示共有38个等位基因,平均每个位点有3.8个,其中在OarCP20位点检测到的等位基因数最多(8个),在CSRD247、OarCP34、McM527和HSC四个位点检测到的等位基因数最少(2个)。CSRD247位点的基因分析的结果如图1所示。 图1 微卫星CSRD247的PCR扩增产物PAGE胶图 2.1.2 微卫星座位的遗传参数 (1)所有微卫星座位上有效等位基因占85.11%,各位点平均有效等位基因数范围在0.3306~0.9151之间,与有效等位基因数相差较大,表明等位基因分布不均匀,可能与人工选育有关。(2)我们选了检测到的3个高度多态性位点和5个中度多态性位点来分析遗传多样性。这在这8个位点中有37.5%的位点偏离了哈代-温伯格平衡,6个位点的期望杂合度都超过观察杂合度(HO 表2 微卫星位点的观察杂合度、期望杂合度、多态信息含量和哈代-温伯格平衡 注:***为0.1%显著性水平偏离哈代-温伯格平衡,**为1%显著性水平偏离哈代-温伯格平衡,*为5%显著性水平偏离哈代-温伯格平衡,NS为不显著偏离哈代-温伯格平衡;ND 没有做H-W检测 FST值代表整个群体不同亚群间的遗传分化程度,在8个所选的位点中,FST值在0.015到0.278之间,平均值为0.0842;群内近交系数(FIS)显示大部分位点的近交系数为正值,平均值为0.1784;FIT值用来表示总群体的近交系数,其平均值为0.2466(表3)。 表3 群体的遗传多样性参数 2.2.1 mtDNA D-loop区序列扩增 根据普通绵羊线粒体DNA序列设计的引物在大尾寒羊基因组中得到较好的扩增(图2)。PCR产物片段单一,扩增片段大小与预期的产物片段大小一致,可用于后续的测序分析。 图2 大尾寒羊mtDNA D-loop区的扩增结果 2.2.2 mt DNA D-loop序列的遗传多样性 测序分析了其中38只D-loop区扩增结果较为理想的个体。结果显示38个个体分别属于23种单倍型(表4)。D-loop区碱基组成A+T的平均含量(62.0%)明显高于G+C的平均含量(34.6%),转换/颠换值为2.90,遗传多样性指数如表4所示。 表4 大尾寒羊mt DNA D-loop部分序列的遗传多样性指数 应用MEGA软件,根据线粒体D-loop序列计算了23个单倍型之间的遗传距离,单倍型的平均遗传距离为0.37,其中有8组遗传距离≤0.01,单倍型9、11的遗传距离最大,为20.294。单倍型12、13为最小遗传距离0.0055,绝大部分的遗传距离在0~2之间(表5)。通过软件构建了大尾寒羊单倍型间的UPGMA分子系统树(Bootstrap检验重复为1000次)。由图3可以看出,23种单倍型最终聚为3大分支。单倍型8与其他单倍型的同源性都有较远距离。 图3 大尾寒羊群体的UPGMA分子系统树 表5 大尾寒羊mt DNA D-loop部分序列单倍型间的遗传距离 (1)利用微卫星标记来探究种群的遗传多样性时,杂合度能够反映群体在多个基因座位上的变异,它是衡量群体遗传变异的一个最适参数[3~ 13]。在本研究中,10个标记的平均观察杂合度为0.3631,期望杂合度均在0.3以上的座位有8个,观察杂合度在0.3以上的座位有7个,与Takezaki和Nei(1996)认为微卫星计算出的杂合度范围为0.3~ 0.8基本吻合[14]。多态信息含量(PIC)是基因丰富度的一个指标,多态信息含量的高低表明了品种遗传基础的丰富多样性,用多态信息含量来描述种群在微卫星位点上的变异程度,当PIC>0.5时,该位点为高度多态;0.5>PIC>0.25时,该位点中度多态;PIC<0.25时,为低度多态[15, 16]。在所选用的10个微卫星多态位点的多态信息含量中HSC为和McM527低度多态,MAF214、OarFCB20和OarCP20为高度多态,其余基因座的多态信息含量为中度多态,所有位点的平均PIC值为0.416,说明所选择的微卫星基因座位的多态性中等。(2)由于上述10个微卫星位点不论是PIC值还是杂合度,变化范围都很大,不利于对大尾寒羊群体的遗传多样性进行评估。又分析了FIT、FST和FIS3个遗传参数。FST值代表整个群体不同亚群间的遗传分化程度,取值范围从0~1,其值越大,表明亚群间遗传分化越明显。一般评价标准是FST<0.05,代表群体分化较小;0.05 本实验由于保种场内样品数量有限,品种间亲缘关系混杂,抽样效率低,继而利用线粒体DNA D-loop区部分序列进行了分析,明确种群内个体的亲缘关系及遗传多样性。应用mtDNA测序技术来研究物种的遗传多样性时,通常用两个重要指标来衡量一个群体mtDNA的遗传变异程度,单倍型间的平均遗传距离(P)和核苷酸多样性(π)[7, 20]。一般来说大多数哺乳动物的P值都在0.01以上时被认为变异大[21]。本研究显示,群体单倍型间的平均遗传距离为0.37,大于0.01,这说明中大尾寒羊的遗传变异较大。核苷酸多样性(π)是指群体内两个随机个体mtDNA序列间平均每个位点的核苷酸差异数目。Nei等定义,给定群体内两个随机选取的mtDNA序列间π值越小,表明群体的遗传多样性越低。本研究中,中大尾寒羊群体mtD NAD-loop区的核苷酸多样性(π)为0.02815,高于Lan等认为的低遗传多样性指标(π值在0.0015 ~0.0047)[23],但低于山东其他绵羊品种,小尾寒羊(0.0324)与洼地绵羊(0.03172)[24],就其分子进化树来看,三个分支内部的遗传距离相对较近,存在近交现象。 总的来说该大尾寒羊种群虽然遗传多样性相对较高,但群体内部存在一定程度的近交现象,在大尾寒羊育种工作中,应避免遗传距离较近的家系间的交配,进一步加大大尾寒羊保护、开发和利用力度,有效维持大尾寒羊这一国家保护品种的基本数量与质量。 [1] 王俊海, 姜永红, 肖延光等. 大尾寒羊品种资源现状与保护利用对策[J]. 山东畜牧兽医, 2010, 31(5): 60-61+63. [2] 吴诗, 姜勛平. 基于GIS的中国山羊品种资源管理系统研制[J]. 中国草食动物, 2011, 31(4): 71-75. [3] 吴诗, 姜勋平, 蔡忠亮. 基于GIS的中国山羊品种资源管理系统研制[J]. 中国草食动物, 2011, 31(4): 71-75. [4] Beck N R, Double M C, Cockburn A. Microsatellite Evolution at Two Hypervariable Loci Revealed by Extensive Avian Pedigrees[J]. Molecular Biology and Evolution, 2003, 20(1): 54-61. [5] Zeveren A V, Peelman L, Weghe A V D, et al. A genetic study of four Belgian pig populations by means of seven microsatellite loci[J]. Journal of Animal Breeding and Genetics, 1995, 112(1-6): 191-204. [6] Markowitz S D, Wang J Y, Myeroff L L, et al. Markowitz, S. et al. Inactivation of the type II TGF- receptor in colon cancer cells with microsatellite instability. Science 268, 1336-1338[J]. Science, 1995, 268(5215): 1336-1338. [7] 黄雪贞, 钱国英, 李彩燕. 中华鳖3个地理群体线粒体基因D-loop区遗传多样性分析[J]. 水产学报, 2012, 36(1): 17-24. [8] 李齐发, 李隐侠, 赵兴波等. 牦牛线粒体DNA D-loop区序列测定及其在牛亚科中分类地位的研究[J]. 畜牧兽医学报, 2008, 39(1): 1-6. [9] Cann R L, Brown W M, Wilson A C. Polymorphic sites and the mechanism of evolution in human mitochondrial DNA[J]. Genetics, 1984, 106(3): 479-499. [10] Glenn T C, Staton J L, Vu A T, et al. Low mitochondrial DNA variation among American alligators and a novel non-coding region in crocodilians[J]. Journal of Experimental Zoology, 2002, 294(4): 312-24. [11] Sankoff D, Bryant D, Deneault M, et al. Early eukaryote evolution based on mitochondrial gene order breakpoints[C]// International Conference on Computational Molecular Biology. ACM, 2000: 254-262. [12] Saccone C, Gissi C, Lanave C, et al. Evolution of the mitochondrial genetic system: an overview[J]. Gene, 2000, 261(1): 153-9. [13] 包文斌, 束婧婷, 许盛海等. 样本量和性比对微卫星分析中群体遗传多样性指标的影响[J]. 中国畜牧杂志, 2007, 43(1): 6-9. [14] Takezaki N, Nei M. Genetic Distances and Recons-truction of Phylogenetic Trees From Microsatellite DNA[J]. Genetics, 1996, 144(1): 389-99. [15] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms.[J]. American Journal of Human Genetics, 1980, 32(3): 314. [16] 张爱玲, 马月辉, 李宏滨等. 利用微卫星标记分析6个山羊品种遗传多样性[J]. 农业生物技术学报, 2006, 14(1): 38-44. [17] Balloux F, Lugon‐Moulin N. The estimation of population differentiation with microsatellite markers.[J]. Molecular Ecology, 2002, 11(2): 155-65. [18] 孙俊丽, 张冰, 潘天彪等. 基于微卫星位点研究7个广西地方猪种的遗传关系[J]. 西南农业学报, 2016, 29(6): 1475-1480. [19] 毛永江, 常洪, 杨章平等. 3个中国地方黄牛品种遗传结构及其遗传分化的研究[J]. 畜牧兽医学报, 2007, 38(2): 125-132. [20] Neigel J E, Avise J C. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation.[J]. 1994, 135(4): 1209-1220. [21] Lan H, Shi L. The origin and genetic differentiation of native breeds of pigs in southwest China: an approach from mitochondrial DNA polymorphism.[J]. Biochemical Genetics, 1993, 31(1): 51-60. [22] Neigel J E, Avise J C. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation.[J]. 1994, 135(4):1209-1220. [23] Lan H, Shi L. The origin and genetic differentiation of native breeds of pigs in southwest China: an approach from mitochondrial DNA polymorphism.[J]. Biochemical Genetics, 1993, 31(1): 51-60. [24] 黄庆华, 张果平, 王金文等. 山东省地方绵羊品种的mtDNA D-loop序列多态性及系统进化研究[J]. 山东农业科学, 2012(5): 5-8. (2019–04–09) S813.3 A 1007-1733(2019)07-0001-051 材料和方法
1.1 试验动物
1.2 方法

2 结果
2.1 微卫星DNA的多态性分析


2.2 mtDNA D-loop区多态性分析


2.3 单倍型之间的遗传距离及分子系统树
3 讨论
3.1 微卫星DNA的多态性分析
3.2 mtDNA D-loop区多态性分析
